滑液囊支原体(Mycoplasma synoviae,MS)是一种危害禽类和鸟类健康的重要病原,可引起滑液囊支原体病,又称传染性滑膜炎[1]。该病可通过直接接触水平传播或经卵垂直传播,不同发育阶段的鸡均可感染,导致采食量下降,饲料转化率下降,生长发育迟缓,胴体降级[2-3];成年鸡关节肿大,滑膜炎,出现呼吸症状,蛋鸡产蛋率下降,蛋壳顶端异常,种蛋孵化率降低[4]。单一的MS感染很少导致鸡死亡,但会降低鸡的免疫力,导致继发感染[5]。Olson等首次在美国发现该病,随后在法国、意大利、澳大利亚及葡萄牙等国家相继报道该病。近年来,国内外多个地区均存在MS感染,且该病有发展及蔓延趋势,给养禽业带来较大的经济损失。马爽等[6]对我国11个省份不同鸡场进行检测,均发现MS感染,鸡群感染率超过69%[7]。MS基因组较细菌小,为750~850 kb,G+C含量为28%左右。NCBI收录的MS全基因组有10条,包括巴西MS53株[8]、美国WVU-1853株[9]、澳大利亚86079/7 NS株[10]、中国河南HN01株[11],以及经86079/7 NS诱导获得的MS-H株[12]。MS-H株是MS弱毒疫苗生产用菌株,是全球通用的MS弱毒疫苗株,但研究表明,MS-H株疫苗仅对未感染MS的鸡群有效,对已感染MS的鸡群几乎无效,甚至可能会加重病情[13]。全基因组测序是了解基因序列信息的一种重要手段,中国目前只有1株分离自河南的HN01全基因组序列,因此,关于我国MS的遗传多样性资料不全面。本实验室于2018—2019年,从川西4个规模2 000只以上三黄鸡肉鸡场分离出7株MS[14],4个场均未免疫MS弱毒疫苗,发病鸡表现为跗关节肿大、胸部囊肿,剖检时可见跗关节腔、爪垫、胸部龙骨滑膜囊有淡黄色胶冻样分泌物,或黄色干酪样分泌物附着。本试验对7株MS进行全基因组测序,通过生物信息学分析,确定MS分离株的基因组成、基因岛等基因组特征;并与NCBI上其他MS株的基因组序列比较分析,明确川西地区MS菌株的遗传进化关系和差异基因,为进一步研究MS的致病机制、开发MS亚单位疫苗和建立特异性检测技术等奠定基础。
1 材料与方法 1.1 MS分离株7株MS四川分离株由西南民族大学动物医学实验室分离鉴定并保存,分别命名为MS251、MS254、MS221、MS231、MS12H、MS1G、MSK1,其中MS251、MS254、MS221、MS231分离自同一鸡场,MS12H、MS1G、MSK1分离自其他3个鸡场。改良Frey氏培养基购自北京中海生物科技有限公司。
1.2 MS分离株培养和DNA提取分别将7株MS株各200 μL菌液接种到1.8 mL改良Frey氏液体培养基中,于37 ℃、5% CO2恒温培养箱中培养,待培养基颜色变黄时收集菌液即为MS分离株种子菌液。经半固体培养基培养及菌落形态观察确定是否符合MS的培养特征,并将经特异性PCR扩增鉴定的种子菌液扩大培养后提取MS基因组DNA。要求基因组DNA完整且没有降解,在23 kb以上,OD260 nm/OD280 nm值为1.8~2.0;DNA浓度>30 ng·μL-1。
1.3 全基因组测序、原始数据质量评估和质量控制7株MS全基因组测序由北京赛默百合生物科技有限公司完成,采用Illumina HiSeq平台测序,构建PE测序文库,得到的原始图像数据经过Base Calling处理后,FASTQ格式文件存储读长(read)的碱基及其质量信息。对原始测序数据使用FastQC(版本0.11.5)进行碱基质量统计,并使用R统计软件对结果进行可视化。使用Trimmomatic(版本0.36)对序列进行修剪和去除接头序列。
1.4 7株MS的基因从头组装、基因预测和注释利用Edena软件,通过设置默认参数将所有过滤后的reads进行组装,得到最终的组装结果并统计长度信息。使用经典的OLC(overlap-layout-consensus)算法架构的组装工具,将经过算法处理后的reads进行组装。组装后,得到多条Contig。使用Prodigal对CDS进行预测,使用SignalP对信号肽进行预测,使用infernal+Rnammer对tRNA、rRNA进行预测。将所有的CDS序列利用blast比对到KEGG、Swissport、Nr、Nt、eggNOG数据库,并进行注释,e value阈值统一为1×10-35。
1.5 MS全基因组比较分析在进行全基因组比较时,引用了NCBI上收录的8株MS的全基因组序列,分别为86079-7 NS(NZ_CP012624.1)、86079/7 NS (NZ_CP029258.1)、HN01(NZ_CP034544.1)、MS53(NC_007294.1)、MS-H(NZ_KP704286.1)、MS-H(NZ_CP021129.1)、NCTC10124(NZ_LS991953.1)、WVU1853T(NZ_CP011096.1)。使用Xshell软件对注释完成的7株MS四川分离株全基因组进行筛选统计,包括总基因长度、GC含量、编码基因。使用R语言中的genoPlotR包,将7株MS四川分离株的全基因组与NCBI上记录的8株MS全基因组比较,分析分离株与其他野毒株和疫苗株的差异。将差异区域进行线性分析。
2 结果 2.1 提取的MS分离株DNA质量MS分离株DNA提取结果表明MS基因组DNA完整且没有降解,在23 kb以上,测量的OD260 nm/OD280 nm比值为1.8~2.0;DNA浓度大于30 ng·μL-1,符合测序要求。
2.2 全基因组测序及预测使用Xshell软件中shell脚本语言对全基因组筛选统计,结果显示,7株MS全基因总长度在760~ 810 kb,GC含量28.2%~28.44%,编码基因数677~725个,CDS 636~685个,rRNAs 5个,tRNA数34个,tmRNA 1个。详见表 1。
|
|
表 1 7株MS全基因组的基本信息 Table 1 Basic information of the whole genome of 7 MS isolates |
2.3.1 COG文库注释 将所有预测到的蛋白质与EggNOG 4.0数据库进行比对后,取e value 1×10-35为阈值,取best hit one作为映射依据进行COG注释。COG基因功能注释后分类结果显示,7株MS四川分离株MS251、MS254、MS221、MS231、MS12H、MS1G、MSK1功能基因数分别为528、527、527、527、512、514、510个。按照功能分类又可分为三大类,第一类代谢基因,包括氨基酸的运输和代谢、碳水化合物的运输和代谢、辅酶的运输和代谢、无机离子的转运和代谢、脂质转运与代谢、核苷酸的运输和代谢、次生代谢产物生物合成运输和分解代谢、能源生产与转换;第二类信息储存和处理基因,包括转录、复制重组和修复、防御机制;第三类为细胞过程和信号转导基因,包括细胞周期控制、细胞分裂和染色体分割、细胞壁/膜/外壳起源、细胞内运输、细胞运动性,分泌和囊泡运输、翻译后修饰,蛋白质转换,伴侣、信号转导机制、翻译、核糖体结构和生物发生。还含有功能未知基因和预测基因,结果如表 2所示。结果显示,7株MS功能基因种类相同,来自同一鸡场的4个分离株MS251、MS254、MS221、MS231,其功能基因个数几乎相同,而分别来自其他3个鸡场的MS12H、MS1G、MSK1间功能基因个数差异相对较小,但较来自同一鸡场的4株MS碳水化合物的运输和代谢基因数少8~9个,其他部分基因数差异在0~3个之间,如表 2所示。
|
|
表 2 7株MS分离株COG注释结果 Table 2 COG annotation results statistics of 7 MS isolates |
2.3.2 GO文库注释 将所有基因序列比对到Swissport数据库,并使用blast2 GO进行GO Mapping。所有的GO根据GO的有向无环图向上回溯,直到第3层,而后统计第3层GO的分析结果。结果显示,功能基因包括分子功能、生物过程、细胞成分3大部分。分子功能基因包括催化活性、结构分子活性、转运蛋白活性、翻译调节活性、分子功能调节因子、结合因子;生物过程基因包括细胞过程、生物调节、对刺激的反应、代谢过程、生物间相互作用、定位、复制;细胞成分基因包括细胞解剖实体、含蛋白质的复合物。分析结果表明,7株MS四川分离株功能基因种类相同,同场内分离的MS251、MS254、MS221、MS231,功能基因数几乎相同,分别来自3个鸡场的MS分离株MS12H、MS1G、MSK1间在部分功能基因数上差异相对较小,但较同场内分离的4株MS的催化活性基因数少7~8个、细胞过程基因数少4~6个,其他部分基因数差异在0~3个之间,如表 3所示。
|
|
表 3 7株MS分离株GO文库注释结果 Table 3 GO library annotation results of 7 MS isolates |
2.3.3 KEGG文库注释 将所得的序列比对到KEGG数据库并进一步映射到KO(KEGG orthology),并通过KO映射到Pathway,对KEGG每一个功能模块的Pathway映射到的基因数进行统计的结果如下,细胞过程:细胞群落-原核生物、细胞生长和死亡、细胞运动性、运输和分解代谢;环境信息处理:膜运输、信号传导;遗传信息处理:翻译、复制和修复、折叠、分类和降解、转录;新陈代谢:碳水化合物代谢、核苷酸代谢、能量代谢、辅助因子和维生素的代谢、氨基酸代谢、类脂物代谢作用、其他氨基酸的代谢、生物降解和代谢、萜类和多酮的代谢、其他次生代谢产物的生物合成、糖的生物合成和代谢。结果显示,7株MS四川分离株功能基因种类相同,MS251、MS254、MS221、MS231各种功能基因数目几乎相同,MS12H、MS1G、MSK1间部分功能基因数差异相对较小,较4株同场分离MS的膜运输基因少6~9个、碳水化合物代谢基因少12~13个,氨基酸代谢基因少3~6个,能量代谢基因数少2~5个,其他部分基因数相差异在0~3个之间。详见表 4。
|
|
表 4 7株MS分离株KEGG文库注释结果 Table 4 KEGG library annotation results of 7 MS isolates |
2.3.4 Nr和Nt文库注释结果 将所有的基因序列比对到Nr和Nt数据库, 结果显示,7株MS分离株测序基因99.69%以上来源于MS基因,但有少部分基因可能来源于鸡毒支原体。证明7株分离株为MS。
2.4 专业数据库分析2.4.1 PHI-base数据库分析 PHI-base数据库(pathogen host interactions database)从文献中收集了大量致病基因和效应基因的序列。根据数据库中病原基因表型对照分析7株MS分离株的基因表型结果显示,导致病原菌致病能力丧失的基因为3个;导致病原菌致病能力减弱的基因MS251、MS221、MS231为44个,其余菌株45个;对病原菌致病性没有影响的基因均为10个;导致病原致病能力增强的基因MS1G 8个,其余菌株有9个;致病效应基因7株MS均为2个,致死因子基因MS251、MS254、MS221、MS231为3个,MS12H、MS1G、MSK1为2个,抗药和感药基因在各分离株均1个。详见表 5。
|
|
表 5 7株MS分离株PHI文库分析结果 Table 5 PHI library analysis results of 7 MS isolates |
2.4.2 VFDB数据库分析 使用blastp将所有预测出的蛋白质序列比对到VFDB数据库中,以1×10-35作为阈值进行筛选。VFDB记录了大量毒力因子序列信息,结果显示分离的7株MS含有25种毒力因子,msbA、sugC、oppF基因拷贝数均为2,tnp其拷贝数MS251、MS254、MS221、MS231、MS12H、MS1G、MSK1分别为6、5、5、5、3、3、2,VlhA基因高拷贝存在各菌株中,其拷贝数分别为17、16、17、13、14、25、13个,表明VlhA是MS主要的毒力因子,对MS的致病性具有重大作用,剩余毒力基因7株MS拷贝数均为1。详见表 6。
|
|
表 6 7株MS分离株毒力因子种类及数量 Table 6 Types and numbers of virulence factors in 7 MS isolates |
2.5.1 全基因组进化图 用MEGAX软件将7株MS分离株与NCBI上收录的8株MS全基因组比较,发现7株MS四川分离株与NCBI上收录的HN01基因组最接近,而与NCBI上其余7株MS遗传距离较远,7株MS分离株间基因组存在差异,MS251、MS221和MSK1聚为一小支,MS231、MS12H和MS254、MS1G与HN01聚为另一小支。详见图 1。
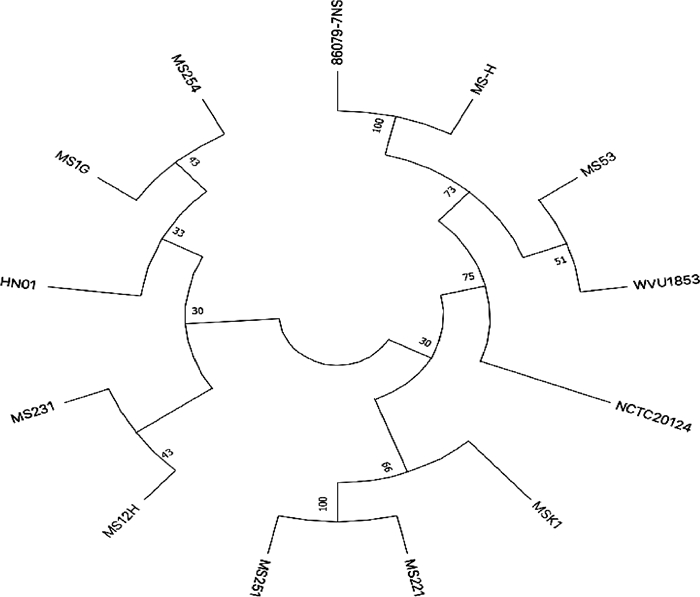
|
图 1 7株MS和8株NCBI登录菌株进化图 Fig. 1 Evolution diagram of 7 MS and 8 NCBI registered strains |
2.5.2 全基因组比较圈图 用MEGAX软件将7株MS分离株与NCBI上收录的8株MS全基因组进行圈图比较(图 2),发现7株MS四川分离株与巴西分离株MS53、美国分离株WVU-1853、澳大利亚分离株86079/7 NS以及疫苗株MS-H差异较大,而与中国河南分离株HN01最相近,但在37~50、190~220、490~500、530~560、680~780、790~820 kb 6处片段区域与HN01存在差异(图 2)。
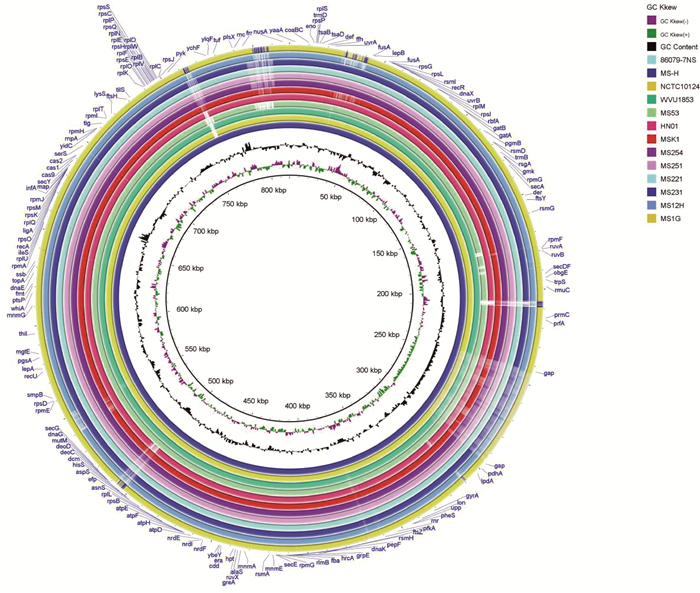
|
图 2 全基因比较圈图 Fig. 2 Comparison circle of the whole gene |
2.5.3 与HN01差异区域线性分析 使用R语言中的genoPlotR作图,将分离株与HN01株6处差异片段区域进行线性比较。比较37~50 kb片段区域, 发现HN01上存在的3个基因lepB、NA和fusA在7株MS四川分离株上分布不一致。lepB基因在MS251、MS254、MS221、MS12H呈长片段对应,但是片段倒位,与HN01方向相反,且位置也发生改变,而在MS231、MS1G和MSK1上呈小片段对应。NA和fusA对应片段都是散射性地分布在7株MS四川分离株间,且片段较短(图 3)。

|
红色线段和红色边框线段代表与HN01的基因方向相反,蓝色线段代表与HN01的基因方向相同,下图同
RuvB. Holliday_junction_ATP-dependent_DNA_helicase_RuvB; Pa. Phosphate_acyltransferase; R3. Ribonuclease_3; aRlmCD. 23S_rRNA_(uracil-C(5))-methyltransferase_aRlmCD; MnmA. tRNA-specific_2-thiouridylase_MnmA; Lld. L-lactate_dehydrogenase; RecO. DNA_repair_protein_RecO; A. DNA_topoisomerase_4_subunit_A; Pgdp1. Putative_glycerophosphoryl_diester_phosphodiesterase_1; IV. DNA_polymerase_IV; Cop1. ComE_operon_protein_1; Ef4. Elongation_factor_4; FtsH. ATP-dependent_zinc_metalloprotease_FtsH; PcrA. ATP-dependent_DNA_helicase_PcrA; RR. Ribonuclease_R; Gtl. Glycine--tRNA_ligase; NrnA. Bifunctional_oligoribonuclease_and_PAP_phosphatase_NrnA The red line segment and the red border line represent the opposite direction to the HN01 gene, the blue line represents the same direction as the HN01 gene, the same as below 图 3 37~50 kb间线性比较图 Fig. 3 Linear comparison between 37-50 kb |
比较190~220 kb序列,发现HN01上的rumC在MS251、MS254、MS221、MS12H上都有对应长片段分布,片段倒位,与HN01方向相反,存在移位。NA在MS251、MS254、MS221上有对应长片段,倒位,有移位。secDF、obgE、trpS则在7株MS四川分离株上呈散在分布的小片段,既有同向的,也有反向的。在7株MS分离株上此基因片段间都有OPPF和sugC 2个毒力因子片段(图 4)。

|
RecO. DNArepair_protein_RecO; RuvB. Holliday_junction_ATP-dependent_DNA_helicase_RuvB; RibF. Putative_riboflavin_biosynthesis_protein_RibF; OppF. Oligopeptide_transport_ATP-binding_protein_OppF; MnmG. tRNA_uridine_5-carboxymethylaminomethyl_modification_enzyme_MnmG; SugC. Trehalose_import_ATP-binding_protein_SugC; NrnA. Bifunctional_oligoribonuclease_and_PAP_phosphatase_NrnA 图 4 190~220 kb间线性比较图 Fig. 4 Linear comparison between 190-220 kb |
490~500 kb间,HN01上的5个基因与7株MS分离株分布不一致。efp在MS231上有长片段对应,在MS251、MS254、MS221、MS12H上有部分片段对应,长度相同,位置对应,基因片段倒位,与HN01株方向相反,有移位。dcm在MS231和MS1G分离株上有长片段对应,倒位,在其余5株上对应片段散在分布,片段较短,方向与HN01上既有相同的也有相反的。NA、aspS和hisS基因在7株MS四川分离株上对应片段散在分布,片段较短(图 5)。
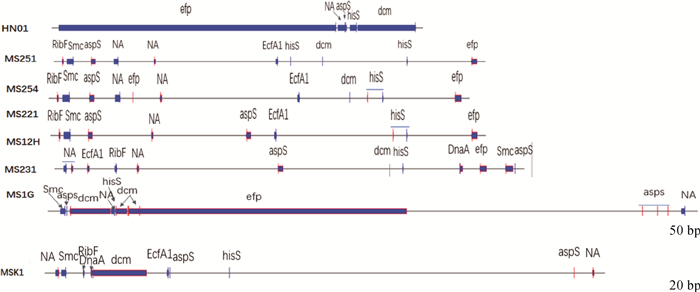
|
RibF. Putative_riboflavin_biosynthesis_protein_RibF; Smc. Chromosome_partition_protein_Smc; EcfA1. Energy-coupling_factor_transporter_ATP-binding_protein_EcfA1; DnaA. Chromosomal_replication_initiator_protein_DnaA; OppF. Oligopeptide_transport_ATP-binding_protein_OppF 图 5 490~500 kb线性比较图 Fig. 5 Linear comparison between 490-500 kb |
530~560 kb间差异片段基因比较结果表明:HN01只有1个基因NA在MS231和MS1G上有长片段对应,倒位,且有位移。在MS251、MS254、MS221、MS12H上有NA基因对应小片段,且位置相同(图 6)。
|
图 6 530~560 kb线性比较图 Fig. 6 Linear comparison between 530-560 kb |
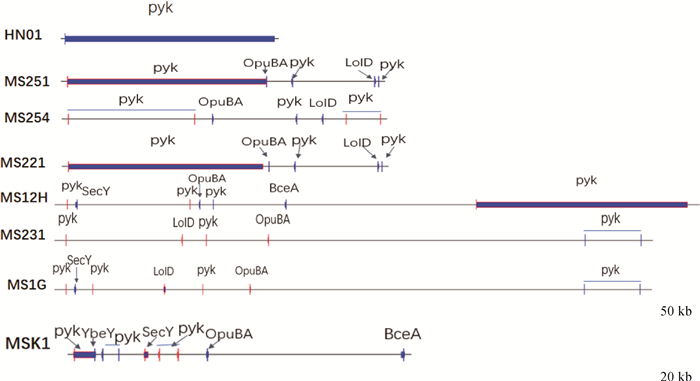
在680~780 kb间,HN01上有1个基因pyk在MS251、MS221、MS12H上有该基因长片段对应,但都倒位,MS251和MS221位置相同,而MS12H位置不同,其他4株MS只有小片段对应,且方向不同(图 7)。
|
OpuBA. Transport_ATP-binding_protein_OpuBA; LolD. Lipoprotein-releasing_system_ATP-binding_protein_LolD; SecY. preprotein_translocase_subunit_SecY; BceA. Bacitracin_export_ATP-binding_protein_BceA; YbeY. Endoribonuclease_YbeY 图 7 680~780 kb线性比较图 Fig. 7 Linear comparison between 680-780 kb |
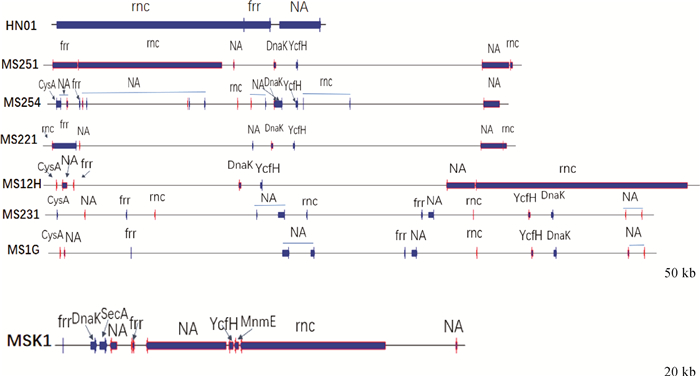
790~820 kb之间,HN01上有3个基因,其中rnc在MS251和MS12H上有大片段对应,倒位,且基因位置不同。NA在MS251、MS254、MS221、MS12H上有基因片段对应,位置相似,但倒位,如图 8所示。
|
DnaK. Chaperone_protein_DnaK; YcfH. putative_deoxyribonuclease_YcfH; CysA. Sulfate/thiosulfate_import_ATP-binding_protein_CysA; SecA. preprotein_translocase_subunit_SecA; MnmE. tRNA_modification_GTPase_MnmE 图 8 790~820 kb线性比较图 Fig. 8 Linear comparison between 790-820 kb |
以上结果表明6段基因线性比较,MS251、MS254、MS221、MS12H结构相似,MS231和MS1G相似,MSK1较短,与其他6株MS差异较大。7株MS分离株中除了HN01的基因外,在6个区域中,还含有其他的基因,包括一些酶、生长因子、毒力因子。其中图 4中,7株MS上都有毒力基因OPPF和sugC。这6个区域的差异对于四川分离株生物学特性及理化性质可能具有影响。
3 讨论近年来,四川地区鸡群MS感染较为严重。本试验前期从四川地区4个暴发MS的鸡场分离出7株MS,并对其致病性和耐药性等生物学特性进行了研究。MS感染对鸡和火鸡的养殖影响严重[15]。MS-H株是世界上唯一的MS弱毒疫苗株[16]。研究发现MS-H对国内鸡群MS的预防有的有效,有的效果差,甚至无效[17]。因此MS流行毒株全基因组测序分析对MS疫苗株的筛选、亚单位疫苗的开发和特异性检测技术的建立是一项基础性的工作。
本研究对7株MS四川分离株进行全基因组测序,eggNOG根据序列的相似性寻找直系同源基因,基因编码蛋白序列功能的注释有COG、GO、KEGG等多数据库形式,多数据库比对保证了基因注释的全面性,各数据库比对结果相似,确定7株分离菌株均为MS,其中来自同一鸡场的4株MS在基因数量及种类上差异较小,而与分离自另外3个鸡场的3株MS差异较大,表明MS四川分离株存在丰富的遗传多样性。
PHI数据库是研究病原与机体相互作用的专业数据库,用于研究致病菌与宿主间的相互作用[18]。PHI数据库分析7株MS导致病原菌致病能力降低的基因,除了共有基因,MS251、MS254、MS221、MS231还含有MoDeam基因,MS254特有glcA基因,MS12H、MS1G、MSK1含有FgTRR (FGSG_00871)和CaNAG1基因。致死因子中,MS251、MS254、MS221、MS231只含有GzOB009、GzOB006,而MS12H、MS1G、MSK1还含有TRR1基因。这些基因的差异可能与各MS分离株的致病性有关,推测MS251、MS254、MS221可能比MS12H、MS1G、MSK1有更强的致病性。TRR1基因表达硫氧还蛋白还原酶,Trr1可以降低Trx2-5,从而控制细胞内的氧化还原状态,深刻影响球孢白僵菌对节肢动物的杀灭潜力。这些基因对MS致病性的影响还需要进一步的试验验证。药敏试验中发现7株分离株对土霉素、大观霉素、泰乐菌素、替米考星等敏感,对红霉素的MIC值较高,但基因库分析7株MS只有1个抗药基因GyrA具有抗氟喹诺酮类药物的能力。
VFDB数据库是记录大量毒力因子序列信息的数据库[19],毒力因子是微生物代谢产生的具有侵袭力和毒力的分子,帮助微生物逃避或抑制宿主免疫反应。本研究将MS四川分离株序列信息导入VFDB数据库分析,发现MS含有25种毒力因子,大多数毒力因子为单拷贝数,但msbA、sugC、oppF、VlhA、tnp为多拷贝数。MS中主要的毒力因子基因大多数与黏附和侵袭力有关。如VlhA在7株MS四川分离株中以多拷贝形式存在,拷贝数最小13,最大25,该基因编码蛋白包含变脂蛋白(MSPB)和可变血凝素蛋白(MSPA),介导MS黏附、侵袭和免疫逃避,是重要的毒力因子[20-21]。除VlhA外,pdhb、eno、CT396、MG-301等毒力基因,都已证明与支原体的黏附有关。前期初步研究显示7株MS分离株对红细胞具有吸附能力,以及对鸡胚和雏鸡有感染能力,但未对菌株间致病力强弱进行比较,因此需要在后续试验中,验证菌株毒力基因差异对致病力的影响。
用MEGAX软件,将7株MS四川分离株与NCBI上收录的8株MS全基因组比较,发现7株MS与中国河南HN01株基因组最近,且与世界各国流行株差异较大,说明中国MS流行株有其独特性[11]。进一步通过全基因组圈图比较,发现7株MS四川分离株和HN01株在全基因组上共有6个差异区域,在6个差异区域内,既包含HN01上的同源基因,还含有其他的差异基因,且同源基因长度相似,但有的出现倒位,这可能对MS的生物学特性产生影响。支原体DNA复制和修复功能的部分缺失可以阻止大的基因组倒位的形成[22],已有研究发现MS-H基因组中存在55 kb的倒位,而MS-H亲本株86079/7 NS的全基因组中也存在相同的倒位,证实基因组倒置不是由化学诱变引起的,而是自然发生的[10, 23]。因此7株MS基因组中基因的倒位也可能是自然发生的。
在与HN01差异区域190~220 kb,7株MS四川分离株都存在毒力基因OppF和sugC,OppF为细胞质ATP酶,为肽的转运提供能量[24-25],对MS在气管黏膜上皮细胞的定殖必不可少[26]。在MS-H疫苗株中oppF的移码突变,导致其编码蛋白的改变,最终导致该蛋白正常功能的丧失[23],而在测序结果中显示OPPF提前终止,没有表达出完整的蛋白。sugC在结核分枝杆菌中编码一种ABC转运蛋白,参与海藻糖的输入,ABC转运蛋白的一个功能是作为ATPase的结构域[27]。除了毒力因子基因片段外,差异区域也包括一些编码酶和其他各种因子的基因,这些差异可能影响MS的生物学特性。比较7株MS四川分离株之间6个差异区域发现,总体上,MS251、MS254、MS221基因结构相似;MS1G和MS231相近;MSK1单独一支,且每个差异区域片段均较短。表明四川分离株在同场和不同场间,都存在遗传多样性。
4 结论目前,中国各个地区均存在MS感染。四川地区MS分离株全基因组比较分析发现,分离株与我国河南分离株HN01相近,与世界上主要MS流行株不同,可称为中国流行株。四川地区MS分离株与HN01株基因组的6个差异基因区域编码基因片段长度和方向倒位等变化,说明四川分离株发生了变异。而7株MS四川分离株间6个差异基因区域的不同,表明四川分离株不仅在不同场间,而且在同场间也存在遗传多样性。
| [1] |
PASCUCCI S, MAESTRINI N, GOVONI S, et al. Mycoplasma synoviae in the guinea-fowl[J]. Avian Pathol, 1976, 5(4): 291-297. DOI:10.1080/03079457608418198 |
| [2] |
KLEVEN S H. Mycoplasmas in the etiology of multifactorial respiratory disease[J]. Poult Sci, 1998, 77(8): 1146-1149. DOI:10.1093/ps/77.8.1146 |
| [3] |
MOREIRA F A, CARDOSO L, COELHO A C. Epidemiological survey on Mycoplasma synoviae infection in Portuguese broiler breeder flocks[J]. Vet Ital, 2015, 51(2): 93-98. |
| [4] |
FEBERWEE A, DE WIT J J, LANDMAN W J M. Induction of eggshell apex abnormalities by Mycoplasma synoviae: field and experimental studies[J]. Avian Pathol, 2009, 38(1): 77-85. DOI:10.1080/03079450802662772 |
| [5] |
陈秀红, 郭亚男, 司朵朵, 等. 宁夏地区鸡滑液囊支原体病流行病学调查与分析[J]. 中国预防兽医学报, 2020, 42(3): 234-238. CHEN X H, GUO Y N, SI D D, et al. Epidemiological investigation and analysis of Mycoplasma synoviae in infection in chickens in Ningxia region[J]. Chinese Journal of Preventive Veterinary Medicine, 2020, 42(3): 234-238. (in Chinese) |
| [6] |
马爽, 郭莉莉, 宋新宇, 等. 鸡滑液囊支原体感染的流行病学调查与分析[J]. 中国家禽, 2016, 38(23): 68-71. MA H, GUO L L, SONG X Y, et al. Epidemiological investigation and analysis of Mycoplasma synovial infection in chickens[J]. China Poultry, 2016, 38(23): 68-71. (in Chinese) |
| [7] |
SUN S K, LIN X, CHEN F, et al. Epidemiological investigation of Mycoplasma synoviae in native chicken breeds in China[J]. BMC Vet Res, 2017, 13(1): 115. DOI:10.1186/s12917-017-1029-0 |
| [8] |
VASCONCELOS A T R, FERREIRA H B, BIZARRO C V, et al. Swine and poultry pathogens: the complete genome sequences of two strains of Mycoplasma hyopneumoniae and a strain of Mycoplasma synoviae[J]. J Bacteriol, 2005, 187(16): 5568-5577. DOI:10.1128/JB.187.16.5568-5577.2005 |
| [9] |
MAY M A, KUTISH G F, BARBET A F, et al. Complete genome sequence of Mycoplasma synoviae strain WVU 1853T[J]. Genome Announc, 2015, 3(3): e00563-15. |
| [10] |
ZHU L, SHAHID M A, MARKHAM J, et al. Comparative genomic analyses of Mycoplasma synoviae vaccine strain MS-H and its wild-type parent strain 86079/7 NS: implications for the identification of virulence factors and applications in diagnosis of M. synoviae[J]. Avian Pathol, 2019, 48(6): 537-548. DOI:10.1080/03079457.2019.1637514 |
| [11] |
XU B, LIU R, TAO L H, et al. Complete genome sequencing of Mycoplasma synoviae strain HN01, isolated from chicken in Henan province, China[J]. Microbiol Resour Announc, 2020, 9(6): e01480-19. |
| [12] |
MARKHAM J F, SCOTT P C, WHITHEAR K G. Field evaluation of the safety and efficacy of a temperature-sensitive Mycoplasma synoviae live vaccine[J]. Avian Dis, 1998, 42(4): 682-689. DOI:10.2307/1592703 |
| [13] |
田亚琴, 文玉康, 王豪举, 等. 鸡滑液囊支原体疫苗研究进展[J]. 中国家禽, 2020, 42(9): 96-102. TIAN Y Q, WEN Y K, WANG H J, et al. Research progress in the development of Mycoplasma synoviae vaccine[J]. China Poultry, 2020, 42(9): 96-102. (in Chinese) |
| [14] |
李凡, 张焕容, 陈云霞. 川西地区鸡滑液囊支原体分离株的部分生物学特性及其VlhA基因遗传进化分析[J]. 中国预防兽医学报, 2020, 42(2): 122-127. LI F, ZHANG H R, CHEN Y X. Partial biological charicteristics of Mycoplasma synoviae isolates from western Sichuan analysis of it VIhA gene[J]. Chinese Journal of Preventive Veterinary Medicine, 2020, 42(2): 122-127. (in Chinese) |
| [15] |
CATANIA S, BILATO D, GOBBO F, et al. Treatment of eggshell abnormalities and reduced egg production caused by Mycoplasma synoviae infection[J]. Avian Dis, 2010, 54(2): 961-964. DOI:10.1637/9121-110309-Case.1 |
| [16] |
NOORMOHAMMADI A H, HEMMATZADEH F, WHITHEAR K G. Safety and efficacy of the Mycoplasma synoviae MS-H vaccine in turkeys[J]. Avian Dis, 2007, 51(2): 550-554. DOI:10.1637/0005-2086(2007)51[550:SAEOTM]2.0.CO;2 |
| [17] |
石晓磊. 鸡滑液囊支原体的分离鉴定及其活疫苗免疫效果的评价[D]. 杨凌: 西北农林科技大学, 2018. SHI X L. Isolation and identification of Mycoplasma synoviae and evaluation of the effect of live-vaccine[D]. Yangling: Northwest A&F University, 2018. (in Chinese) |
| [18] |
URBAN M, CUZICK A, RUTHERFORD K, et al. PHI-base: a new interface and further additions for the multi-species pathogen-host interactions database[J]. Nucleic Acids Res, 2017, 45(D1): D604-D610. DOI:10.1093/nar/gkw1089 |
| [19] |
KLEINHEINZ K A, JOENSEN K G, LARSEN M V. Applying the ResFinder and VirulenceFinder web-services for easy identification of acquired antibiotic resistance and E. coli virulence genes in bacteriophage and prophage nucleotide sequences[J]. Bacteriophage, 2014, 4(2): e27943. DOI:10.4161/bact.27943 |
| [20] |
NOORMOHAMMADI A H, MARKHAM P F, DUFFY M F, et al. Multigene families encoding the major hemagglutinins in phylogenetically distinct mycoplasmas[J]. Infect Immun, 1998, 66(7): 3470-3475. DOI:10.1128/IAI.66.7.3470-3475.1998 |
| [21] |
EL-GAZZAR M M, WETZEL A N, RAVIV Z. The genotyping potential of the Mycoplasma synoviae vlhA gene[J]. Avian Dis, 2012, 56(4): 711-719. DOI:10.1637/10200-041212-Reg.1 |
| [22] |
SUYAMA M, BORK P. Evolution of prokaryotic gene order: genome rearrangements in closely related species[J]. Trends Genet, 2001, 17(1): 10-13. DOI:10.1016/S0168-9525(00)02159-4 |
| [23] |
ZHU L, SHAHID M A, MARKHAM J, et al. Genome analysis of Mycoplasma synoviae strain MS-H, the most common M. synoviae strain with a worldwide distribution[J]. BMC Genomics, 2018, 19(1): 117. DOI:10.1186/s12864-018-4501-8 |
| [24] |
MONNET V. Bacterial oligopeptide-binding proteins[J]. Cell Mol Life Sci, 2003, 60(10): 2100-2114. DOI:10.1007/s00018-003-3054-3 |
| [25] |
GARDAN R, BESSET C, GUILLOT A, et al. The oligopeptide transport system is essential for the development of natural competence in Streptococcus thermophilus strain LMD-9[J]. J Bacteriol, 2009, 191(14): 4647-4655. DOI:10.1128/JB.00257-09 |
| [26] |
MORAES P M R O, SEYFFERT N, SILVA W M, et al. Characterization of the Opp peptide transporter of Corynebacterium pseudotuberculosis and its role in virulence and pathogenicity[J]. BioMed Res Int, 2014, 2014: 489782. |
| [27] |
SABHARWAL N, VARSHNEY K, RATH P P, et al. Biochemical and biophysical characterization of nucleotide binding domain of Trehalose transporter from Mycobacterium tuberculosis[J]. Int J Biol Macromol, 2020, 152: 109-116. DOI:10.1016/j.ijbiomac.2020.02.237 |
(编辑 白永平)



