家禽的就巢性影响着其产蛋性能,就巢性越强的品种产蛋性能越低。卵泡的发育与环境、营养和激素调控有关系,决定着家禽就巢行为的发生和维持,对产蛋性能具有重要影响。卵泡发育取决于细胞的增殖、分化和卵黄质大量沉积,进而促进了卵泡质量的快速增长[1]。在家禽生产过程中,一些与卵泡发育相关的基因参与调控家禽的就巢性。随着家禽选育程度的不断提高,多基因调控研究在家禽遗传改良中得到广泛的应用。
边鸡是肉蛋兼用的中国地方品种,主要分布在内蒙古自治区与山西省北部相毗连的长城内外一带,具有蛋重大、肉质好、适应性强、耐粗饲和抗寒等优点。由于边鸡具有很强的就巢性,产蛋量很少,限制了该品种资源的开发和利用。因此,提高边鸡产蛋性能已受到相关家禽育种研究者的广泛关注。家禽育种研究者通常以卵巢作为研究家禽就巢性的试验材料,从分子水平上研究与就巢性相关基因的差异表达。江红霞等[2]对克氏原螯虾的卵巢和肝胰腺进行转录组测序,共获得53 006个单基因(unigene),卵巢与肝、胰对比分析,发现卵巢中有20 382个差异性表达基因(differentially expressed genes, DEGs),通过GO功能分类分析发现,部分DEGs被注释到繁殖(reproduction)、繁殖过程(reproduction process)、免疫系统过程(immune system process)和生长(growth)GO条目。孟金柱等[3]对贵州白山羊的优势卵泡和从属卵泡进行转录组测序,筛选出233个上调表达基因,并对这些基因进行GO、KEGG分析,最终筛选出4个与卵泡发育有关的基因。郝庆玲等[4]对山西省文水县海福特母牛的不同生理阶段发育卵泡进行转录组分析,共获得608个差异表达基因,其中有9个参与牛卵泡发育的信号通路调控,并筛选出11个直接参与信号通路调节及细胞增殖分化的基因。朱志明等[5]对山麻鸭开产期和产蛋高峰期卵巢组织转录组测序,发现存在1 929个差异表达基因,其中上调基因989个,下调基因940个,并将这些基因在Nr、GO、KEGG数据库进行分析比较,获得了差异表达基因的功能、分类和代谢通路。Shen等[6]使用转录组测序来分析靖黄鸡三种类型的卵泡细胞中circRNA和mRNA,利用生物信息学分析确定了14 502个circRNA,其中5 622个广泛分布在卵泡细胞发育的所有阶段,差异表达分析表明,在与生殖相关的途径中富集了不同表达的circRNA和mRNA,包括TGF-β信号通路、卵母细胞减数分裂和血管平滑肌收缩,为卵泡发育提供了理论依据。董新龙等[7]利用转录组对京海黄鸡卵巢组织进行分析,发现了4 431个新转录本,这些新转录本比对到GO、Nr、KEGG数据库的数量分别为917、1 795、422。与这些数据库的成功比对,为进一步证实新基因的存在提供了基础;此外,经过与KEGG数据库比对,发现丝裂原活化蛋白激酶信号通路(MAPK signaling pathway)排在富集通路的首位。虽然转录组测序已经在畜禽不同生理阶段卵巢组织差异表达基因的研究中广泛应用,但是目前尚无对于边鸡卵巢组织转录组差异表达基因的研究。本研究为了挖掘边鸡卵巢卵泡发育过程中起调节作用的遗传因子,揭示卵巢发育期、开产期、产蛋高峰期和就巢期卵巢组织的差异表达基因,筛选与卵泡发育相关的候选基因,利用转录组测序技术构建不同日龄的边鸡卵巢的转录组文库,对边鸡在不同生理状态下卵巢组织做转录组差异分析、差异表达基因GO功能富集分析和KEGG分析,将差异表达基因进行功能注释,旨在研究与边鸡卵巢发育相关的基因,以揭示边鸡就巢性强的遗传机制。
1 材料与方法 1.1 试验动物和卵巢样本采集试验用边鸡在标准化饲养条件下进行饲养管理,每次取材前进行连续7 d的产蛋观察:产下第一枚蛋起,表示性成熟;产蛋率达到70%以上,代表产蛋高峰;320日龄后产蛋率下降为30%以下,观察记录一些健康的停产并表现出就巢行为的个体。随机选取70日龄(卵巢发育期)、165日龄(开产期)、220日龄(产蛋高峰期)和330日龄(就巢期)健康、体重相近且符合取样条件的边鸡个体,每个日龄5只,颈静脉放血法处死,采集卵巢组织,迅速投入液氮中,转运后置于-80 ℃超低温冰箱中保存备用。
1.2 cDNA文库构建和测序采用TRIzol试剂(美国Invitrogen)提取不同日龄边鸡的卵巢组织总RNA,用Agilent 2100生物分析仪进行总RNA品质检测。用带有poly-T oligo附着的磁珠富集含有poly A的mRNA,使用逆转录酶和随机引物将mRNA反转录合成第一条cDNA链,使用DNA polymerase I和RNase H合成第二条cDNA链。通过PCR扩增方法纯化和富集cDNA,构建边鸡卵巢cDNA文库。在BGISEQ-500平台进行测序(华大基因公司,深圳,中国)。
1.3 生物信息学分析过滤低质量片段,去除带接头污染和未知碱基含量比例过高的片段获得clean reads。Clean reads与参考基因组比对预测新基因和转录本,检测基因剪接事件,识别卵巢样本间的差异表达基因(DEGs),进行聚类分析和功能注释。
1.4 新转录本预测利用HISAT软件将clean reads定位到鸡参考基因组。基因组定位后,用StringTie[8]重建转录本,基于基因组注释信息,用Cuffcompare[9]识别新的转录本,并使用CPC[10]预测新转录本的编码能力。
1.5 基因表达、DEGs检测、GO功能富集和KEGG分析利用Bowtie2[11]将新的编码转录本与转录本合并,将clean reads映射成完整的参考基因组后,利用RSEM[12]计算基因表达水平。使用Cor计算卵巢样本间的Pearson相关性,使用R软件里的hclust函数进行层次聚类分析,图形的绘制采用了R软件中的ggplot2包。
采用PossionDis[13]算法检测不同日龄边鸡卵巢间的差异表达基因。根据GO注释结果,对DEGs进行功能分类和生物通路分类,同时使用R软件中的phyper函数进行GO的功能和KEGG富集,用超几何分布函数计算出P值,计算每个P值的错误发现率(FDR),当FDR≤0.01时则被认为显著富集。
1.6 实时荧光定量PCR(qPCR)验证为了验证转录组测序的结果,从已知参与卵泡发育的差异表达基因中随机选择11个基因,通过qPCR进行验证。将上述从测序同样的个体样本中提取的不同日龄边鸡卵巢的总RNA用MMLV逆转录酶(TaKaRa,大连,中国)将其逆转录为cDNA。通过SYBR方法进行qPCR验证(引物见表 1)。扩增条件为:预变性95 ℃ 30 s;(变性95 ℃ 5 s、退火55 ℃ 30 s、延伸72 ℃ 10 s)×40循环;熔解过程(95 ℃ 1 min、55 ℃ 30 s、55 ℃ 30 s)。内参基因β-actin与目的基因的样本要在同样的条件下进行qPCR,通过2-△△CT法计算目的基因的相对表达量,结果以“平均值±标准差(Means±SD)”表示,用Graph-Pad Prism 8.0软件分析数据,采用one-way ANOVA进行单因素方差分析。每个样本做4次技术重复。
|
|
表 1 引物序列 Table 1 Primers sequences |
通过在BGISEQ-500平台上对不同日龄边鸡卵巢样本进行测序,构建基因表达谱,每个样品约产生11.04 Gb的序列。共鉴定出18 891个基因,其中已知基因18 063个,预测的新基因828个。此外,共检测出21 299个新的转录本,其中14 376个属于已知基因且具有蛋白编码潜力的新转录本,925个属于新基因的具有蛋白编码潜力的新转录本,其余5 998个为长链非编码RNA。
2.2 不同生理阶段边鸡卵巢基因表达及相关性分析通过RSEM分析每组样本的基因表达水平,计算每两个样本之间的Pearson相关性并对所有样本进行层次聚类分析。结果表明,在165与220 d边鸡卵巢基因表达聚类最近,二者间的Pearson值为0.983,说明有显著相关性;而70和330 d与165和220 d边鸡卵巢基因表达均未聚类,且相互间相关性相对较低(图 1)。

|
样品间彼此靠的越近代表样品的表达谱越相似 The closer the samples are to each other, the more similar the expression profiles of the samples are 图 1 不同日龄边鸡卵巢表达基因的层次聚类和Pearson相关性 Fig. 1 Hierarchical clustering and Pearson correlation of genes expressed among the ovaries of Bian chicken at different ages |
通过PossionDis计算差异表达基因水平,70、165、220和330 d边鸡4个发育阶段的卵巢转录组数据两两对比,形成6组差异表达的mRNA(图 2)。通过比较发现,70 d边鸡卵巢转录组与其他阶段差异最大,获得的差异表达基因也相对较多,而且下调基因的数量大于上调基因;而165与330 d相比、220与330 d相比,差异表达基因的上调数量大于下调基因的数量;165与220 d相比,差异表达基因的总量最少。
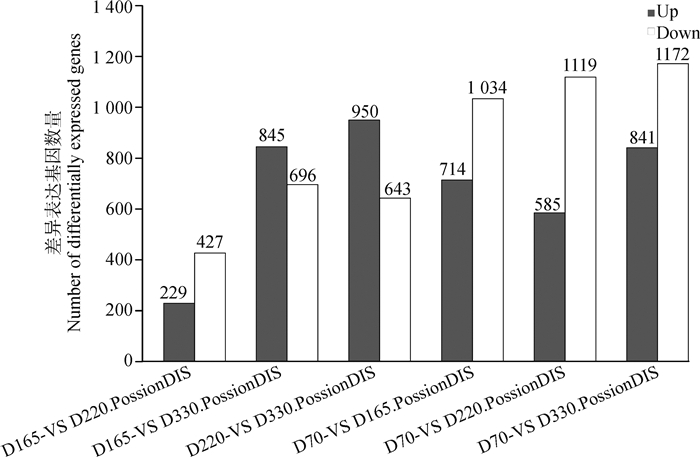
|
图 2 不同日龄边鸡卵巢中的差异表达基因 Fig. 2 Differentially expressed genes in the ovary of Bian chickens at different ages |
以差异倍数>1.5,FDR < 0.01为标准,使用DESeq2R包对转录组数据进行筛选,找出了在不同生理阶段与发育卵泡数变化相关的差异表达基因40个(表 2)。结果表明,有18个基因参与卵泡发育的整个过程,有22个基因仅在220 d前表达。其中35个基因的表达量与发育卵泡数成正相关,FDX1L在70、220、165和330 d边鸡卵巢中表达量依次递减,NDRG4在70、165、220和330 d边鸡卵巢中表达量随日龄增加呈递减趋势,MC3R和PRLHR在70和330 d边鸡卵巢中表达量高于165和220 d的表达量,VIP仅在70、165和220 d边鸡卵巢中表达。
|
|
表 2 不同日龄边鸡卵巢中可能与卵泡发育相关的差异表达基因及其表达量(RPKM值) Table 2 The number and expression level of DEGs (RPKM value) which were potentially related to the follicle development in ovaries of Bian chickens with different ages |
GO分为三大类,即生物过程、细胞组分、分子功能。将4个混合样本相互组合形成6个对比组,对筛选获得的差异表达基因进行GO功能富集分析并绘制差异表达基因GO富集柱状图(图 3)。结果表明:70与165 d相比,共有1 748个差异基因,其中有615个得到功能注释;70与220 d相比,有1 704个差异基因,其中596个得到功能注释;70与330 d相比,有2 013个差异基因,其中有622个得到功能注释;165与220 d相比,有656个差异基因,其中169个得到功能注释;165与330 d相比,有1 541个差异基因,其中有480个得到功能注释;220与330 d相比,有1 593个差异基因,其中有474个得到功能注释。差异基因主要集中在“生物过程”,其次是“细胞组分”,最后是“分子功能”。对比6个组合的差异基因功能富集GO term,其差异基因在“生物过程”分类中,细胞过程(cellular process)所占比例最多,其次是生物调控(biological regulation)和生物过程调控(regulation of biological process),在“细胞组分”分类中,细胞(cell)占的比例最大,其次是细胞部分(cell part)和细胞膜等;在“分子功能”分类中,结合(binding)占的比例最多,其次是催化活性(catalytic activity)。此外,可以看到“生物过程”分类中与发育繁殖相关的有发育过程(developmental process)、繁殖(reproduction)、繁殖过程(reproductive process)。在70与165 d相比,共涉及272个与发育繁殖相关的差异表达基因;70与220 d相比,共涉及269个与发育繁殖相关的差异表达基因;70与330 d相比,共涉及262个与发育繁殖相关的差异表达基因;165与220 d相比,共涉及57个与发育繁殖相关的差异表达基因;165与330 d相比,共涉及194个与发育繁殖相关的差异表达基因;220 d与330 d相比,共涉及188个与发育繁殖相关的差异表达基因。
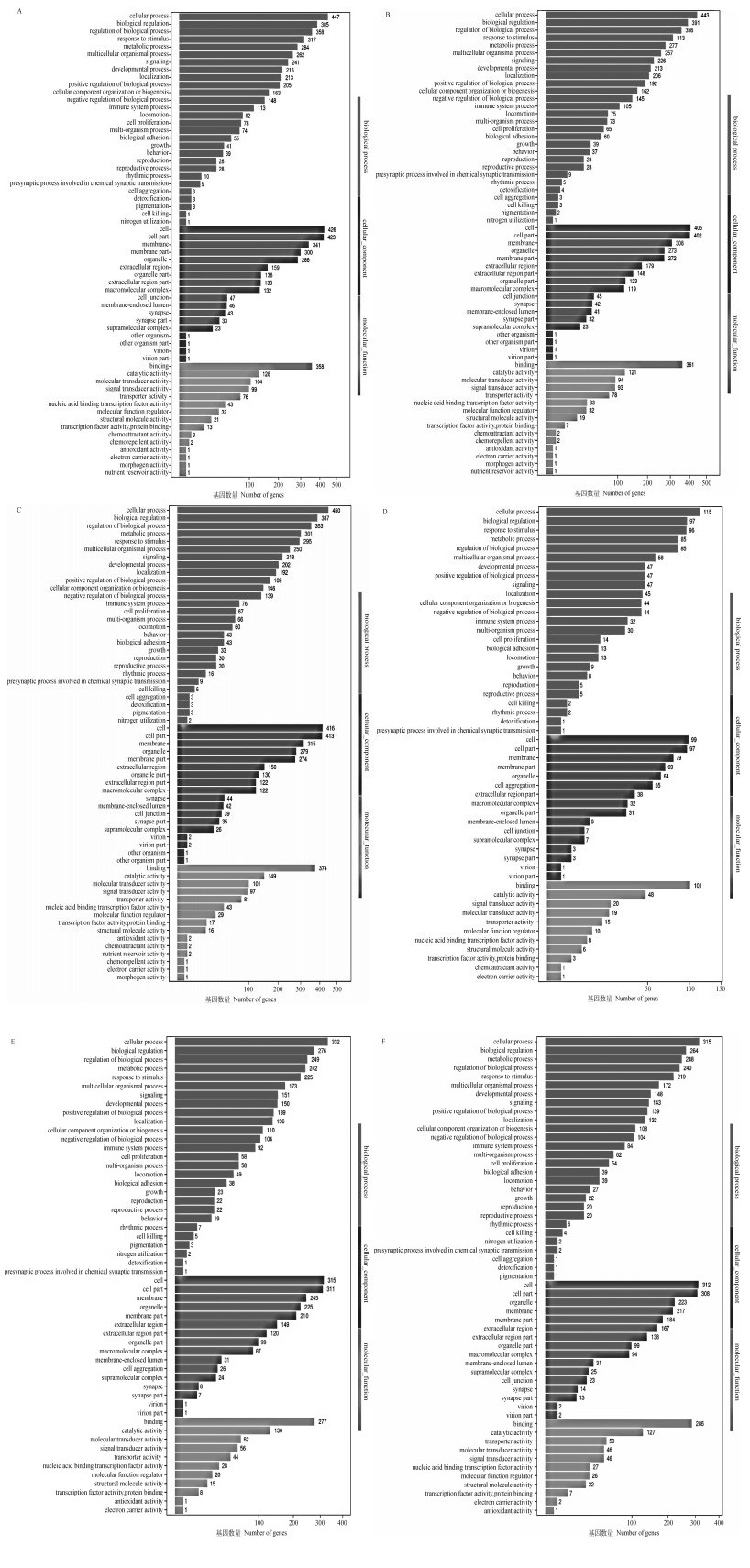
|
A、B、C、D、E、F分别是70 d vs. 165 d、70 d vs. 220 d、70 d vs. 330 d、165 d vs. 220 d、165 d vs. 330 d、220 d vs. 330 d; X轴表示DEGs数目,Y轴表示GO功能分类 A, B, C, D, E, F are 70 d vs. 165 d, 70 d vs. 220 d, 70 d vs. 330 d, 165 d vs. 220 d, 165 d vs. 330 d, 220 d vs. 330 d, respectively; X axis represents the number of DEGs, and Y axis represents GO functional classification 图 3 不同日龄边鸡卵巢差异表达基因GO富集柱状图 Fig. 3 Column map of GO enrichment of DEGs in ovary of Bian chickens at different ages |
通过对卵泡发育的不同生理阶段的边鸡卵巢差异表达基因KEGG通路比较均可发现信号转导、免疫系统以及信号分子与相互作用路径上富集最多(表 3)。
|
|
表 3 不同日龄边鸡卵巢中差异表达基因在KEGG路径富集较多的数量比较 Table 3 The numbers of DEGs in ovaries of Bian chickens with different ages, which were most enriched on KEGG pathways |
为进一步验证转录组测序的可靠性,本研究通过qPCR随机验证了11个差异基因的表达。结果表明,所选基因的mRNA表达趋势与转录组测序数据一致(图 4),可以证明转录组测序的结果可靠。
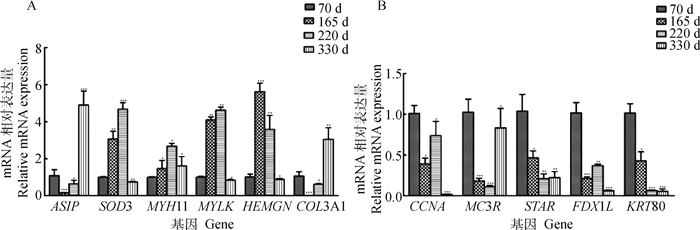
|
图 4 qPCR验证不同日龄边鸡卵巢中的差异表达基因 Fig. 4 Differentially expressed genes in the ovary of Bian chicken at different ages verified by the qPCR |
家禽的就巢行为与卵巢发育有直接联系,就巢行为的发生会引起产蛋性能的下降。转录组测序以它检测范围广、测序通量高、检测成本低、分辨率好和灵敏度高等特点,已经被广泛应用[14],在金定鸭的卵巢[15]、京海黄鸡卵巢[16]中都有应用。本研究对边鸡卵巢组织的转录组测序,共鉴定出18 891个基因,其中已知基因18 063个,预测的新基因828个。此外,共检测出21 299个新的转录本。本研究挖掘了边鸡卵巢组织转录组信息,为开展边鸡就巢性状相关基因的研究及分子调控机制研究奠定基础。
对所有样本间的相关性进行层次聚类分析,用pearson法计算每两个样本间所有基因表达量的相关系数,相关系数越接近1,表明这两个样本间的基因表达的相似度越高。本研究发现,在165和220 d之间的相关系数为0.983,基因表达相似度最高,说明卵巢发育成熟后,开始排卵和排卵高峰两个样本卵巢组织基因表达相关性最强,与层次聚类分析表达谱结果一致。从边鸡不同生理阶段卵巢组织差异表达基因数量显示,存在上调基因和下调基因,165与220 d相比,差异表达基因的总量最少(656个),与样本间的相关性进行层次聚类分析结果一致,这些差异表达基因可能促进卵泡发育。
本研究使用DESeq2R包对转录组数据进行筛选,以样本间的相关性和不同生理阶段卵泡数为筛选基础,找出了40个可能与卵泡发育相关的差异表达基因,qPCR随机验证所选基因的mRNA表达趋势与转录组测序数据一致,证明了转录组测序的结果可靠。其中35个基因的表达量与发育卵泡数成正相关,5个基因表达量与发育卵泡数不成正相关(铁氧还蛋白-1L(FDX1L)、N-Myc下游调控基因-4(NDRG4)、黑素皮质素受体(MC3R)、催乳素释放激素受体(PRLHR)和血管活性肠肽(VIP))。FDX1L基因编码一种183个氨基酸的线粒体蛋白Fdx2,它在铁-硫簇生物发生过程中起重要作用[17]。它们通常通过半胱氨酸的巯基侧链与假体蛋白结合,并作为电子受体或供体,在关键的细胞活动中发挥重要作用[18]。本试验中发现,FDX1L在70、220、165和330 d边鸡卵巢中表达量依次递减,表明该基因可能参与卵巢发育和调控排卵过程,作用机理需进一步研究探讨。NDRG4在多种癌症中被认为与肿瘤细胞增殖能力相关[19-20],NDRG4表达下调后卵巢癌细胞增殖能力增高、凋亡减少,其机制可能与调节Bcl-2/Bax表达及P38 /MAPK信号通路活性有关[21-22],NDRG4通过Akt/CREB激活促进肌原性分化[23],支持激活细胞外信号调节激酶(ERK)促进神经元分化,并减少参与神经元生长的Elk-1的激活[24]。现已有报道称Bcl-2与鹅就巢相关[25],虽在本试验中未发现Bcl-2在不同卵泡发育阶段的边鸡卵巢中的表达量呈差异性,但发现NDRG4在边鸡卵巢中的表达量随日龄增加呈递减趋势,NDRG4基因下调可能促进卵巢机能衰老,与上述研究中NDRG4基因下调后卵巢癌细胞增殖能力增高、凋亡减少的信号通路可能相关,卵巢机能衰老与卵巢癌细胞增殖能力的相关性需要进一步研究探讨。黑素皮质素受体(melanocortin receptors,MCRs)是G-蛋白偶联受体(G protein-coupled receptors,GPCRs)超家族成员之一[26]。人、鼠和鸡MCRs由5个成员组成,分别是MC1R、MC2R、MC3R、MC4R和MC5R,其中,MC3R基因不仅在外周组织中有一定表达,而且在大脑中呈现高表达,因此,又被称为神经性黑素皮质素受体基因[27-28]。现有研究发现,MC3R主要参与调控动物的体重和能量代谢的过程[29],也有研究发现,MC3R在人的心、乳腺、骨骼肌和肾以及睾丸和卵巢中表达[30],在这些组织中表达很可能与免疫功能相关[31]。有关MC3R表达对禽类繁殖性能的研究尚未见报道,本试验中发现,MC3R基因在边鸡卵巢70和330 d表达量高于165和220 d的表达量,推测MC3R可能起到抑制卵泡发育的作用,能够促进边鸡卵巢就巢性,这一发现为进一步研究卵泡发育及排卵调控机制奠定基础。李国辉等[32]发现,催乳素(PRL)和神经肽Y(NPY)基因作为鸡繁殖性能的候选基因,为多基因聚合育种技术在家禽遗传改良方面的应用。在本试验中虽未发现PRL,但发现催乳素释放激素受体(PRLHR)在卵泡发育期即165和220 d低表达,分别为1.14和1.16,而在未发育期70 d和就巢期330 d表达量相对较高,分别为3.13和2.72,PRLHR与NPY受体(NPYR)结合,从而影响PRL释放,进而调控边鸡的就巢性。Hosseinpour等[33]发现,血管活性肠肽(VIP)及其受体(VIPR-1)的SNP与火鸡产蛋性能相关,但未报道与就巢性相关。本试验发现,在70、165和220 d的边鸡卵巢中VIP呈差异性表达,但在就巢期即330 d的边鸡卵巢中未发现VIP表达,该基因表达可能参与卵巢发育,与Hosseinpour等[33]的结果一致。
本研究通过GO功能分析,发现边鸡不同日龄卵巢组织差异表达基因功能主要集中在“生物过程”。在“生物过程”中,与发育繁殖相关的通路有发育过程、繁殖和繁殖过程,富集在这些通路的差异表达基因可能参与卵泡的发育和排卵调控机制;而边鸡卵巢成熟开始排卵期间,即165与220 d相比,与发育繁殖相关的差异表达基因仅57个且数量最少,与本研究中卵巢基因差异表达相关性分析结果一致,这些少量的差异表达基因可能参与发育成熟卵泡数量的调控;220与330 d相比,共涉及188个与发育繁殖相关的差异表达基因,这些差异表达基因可能与边鸡就巢性的调控相关。对不同生理阶段的边鸡卵巢差异表达基因KEGG通路比较,均可发现这些差异基因在信号转导、免疫系统以及信号分子与相互作用的路径上富集最多。通过揭示这些差异表达基因的功能,为多基因聚合育种改良边鸡就巢性提供了可能。
4 结论本研究通过构建基因表达谱,获取边鸡卵巢组织转录组信息,筛选出40个与卵巢发育相关的差异表达基因,通过GO功能富集和KEGG分析,获得了差异表达基因的功能分类和通路,为开展边鸡卵泡发育相关基因的研究及分子调控机制研究奠定基础。
| [1] |
林欣. 蛋鸡退化卵泡衰退的机制及其功能的研究[D]. 杭州: 浙江大学, 2018. LIN X. Degradation and functions of the regressed ovarian follicles in the laving chickens[D]. Hangzhou: Zhejiang University, 2018. (in Chinese) |
| [2] |
江红霞, 刘慧芬, 马晓, 等. 转录组测序筛选克氏原螯虾卵巢发育、免疫和生长相关基因[J]. 水产学报, 2021, 45(3): 396-414. JIANG H X, LIU H F, MA X, et al. Transcriptome analysis of Procambarus clarkii to screen genes related to ovary development, immunity and growth[J]. Journal of Fisheries of China, 2021, 45(3): 396-414. (in Chinese) |
| [3] |
孟金柱, 安清明, 赵成刚, 等. 基于转录组测序筛选山羊卵泡发育相关上调基因[J]. 中国畜牧杂志, 2020, 56(8): 190-195. MENG J Z, AN Q M, ZHAO C G, et al. Transcriptome sequencing screened the up-regulated genes related to follicle development in goats[J]. Chinese Journal of Animal Science, 2020, 56(8): 190-195. (in Chinese) |
| [4] |
郝庆玲, 景炅婕, 朱芷葳, 等. 基于Illumina平台转录组测序筛选牛卵泡发育调控基因[J]. 湖南农业大学学报: 自然科学版, 2019, 45(1): 60-67. HAO Q L, JING J J, ZHU Z W, et al. Screening follicular development regulation genes in bovine using Illumina transcriptome sequencing[J]. Journal of Hunan Agricultural University: Natural Sciences, 2019, 45(1): 60-67. (in Chinese) |
| [5] |
朱志明, 陈红萍, 林如龙, 等. 山麻鸭开产期和产蛋高峰期卵巢组织转录组分析[J]. 中国农业科学, 2016, 49(5): 998-1007. ZHU Z M, CHEN H P, LIN R L, et al. Transcriptome analysis of ovary tissue in early laying period and egg laying peak Period of Shanma ducks[J]. Scientia Agricultura Sinica, 2016, 49(5): 998-1007. (in Chinese) |
| [6] |
SHEN M M, WU P, LI T T, et al. Transcriptome analysis of circRNA and mRNA in theca cells during follicular development in chickens[J]. Genes (Basel), 2020, 11(5): 489. DOI:10.3390/genes11050489 |
| [7] |
董新龙, 张向前, 韩昆鹏, 等. 基于RNA-Seq技术的京海黄鸡卵巢组织转录本预测及新基因挖掘[J]. 中国畜牧兽医, 2017, 44(2): 497-504. DONG X L, ZHANG X Q, HAN K P, et al. Prediction of novel transcripts and discovery of novel genes on RNA-Seq analysis of Jinghai Yellow chicken ovary[J]. China Animal Husbandry & Veterinary Medicine, 2017, 44(2): 497-504. (in Chinese) |
| [8] |
PERTEA M, PERTEA G M, ANTONESCU C M, et al. StringTie enables improved reconstruction of a transcriptome from RNA-seq reads[J]. Nat Biotechnol, 2015, 33(3): 290-295. DOI:10.1038/nbt.3122 |
| [9] |
MARONGIU M, DEIANA M, MARCIA L, et al. Novel action of FOXL2 as mediator of Col1a2 gene autoregulation[J]. Dev Biol, 2016, 416(1): 200-211. DOI:10.1016/j.ydbio.2016.05.022 |
| [10] |
KONG L, ZHANG Y, YE Z Q, et al. CPC: assess the protein-coding potential of transcripts using sequence features and support vector machine[J]. Nucleic Acids Res, 2007, 35(S2): W345-W349. |
| [11] |
LANGMEAD B, SALZBERG S L. Fast gapped-read alignment with Bowtie 2[J]. Nat Methods, 2012, 9(4): 357-359. DOI:10.1038/nmeth.1923 |
| [12] |
LI B, DEWEY C N. RSEM: accurate transcript quantification from RNA-Seq data with or without a reference genome[J]. BMC Bioinformatics, 2011, 12(1): 323. DOI:10.1186/1471-2105-12-323 |
| [13] |
AUDIC S, CLAVERIE J M. The significance of digital gene expression profiles[J]. Genome Res, 1997, 7(10): 986-995. DOI:10.1101/gr.7.10.986 |
| [14] |
李元曦, 夏应菊, 徐璐, 等. 基于RNA-seq对猪瘟病毒感染宿主的转录组学研究进展[J]. 中国兽药杂志, 2020, 54(11): 79-85. LI Y X, XIA Y J, XU L, et al. Research progress of transcriptome of classical swine fever virus after infection[J]. Chinese Journal of Veterinary Drug, 2020, 54(11): 79-85. (in Chinese) |
| [15] |
朱志明, 缪中纬, 章琳俐, 等. 金定鸭卵巢组织转录组SNPs和可变剪接分析[J]. 中国家禽, 2020, 42(2): 11-16. ZHU Z M, MIAO Z W, ZHANG L L, et al. Analysis on SNPs and alternative splicing of Jinding duck ovary with RNA-Seq[J]. China Poultry, 2020, 42(2): 11-16. (in Chinese) |
| [16] |
韩昆鹏. 高、低产蛋量京海黄鸡卵巢组织转录组学分析[D]. 扬州: 扬州大学, 2016. HAN K P. Transcriptomics analysis of ovaries of high and low egg production Jinghai Yellow chicken[D]. Yangzhou: Yangzhou University, 2016. (in Chinese) |
| [17] |
SHI Y B, GHOSH M, KOVTUNOVYCH G, et al. Both human ferredoxins 1 and 2 and ferredoxin reductase are important for iron-sulfur cluster biogenesis[J]. Biochim Biophys Acta Mol Cell Res, 2012, 1823(2): 484-492. DOI:10.1016/j.bbamcr.2011.11.002 |
| [18] |
SPIEGEL R, SAADA A, HALVARDSON J, et al. Deleterious mutation in FDX1L gene is associated with a novel mitochondrial muscle myopathy[J]. Eur J Human Genet, 2014, 22(7): 902-906. DOI:10.1038/ejhg.2013.269 |
| [19] |
AGOSTA C, LAUGIER J, GUYON L, et al. MiR-483-5p and miR-139-5p promote aggressiveness by targeting N-myc downstream-regulated gene family members in adrenocortical cancer[J]. Int J Cancer, 2018, 143(4): 944-957. DOI:10.1002/ijc.31363 |
| [20] |
ZHANG Z X, SHE J J, YANG J Y, et al. NDRG4 in gastric cancer determines tumor cell proliferation and clinical outcome[J]. Mol Carcinog, 2018, 57(6): 762-771. DOI:10.1002/mc.22798 |
| [21] |
刘巍, 贾朝阳, 潘文婧, 等. 下调NDRG4表达对卵巢癌细胞增殖、凋亡的影响及机制[J]. 山东医药, 2019, 59(17): 5-8. LIU W, JIA Z Y, PAN W J, et al. Effects of down-regulating NDRG4 expression on proliferation and apoptosis of ovarian cancer cells[J]. Shandong Medical Journal, 2019, 59(17): 5-8. DOI:10.3969/j.issn.1002-266X.2019.17.002 (in Chinese) |
| [22] |
曹雪娇, 董丽娜, 谭文华, 等. NDRG4在卵巢癌中的表达[J]. 哈尔滨医科大学学报, 2017, 51(5): 434-437. CAO X J, DONG L N, TAN W H, et al. Expression of NDRG4 in ovarian cancer[J]. Journal of Harbin Medical University, 2017, 51(5): 434-437. DOI:10.3969/j.issn.1000-1905.2017.05.012 (in Chinese) |
| [23] |
ZHU M F, ZHENG R, GUO Y W, et al. NDRG4 promotes myogenesis via Akt/CREB activation[J]. Oncotarget, 2017, 8(60): 101720-101734. DOI:10.18632/oncotarget.21591 |
| [24] |
HONGO S, WATANABE T, TAKAHASHI K, et al. Ndrg4 enhances NGF-induced ERK activation uncoupled with Elk-1 activation[J]. Cell Biochem, 2006, 98(1): 185-193. DOI:10.1002/jcb.20763 |
| [25] |
热依拉·普拉提. 巴什拜羊PBDC1、MC3R、NF1和A-FABP基因多态性与其生长性状相关性研究[D]. 乌鲁木齐: 新疆农业大学, 2018. REYILA. P L T. Study on the correlation between PBDC1, MC3R, NF1 and A-FABP gene polymorphisms and growth traits of Bashbai Sheep[D]. Urumqi: Xinjiang Agricultural University, 2018. (in Chinese) |
| [26] |
HOU L E, JI W Y, GU T T, et al. MiR-34c-5p promotes granulosa cells apoptosis by targeting Bcl2 in broody goose (Anser cygnoides)[J]. Animal Biotechnol, 2021. DOI:10.1080/10495398.2021.1886943 |
| [27] |
付晶, 宁方勇, 魏来, 等. 鹌鹑MC3R基因的SNPs及其与生长和屠体性状的相关研究[J]. 中国家禽, 2011, 33(20): 13-17. FU J, NING F Y, WEI L, et al. Single nucleotide polymorphisms of quail melanocortin-3 receptor (MC3R) gene and its association analysis with growth and slaughter traits[J]. China Poultry, 2011, 33(20): 13-17. (in Chinese) |
| [28] |
张心扬, 霍明东, 王守志, 等. 鸡MC3R基因多态性与体重和体组成性状的相关研究[J]. 中国家禽, 2015, 37(10): 12-15. ZHANG X Y, HUO M D, WANG S Z, et al. Correlation analysis between polymorphism of MC3R gene and growth and body composition traits in chicken[J]. China Poultry, 2015, 37(10): 12-15. (in Chinese) |
| [29] |
徐邢宾. 利用CRISPR/Cas9系统构建MC3R基因敲除猪[D]. 秦皇岛: 河北科技师范学院, 2016. XU X B. Construction of MC3R knock-out pigs by CRISPR/Cas9 system[D]. Qinhuangdao: Hebei Normal University of Science, 2016. (in Chinese) |
| [30] |
CHHAJLANI V. Distribution of cDNA for melanocortin receptor subtypes in human tissues[J]. Biochem Mol Biol Int, 1996, 38(1): 73-80. |
| [31] |
NI X P, BUTLER A A, CONE R D, et al. Central receptors mediating the cardiovascular actions of melanocyte stimulating hormones[J]. J Hypertens, 2006, 24(11): 2239-2246. DOI:10.1097/01.hjh.0000249702.49854.fa |
| [32] |
李国辉, 张学余, 韩威, 等. 催乳素和神经肽Y基因及基因聚合对白耳鸡产蛋数的遗传效应[J]. 湖北农业科学, 2010, 49(5): 1029-1032. LI G H, ZHANG X Y, HAN W, et al. Effect of single and pyramiding genotypes of PRL and NPY genes on baier chicken egg production[J]. Hubei Agricultural Sciences, 2010, 49(5): 1029-1032. DOI:10.3969/j.issn.0439-8114.2010.05.002 (in Chinese) |
| [33] |
HOSSEINPOUR L, NIKBIN S, HEDAYAT-EVRIGH N, et al. Association of polymorphisms of vasoactive intestinal peptide and its receptor with reproductive traits of turkey hens[J]. South Afr J Animal Sci, 2020, 50(3): 345-352. DOI:10.4314/sajas.v50i3.1 |
(编辑 范子娟)



