2. 山西省畜禽繁育工作站, 太原 030001;
3. 广灵县优种驴场, 广灵 037500;
4. 山西农业大学动物医学学院, 太谷 030801
2. Shanxi Livestock Breeding Station, Taiyuan 030001, China;
3. Guangling Donkey Breeding Farm, Guangling 037500, China;
4. College of Veterinary Medicine, Shanxi Agricultural University, Taigu 030801, China
驴是一种重要的经济动物,其驯化史可追溯到距今6 000~6 500年前的古埃及[1-2]。目前广泛认为,家驴起源于非洲[3-4],但其确切的驯化地仍未知。我国有4 000年的驴驯养史[5-6],驴存栏量占世界家驴数量的5.89%[7],并形成了24个适应不同气候环境、地形地貌的地方品种[5]。广灵驴是我国优良的地方品种,具有体型高大、耐粗饲、抗病抗逆性强等特点,是改良中小型驴种的重要品种之一,也是生产驴肉、驴皮的优良品种。广灵驴主要分布在山西省广灵、灵邱两县及其周围各县的边缘地区[8],以广灵县南村镇、壶泉镇、加斗乡分布最多,质量最好,其品种形成具有200多年的历史[9]。之后,随着驴役用性能逐渐退出历史舞台,广灵驴的存栏量逐年下降。据《中国畜禽遗传资源志—马驴驼志》报道,2006年末广灵驴共存栏4 808头,其中母驴1 700头,种用公驴220头[10]。2020年报道广灵驴存栏量为3 000头左右[11],种群数目萎缩,保种形式严峻。
保种工作的总目标是保持种群的遗传多样性,其核心内容是“原封不动的保存全部基因”,其指导思想是控制群体近交系数的上升速率,避免基因由于遗传漂变而丢失[12-13]。而保种的基础工作是种群具有完整、准确、清楚的系谱记录。广灵驴的保种群体主要饲养于广灵县优种驴场,该场建成于1960年,为国家级保种场,饲养种驴150余头。但由于保种场地处偏僻,加之建场年代久远,饲养员更换频繁,存在种驴系谱错记、漏记、缺失等情况,给种群的扩繁、选配方案的制订带来了一定困难,也为今后的品系培育带来了不利影响。因此,利用分子标记对系谱的校正和完善以及群体遗传结构的分析具有一定意义。
目前,用于亲缘关系分析、评估群体遗传多样性水平、遗传图谱构建的分子标记主要为微卫星标记和SNP标记。联合国粮农组织(FAO)在动物遗传资源管理和可持续发展战略计划中,将微卫星标记作为优先推荐的分析工具,并制订了家畜品种之间遗传距离测定的全球方案[14]。采用微卫星标记进行的亲缘关系鉴定准确率可达99%以上,且具有多态性丰富、高保守性、结果重复性好等优点,在畜禽遗传资源多样性评估、亲缘关系鉴定、品种起源进化方面发挥了重要作用[14]。
本研究以广灵县优种驴场保种群为研究群体,采用FAO和国际动物遗传协会(ISAG)共同推荐的马属动物遗传多态性标记中的13个微卫星标记,进行保种群的亲缘关系鉴定和分子系谱构建,并进一步对群体进行遗传结构分析和系统发育树(NJ树)构建。
1 材料与方法 1.1 材料1.1.1 试验动物的选取及采样 选取山西省广灵县优种驴场种公驴13头,种母驴94头,采集颈静脉血10 mL,采用EDTA法抗凝,充分混匀后立即放入4 ℃保温箱内冷藏,在24 h内离心并分离白细胞层,置于-80 ℃下保存。
1.1.2 微卫星引物 用于亲缘关系分析的微卫星引物序列参考自王敏等[15]鉴定德州驴亲缘关系时所用的13个标记引物序列,经预扩增后选择12个标记进行后续试验。微卫星荧光引物(表 1)由上海生工生物工程公司合成。
|
|
表 1 12对微卫星引物的碱基序列及PCR反应条件 Table 1 The sequences and PCR conditions of 12 SSR primers |
1.1.3 试剂与耗材 动物血液DNA提取试剂盒购自OMEGA公司,PCR反应用Mix购自北京全式金公司,琼脂糖凝胶购自BBI公司,其余耗材购自Axygen公司。
1.1.4 主要仪器设备 PCR仪(BioRad公司,美国),台式高速冷冻离心机(Eppendorf公司,德国),凝胶成像系统(Alpha Innotech,美国),移液枪(Eppendorf公司,美国),电泳仪(北京六一仪器厂)。
1.2 方法1.2.1 DNA提取和纯化 DNA提取按照OMEGA Blood DNA Mini Kit(D3392)步骤进行。DNA纯化按照OMEGA MicroElute©DNA Clean Up Kit(D6296)步骤进行。
1.2.2 PCR扩增 PCR反应体系为20.0 μL:模板DNA(浓度为50 ng左右)2.0 μL,上游引物1.0 μL,下游引物1.0 μL,PCR Mix 10.0 μL,ddH2O 6.0 μL。
PCR反应条件:94 ℃预变性5 min;94 ℃变性30 s,50~ 60 ℃退火30 s,72 ℃延伸30 s,40循环;72 ℃终末延伸10 min,产物保存在4 ℃。
1.2.3 微卫星产物的分型 微卫星扩增体系为每孔内加入分子量内标和甲酰胺混合液(0.5 ∶8.5)9.0 μL,与PCR产物1.0 μL混合后95 ℃变性3 min,ABI3730毛细管电泳检测。将检测得到的原始数据文件导入到分析软件GeneMapper 3.2中分析试验结果。
1.2.4 统计分析1.2.4.1 微卫星标记遗传参数的计算 12个微卫星标记的等位基因数(N)、观测杂合度(HO)、期望杂合度(HE)、多态信息含量(PIC)等参数和标记的哈代温伯格平衡情况采用Cervus2.0软件计算。
1.2.4.2 亲缘关系分析及系谱构建 采用Cervus2.0软件中的Parentage Analysis模块分别对每头子代的最似父亲和最似母亲进行分析,并与保种场提供的原始系谱记录进行比对,计算系谱错误率。采用Pedigraph软件对保种场各家系进行分子系谱的构建。
1.2.4.3 遗传距离及聚类分析 采用STRUCTURE软件(版本:2.3)和R语言中hclust函数对各公驴家系进行Nei’s遗传距离(DA和DS)的计算,聚类分析以及NJ树的构建,结合纸质系谱,判断家系数目和亲缘关系远近。
1.2.4.4 群体F统计量的计算 采用Fstat软件计算不同公驴家系内和家系间遗传变异(FIS、FIT和FST)和近交情况。
2 结果 2.1 PCR扩增结果及多态性对广灵县优种驴场107头种驴(公驴13头,母驴94头)在12个微卫星座位上的多态性进行分析,统计出12个微卫星标记中等位基因数最多的为11个,最少为2个。
2.2 各微卫星标记的群体遗传学参数利用微卫星标记图形分析软件Genemapper 3.2对每个采样个体的荧光PCR产物进行基因型判读,并通过软件导出测序峰图后进行各等位基因片段的统计,并统计出标记的等位基因数(N)、观测杂合度(HO)、期望杂合度(HE)和多态信息含量(PIC),对每个微卫星位点进行哈代-温伯格平衡(H-W)检测(表 2)。
|
|
表 2 广灵驴群体在12个微卫星座位上的群体遗传学参数 Table 2 Population genetic parameters of 12 SSR in Guangling population |
根据表 2,上述12个微卫星标记的等位基因数在2~11个之间,多态信息含量(PIC)从大到小依次为:HTG7(0.780 3)、TKY297(0.770 4)、ASB23(0.739 1)、HMS18(0.733 6)、AHT4(0.715 2)、HMS2(0.664 4)、HMS3(0.540 7)、TKY343(0.537 7)、TKY312(0.520 2)、TKY337(0.472 7)、HMS6(0.363 6)、HMS7(0.289 4)。除HMS7外,标记杂合度较高,适于做亲缘关系鉴定。对不同微卫星位点进行哈代-温伯格平衡检测结果可知,10个微卫星标记均处于平衡状态,说明群体处于随机交配状态,基本未经过选育。
2.3 群体中个体的亲缘关系分析采用Cervus2.0软件中的Simulation模块模拟10 000个后代在宽松阈值条件(0.90)下和严格阈值(0.99)条件下的阈值(LOD值)分别为2.81和6.65。然后采用该软件中的Parentage Analysis模块分别对每头子代的最似父亲和最似母亲进行分析,算法采用最大似然法。
由表 3可知,在107头参与亲子关系鉴定的个体中,最终通过微卫星标记分析确定父子关系的子代个体为30头,确定母子关系的子代个体为7头,准确度均达到90%(表 3)。进一步将确认亲子关系个体进行纸质系谱的比对,在30个个体中,原系谱记录中亲本缺失个体为17个,占57%;原系谱记录错误的个体为7个,占23%。说明原始系谱记录存在较多缺失和错记情况。
|
|
表 3 通过Cervus2.0软件确认子代的父母 Table 3 Parents confirmed through Cervus 2.0 software |
采用Pedigraph2.4软件绘制已经确定亲缘关系个体的系谱图,共检测到7个公驴家系,分别为:201109010013、201109010009、201204040019、201307050021、201301060031、201504070035、201506040043,确定了30头子代的父亲和7头子代的母亲(表 3),占采样总数的40%左右。由图 1可知,201307050021是最大的一个公驴家系,包括1头雄性子代和9头雌性子代。而201301060031以及201504070035是最小的公驴家系,均检测出1个后代。
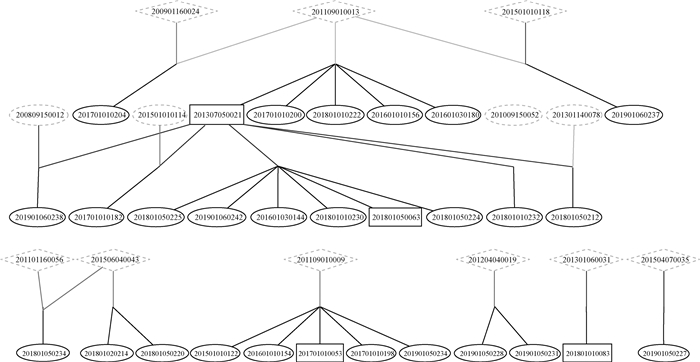
|
图中数字为确定亲缘关系的亲本及子代的电子耳号。菱形表示亲本,椭圆表示子代雌性个体,长方形表示子代雄性个体 Numbers in the figure are electronic labels of parents and offspring with confirmed relationships. The diamonds represent the parents, the ellipses represent the female offspring, and the rectangles represent the male offspring 图 1 广灵驴群体的分子系谱 Fig. 1 Molecular pedigree of Guangling donkey population |
采用STRUCTURE软件(版本2.3)计算各家系间的Nei’s遗传距离,设定参数值K=7。采用R语言中的hclust函数构建广灵驴7个家系的系统发育邻接树(NJ树)。结果如图 2所示。

|
图中编号代表进行NJ树构建的7个公驴家系公驴及其后代的顺序号;纵坐标为相对遗传距离 The numbers in the figure are sequential numbers of 7 male donkey families and their offspring for NJ tree construction; Y-axis is the relative genetic distance 图 2 采用R语言构建的子代及其父亲(n=37)的系统发育树 Fig. 2 NJ tree for offspring and their fathers using R software(n=37) |
由图 2可知,使用R语言hclust函数绘制7头公驴及其后代的NJ树,发现它们聚为两大分支。其中1号(201109010013)、9号(201307050021)、21号(201109010009)、27号(201506040043)、31号(201204040019)、34号(201301060031)、36号(201504070035)为亲本公驴,其余编号个体为子代。
2.6 群体遗传结构分析采用Fstat软件可通过共显性遗传标记来估计和检验基因多样性和基因分化情况,表 4是所有微卫星基因座群体遗传结构参数的计算结果。其中,FIS表示个体(I)相对于亚群(S)的近交系数,即亚群的平均近交系数;FST表示亚群(S)相对于总群体(T)的近交系数,即有亲缘关系亚群间的平均近交系数; FIT值代表各亚群间近交系数的平均值,反映了保种群全群的近交程度[16]。
|
|
表 4 微卫星基因座群体遗传参数 Table 4 Population genetic parameters of SSR locus |
由表 4可知,所采用标记的平均观测杂合度为0.648,说明标记具有较高多态性,具有较好的保种潜力。群体分化指数FST和群体近交系数FIS在群体遗传学中均称为F-Statistics,其含义是衡量种群中基因型实际频率是否偏离哈-温平衡理论比例的指标。F-Statistics也可以被认为是在分层群体中不同亚群间基因相关性的度量。由于FST的取值在0 ~ 1之间,数值越大,说明等位基因在各自亚群中越固定,群体分化程度越大[16]。上述群体FST平均值为0.184,说明各亚群之间存在较大的遗传分化,具有不同的来源。
FIT由标记计算结果为0.033,采用所有个体系谱计算结果为0.02,两者计算的近交系数接近。当FIT值大于0.375时,说明群体为近交系[17]。上述计算结果说明群体基本处于随机交配状态,群体内存在很弱的近交。
由上述结果可知,大多数FIS为负值,平均值为-0.238。上述计算结果与FIT值评估结果较为一致,说明群体内个体间存在很弱的近交。
3 讨论 3.1 广灵驴群体的遗传多样性遗传多样性是生命进化和物种分化的基础,在一定程度上决定了物种在自然或人为条件下对环境改变的适应能力[17]。本研究中所用的微卫星标记平均等位基因数(N)为6.25,平均观测杂合度(HO)为0.676 5,平均多态含量(PIC)为0.593 9。其中,杂合度和PIC值反映了种群的遗传多样性程度,当杂合度>0.5,则意味着该群体未受到高强度的选择,具有较为丰富的遗传多样性;PIC值则代表了标记位点变异程度的大小,PIC>0.5为高度多态性位点,可提供足够的信息量。上述12个微卫星标记的PIC值除HMS7外均具有高多态含量,可用于亲缘关系鉴定和遗传多样性评估。根据纪志宾等[18]在采用微卫星标记构建崂山奶山羊分子系谱中的分析结果,发现当标记的平均PIC值在0.604~0.687时,需要11个和7个微卫星标记才能够满足系谱构建的可靠性在0.99以上。本研究标记的平均PIC值在0.594左右,采用12个标记,符合可靠性要求。
王敏等[15]采用13个微卫星标记评估了53头德州驴的群体多样性和亲缘关系,13个微卫星标记的等位基因数(N)、观测杂合度(HO)、多态信息含量(PIC)分别为6.850, 0.689和0.625, 与本研究结果相似。刘艳艳等[19]采用13个微卫星标记对39头三粉驴和16头乌头驴进行了遗传多样性分析,其中乌头驴群体平均观测杂合度为0.27,三粉驴群体平均观测杂合度为0.33,低于本研究。杨虎等[20]则采用8个微卫星标记评估了3个新疆地方驴品种的多态性,并对其进行了聚类分析,发现3个品种平均杂合度为0.784 1,PIC值为0.756 8,均处于较高水平。Jordana等[21]研究了美国驴的遗传多样性和遗传结构,他们采用14个微卫星标记对350头来源于13个国家的美国驴群体进行分析,最终将其划分为南方和北方两个群体。Zeng等[22]采用12个荧光标记的微卫星对504头12个中国地方驴品种的遗传多样性进行分析,发现其标记的杂合度为0.631 5~0.699 9,多态信息含量为0.660 0,与本研究的标记观测杂合度相似,但此研究分析的12个地方品种未包括广灵驴。相比上述研究,可得出广灵驴群体内遗传变异程度高,具有较好的保种和品种开发潜力。
相比SNP标记,微卫星标记具有多态信息含量高,保守性好的特点,被广泛使用在群体结构多样性分析中。但SNP标记在家畜基因组上的分布更加广泛、密集,且检测方便,更适合现代高通量检测平台,也是群体遗传多样性分析的理想标记。Dell等[23]采用15个微卫星标记研究了一个英国濒危的马品种—克利夫兰湾马(Cleveland Bay horse)的遗传多样性和系谱关系,在402匹马中发现3个系祖,可以解释70%的母系遗传变异,群体近交系数为7.8%,标记观测杂合度为0.534 1。
目前,尚未有在驴中使用中高密度芯片进行群体遗传分析的报道。在马中,已经先后开发出50K[24]、65K[25]和74K[26]芯片,最近又开发出极高密度的670K[27-28]芯片,用于赛马品种选育、种群分化和特定功能基因研究[29-30]。Nolte等[31]采用Illumina Equine50K芯片检测了4个德国赛马品种共942个个体的选择信号,获得了一系列影响赛马肌肉功能(TPM1、TMOD2-3、MYO5A、MYO5C),肌肉生长和能量代谢(AEBP1、RALGAPA2、IGFBP1、IGFBP3-4),胚胎发育和繁殖方面的基因。2013年,Petersen等[27]通过对33个品种的744匹马采用50K SNP芯片检测了其基因组的变异情况,发现MSTN受到强烈选择,同时发现马的体型大小与ECA11基因有关。Avila等[32]采用65K SNP芯片对夸特马(Quarter horse)群体中经历强选择的基因组区段和单倍型进行了分析,发现与肌肉发育和代谢以及中枢神经系统有关的基因区段存在选择印记。Gurgul等[33]采用Illumina芯片对波兰6个马品种共571个个体进行选择信号分析,发现了一系列与肌肉力量、心脏功能、疾病抗性、繁殖性能和协调性有关的基因。因此,进一步开发家驴中高密度的基因芯片将对驴的起源进化研究和特定品种功能基因挖掘起到极大的推动作用。另外,也可利用基因组重测序数据进行群体分化差异和位点的连锁不平衡检测,进而寻找候选基因。
3.2 群体亲缘关系在亲缘关系鉴定中,累计非父排除概率(CEP)可作为标记是否可靠的衡量标准[34]。本研究所采用标记的CEP平均值在97%以上,所鉴定获得的亲子关系可靠性约为95%,结合原始系谱,基本可以获得准确的亲子关系。
从分子系谱分析结果可知,各公驴家系的后代数从1到10头,存在较大的变异。从长期保种的角度来看,各家系留种后代不一致会导致有效群体数量降低,遗传漂变等不利情况,所以应尽量采用各家系等量留种的方式。图 1和表 3中,确定母子关系的亲子对只有7对,这可能是由于场内母驴转出或售卖的情况较多,周转较快,导致子代的真实母亲不在采样群体中。此次分子系谱构建仅获得了30个父子对和7个母子对的可靠亲子关系,占到总采样群体的约40%,说明还有近一半个体未找到其亲本。原纸质系谱中包含了16头公驴,而本次检测仅检测到7个公驴家系,说明尚有一些公驴被淘汰或出售。
固定指数FIS通常代表了群体内个体间的近交系数[35]。当群体内HO小于HE,同时FIS为正值时,表明群体内近交程度较严重; 而FIS为负值,群体内HO大于HE,则表示群体内存在远缘繁殖。本研究群体的HO(0.676 5)> HE(0.642 9),且群体内平均近交系数为-0.238,说明群体基本不存在家系内近交,为远缘繁殖。这也与标记的哈代-温伯格平衡检验结果(表 3)相一致,即群体处于随机交配状态。这一结果也与群体分化系数FST和群体平均近交系数FIT所得出的结果相吻合。
3.3 群体遗传结构与分化情况Wright建议,实际研究中FST为0~0.05,表明群体间遗传分化很小,可以不考虑;FST为0.05~0.15,表明群体间存在中等程度的遗传分化;FST为0.15~0.25,群体间遗传分化较大;FST为0.25以上,表明群体间有很大的遗传分化。本研究通过12个微卫星标记所获得的FST值为0.184,说明各家系间具有较大的遗传分化,可能具有不同来源[17]。通过查询原始系谱资料可知,图 1中的1号公驴购自广灵县壶泉镇,36号公驴购自广灵县蕉山乡,两地相距8 km;21和34号公驴原始记录丢失,不能判断其来源;9号公驴则购自龙虎岩村;21和37号公驴则都来自浮图村,该地位于河北省蔚县,距离广灵县26 km,蔚县也是阳原驴的产区,可能会出现品种混杂的情况,对其血统还需要进行更进一步的分析。
本研究采用基于Nei’s遗传距离构建系统发生树,以期得到各家系间亲缘关系的远近。Nei’s标准遗传距离是根据基因频率估算每个位点平均密码子的差数,可用于推断两个家系的分化时间[36]。但此公式的基本假定是群体为恒定的大群体,每个基因每年的突变率恒定,且所有突变均为中性突变,不受选择的影响[37]。在构建系统发生树的过程中,除采用邻接法外(NJ法),还可采用UPGMA法构建无根树,但此法假设各分支的进化速率一致,在参考序列较短时,误差较大,故采用NJ法,且得到了较为合理的结果。
一些学者对驴线粒体DNA D-loop区进行测序,分析不同地方品种之间的亲缘关系和家驴的起源进化[38-39]。其中葛庆兰等[40]证明,中国家驴的祖先为非洲野驴,这与王全喜等[41]的研究结果一致。Xia等[42]研究了南美地方驴品种的母系起源,采用D-loop区489 bp的序列分析了323头来自南美4个国家的驴群体,结果证明4个群体聚为两个大的分支,但它们之间的遗传交流很少。Özkan Ünal等[43]采用mtDNA D-loop区对16个土耳其地方驴品种的母系起源进行了研究,发现这些品种聚为两个分支,分别为索马里分支和努比亚分支。Ma等[44]比较了世界范围内700余条不同驴品种的mt D-loop区序列,对两个大分支的驯化史进行了详细分析,发现撒哈拉以南地区的驴来源于分支Ⅰ,而非洲以东和以北的驴来源于分支Ⅱ。进一步应采用多种标记对广灵驴的起源和选择进化过程进行研究。
4 结论本研究采用12个微卫星标记对广灵县优种驴场保种群进行了遗传多样性分析和分子系谱构建,可靠性和准确性较高。目前,该群体基本不存在严重近交的情况,具有较丰富的遗传多态性,具有较好的保种潜力。
| [1] |
ROSSEL S, MARSHALL F, PETERS J, et al. Domestication of the donkey: timing, processes, and indicators[J]. Proc Natl Acad Sci U S A, 2008, 105(10): 3715-3720. DOI:10.1073/pnas.0709692105 |
| [2] |
BLENCH R M, MACDONALD K C. The origins and development of African livestock: archaeology, genetics, linguistics and ethnography[M]. London: University College London Press, 2000.
|
| [3] |
WANG C F, LI H J, GUO Y, et al. Donkey genomes provide new insights into domestication and selection for coat color[J]. Nat Commun, 2020, 11(1): 6014. DOI:10.1038/s41467-020-19813-7 |
| [4] |
BEJA-PEREIRA A, ENGLAND P R, FERRAND N, et al. African origins of the domestic donkey[J]. Science, 2004, 304(5678): 1781. DOI:10.1126/science.1096008 |
| [5] |
谢成侠. 中国马驴品种志[M]. 上海: 上海科学技术出版社, 1987. XIE C X. Horse and ass breeds in China[M]. Shanghai: Shanghai Scientific & Technical Publishers, 1987. (in Chinese) |
| [6] |
HAN H, CHEN N, JORDANA J, et al. Genetic diversity and paternal origin of domestic donkeys[J]. Anim Genet, 2017, 48(6): 708-711. DOI:10.1111/age.12607 |
| [7] |
HAN L, ZHU S B, NING C, et al. Ancient DNA provides new insight into the maternal lineages and domestication of Chinese donkeys[J]. BMC Evol Biol, 2014, 14(1): 246. DOI:10.1186/s12862-014-0246-4 |
| [8] |
聂鸿瑶. 广灵驴简介[J]. 辽宁畜牧兽医, 1983(5): 16-19. NIE H Y. Brief introduction of Guangling donkey[J]. Liaoning Animal Husbandry and Veterinary Medicine, 1983(5): 16-19. (in Chinese) |
| [9] |
梁全. 广灵驴品种的保护与开发[J]. 中国畜牧业, 2015(8): 53-54. LIANG Q. Breed protection and development of Guangling donkey[J]. China Animal Industry, 2015(8): 53-54. DOI:10.3969/j.issn.2095-2473.2015.08.026 (in Chinese) |
| [10] |
国家畜禽遗传资源委员会. 中国畜禽遗传资源志—马驴驼志[M]. 北京: 中国农业出版社, 2011. National livestock and Poultry Genetic Resources Committee. Animal genetic resources in China horses, donkeys, camels[M]. Beijing: China Agriculture Press, 2011. (in Chinese) |
| [11] |
闫俊峰. 广灵驴特性和生长规律[J]. 农业技术与装备, 2020(11): 165-166. YAN J F. Characteristics and growth Law of Guangling donkey[J]. Agricultural Technology & Equipment, 2020(11): 165-166. DOI:10.3969/j.issn.1673-887X.2020.11.078 (in Chinese) |
| [12] |
常洪. 动物遗传资源学[M]. 北京: 科学出版社, 2009. CHANG H. Animal genetic resources[M]. Beijing: Science Press, 2009. (in Chinese) |
| [13] |
GICQUEL E, BOETTCHER P, BESBES B, et al. Impact of conservation measures on demography and genetic variability of livestock breeds[J]. Animal, 2020, 14(4): 670-680. DOI:10.1017/S1751731119002672 |
| [14] |
张勇, 肖礼华, 陈祥, 等. 利用微卫星标记分析贵州地方鸡种的遗传多样性及亲缘关系[J]. 畜牧兽医学报, 2006, 37(12): 1274-1281. ZHANG Y, XIAO L H, CHEN X, et al. Analysis of genetic diversity and relationship among local chicken breeds in Guizhou by using microsatellites marker[J]. Acta Veterinaria et Zootechnica Sinica, 2006, 37(12): 1274-1281. DOI:10.3321/j.issn:0366-6964.2006.12.007 (in Chinese) |
| [15] |
王敏, 张新浩, 崔冉, 等. 利用微卫星标记鉴定德州驴亲子关系[J]. 中国畜牧兽医, 2019, 46(7): 2003-2011. WANG M, ZHANG X H, CUI R, et al. Parentage testing of Dezhou Donkey with microsatellite marker[J]. China Animal Husbandry & Veterinary Medicine, 2019, 46(7): 2003-2011. (in Chinese) |
| [16] |
NEI M. Molecular population genetics and evolution[M]. New York: North-Holland Publishing Company, 1975.
|
| [17] |
FRANKHAM R, BALLOU J D, BRISCOE D A. Introduction to conservation genetics[M]. Cambridge: Cambridge University Press, 2002.
|
| [18] |
纪志宾, 王桂芝, 王金凤, 等. 崂山奶山羊分子系谱的构建及家系遗传特征分析[J]. 中国农业科学, 2011, 44(13): 2796-2807. JI Z B, WANG G Z, WANG J F, et al. Construction of molecular genealogy and analysis of family genetic characteristics in Laoshan dairy goat[J]. Scientia Agricultura Sinica, 2011, 44(13): 2796-2807. DOI:10.3864/j.issn.0578-1752.2011.13.018 (in Chinese) |
| [19] |
刘艳艳, 谭晴晴, 范阳阳, 等. 乌头驴和三粉驴微卫星遗传多态性分析[J]. 中国畜牧杂志, 2017, 53(1): 44-50. LIU Y Y, TAN Q Q, FAN Y Y, et al. Genetic diversity analysis of Wutou Donkey and Sanfen Donkey[J]. Chinese Journal of Animal Science, 2017, 53(1): 44-50. (in Chinese) |
| [20] |
杨虎, 阿吉, 王金富, 等. 新疆3个地方品种驴微卫星遗传分析[J]. 中国畜牧杂志, 2008, 44(1): 8-10. YANG H, A J, WANG J F, et al. Genetic analysis of three ingenious breeds of Xinjiang Province[J]. Chinese Journal of Animal Science, 2008, 44(1): 8-10. (in Chinese) |
| [21] |
JORDANA J, FERRANDO A, MIRÓ J, et al. Genetic relationships among American donkey populations: insights into the process of colonization[J]. J Anim Breed Genet, 2016, 133(2): 155-164. DOI:10.1111/jbg.12180 |
| [22] |
ZENG L L, DANG R H, DONG H, et al. Genetic diversity and relationships of Chinese donkeys using microsatellite markers[J]. Arch Anim Breed, 2019, 62(1): 181-187. DOI:10.5194/aab-62-181-2019 |
| [23] |
DELL A, CURRY M, YARNELL K, et al. Genetic analysis of the endangered Cleveland Bay horse: a century of breeding characterised by pedigree and microsatellite data[J]. PLoS One, 2020, 15(10): e0240410. DOI:10.1371/journal.pone.0240410 |
| [24] |
MCCUE M E, BANNASCH D L, PETERSEN J L, et al. A high density SNP array for the domestic horse and extant Perissodactyla: utility for association mapping, genetic diversity, and phylogeny studies[J]. PLoS Genet, 2012, 8(1): e1002451. DOI:10.1371/journal.pgen.1002451 |
| [25] |
MCCOY A M, MCCUE M E. Validation of imputation between equine genotyping arrays[J]. Anim Genet, 2014, 45(1): 153. DOI:10.1111/age.12093 |
| [26] |
SCHUBERT M, JÓNSSON H, CHANG D, et al. Prehistoric genomes reveal the genetic foundation and cost of horse domestication[J]. Proc Natl Acad Sci U S A, 2014, 111(52): E5661-E5669. |
| [27] |
PETERSEN J L, MICKELSON J R, COTHRAN E G, et al. Genetic diversity in the modern horse illustrated from genome-wide SNP data[J]. PLoS One, 2013, 8(1): eE5669. |
| [28] |
SCHAEFER R J, SCHUBERT M, BAILEY E, et al. Developing a 670k genotyping array to tag ~2M SNPs across 24 horse breeds[J]. BMC Genomics, 2017, 18(1): 565. DOI:10.1186/s12864-017-3943-8 |
| [29] |
FAWCETT J A, SATO F, SAKAMOTO T, et al. Genome-wide SNP analysis of Japanese Thoroughbred racehorses[J]. PLoS One, 2019, 14(7): e0218407. DOI:10.1371/journal.pone.0218407 |
| [30] |
HAUSWIRTH R, HAASE B, BLATTER M, et al. Mutations in MITF and PAX3 cause "splashed white" and other white spotting phenotypes in horses[J]. PLoS Genet, 2012, 8(4): e1002653. DOI:10.1371/journal.pgen.1002653 |
| [31] |
NOLTE W, THALLER G, KUEHN C. Selection signatures in four German warmblood horse breeds: tracing breeding history in the modern sport horse[J]. PLoS One, 2019, 14(4): e0215913. DOI:10.1371/journal.pone.0215913 |
| [32] |
AVILA F, MICKELSON J R, SCHAEFER R J, et al. Genome-wide signatures of selection reveal genes associated with performance in American Quarter Horse subpopulations[J]. Front Genet, 2018, 9: 249. DOI:10.3389/fgene.2018.00249 |
| [33] |
GURGUL A, JASIELCZUK I, SEMIK-GURGUL E, et al. A genome-wide scan for diversifying selection signatures in selected horse breeds[J]. PLoS One, 2019, 14(1): e0210751. DOI:10.1371/journal.pone.0210751 |
| [34] |
王杰, 王永, 许期树, 等. 微卫星DNA在高原型藏山羊亲子鉴定中的应用研究[J]. 西南民族大学学报: 自然科学版, 2010, 36(1): 74-79. WANG J, WANG Y, XU Q S, et al. The application of microsatellite DNA in paternity testing for Tibetan goats[J]. Journal of Southwest University for Nationalities: Natural Science Edition, 2010, 36(1): 74-79. DOI:10.3969/j.issn.1003-2843.2010.01.018 (in Chinese) |
| [35] |
李成渤, 王桂芝, 纪志宾, 等. 崂山奶山羊微卫星标记与泌乳性状的相关分析[J]. 中国农业科学, 2009, 42(10): 3639-3646. LI C B, WANG G Z, JI Z B, et al. Correlation analysis between microsatellite markers and milk performance traits in Laoshan Dairy Goat[J]. Scientia Agricultura Sinica, 2009, 42(10): 3639-3646. DOI:10.3864/j.issn.0578-1752.2009.10.0032 (in Chinese) |
| [36] |
马建岗, 路兴中. 关于用Nei氏标准遗传距离推算畜禽分化时间的探讨[J]. 遗传, 1992, 14(3): 37-39. MA J G, LU X Z. Discussion about estimating the time of divergence between animals with Nei's standard genetic distance[J]. Hereditas, 1992, 14(3): 37-39. DOI:10.3321/j.issn:0253-9772.1992.03.013 (in Chinese) |
| [37] |
WRIGHT S. Evolution and the genetics of Populations. Vol. 4. Variability within and among natural populations[M]. Chicago: University of Chicago Press, 1978: 35-49.
|
| [38] |
雷初朝, 陈宏, 杨公社, 等. 中国驴种线粒体DNA D-loop多态性研究[J]. 遗传学报, 2005, 32(5): 481-486. LEI C Z, CHEN H, YANG G S, et al. Study on mitochondrial DNA D-loop polymorphism in Chinese donkeys[J]. Acta Genetica Sinica, 2005, 32(5): 481-486. (in Chinese) |
| [39] |
雷初朝, 陈宏, 王德解, 等. 关中驴线粒体DNA D-loop多态性分析[J]. 中国畜牧杂志, 2004, 40(4): 10-12. LEI C Z, CHEN H, WANG D J, et al. Analysis on Polymorphism of mtDNA D-loop Region in Guanzhong Donkey[J]. Chinese Journal of Animal Science, 2004, 40(4): 10-12. DOI:10.3969/j.issn.0258-7033.2004.04.004 (in Chinese) |
| [40] |
葛庆兰, 雷初朝, 蒋永青, 等. 中国家驴mtDNA D-loop遗传多样性与起源研究[J]. 畜牧兽医学报, 2007, 38(7): 641-645. GE Q L, LEI C Z, JIANG Y Q, et al. Mitochondrial DNA D-loop genetic diversity and origin of chinese domestic donkeys[J]. Acta Veterinaria et Zootechnica Sinica, 2007, 38(7): 641-645. DOI:10.3321/j.issn:0366-6964.2007.07.004 (in Chinese) |
| [41] |
王全喜, 陈建兴, 闵令江, 等. 中国家驴品种遗传多样性及母系起源研究[J]. 内蒙古农业大学学报, 2012, 33(2): 7-11. WANG Q X, CHEN J X, MIN L J, et al. Genetic Civersith and maternal origin of Chinese donkey breeds[J]. Journal of Inner Mongolia Agricultural University, 2012, 33(2): 7-11. (in Chinese) |
| [42] |
XIA X, YU J, ZHAO X, et al. Genetic diversity and maternal origin of Northeast African and South American donkey populations[J]. Anim Genet, 2019, 50(3): 266-270. DOI:10.1111/age.12774 |
| [43] |
ÖZKAN VNAL E, ÖZDIL F, KAPLAN S, et al. Phylogenetic relationships of Turkish indigenous donkey populations determined by mitochondrial DNA D-loop region[J]. Animals, 2020, 10(11): 1970. DOI:10.3390/ani10111970 |
| [44] |
MA X Y, NING T, ADEOLA A C, et al. Potential dual expansion of domesticated donkeys revealed by worldwide analysis on mitochondrial sequences[J]. Zool Res, 2020, 41(1): 51-60. DOI:10.24272/j.issn.2095-8137.2020.007 |
(编辑 郭云雁)



