奶业是现代农牧业和食品行业发展最快、最有活力的产业之一。牛奶及各类奶制品已成为人民生活中必不可缺的一部分,牛奶品质则与人们的健康密不可分。奶牛乳房炎是影响奶业可持续发展的重要疾病之一,发病的主要原因是病原微生物通过奶牛的乳头导管以及乳房皮肤外伤等进入奶牛的乳腺组织,引起乳腺发生炎症反应。根据临床表现,可将奶牛乳房炎分为临床乳房炎和隐性乳房炎。据报道,全球荷斯坦牛群隐性乳房炎发病率高达48%,我国为25%~70%[1-2]。隐性乳房炎不仅严重影响牛奶产量和品质,还会极大增加妊娠牛的流产率和淘汰率,给现代奶业带来严重的经济损失。本文针对近年来有关奶牛乳房炎及其在转录组以及表观基因组等方面的研究进展进行了综述。
1 奶牛乳房炎研究进展奶牛乳房炎[3]是乳业生产中花费最高的疾病之一,提高乳房炎抗性已成为奶牛育种的重要目标。但是,乳房炎抗性通常涉及多个过程的多基因调控,包括对感染、炎症和感染后愈合的反应。低遗传力、环境变异和农场管理差异使得鉴定遗传变异与乳房炎抗性的关系变得更为复杂和困难。临床乳房炎可以通过乳房、乳汁的异常表现检测到;而隐性乳房炎的特征是体细胞数(somatic cell counts,SCCs)增加,但乳房或者乳汁中没有任何感官异常。由于较高的发病率,隐性乳房炎造成的经济损失要更大。
1.1 奶牛隐性乳房炎抗性相关基因研究进展奶牛隐性乳房炎抗病育种研究主要利用QTL(quantitative trait locus)定位、候选基因及全基因组关联分析等方法,挖掘与隐性乳房炎抗性相关的候选基因或QTLs。已有多项研究定位到大量与SCCs及体细胞评分(somatic cell score,SCS)显著相关的常染色体QTLs。2012年,Meredith等[4]通过全基因组关联分析鉴定到与产奶性状和体细胞评分显著相关的276个单核苷酸多态性(single nucleotide polymorphisms, SNPs)。2013年,王晓等[5]利用Illumina牛54K芯片对2 093头中国荷斯坦牛进行SNP检测,基于case-control分组策略进行GWAS(genome-wide association study)分析,发现了6个与奶牛乳房炎抗性关联显著的SNPs及其相邻基因可作为奶牛乳房炎抗性的分子标记及候选基因;2015年,基于上述2 093头奶牛SCCs育种值,利用GWAS鉴定分析了与奶牛乳房炎抗性及易感性相关的SNPs和候选基因[6],发现TRAPPC9可作为潜在候选基因;冯文等[7]进一步将TRAPPC9作为抗性候选基因,与奶牛金葡菌乳房炎的遗传效应进行关联分析,发现TRAPPC9可作为中国荷斯坦牛金葡菌乳房炎抗性的分子遗传标记。2020年,Kirsanova等[8]在挪威红牛群体,对基于测定日体细胞数阈值定义的12个隐性乳房炎性状进行了全基因组关联分析,鉴定到ACOT2、ACOT4、FOS基因和一系列趋化因子基因(C-X-C motif)等可作为挪威红牛隐性乳房炎的潜在候选基因。
1.2 奶牛金葡菌乳房炎研究进展金黄色葡萄球菌(Staphylococcus aureus,S. aureus,简称金葡菌)是导致奶牛隐性乳房炎的主要接触传染性致病菌[9-10]。金葡菌是一种重要的人兽共患细菌,对全球人类和动物的健康有害[11]。约有25%~30%的健康人群黏膜上携带金葡菌,正常情况下不会引发疾病,但当出现创伤时,金葡菌将造成皮肤和软组织感染,主要包括脓疱病、毛囊炎、鼻炎、溃疡和伤口感染等。在金葡菌感染过程中,金葡菌会产生多种酶(蛋白酶、脂肪酶和弹性蛋白酶等),这些酶会强化其攻击力,破坏宿主的组织并且向其他位置扩展和进攻(图 1)[14]。与其它细菌相比,金葡菌极强的耐药性使其具有极低的治愈率和极高的致死率,其中,耐甲氧西林金葡菌(Methicillin-resistant S. aureus,MRSA)是迄今为止最不易治疗的“超级细菌”之一[15]。
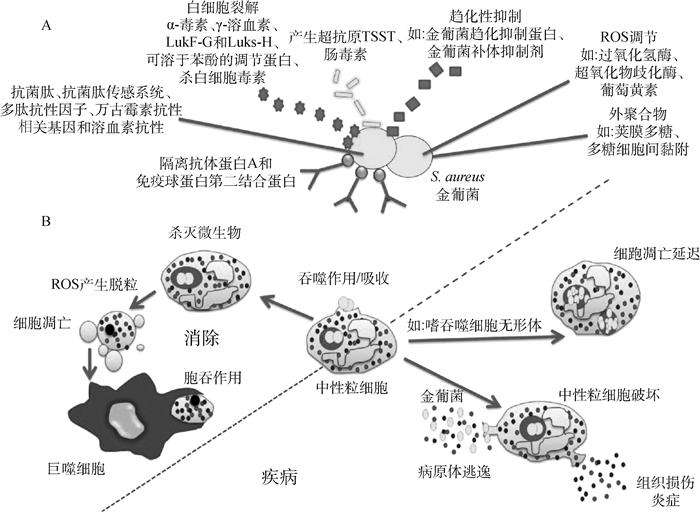
|
A.金葡菌通过防止识别、抑制免疫细胞趋化性、调节ROS、抗Amp及直接溶解白细胞等途径攻击免疫系统;B. 中性粒细胞吞噬细菌会导致ROS和脱颗粒的增加,有助于杀死所摄入的微生物并导致中性粒细胞凋亡,巨噬细胞可以将其清除以帮助解决感染。或者,细菌通过加速细胞凋亡的延迟或增加中性粒细胞的损伤,使得病原体逃逸并进入组织,进而导致疾病的发生 A. S. aureus attacks the immune system by various trials as preventing identification, preventing chemotaxis, regulating ROS, resistance to Amp, and directly lysis of leukocytes; B. Phagocytosis of bacteria by neutrophil leads to increased ROS and degranulation, which help in killing the ingested microorganism and resulted in apoptosis of neutrophil that can be removed by macrophage to aid in the resolution of infection. Alternatively, bacteria may change in normal neutrophil by accelerating a delay of apoptosis or enhanced neutrophil damage, helping the pathogen escape and get into the tissue, which leads to the occurence of disease 图 1 宿主免疫系统对抗金葡菌感染的机制[14] Fig. 1 Mechanisms of the immune system against S. aureus infection[14] |
奶牛金葡菌乳房炎通常表现出潜伏期长、高接触性传染、持续性感染、呈隐性发作、容易产生耐药性和导致乳腺坏死等特征,目前主要使用抗生素对其进行治疗[16]。但是由于金葡菌的耐药性及牛奶中抗生素残留等问题,国内外学者们正在努力探索通过遗传学手段降低奶牛金葡菌乳房炎的发生率,并通过候选基因法、全基因组关联分析(genome-wide association study, GWAS)等研究方法和转录组、蛋白质组、表观基因组等测序技术手段筛选到了许多与奶牛金葡菌乳房炎相关的抗性候选基因及重要的信号通路,为奶牛金葡菌乳房炎的抗病遗传育种提供了宝贵的数据支持。
2014—2015年,Usman等[17-18]通过候选基因法分别分析了JAK2、STAT5A和STAT5B基因SNPs与中国荷斯坦牛血清细胞因子、乳房炎和产奶性状之间的关系,认为JAK2、STAT5A和STAT5B基因可作为奶牛金葡菌乳房炎抗性的候选基因,显著的SNPs可作为潜在的分子遗传标记。目前也有研究发现JAK-STAT信号通路参与机体的免疫应答反应[19],并且与炎症、癌症和神经退行性疾病相关,但是该通路在奶牛金葡菌隐性乳房炎中的功能研究未见报道。2015年,Pawlik等[20]对CXCR1基因及该基因SNPs与金葡菌乳房炎易感性及SCCs相关性进行了研究,结果发现测定日SCCs与CXCR1的1个SNP显著相关,而CXCR1基因多态性与金葡菌乳房炎易感性相关,但是未达统计学显著水平。同年,Mazzilli等[21]通过芯片技术调查研究了33个奶牛场中分离到的94株金葡菌毒力基因类型和免疫逃逸基因分布情况,并发现与SCCs升高显著相关的一系列基因(ssl5、ssl7、Sak和SCIN等),可作为进一步研究新型抗金葡菌疫苗的候选标记基因。
2 奶牛乳房炎抗病遗传育种与转录组学研究奶牛乳房炎抗性的众多遗传变异已经得到证实,主要通过估计与乳房炎相关的表型性状(如体细胞数和临床记录等)的多基因变异的遗传力和遗传相关进行研究。从经济角度和动物福利与食品安全的考虑,加之消费者与育种工作者的关注,综合各方面因素,已将乳房炎抗性纳入到奶牛的育种目标中。
2.1 转录组测序及其在奶牛乳房炎中的研究进展随着高通量测序技术的发展,RNA-seq的应用越来越广泛。通常情况下,RNA将被转录为cDNA文库,并在其一端或两端连接衔接子(adaptors)并对其进行测序,从而获得高质量的Reads。RNA-seq是一种集合实验方法和计算机手段的技术,它可以确定生物样品中RNA序列的特征和表达丰度,即可以通过RNA测序技术识别出每条单链RNA分子中的核糖核苷酸残基的组成序列。RNA测序技术自诞生起就应用于分子生物学,用于解读各个层面的基因功能。
在牛的研究上,转录组测序技术的应用越来越广泛。2014年,McLoughlin等[22]对牛分枝杆菌感染牛外周血白细胞的转录组进行分析,并与表达芯片结果进行比较,发现RNA-seq结果具有更高的动态范围,可以更精确、更灵敏地鉴定和定量差异表达基因。大肠杆菌感染奶牛乳腺后的转录组学研究揭示了一系列差异表达基因及其显著富集的免疫反应相关通路[23-24]。某法国课题组对乳房炎易感绵羊和乳房炎抗性绵羊的乳汁体细胞进行金葡菌和表皮链球菌感染试验,并进行转录组学研究,发现乳房炎抗性的绵羊具有更活跃的免疫反应[25]。相对于乳房炎易感奶牛来说,在应对金葡菌或大肠杆菌攻击时,乳房炎抗性奶牛的乳腺上皮细胞转录组水平上的反应更强烈[26]。上述结果说明,在抵抗微生物入侵时奶牛乳腺对细菌的易感性起到重要作用,因此奶牛乳房炎抗性的选育工作至关重要。
2.2 奶牛金葡菌乳房炎抗性的转录组学研究进展自20世纪90年代以来,微阵列表达芯片逐步应用于生物学研究,在乳房炎研究上也发挥重要作用。随着高通量测序技术的发展,RNA-seq已成为了解宿主与病原体关系、探究乳房炎抗性机制的主要工具。一些转录组学研究探究了在奶牛乳房炎中乳腺组织对金黄色葡萄球菌和大肠杆菌的应答反应,以及肝组织对大肠杆菌感染和脂多糖(lipopolysaccharides, LPS)的反应。奶牛乳腺上皮细胞在乳腺的先天性免疫防御中起重要作用,有研究发现感染区与对照组的乳汁中体细胞,以及不同病原体引起的乳房炎分离的乳汁中体细胞之间广泛存在基因表达的差异性[27]。
转录组分析可以鉴定病原菌感染乳腺组织过程中对部分基因表达和通路的调控作用。不同病原体引起的先天性免疫反应通常不同,通常大肠杆菌乳房炎中的细胞因子反应更为强烈,而引起慢性感染可能是金葡菌乳房炎中炎性细胞因子上调水平较低造成的。2016年,He等[28]对健康奶牛和金葡菌乳房炎奶牛的外周血淋巴细胞进行了数字基因表达谱测序,筛选到一些参与到JAK-STAT信号通路、细胞因子-细胞因子受体互作、T细胞受体信号通路和代谢通路相关的基因与金葡菌乳房炎密切相关,可作为奶牛金葡菌乳房炎抗性候选基因。同年,Wang等[29]对健康奶牛和金葡菌乳房炎奶牛的乳腺组织进行转录组测序,筛选到一系列免疫相关基因ITGB6、MYD88、ADA、ACKR1等可作为奶牛金葡菌乳房炎抗性候选基因。2016年,Song等[30]通过DNA甲基化组和转录组的联合分析,发现了NRG1、MST1、NAT9三个基因可作为金葡菌乳房炎抗性的潜在候选基因。转录组表达数据与QTL数据的整合分析可以更好地定位QTL区域基因,在应对遗传力相对较低的复杂性状(如奶牛乳房炎)时更有效率。Fang等[31]通过检查高、低浓度金葡菌攻菌24 h后奶牛乳腺组织转录组的改变,鉴定到194个基因与高浓度金葡菌感染有关,这些基因主要参与先天性免疫反应;通过转录组与QTL数据库的联合分析,最终确定了28个与金葡菌乳房炎抗性相关的候选基因。
3 奶牛乳房炎抗病遗传育种与表观遗传学研究炎症反应受到遗传因素和非遗传因素的调控,表观遗传在炎症的发生发展中起到重要的调控作用[32]。在奶牛乳房炎的防治中,除了抗生素治疗,国内外学者正努力通过抗病遗传育种手段来提高奶牛乳房炎抗性。但是由于低遗传力和微效多基因效应,许多与乳房炎抗性相关的候选基因并不能得到有效利用。因此,仅从病原微生物或遗传学角度,均难以有效实现奶牛乳房炎的防控。表观遗传学建立了病原微生物(环境)与疾病抗性(表型)之间的联系[33](图 2),从表观遗传修饰与调控方面对炎症调控作用的研究,为金葡菌乳房炎带来了新的思路和挑战。DNA甲基化[34]是最早被发现、且目前研究最为深入的表观遗传调控机制之一,并且在细胞分化、胚胎发育和疾病发生等过程中都发挥重要作用,是当前表观遗传学研究的热点之一[35]。近年来,陆续有研究发现,DNA甲基化变动与多种癌症有关,例如肝细胞癌、胶质母细胞瘤、乳腺癌、鳞状细胞肺癌、甲状腺癌和白血病等[36-37]。同时,也有很多研究发现其与多种慢性病、代谢紊乱和神经系统疾病等有关[38-39]。
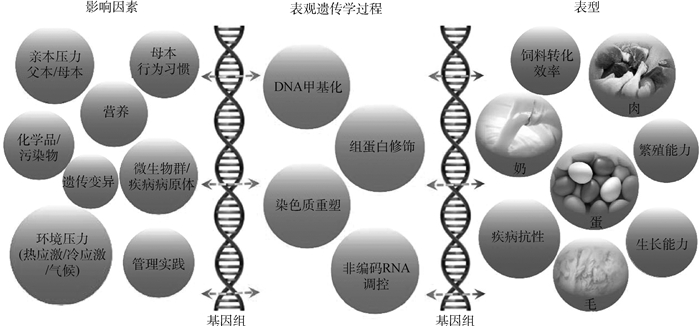
|
图为不同影响因素(左)与表观遗传学过程(中)和基因组(垂直的DNA螺旋结构)相互作用对表型(右)的影响 The figure show the common impact factors (left) that interact with epigenetic processes (center) and the genome (vertical DNA helix structures) to influence phenotypic outcome (right) 图 2 表观遗传学调控联系不同影响因素和动物表型[33] Fig. 2 The epigenetic regulatory links different impact factors and animal phenotypes[33] |
奶牛乳房炎是一种复杂疾病,受环境与病原菌的共同影响,同时也与个体自身的耐受力相关,其遗传力较低,且目前寻找到的乳房炎相关候选基因的效应较小,在不同群体中效应也有所不同,难以得到有效利用。通过抗病遗传育种的手段去控制奶牛乳房炎还有一定的难度,仅从病原菌角度或仅从遗传学角度均难以有效实现奶牛乳房炎的防控。“遗传-表观遗传假说”认为,环境因素致病(如病原菌、病毒、激素等)主要通过DNA甲基化和组蛋白修饰等表观基因组起作用,表观遗传学对炎症调控作用的研究从表观遗传修饰与调控方面为奶牛乳房炎的研究带来了新的思路与研究手段。
为应对炎症反应,宿主机体会分泌相应的细胞因子调控基因表达。DNA甲基化在调控基因表达过程中起到关键作用,且DNA甲基化与奶牛乳房炎的发生发展相关[40-41],Song等[30]在金葡菌乳房炎奶牛与健康奶牛之间观察到外周血淋巴细胞的DNA甲基化差异,发现NRG1、MST1和NAT9基因与金葡菌乳房炎的发展密切相关,是提高奶牛金葡菌乳房炎抗性的表观遗传学标记。2020年,Ju等[42]通过RRBS和RNA-seq方法首次探究了自然感染大肠杆菌乳房炎奶牛的血液中性粒细胞全基因组DNA甲基化和转录组模式,并发现在大肠杆菌乳房炎感染中起到关键作用的基因(CITED2、SLC40A1和LGR4)。这些基因通过DNA甲基化在大肠杆菌乳房炎奶牛中调控基因的差异表达,可作为大肠杆菌乳房炎抗性候选表观遗传标记。
此外,DNA甲基化对miRNA的表达具有重要影响,大约10%的miRNAs表达受到DNA甲基化的调控[43]。有研究发现差异甲基化的bta-mir-15a和bta-miR-146a在乳腺炎症的调节中起重要作用[42-44]。DNA甲基化还参与基因可变剪接事件的调控[45],2018年,Zhang等[41]报道IL6R基因的第二外显子区域的甲基化水平在乳房炎奶牛的乳腺组织中显著升高,从而促进了IL6R基因可变剪接事件的发生。Ju等[42]的研究发现,LGR4基因的一个的可变剪接体中第五外显子存在一个139 bp序列的缺失,且乳房炎奶牛中LGR4基因第五外显子的甲基化水平显著降低,推测LGR4基因第五外显子的DNA甲基化可能调节该基因的可变剪接,从而调控基因的表达。
3.2 奶牛金葡菌乳房炎抗性的表观遗传学研究进展在牛中,存在多基因相关的复杂疾病与表观遗传修饰异常有关,如奶牛乳房炎、布鲁氏杆菌病、繁殖障碍综合征等。多基因相关疾病的遗传基础较为复杂,遗传-病原体-环境间的互作是导致这类复杂疾病的主要原因。而炎症是哺乳动物复杂疾病研究最多的话题之一,同时关于炎症与癌症之间错综复杂关系的研究正在成为科学界尤其是医学界的热门领域,一直备受研究者的关注。目前关于炎症研究最为集中的是炎症相关癌症微环境,即病原——慢性炎症——癌症。
金葡菌由于其免疫逃逸机制[46-47],可以迅速适应乳腺环境并致使牛只感染隐性乳房炎[48-49]。相较于其他细菌感染,金葡菌乳房炎症状更轻,甚至呈现无症状感染,但是金葡菌乳房炎的感染持续时间长,并且表现出显著的抗药性[50]。此外,在挤奶过程中金葡菌会在牛群中进行传播[51],使得金葡菌乳房炎的防控变得难上加难。金葡菌乳房炎在给奶业带来巨大经济损失的同时,也会通过牛奶带来重大的公共健康隐患。
近年来,很多研究致力于金葡菌乳房炎的转录组和转录调控机制的探索,旨在从不同角度寻找生物标记,应用于基因组选择、分子诊断和治疗等[30-31]。miRNA是一种重要的基因转录后调控机制,Jin等[52]对奶牛乳腺上皮细胞系Mac-T细胞进行转录组测序,发现4个差异调控的miRNAs(bta-miR-2339、miR-499、miR-23a和miR-99b)可作为奶牛金葡菌乳房炎诊断和防控的生物标志物。DNA甲基化是哺乳动物基因组中最稳定的表观遗传修饰。在不同组织中鉴定表观基因组模式有助于了解表观遗传调控在家养动物健康中的作用,表观遗传修饰会动态调节病原菌感染引起的机体免疫反应(表 1)。在牛乳腺上皮细胞中,低剂量的LPS(1~10 EU·mL-1(endotoxin units per milliliter))引起的DNA高甲基化促进了脂类和氨基酸代谢等基因的表达(如ACACA、ACSS2和S6K1);而高剂量的LPS(>10 EU·mL-1)则会引起免疫应答通路中基因的低甲基化[53]。此外,与LPS对牛乳腺上皮细胞的刺激相比,LPS、肽聚糖(peptidoglycan, PGN)和脂磷壁酸(lipoteichoic acid, LTA)的共同刺激会显著降低DNA甲基化水平,导致转录组剧烈变化,炎症反应增强[54]。全基因范围内CmCGG的DNA甲基化模式与金葡菌诱导的奶牛乳房炎的免疫反应显著相关,并发现IL6R、TNF、BTK、IL1R2和TNFSF8等基因可作为奶牛金葡菌乳房炎的潜在表观遗传标记[55]。Song等[30]对金葡菌乳房炎奶牛的外周血淋巴细胞进行全基因启动子区DNA甲基化模式及修饰差异进行探究,从表观基因组和分子遗传方面深入挖掘金葡菌乳房炎抗性候选基因,并在细胞水平进行功能验证。DNA甲基化作为一种重要的表观遗传调控方式,有研究表明其可以调节乳腺健康,Wang等[56]报道CD4基因启动子区的超甲基化在临床乳房炎奶牛中抑制基因的表达。2018年,Zhang等[40]研究发现,在乳房炎奶牛中,IL6R基因的异常表达是由于DNA甲基化的变化而非基因突变引起。此外,Nckap5和转座子MTD的甲基化变化与金葡菌乳房炎的发展有关[57],而在金葡菌感染的牛乳腺上皮细胞中,整体DNA甲基化水平的下降抑制了DNA转移酶活性[58]。2020年,Wang等[55]对金葡菌乳房炎奶牛的乳腺组织进行全基因组DNA甲基化分析,发现DNA甲基化是金葡菌感染过程中调控基因表达的机制之一,并发现一系列与免疫相关的差异甲基化基因可能成为金葡菌乳房炎的候选基因。表观遗传标记可以用于提高疾病诊断的效率,节约时间和金钱成本,随着表观遗传修饰与奶牛乳房炎的联系越来越多的得到证实,表观遗传标记在奶牛金葡菌乳房炎的抗病遗传育种中的应用价值将会越来越高。
|
|
表 1 乳房炎中的表观基因组变化 Table 1 Epigenetic changes involving in mastitis |
奶牛乳房炎频发给奶牛的健康状况、生产性能及繁殖性能带来极为严重的损害,近年来,包括乳房炎抗性在内的健康性状已逐步纳入现代奶业的育种目标。本文总结了近年来有关奶牛乳房炎,特别是金葡菌乳房炎的研究进展,详细综述了奶牛乳房炎抗病遗传育种在转录组学和表观基因组学方面的研究进展。为奶牛金葡菌隐性乳房炎发病的分子标记及抗病遗传育种工作,和更好地理解奶牛金葡菌乳房炎发病的复杂过程提供参考。当然, 显著改善奶牛乳腺健康状况需要进行更广泛和深入地研究,且奶牛乳房炎的治理应该本着防大于治的思路和原则,为牧场的生产管理提供便捷。
| [1] |
MIDDLETON J R, SAEMAN A, FOX L K, et al. The national mastitis council: a global organization for mastitis control and milk quality, 50 years and beyond[J]. J Mammary Gland Biol Neoplasia, 2014, 19(3-4): 241-251. DOI:10.1007/s10911-014-9328-6 |
| [2] |
WANG X, WANG X, WANG Y, et al. Antimicrobial resistance and toxin gene profiles of Staphylococcus aureus strains from Holstein milk[J]. Lett Appl Microbiol, 2014, 58(6): 527-534. DOI:10.1111/lam.12221 |
| [3] |
EL-SAYED A, KAMEL M. Bovine mastitis prevention and control in the post-antibiotic era[J]. Trop Anim Health Prod, 2021, 53(2): 236. DOI:10.1007/s11250-021-02680-9 |
| [4] |
MEREDITH B K, KEARNEY F J, FINLAY E K, et al. Genome-wide associations for milk production and somatic cell score in Holstein-Friesian cattle in Ireland[J]. BMC Genet, 2012, 13: 21. |
| [5] |
王晓, 解小莉, 王胜, 等. 中国荷斯坦牛乳房炎易感性及抗性的全基因组关联分析[J]. 畜牧兽医学报, 2013, 44(12): 1907-1912. WANG X, XIE X L, WANG S, et al. Genome-wide association study for mastitis susceptibility and resistance in Chinese Holsteins[J]. Acta Veterinaria et Zootechnica Sinica, 2013, 44(12): 1907-1912. (in Chinese) |
| [6] |
WANG X, MA P P, LIU J F, et al. Genome-wide association study in Chinese Holstein cows reveal two candidate genes for somatic cell score as an indicator for mastitis susceptibility[J]. BMC Genet, 2015, 16: 111. |
| [7] |
冯文, 董易春, 王晓, 等. TRAPPC9基因对奶牛金葡菌乳房炎抗性性状的遗传效应[J]. 畜牧兽医学报, 2016, 47(2): 276-283. FENG W, DONG Y C, WANG X, et al. The genetic effect of TRAPPC9 on mastitis resistance to S. aureus in dairy cows[J]. Acta Veterinaria et Zootechnica Sinica, 2016, 47(2): 276-283. (in Chinese) |
| [8] |
KIRSANOVA E, HERINGSTAD B, LEWANDOWSKA-SABAT A, et al. Identification of candidate genes affecting chronic subclinical mastitis in Norwegian Red cattle: combining genome-wide association study, topologically associated domains and pathway enrichment analysis[J]. Anim Genet, 2020, 51(1): 22-31. DOI:10.1111/age.12886 |
| [9] |
RUEGG P L. A 100-Year Review: mastitis detection, management, and prevention[J]. J Dairy Sci, 2017, 100(12): 10381-10397. DOI:10.3168/jds.2017-13023 |
| [10] |
MAGRO G, REBOLINI M, BERETTA D, et al. Methicillin-resistant Staphylococcus aureus CC22-MRSA-Ⅳ as an agent of dairy cow intramammary infections[J]. Vet Microbiol, 2018, 227: 29-33. DOI:10.1016/j.vetmic.2018.10.021 |
| [11] |
ZHOU K X, LI C, CHEN D M, et al. A review on nanosystems as an effective approach against infections of Staphylococcus aureus[J]. Int J Nanomedicine, 2018, 13: 7333-7347. DOI:10.2147/IJN.S169935 |
| [12] |
FOSTER T J. Immune evasion by Staphylococci[J]. Nat Rev Microbiol, 2005, 3(12): 948-958. DOI:10.1038/nrmicro1289 |
| [13] |
GRUNDMANN H, AIRES-DE-SOUSA M, BOYCE J, et al. Emergence and resurgence of meticillin-resistant Staphylococcus aureus as a public-health threat[J]. Lancet, 2006, 368(9538): 874-885. DOI:10.1016/S0140-6736(06)68853-3 |
| [14] |
ALGHARIB S A, DAWOOD A, XIE S Y. Nanoparticles for treatment of bovine Staphylococcus aureus mastitis[J]. Drug Deliv, 2020, 27(1): 292-308. DOI:10.1080/10717544.2020.1724209 |
| [15] |
WANG D F, WANG Z C, YAN Z T, et al. Bovine mastitis Staphylococcus aureus: antibiotic susceptibility profile, resistance genes and molecular typing of methicillin-resistant and methicillin-sensitive strains in China[J]. Infect Genet Evol, 2015, 31: 9-16. DOI:10.1016/j.meegid.2014.12.039 |
| [16] |
SCHERPENZEEL C G M, HOGEVEEN H, MAAS L, et al. Economic optimization of selective dry cow treatment[J]. J Dairy Sci, 2018, 101(2): 1530-1539. DOI:10.3168/jds.2017-13076 |
| [17] |
USMAN T, WANG Y, LIU C, et al. Association study of single nucleotide polymorphisms in JAK2 and STAT5B genes and their differential mRNA expression with mastitis susceptibility in Chinese Holstein cattle[J]. Anim Genet, 2015, 46(4): 371-380. DOI:10.1111/age.12306 |
| [18] |
USMAN T, YU Y, LIU C, et al. Genetic effects of single nucleotide polymorphisms in JAK2 and STAT5A genes on susceptibility of Chinese Holsteins to mastitis[J]. Mol Biol Rep, 2014, 41(12): 8293-8301. DOI:10.1007/s11033-014-3730-4 |
| [19] |
OWEN K L, BROCKWELL N K, PARKER B S. JAK-STAT signaling: a double-edged sword of immune regulation and cancer progression[J]. Cancers (Basel), 2019, 11(12): 2002. DOI:10.3390/cancers11122002 |
| [20] |
PAWLIK A, SENDER G, KAPERA M, et al. Association between interleukin 8 receptor α gene (CXCR1) and mastitis in dairy cattle[J]. Cent Eur J Immunol, 2015, 40(2): 153-158. |
| [21] |
MAZZILLI M, PICCININI R, SCALI F, et al. Pattern characterization of genes involved in non-specific immune response in Staphylococcus aureus isolates from intramammary infections[J]. Res Vet Sci, 2015, 103: 54-59. DOI:10.1016/j.rvsc.2015.09.007 |
| [22] |
MCLOUGHLIN K E, NALPAS N C, RUE-ALBRECHT K, et al. RNA-seq transcriptional profiling of peripheral blood leukocytes from cattle infected with Mycobacterium bovis[J]. Front Immunol, 2014, 5: 396. |
| [23] |
MOYES K M, SØRENSEN P, BIONAZ M. The impact of intramammary Escherichia coli challenge on liver and mammary transcriptome and cross-talk in dairy cows during early lactation using RNAseq[J]. PLoS One, 2016, 11(6): e0157480. DOI:10.1371/journal.pone.0157480 |
| [24] |
HEIMES A, BRODHAGEN J, WEIKARD R, et al. Hepatic transcriptome analysis identifies divergent pathogen-specific targeting-strategies to modulate the innate immune system in response to intramammary infection[J]. Front Immunol, 2020, 11: 715. DOI:10.3389/fimmu.2020.00715 |
| [25] |
BONNEFONT C M D, TOUFEER M, CAUBET C, et al. Transcriptomic analysis of milk somatic cells in mastitis resistant and susceptible sheep upon challenge with Staphylococcus epidermidis and Staphylococcus aureus[J]. BMC Genom, 2011, 12: 208. DOI:10.1186/1471-2164-12-208 |
| [26] |
BRAND B, HARTMANN A, REPSILBER D, et al. Comparative expression profiling of E. coli and S. aureus inoculated primary mammary gland cells sampled from cows with different genetic predispositions for somatic cell score[J]. Genet Sel Evol, 2011, 43(1): 24. DOI:10.1186/1297-9686-43-24 |
| [27] |
PISONI G, MORONI P, GENINI S, et al. Differentially expressed genes associated with Staphylococcus aureus mastitis in dairy goats[J]. Vet Immunol Immunopathol, 2010, 135: 208-217. DOI:10.1016/j.vetimm.2009.11.016 |
| [28] |
HE Y H, SONG M Y, ZHANG Y, et al. Whole-genome regulation analysis of histone H3 lysin 27 trimethylation in subclinical mastitis cows infected by Staphylococcus aureus[J]. BMC Genom, 2016, 17: 565. DOI:10.1186/s12864-016-2947-0 |
| [29] |
WANG X G, JU Z H, HOU M H, et al. Deciphering transcriptome and complex alternative splicing transcripts in mammary gland tissues from cows naturally infected with Staphylococcus aureus mastitis[J]. PLoS One, 2016, 11(7): e0159719. DOI:10.1371/journal.pone.0159719 |
| [30] |
SONG M Y, HE Y H, ZHOU H K, et al. Combined analysis of DNA methylome and transcriptome reveal novel candidate genes with susceptibility to bovine Staphylococcus aureus subclinical mastitis[J]. Sci Rep, 2016, 6: 29390. DOI:10.1038/srep29390 |
| [31] |
FANG L Z, HOU Y L, AN J, et al. Genome-wide transcriptional and post-transcriptional regulation of innate immune and defense responses of bovine mammary gland to Staphylococcus aureus[J]. Front Cell Infect Microbiol, 2016, 6: 193. |
| [32] |
RAMOS-LOPEZ O, MILAGRO F I, RIEZU-BOJ J I, et al. Epigenetic signatures underlying inflammation: an interplay of nutrition, physical activity, metabolic diseases, and environmental factors for personalized nutrition[J]. Inflamm Res, 2021, 70(1): 29-49. DOI:10.1007/s00011-020-01425-y |
| [33] |
WANG M Q, IBEAGHA-AWEMU E M. Impacts of epigenetic processes on the health and productivity of livestock[J]. Front Genet, 2021, 11: 613636. DOI:10.3389/fgene.2020.613636 |
| [34] |
HALUŠKOVÁ J, HOLEČKOVÁ B, STANIČOVÁ J. DNA methylation studies in cattle[J]. J Appl Genet, 2021, 62(1): 121-136. DOI:10.1007/s13353-020-00604-1 |
| [35] |
GREENBERG M V C, BOURC'HIS D. The diverse roles of DNA methylation in mammalian development and disease[J]. Nat Rev Mol Cell Biol, 2019, 20(10): 590-607. DOI:10.1038/s41580-019-0159-6 |
| [36] |
ENNOUR-IDRISSI K, DRAGIC D, DUROCHER F, et al. Epigenome-wide DNA methylation and risk of breast cancer: a systematic review[J]. BMC Cancer, 2020, 20(1): 1048. DOI:10.1186/s12885-020-07543-4 |
| [37] |
REID B M, FRIDLEY B L. DNA methylation in ovarian cancer susceptibility[J]. Cancers (Basel), 2020, 13(1): 108. DOI:10.3390/cancers13010108 |
| [38] |
ZHANG B, ZHOU T, WU H J, et al. Difference of IFI44L methylation and serum IFN-a1 level among patients with discoid and systemic lupus erythematosus and healthy individuals[J]. J Transl Autoimmun, 2021, 4: 100092. DOI:10.1016/j.jtauto.2021.100092 |
| [39] |
MAHONY C, O'RYAN C. Convergent canonical pathways in autism spectrum disorder from proteomic, transcriptomic and DNA methylation data[J]. Int J Mol Sci, 2021, 22(19): 10757. DOI:10.3390/ijms221910757 |
| [40] |
CHANG G J, PETZL W, VANSELOW J, et al. Epigenetic mechanisms contribute to enhanced expression of immune response genes in the liver of cows after experimentally induced Escherichia coli mastitis[J]. Vet J, 2015, 203(3): 339-341. DOI:10.1016/j.tvjl.2014.12.023 |
| [41] |
ZHANG Y, WANG X G, JIANG Q, et al. DNA methylation rather than single nucleotide polymorphisms regulates the production of an aberrant splice variant of IL6R in mastitic cows[J]. Cell Stress Chaperones, 2018, 23(4): 617-628. DOI:10.1007/s12192-017-0871-0 |
| [42] |
JU Z H, JIANG Q, WANG J P, et al. Genome-wide methylation and transcriptome of blood neutrophils reveal the roles of DNA methylation in affecting transcription of protein-coding genes and miRNAs in E. coli-infected mastitis cows[J]. BMC Genom, 2020, 21(1): 102. DOI:10.1186/s12864-020-6526-z |
| [43] |
HAN L F, WITMER P D, CASEY E, et al. DNA methylation regulates MicroRNA expression[J]. Cancer Biol Ther, 2007, 6(8): 1284-1288. |
| [44] |
LI Z X, ZHANG H L, SONG N, et al. Molecular cloning, characterization and expression of miR-15a-3p and miR-15b-3p in dairy cattle[J]. Mol Cell Probes, 2014, 28(5-6): 255-258. DOI:10.1016/j.mcp.2014.06.003 |
| [45] |
LEV MAOR G, YEARIM A, AST G. The alternative role of DNA methylation in splicing regulation[J]. Trends Genet, 2015, 31(5): 274-280. DOI:10.1016/j.tig.2015.03.002 |
| [46] |
DARISIPUDI M N, BRÖKER B M. How S. aureus blinds the inflammasome to escape immune control[J]. EBioMedicine, 2021, 71: 103549. DOI:10.1016/j.ebiom.2021.103549 |
| [47] |
LIAO X Y, HU W C, LIU D H, et al. Stress resistance and pathogenicity of nonthermal-plasma-induced viable-but-nonculturable Staphylococcus aureus through energy suppression, oxidative stress defense, and immune-escape mechanisms[J]. Appl Environ Microbiol, 2021, 87(2): e02380-20. |
| [48] |
BRADLEY A J. Bovine mastitis: an evolving disease[J]. Vet J, 2002, 164(2): 116-128. DOI:10.1053/tvjl.2002.0724 |
| [49] |
SCHUKKEN Y H, GVNTHER J, FITZPATRICK J, et al. Host-response patterns of intramammary infections in dairy cows[J]. Vet Immunol Immunopathol, 2011, 144(3-4): 270-289. DOI:10.1016/j.vetimm.2011.08.022 |
| [50] |
JENSEN K, GVNTHER J, TALBOT R, et al. Escherichia coli- and Staphylococcus aureus-induced mastitis differentially modulate transcriptional responses in neighbouring uninfected bovine mammary gland quarters[J]. BMC Genom, 2013, 14(1): 36. DOI:10.1186/1471-2164-14-36 |
| [51] |
RADOSTITS O M. Herd health: food animal production medicine[M]. 3rd ed. Philadelphia: W. B. Saunders, 2001.
|
| [52] |
JIN W W, IBEAGHA-AWEMU E M, LIANG G X, et al. Transcriptome microRNA profiling of bovine mammary epithelial cells challenged with Escherichia coli or Staphylococcus aureus bacteria reveals pathogen directed microRNA expression profiles[J]. BMC Genom, 2014, 15: 181. DOI:10.1186/1471-2164-15-181 |
| [53] |
CHEN J B, WU Y J, SUN Y W, et al. Bacterial lipopolysaccharide induced alterations of genome-wide DNA methylation and promoter methylation of lactation-related genes in bovine mammary epithelial cells[J]. Toxins (Basel), 2019, 11(5): 298. DOI:10.3390/toxins11050298 |
| [54] |
WU Y J, SUN Y W, DONG X W, et al. The synergism of PGN, LTA and LPS in inducing transcriptome changes, inflammatory responses and a decrease in lactation as well as the associated epigenetic mechanisms in bovine mammary epithelial cells[J]. Toxins (Basel), 2020, 12(6): 387. DOI:10.3390/toxins12060387 |
| [55] |
WANG M Q, LIANG Y, IBEAGHA-AWEMU E M, et al. Genome-wide DNA methylation analysis of mammary gland tissues from chinese holstein cows with Staphylococcus aureus induced mastitis[J]. Front Genet, 2020, 11: 550515. DOI:10.3389/fgene.2020.550515 |
| [56] |
WANG X S, ZHANG Y, HE Y H, et al. Aberrant promoter methylation of the CD4 gene in peripheral blood cells of mastitic dairy cows[J]. Genet Mol Res, 2013, 12(4): 6228-6239. DOI:10.4238/2013.December.4.10 |
| [57] |
WANG D, WEI Y Y, SHI L Y, et al. Genome-wide DNA methylation pattern in a mouse model reveals two novel genes associated with Staphylococcus aureus mastitis[J]. Asian-Australas J Anim Sci, 2020, 33(2): 203-211. DOI:10.5713/ajas.18.0858 |
| [58] |
WU Y J, CHEN J B, SUN Y W, et al. PGN and LTA from Staphylococcus aureus induced inflammation and decreased lactation through regulating DNA methylation and histone H3 acetylation in bovine mammary epithelial cells[J]. Toxins (Basel), 2020, 12(4): 238. DOI:10.3390/toxins12040238 |
| [59] |
WANG J, YAN X X, NESENGANI L T, et al. LPS-induces IL-6 and IL-8 gene expression in bovine endometrial cells "through DNA methylation"[J]. Gene, 2018, 677: 266-272. DOI:10.1016/j.gene.2018.07.074 |
| [60] |
USMAN T, YU Y, WANG Y. P2001 CD4 promoter hyper methylation is associated with lower gene expression in clinical mastitis cows and vice versa in the healthy controls[J]. J Anim Sci, 2016, 94(S4): 38. |
| [61] |
VANSELOW J, YANG W, HERRMANN J, et al. DNA-remethylation around a STAT5-binding enhancer in the αS1-casein promoter is associated with abrupt shutdown of αS1-casein synthesis during acute mastitis[J]. J Mol Endocrinol, 2006, 37(3): 463-477. DOI:10.1677/jme.1.02131 |
(编辑 郭云雁)



