细菌性脑膜炎是一种较为严重的感染性疾病,会危害人类健康,影响畜禽养殖业的健康发展[1]。在人医方面,细菌性脑膜炎是十大引发高死亡率的感染性疾病之一,且大部分幸存者会有不同程度的神经系统后遗症[2-3]。在兽医方面,养殖动物由于患脑膜炎失去了其经济价值,给畜牧业造成较大经济损失。
鸭疫里默氏杆菌(Riemerella anatipestifer,RA) 是一种革兰阴性菌,主要感染雏鸭,导致鸭浆膜炎和脑膜炎。RA也可以感染鸡、火鸡、鹅、部分野生水禽等多种禽类。感染RA的动物,尤其是易感鸭会出现典型的神经症状,包括头颈震颤或痉挛性点头、共济失调、头颈歪斜、角弓反张、转圈等,最后衰竭死亡[4]。目前已经确定的RA血清型有21种,但每种血清型之间缺乏交叉保护,不利于疫苗免疫的普及。养鸭场在日常饲养中添加抗生素依然是最常用的防治RA的手段,但随着抗生素的应用,耐药菌株不断出现和播散[5],给RA病的防控带来极大挑战。基于此,筛选并鉴定RA突破血脑屏障(blood brain barrier,BBB)而导致脑膜炎的相关毒力因子,研发出针对关键毒力因子的高免血清,并将其作为阻断RA感染脑组织的一种生物制品,是一种可行的研究思路。
已有的研究对RA的致病机制了解甚少,仅仅发现了几种与RA毒力相关的基因,包括OmpA[6]、VapD[7]、Yb2株的AS87_04050(参与脂多糖合成)[8]和AS87_03730[9]、CH-1株的B739_2187[10]和IX型分泌系统组分[11]。研究者发现, 这些基因缺失或突变后,菌株对雏鸭的致病力有不同程度的减弱,但并未说明这些毒力基因是否与RA导致的脑膜炎相关。对于介导RA突破BBB感染脑组织,进而导致脑膜炎的相关毒力因子的研究还未见报道。
当前研究表明,烯醇化酶(Enolase)是介导猪链球菌2型突破BBB的毒力因子[12-14]。肺炎链球菌Enolase除了利用宿主的纤溶系统促进病原对宿主的入侵外,还可与宿主细胞来源的胞外RNA(eRNA)结合,进而促进细菌对肺泡上皮细胞的入侵[15]。金黄色葡萄球菌Enolase定位于细菌表面,与宿主细胞外基质的一种主要成分层黏连蛋白相互作用,参与细菌的扩散和侵染过程[16]。
迄今为止,关于RA Enolase的生物学功能还未见报道。本研究通过构建enolase基因缺失株和回复株,测定菌株的生长速率,对鸭脑微血管内皮细胞(DBMEC)的黏附和入侵能力,感染动物的血液及脑组织载菌量,以期明确enolase基因在RA突破BBB中是否发挥作用。
1 材料和方法 1.1 材料1.1.1 菌株和质粒 鸭疫里默氏杆菌RA-LZ01菌株(GenBank登录号:NZ_CP045564.1)保存于中国农业科学院兰州兽医研究所草食动物细菌病创新团队;大肠杆菌ATCC25922购自美国菌种保藏中心(American type culture collection);大肠杆菌X7213和自杀质粒pRE112均购自NTCC国家典型培养物保藏中心;穿梭质粒pCPRA由本实验室构建;Trans-1感受态细胞购自北京全式金公司。
1.1.2 培养基和主要试剂 胰蛋白酶大豆肉汤(TSB)、胰蛋白酶大豆琼脂(TSA)均购自美国BD difcoTM公司;LB肉汤购自英国Oxoid公司;琼脂粉购自北京Solarbio公司;2, 6-二氨基更二酸(DPA)购自东京化成工业株式会社;抗生素购自北京Solarbio公司;限制性内切酶SacI、SphI,质粒提取试剂盒,胶回收试剂盒均购自TaKaRa公司;0.22 μm无菌硝酸纤维素膜和过滤器购自美国Millipore公司。
1.1.3 主要仪器 紫外分光光度计和台式高速离心机购自美国Beckman公司;PCR热循环仪购自杭州博日科技公司;CO2恒温培养箱购自美国Thermo公司;摇床购自上海苏坤实业有限公司;凝胶成像仪购自美国Bio-Rad公司。
1.2 方法1.2.1 引物设计 引物均用DNASTAR-Editseq软件进行设计,由西安擎科生物有限公司合成。本研究中所使用的引物和扩增目的片段如表 1所示。
|
|
表 1 PCR引物名称和序列 Table 1 PCR primers and their sequences used in this study |
1.2.2 鸭脑微血管内皮细胞(DBMEC)的培养和鉴定 从樱桃谷鸭胚中分离出大脑皮质,除去脑膜等,反复用PBS冲洗干净,将组织块剪成约0.1 cm3的小块,置于培养瓶中,加入2 mL DBMEC完全培养基,确保不能将组织块悬浮,倒置于5% CO2细胞培养箱中2 h,然后将细胞培养瓶正置,待组织块周围生长的细胞融合成片,即可去除组织块,用胰蛋白酶消化细胞,重新铺瓶,传3~4代,得到符合BMEC形态的细胞。利用血管假性血友病因子(vWF)免疫荧光染色进行鉴定,得到合适的DBMEC;SV40过表达慢病毒转染DBMEC,利用嘌呤霉素(2 mg·L-1)的筛选培养基进行筛选。以未转染的DBMEC细胞作为对照,该细胞被全部杀灭,而在同一时间点存活的转染了SV40过表达慢病毒的细胞为转染成功的DBMEC,将细胞传代至12代以上,得到永生化的DBMEC。
1.2.3 enolase基因缺失株的构建 如表 1所示,以亲本株RA-LZ01菌株基因组作为模板,用Enolase-p1/Enolase-p2和Enolase-p5/Enolase-p6引物扩增出enolase基因的Enolase-UP和Enolase-DO;以LJW-2菌株基因组为模板,用ErmF-p3/ErmF-p4引物扩增出ermF抗性基因;通过重叠PCR将Enolase-UP、ermF抗性基因和Enolase-DO依次进行融合,构建出打靶片段;用SacΙ和SphΙ将打靶片段克隆进自杀质粒pRE112,转化进大肠杆菌X7213,利用含DPA和氯霉素的LB平板筛选阳性供体菌,再与RA-LZ01菌株共培养,利用含红霉素和多黏菌素B的TSA平板筛选缺失株,具体方法参考文献[17-18]。得到的菌株用RA保守引物OmpA-F/OmpA-R、Enolase-p1/Enolase-p6、ErmF-p3/ErmF-p4、Enolase-F/Enolase-R进行PCR鉴定,并对Enolase-p1/Enolase-p6扩增得到的片段进行测序,得到的enolase基因缺失株命名为ΔEnolase。
1.2.4 enolase基因回复株的构建 用表 1中cEnolase-p1/cEnolase-p2引物,PCR扩增出pro+Enolase基因目的条带(含有enolase基因的启动子)。将扩增出的片段用PstI和SphI克隆进pCPRA载体中,并转化进X7213菌株中,利用氨苄青霉素抗性筛选供体菌,与ΔEnolase共培养,再利用头孢西丁和多黏菌素B抗性筛选回复株,方法参考文献[17-18]。用引物cEnolase-p1/cEnolase-p2、ErmF-p3/ErmF-p4和OmpA-F/OmpA-R进行鉴定,得到的回复株命名为cΔEnolase。
1.2.5 菌株生长曲线的测定 测定亲本株RA-LZ01、缺失株ΔEnolase和回复株cΔEnolase 12 h内的生长曲线,具体方法参考文献[17-18]。
1.2.6 菌株对DBMEC的黏附和入侵 将亲本株RA-LZ01、缺失株ΔEnolase以及回复株cΔEnolase分别增菌至OD600约为1.0。用无菌0.01 mol·L-1 PBS(pH 7.4)调整至OD600值均为1.0,测定每株菌的菌落形成单位(colony forming unit,CFU)。用无血清内皮细胞基础培养基稀释菌液,用100个MOI(multiplicity of infection)将菌液(1 mL·孔-1)接入铺满了单层DBMEC的24孔细胞培养板中,置于37 ℃,5% CO2培养箱中孵育2 h,弃上清后用无菌PBS重复洗涤3次,每孔加入1 mL 0.25%的胰酶消化细胞,转移进1.5 mL离心管中,5 000 r·min-1离心后弃上清,并用PBS重复洗涤2次,加入0.5 mL无菌双蒸水,室温作用30 min,用PBS以10倍梯度稀释,涂板于含多黏菌素B的TSA平板上,在CO2培养箱中培养至长出单菌落,计算黏附菌株的CFU。每株菌做3个重复,进行3次独立试验。
另外,菌液(1 mL·孔-1)接入铺满单层DBMEC的24孔细胞培养板中后,置于37 ℃,5% CO2培养箱中孵育3 h,用无菌PBS充分洗涤3次后,加入用内皮细胞基础培养基稀释好的终浓度为100 mg·L-1的氨苄青霉素钠,0.5 mL·孔-1,孵育1 h后,弃上清,经过胰酶消化、PBS洗涤以及双蒸水作用后,测定入侵菌株的CFU。每株菌做3个重复,进行3次独立试验[19-20]。
黏附率和入侵率的计算:黏附率(%)=黏附菌株的CFU/接种菌株的CFU×100;入侵率(%)=入侵菌株的CFU/接种菌株的CFU×100[21]。菌株之间的差异以相对黏附率和入侵率进行比较,把亲本株RA-LZ01对DBMEC的黏附率和入侵率设置为100%[21]。
1.2.7 动物试验 30只7日龄樱桃谷雏鸭随机分为3组,每组10只,用亲本株RA-LZ01、缺失株ΔEnolase和回复株cΔEnolase对雏鸭进行攻毒,攻毒剂量为107 CFU·只-1,攻毒后40 h检测雏鸭血液和脑组织中的载菌量[4]。
1.3 统计学分析试验数据用GraphPad Prism 6.0软件进行分析,所有数据由3次重复试验结果得到,数据采用One-way ANOVA进行分析,P < 0.05认为差异显著,具有生物统计学意义。
2 结果 2.1 DBMEC的培养和鉴定利用vWF免疫荧光对传至12代以上的DBMEC进行鉴定后,对照细胞Vero无绿色荧光出现,而DBMEC观察到绿色荧光,说明DBMEC携带vWF,为血管内皮细胞,培养成功永生化的DBMEC,细胞形态如图 1所示。
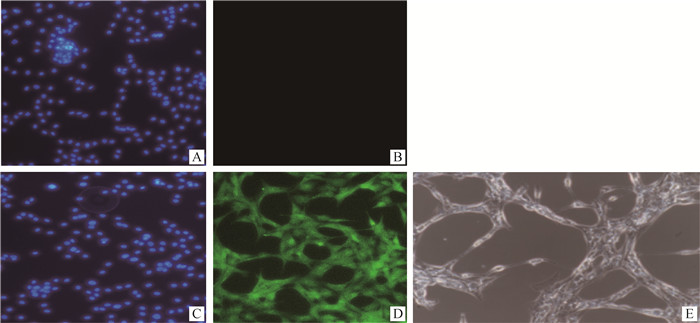
|
A. Vero细胞DAPI染色(200×);B. Vero细胞vWF免疫荧光(200×);C. DBMEC DAPI染色(200×);D. DBMEC vWF免疫荧光(200×);E. DBMEC(200×) A. DPAI staining of Vero cells (200×); B. vWF immunofluorescence from Vero cell (200×); C. DPAI staining of DBMEC (200×); D. vWF immunofluorescence from DBMEC (200×); E. DBMEC (200×) 图 1 DBMEC的鉴定 Fig. 1 Identification of DBMEC |
用Enolase-p1/Enolase-p6引物扩增出2 025 bp大小的目的条带(经测序确定为打靶片段);用ErmF-p3/ErmF-p4引物扩增出801 bp大小的ermF基因;用OmpA-F/OmpA-R引物扩增出1 467 bp大小的目的条带;用Enolase-F/Enolase-R引物未扩增出条带,说明缺失株ΔEnolase构建成功(图 2A)。

|
A. enolase基因缺失株ΔEnolase的鉴定;M. DL5000分子质量标准;1. Enolas-p1/Enolase-p6引物鉴定(2 025 bp);2. ErmF-p3/ErmF-p4引物鉴定(801 bp);3. OmpA-F/OmpA-R引物鉴定(1 467 bp);4. Enolase-F/Enolase-R引物鉴定。B. enolase基因回复株cΔEnolase的鉴定;M. DL2000分子质量标准;1. cEnolase-p1/cEnolase-p2引物鉴定(1 829 bp);2. OmpA-F/OmpA-R引物鉴定(1 467 bp);3. ErmF-p3/ErmF-p4引物鉴定(801 bp) A. Identification of enolase gene deletion mutant ΔEnolase; M. DL5000 relative molecular mass standard; 1. Enolase-p1/Enolase-p6 primer identification (2 025 bp); 2. ErmF-p3/ErmF-p4 primer identification (801 bp); 3. OmpA-F/OmpA-R primer identification (1 467 bp); 4. Enolase-F/Enolase-R primer identification. B. Identification of the complemented strain cΔEnolase; M. DL2000 relative molecular mass standard; 1. cEnolase-p1/cEnolase-p2 primer identification (1 829 bp); 2. OmpA-F/OmpA-R primer identification (1 467 bp); 3. ErmF-p3/ErmF-p4 primer identification (801 bp) 图 2 enolase基因缺失株ΔEnolase和回复株cΔEnolase的鉴定 Fig. 2 Identification of enolase gene deletion mutant ΔEnolase and complemented strain cΔEnolase |
用cEnolase-p1/cEnolase-p2引物扩增出1 829 bp大小的目的条带(测序确定);用ErmF-p3/ErmF-p4引物扩增出801 bp大小的ermF基因;用OmpA-F/OmpA-R引物扩增出1 467 bp大小的目的条带,说明回复株cΔEnolase构建成功(图 2B)。
2.3 生长曲线的测定与亲本株RA-LZ01相比,在测定的12 h内,缺失株ΔEnolase在4~10 h内的生长速率显著下降(P < 0.01, 图 3);回复株cΔEnolase的生长速率与亲本株相比慢约1 h(达到基本一致的OD600 nm值的时间),相较缺失株其生长速率稍有提高,但没有完全恢复。
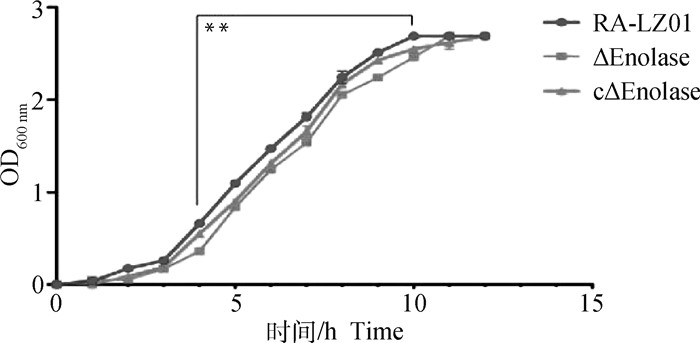
|
**. P<0.01,下同 **. P < 0.01, the same as below 图 3 亲本株、缺失株及回复株生长曲线 Fig. 3 Growth curves of parent strain, deletion mutant and complemented strain |
与亲本株RA-LZ01相比,缺失株ΔEnolase对DBMEC的相对黏附率和入侵率均极显著降低(P < 0.001,P < 0.0001),分别为亲本株黏附率的5.9%和入侵率的2.5%;回复株cΔEnolase显著恢复了对DBMEC的黏附能力,为亲本株黏附率的79.6%;几乎完全恢复了对DBMEC的入侵能力,为亲本株入侵率的97.5%(图 4)。
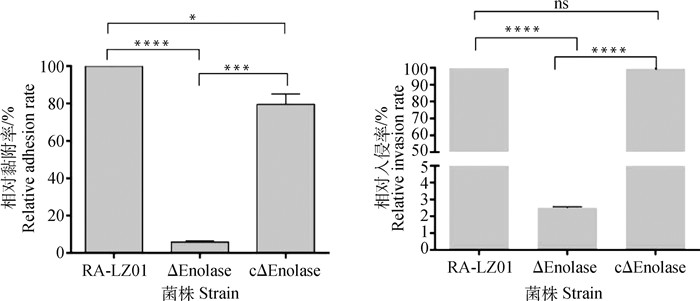
|
*. P<0.05;***. P<0.001;****. P<0.000 1;ns. 无显著差异。下同*. P < 0.05; ***. P < 0.001; ****. P < 0.000 1; ns. No significant difference. The same as below 图 4 RA-LZ01、ΔEnolase和cΔEnolase菌株对DBMEC的相对黏附率和入侵率 Fig. 4 Relative adhesion and invasion rates of RA-LZ01, ΔEnolase and cΔEnolase to DBMEC |
感染亲本株RA-LZ01、缺失株ΔEnolase及回复株cΔEnolase的雏鸭,其血液载菌量差异不显著;但感染ΔEnolase的雏鸭,其脑组织中的载菌量极显著降低(P < 0.01, 图 5)。

|
A.血液载菌量;B.脑组织载菌量 A. The amount of bacteria in blood; B. Bacterial count in brain 图 5 RA-LZ01、ΔEnolase和cΔEnolase菌株感染雏鸭血液及脑组织载菌量的测定 Fig. 5 Determination of bacteria loads in blood and brain of duckling infected by RA-LZ01, ΔEnolase and cΔEnolase strains, respectively |
细菌导致宿主发生脑膜炎,首先要突破宿主的血脑屏障(BBB),进而引起中枢神经系统感染和脑膜炎。BBB是指脑毛细血管壁与神经胶质细胞形成的血液与脑组织之间,以及由脉络丛形成的血液和脑脊液之间的屏障,BBB也是机体参与固有免疫的内部屏障之一[22-23]。BBB的细胞组分主要包括脑微血管内皮细胞(BMEC)、周细胞和星形胶质细胞。大量研究表明,BMEC是研究细菌突破BBB的经典模式细胞,以宿主的BMEC作为体外细胞模型,已经发现了多个与细菌侵袭血脑屏障相关的毒力因子[12-13, 20-21]。本研究成功培养了DBMEC,用于筛选并鉴定鸭疫里默氏杆菌侵袭鸭BBB的相关毒力因子。
Enolase是糖酵解过程中的关键酶,在细胞糖酵解过程中发挥催化作用,为细菌细胞的生长提供能量[24-25],广泛存在于真核和原核生物中。Enolase有3种异构体(α、β、γ),原核生物中一般仅存在α-enolase,因此多种细菌表面存在α-enolase,通常称为Enolase。现有研究表明,Enolase是多种细菌的毒力因子,可介导细菌与宿主靶细胞发生黏附和结合,在细菌感染宿主过程中发挥着重要作用。尤其是有研究表明,Enolase可与猪链球菌2型(SS2)的BMEC结合,直接介导SS2对宿主BBB的侵袭;还可以促进IL-8细胞因子的释放,增强BBB的通透性[12-14]。在肺炎链球菌中,Enolase除了能够利用宿主的纤溶系统促进病原对宿主的入侵外,还可与宿主细胞的胞外RNA(eRNA)结合,从而促进了细菌对肺泡上皮细胞的入侵[15]。对福赛斯坦纳菌Enolase的研究表明,Enolase在细菌感染机体诱导炎症的过程中发挥了重要作用[26]。金黄色葡萄球菌的Enolase定位于细菌的表面,与宿主细胞外基质的一种主要成分层黏连蛋白相互作用,参与细菌的扩散和侵染过程[16]。Enolase是铜绿假单胞菌RNA降解体的重要组成成分之一,参与铜绿假单胞菌RNA的加工和基因的调节过程;Enolase也是铜绿假单胞菌的重要毒力因子,在诱导小鼠急性肺炎模型中发挥重要作用,缺失enolase基因使铜绿假单胞菌对氧化应激的耐受能力减弱,使得铜绿假单胞菌更容易被宿主体内的中性粒细胞所杀伤[27]。
Enolase在已知的测通全序的鸭疫里默氏杆菌中高度保守,核苷酸序列同源性为97.37%~100%,氨基酸序列同源性为93.95%~100%,目前对于该基因的生物学功能还未见报道。本研究构建了鸭疫里默氏杆菌的enolase基因缺失株和回复株,对缺失株的生物学表型进行了研究,发现与亲本株RA-LZ01相比,缺失株ΔEnolase的生长速率显著变慢,说明enolase基因缺失后会影响RA-LZ01菌株的生长,在RA-LZ01的生长中发挥重要作用。
对DBMEC的黏附侵袭试验中,发现ΔEnolase对DBMEC的相对黏附率和相对入侵率均极显著降低,而cΔEnolase对DBMEC的黏附侵袭均有回补作用,且其相对入侵率与亲本株相比差异不显著,说明Enolase在RA-LZ01黏附和侵袭DBMEC过程中发挥重要作用。
为进一步验证enolase基因缺失后菌株在黏附和侵袭DBMEC中发生的表型改变,对樱桃谷雏鸭进行了攻毒试验,确定各菌株对雏鸭BBB的侵袭能力。试验结果发现,感染亲本株RA-LZ01、缺失株ΔEnolase及回复株cΔEnolase的雏鸭,其血液中的载菌量没有显著差异,说明enolase基因不影响菌株在雏鸭血液中的存活;但与感染亲本株和回复株的雏鸭相比,感染缺失株的雏鸭脑组织中的载菌量极显著下降,且雏鸭的精神状态相对较好,说明enolase基因缺失致弱了菌株入侵脑组织的能力,提示enolase基因在鸭疫里默氏杆菌突破BBB中发挥重要作用。
4 结论成功构建了鸭疫里默氏杆菌enolase基因缺失株ΔEnolase和回复株cΔEnolase,发现enolase基因参与菌株的生长,显著影响菌株对DBMEC的黏附、入侵以及对雏鸭脑组织的入侵,在鸭疫里默氏杆菌突破鸭BBB中发挥重要作用。
| [1] |
LIU L, JOHNSON H L, COUSENS S, et al. Global, regional, and national causes of child mortality: An updated systematic analysis for 2010 with time trends since 2000[J]. Lancet, 2012, 379(9832): 2151-2161. DOI:10.1016/S0140-6736(12)60560-1 |
| [2] |
DE GANS J, VAN DE BEEK D. Dexamethasone in adults with bacterial meningitis[J]. N Engl J Med, 2002, 347(20): 1549-1556. DOI:10.1056/NEJMoa021334 |
| [3] |
VAN DE BEEK D, DE GANS J, SPANJAARD L, et al. Clinical features and prognostic factors in adults with bacterial meningitis[J]. N Engl J Med, 2004, 351(18): 1849-1859. DOI:10.1056/NEJMoa040845 |
| [4] |
LI S D, GONG X W, CHEN Q W, et al. Threshold level of Riemerella anatipestifer crossing blood-brain barrier and expression profiles of immune-related proteins in blood and brain tissue from infected ducks[J]. Vet Immunol Immunopathol, 2018, 200: 26-31. DOI:10.1016/j.vetimm.2018.04.005 |
| [5] |
吴彩艳, 程淑琴, 张建峰, 等. 我国鸭疫里氏杆菌病流行概述[J]. 动物医学进展, 2017, 38(6): 86-90. WU C Y, CHENG S Q, ZHANG J F, et al. Introduction to epidemiology of Riemerellosis anatipestifer disease in China[J]. Progress in Veterinary Medicine, 2017, 38(6): 86-90. DOI:10.3969/j.issn.1007-5038.2017.06.020 (in Chinese) |
| [6] |
HU Q H, HAN X A, ZHOU X J, et al. OmpA is a virulence factor of Riemerella anatipestifer[J]. Vet Microbiol, 2011, 150(3-4): 278-283. DOI:10.1016/j.vetmic.2011.01.022 |
| [7] |
WENG S C, LIN W H, CHANG Y F, et al. Identification of a virulence-associated protein homolog gene and ISRa1 in a plasmid of Riemerella anatipestifer[J]. FEMS Microbiol Lett, 1999, 179(1): 11-19. DOI:10.1111/j.1574-6968.1999.tb08701.x |
| [8] |
WANG X L, DING C, WANG S H, et al. The AS87_04050 gene is involved in bacterial lipopolysaccharide biosynthesis and pathogenicity of Riemerella anatipestifer[J]. PLoS One, 2014, 9(10): e109962. DOI:10.1371/journal.pone.0109962 |
| [9] |
WANG X L, YUE J P, DING C, et al. Deletion of AS87_03730 gene changed the bacterial virulence and gene expression of Riemerella anatipestifer[J]. Sci Rep, 2016, 6: 22438. DOI:10.1038/srep22438 |
| [10] |
ZHAO X X, LIU Q, ZHANG J, et al. Identification of a gene in Riemerella anatipestifer CH-1 (B739-2187) that contributes to resistance to polymyxin B and evaluation of its mutant as a live attenuated vaccine[J]. Microb Pathog, 2016, 91: 99-106. DOI:10.1016/j.micpath.2015.12.001 |
| [11] |
GUO Y Q, HU D, GUO J, et al. Riemerella anatipestifer type IX secretion system is required for virulence and gelatinase secretion[J]. Front Microbiol, 2017, 8: 2553. DOI:10.3389/fmicb.2017.02553 |
| [12] |
LIU H T, ZHU S, SUN Y Y, et al. Selection of potential virulence factors contributing to Streptococcus suis serotype 2 penetration into the blood-brain barrier in an in vitro Co-culture model[J]. J Microbiol Biotechnol, 2017, 27(1): 161-170. DOI:10.4014/jmb.1606.06018 |
| [13] |
SUN Y Y, LI N, ZHANG J, et al. Enolase of Streptococcus Suis serotype 2 enhances blood-brain barrier permeability by inducing IL-8 release[J]. Inflammation, 2016, 39(2): 718-726. DOI:10.1007/s10753-015-0298-7 |
| [14] |
XIA X J, QIN W H, ZHU H L, et al. How Streptococcus suis serotype 2 attempts to avoid attack by host immune defenses[J]. J Microbiol Immunol Infect, 2019, 52(4): 516-525. DOI:10.1016/j.jmii.2019.03.003 |
| [15] |
ZAKRZEWICZ D, BERGMANN S, DIDIASOVA M, et al. Host-derived extracellular RNA promotes adhesion of Streptococcus pneumoniae to endothelial and epithelial cells[J]. Sci Rep, 2016, 6: 37758. DOI:10.1038/srep37758 |
| [16] |
CARNEIRO C R W, POSTOL E, NOMIZO R, et al. Identification of enolase as a laminin-binding protein on the surface of Staphylococcus aureus[J]. Microbes Infect, 2004, 6(6): 604-608. DOI:10.1016/j.micinf.2004.02.003 |
| [17] |
秦明星, 宫晓炜, 陈启伟, 等. 鸭疫里默氏杆菌RA-LZ01株GE296_RS01450基因介导其对重金属离子产生耐受性的分析[J]. 畜牧兽医学报, 2021, 52(10): 2864-2873. QIN M X, GONG X W, CHEN Q W, et al. Study on the GE296_RS01450 gene-mediated tolerance of Rimerella anatipestifer strain RA-LZ01 to heavy metal ions[J]. Acta Veterinaria et Zootechnica Sinica, 2021, 52(10): 2864-2873. DOI:10.11843/j.issn.0366-6964.2021.010.017 (in Chinese) |
| [18] |
权衡, 陈启伟, 宫晓炜, 等. 鸭疫里默氏杆菌MFS外排泵rant基因缺失株的构建及其介导的耐药性[J]. 畜牧兽医学报, 2021, 52(7): 1991-1999. QUAN H, CHEN Q W, GONG X W, et al. Construction of MFS efflux pump rant gene deletion mutant of Riemerella anatipestifer and its contribution to drug resistance[J]. Acta Veterinaria et Zootechnica Sinica, 2021, 52(7): 1991-1999. (in Chinese) |
| [19] |
薛亚飞, 宫艳杉, 祁晶晶, 等. 鸭疫里默氏杆菌对鸭胚肝细胞粘附入侵能力的研究[J]. 中国动物传染病学报, 2019, 27(1): 37-41. XUE Y F, GONG Y S, QI J J, et al. Adhesion and invasion of Riemerella anatipestifer to duck embryo liver cells[J]. Chinese Journal of Animal Infectious Diseases, 2019, 27(1): 37-41. (in Chinese) |
| [20] |
ZHAO W D, LIU D X, WEI J Y, et al. Caspr1 is a host receptor for meningitis-causing Escherichia coli[J]. Nat Commun, 2018, 9(1): 2296. DOI:10.1038/s41467-018-04637-3 |
| [21] |
TENG C H, CAI M, SHIN S, et al. Escherichia coli K1 RS218 interacts with human brain microvascular endothelial cells via type 1 fimbria bacteria in the fimbriated state[J]. Infect Immun, 2005, 73(5): 2923-2931. DOI:10.1128/IAI.73.5.2923-2931.2005 |
| [22] |
SAUNDERS N R, DREIFUSS J J, DZIEGIELEWSKA K M, et al. The rights and wrongs of blood-brain barrier permeability studies: A walk through 100 years of history[J]. Front Neurosci, 2014, 8: 404. |
| [23] |
PANDEY P K, SHARMA A K, GUPTA U. Blood brain barrier: An overview on strategies in drug delivery, realistic in vitro modeling and in vivo live tracking[J]. Tissue Barriers, 2016, 4(1): e1129476. DOI:10.1080/21688370.2015.1129476 |
| [24] |
KRUCINSKA J, FALCONE E, ERLANDSEN H, et al. Structural and functional studies of bacterial enolase, a potential target against Gram-negative pathogens[J]. Biochemistry, 2019, 58(9): 1188-1197. DOI:10.1021/acs.biochem.8b01298 |
| [25] |
ANTIKAINEN J, KUPARINEN V, LÄHTEENMÄKI K, et al. Enolases from Gram-positive bacterial pathogens and commensal Lactobacilli share functional similarity in virulence-associated traits[J]. FEMS Immunol Med Microbiol, 2007, 51(3): 526-534. DOI:10.1111/j.1574-695X.2007.00330.x |
| [26] |
LEE J Y, JUNG Y J, JUN H K, et al. Pathogenic potential of Tannerella forsythia enolase[J]. Mol Oral Microbiol, 2016, 31(2): 189-203. DOI:10.1111/omi.12115 |
| [27] |
WENG Y D, CHEN F, LIU Y W, et al. Pseudomonas aeruginosa enolase influences bacterial tolerance to oxidative stresses and virulence[J]. Front Microbiol, 2016, 7: 1999. |
(编辑 范子娟)



