绵羊在我国是一种重要的家畜,能够为人们提供羊肉、羊奶、羊毛和羊皮等优质的畜产品。随着生活水平的提高,羊肉消费量日渐增长,但我国现有绵羊存栏量和羊肉产量虽居前列,出栏量和个体产肉量却低于世界平均水平,目前很难满足国内的需求[1-2],这也使得人们对绵羊繁殖能力进行深入的研究与探索。卵巢是雌性动物重要的生殖器官,其主要功能是产生并排出能够完成受精的健康卵子,以及分泌性激素、黄体酮等类固醇激素以维持雌性动物性征和周期性繁殖活动,对动物繁殖具有关键性作用。
lncRNA是一种长度超过200 nt的非编码RNA分子(non-coding RNA,ncRNA),在多种生物过程中发挥着多种作用,是继mRNA和miRNA之后最热门的分子生物学研究领域之一[3-4]。研究发现,哺乳动物基因组约70%被转录,但实际上能编码蛋白质的只有约2%,其余被转录为非编码RNA,其中lncRNA被认为是动物中表达量最高的ncRNA之一[5],最初被人们当作基因组转录过程中形成的“噪音”,并不具有生物学功能[6]。但随后的研究逐渐表明lncRNA通过表观遗传调控、转录调控和转录后调控[7]等方面来调控基因表达,在细胞增殖分化[8]、信号转导[9]、免疫调节[10]等多种生物学过程中发挥着重要作用。在繁殖方面,关于lncRNA的报道很多,李耀坤等[11]在高、低繁殖力川中黑山羊卵巢组织中发现ENSCHIG00000001479等lncRNA可能对山羊生殖发育过程中的卵巢发育及排卵等进程具有重要的调控作用, 认为鉴定出的差异表达lncRNA与山羊产羔数有关。然后,Feng等[12]分别在高产和低产的湖羊卵巢中鉴定出5个lncRNA和76个mRNA。Su等[13]通过高通量测序筛选梅山猪和约克夏猪早期妊娠过程中差异表达lncRNA,得出XLOC-2222497及其靶标AKR1C1可以与猪子宫内膜中的孕酮相互作用以控制妊娠维持。Chen等[14]在具有不同FecB基因型的绵羊卵泡期和黄体期的下丘脑中鉴定到53个差异表达mRNAs和40个差异表达lncRNAs。这些研究证明了lncRNA在生殖组织中的存在和作用。
本研究利用高通量测序技术和生物信息分析方法对绵羊黄体期和卵泡期卵巢组织中的lncRNA进行测序,鉴定和预测不同时期差异表达基因lncRNA,通过GO和KEGG进行功能富集分析,筛选其中与繁殖相关的lncRNA及其靶基因参与的生物学过程,以期为探索lncRNA在绵羊繁育方面的作用提供理论基础。
1 材料与方法 1.1 试验动物和样品采集试验动物为河南省洛宁农本畜牧科技开发有限公司提供的绵羊,品种为湖羊。选取年龄在1.5~2.5岁,体重43 kg左右,体型均一,发育良好的母羊阴道放置阴道栓(60 mg甲羟孕酮),12 d撤掉阴道栓。在试情两次确认发情并撤栓后的24 h(卵泡期)和10 d(黄体期)屠宰采样,2个时期绵羊各3只。将屠宰后采集的绵羊卵巢组织放在1.2 mL无酶冻存管内,迅速放入液氮中保存并带回,以备提取lncRNA。
1.2 卵巢组织总RNA的抽提利用TRIzol法分别提取每个卵巢组织的总RNA,1.2%琼脂糖凝胶电泳测定RNA完整性及是否存在DNA污染,Nanodorp 2000检测RNA浓度。将测定合格的样品送至北京诺禾致源公司进行RNA-seq。
1.3 文库构建、测序及转录本拼装按照要求进行lncRNA文库构建,库检合格后使用Illumina上机测序。高通量测序平台Illumina测序得到的原始数据(Raw Reads)经过质控并去除低质量、有污染、含有接头等序列后的数据(Clean Reads)用作后续分析。使用HISAT2[15]对过滤后的reads进行参考基因组的比对分析。使用StringTie[16]对HISAT2比对的结果进行转录本的拼接,得到尽可能小的转录本集合,并对转录本进行定量分析。
1.4 差异表达lncRNA的筛选基于转录组拼接结果,使用Cuffmerge软件得到合并的转录本集合,根据lncRNA的结构特点以及不编码蛋白的功能特点,设置一系列严格的筛选条件,通过5步筛选:外显子数筛选、转录本长度筛选、转录本已知注释筛选、转录本表达量筛选、编码潜能筛选,取CPC2、CNCI、PFAM三种工具预测结果的交集,作为本次分析预测得到的lncRNA数据集进行后续的分析。使用StringTie-eB软件对包括mRNA、lncRNA以及TUCP在内的转录本进行定量分析,得到各样本转录本的FPKM信息。使用cuffdiff对不同类型转录本整体进行差异分析,筛选阈值设置为log2(fold change) ≥2且q-value < 0.05。
1.5 lncRNA靶向mRNA预测lncRNA主要通过调控mRNA来实现功能。本研究通过lncRNA与蛋白编码基因的位置关系(co-location)和表达相关性(co-expression)预测其生物学功能。将co-location的阈值设定为lncRNA上、下游100 kb,后续再通过对lncRNA共定位的mRNA基因进行功能富集分析预测lncRNA的主功能。采用Pearson相关系数法分析样本间lncRNA与mRNA的相关性,取相关性绝对值大于0.95的mRNA基因进行功能富集分析,预测与mRNA共表达的lncRNA的主要功能。
1.6 GO富集和KEGG通路分析Gene Ontology(简称GO,www.geneontology.org)是基因功能国际标准分类体系,使用GOseq[17]对差异lncRNA的靶基因进行GO富集分析。KEGG (Kyoto Encyclopedia of Genes and Genomes) 是有关生物学通路的主要公共数据库[18],使用KOBAS(2.0)[19]进行通路富集分析,FDR≤0.05为显著富集。
1.7 lncRNA的荧光定量PCR验证为验证测序结果的可靠性,随机选取5个差异表达的lncRNA进行qRT-PCR验证。使用Primer 5.0设计引物,GAPDH作为内参基因,引物均由北京擎科生物科技有限公司合成,引物序列信息如表 1所示。PCR反应体系(20 μL):10 μL lncRNA PreMix,上、下游引物各0.5 μL,1 μL cDNA和8 μL RNase-Free ddH2O。PCR反应条件:95 ℃ 3 min;95 ℃ 5 s,57 ℃ 10 s,72 ℃ 15 s,40个循环。使用2-ΔΔCt方法计算lncRNA的相对表达量,并与转录组结果进行对比验证。
|
|
表 1 lncRNA引物序列信息 Table 1 lncRNA primers sequence information |
对测序下机的Raw Reads进行质控过滤,如表 2所示,卵泡期湖羊卵巢(OAF)文库和黄体期湖羊卵巢(OAL)文库的Q20和Q30比例均在93%以上。对质控过滤后的reads进行参考基因组的比对分析,如表 3所示,测序数据与基因组比对率在87%以上,79%以上的序列在参考序列上有唯一比对位置。各项数据表明测序数据质量较好,满足后续分析的要求。
|
|
表 2 数据产出质量情况 Table 2 Statement of data output quality |
|
|
表 3 Reads与参考基因组比对情况 Table 3 Comparison of reads and reference genome |
对OAF和OAL的转录本进行分步筛选,鉴定得到候选lncRNAs转录本数目为21 085个,其中包括221个已知lncRNAs和20 864个新lncRNAs。进一步对候选lncRNA进行分类,如图 1A所示,基因间型lncRNAs占46.5%,反义型lncRNAs占8.6%,内含子型的lncRNAs占44.9%。

|
A. lncRNA类型分布; B. lncRNA与mRNA外显子数目;C. lncRNA与mRNA的转录本长度;D. lncRNA与mRNA ORF长度 A. Distribution of lncRNA types; B. lncRNA and mRNA exon number; C. lncRNA and mRNA transcripts length; D. lncRNA and mRNA ORF length 图 1 lncRNA类型分布及其与mRNA的结构特征比较分析 Fig. 1 Distribution of lncRNA types and structural characteristics comparison with mRNA |
将lncRNA与mRNA进行结构比较分析,得到lncRNA与mRNA的转录本长度、外显子个数、ORF长度(图 1D)的对比情况。结果发现,lncRNA和mRNA的长度整体趋势基本一致(图 1B);lncRNA的外显子个数多为2个,显著低于mRNA(图 1C),这与马骏杰等[20]的研究结果一致;mRNA的ORF长度明显大于lncRNA,这提示lncRNA在转录及转录后等表观调控方面起着重要作用[21]。
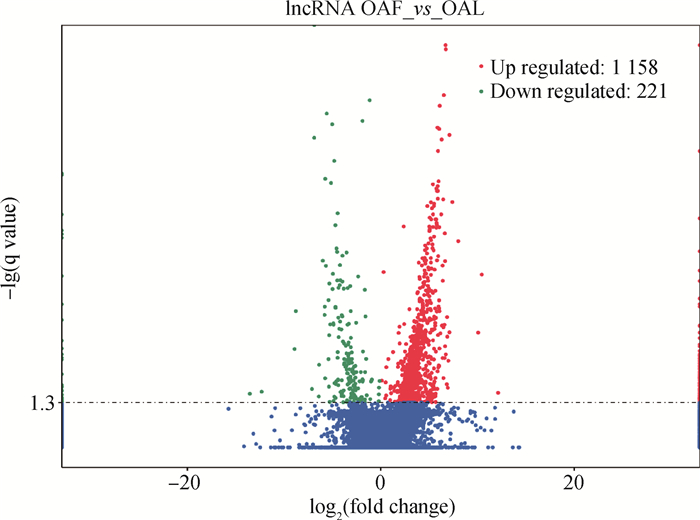
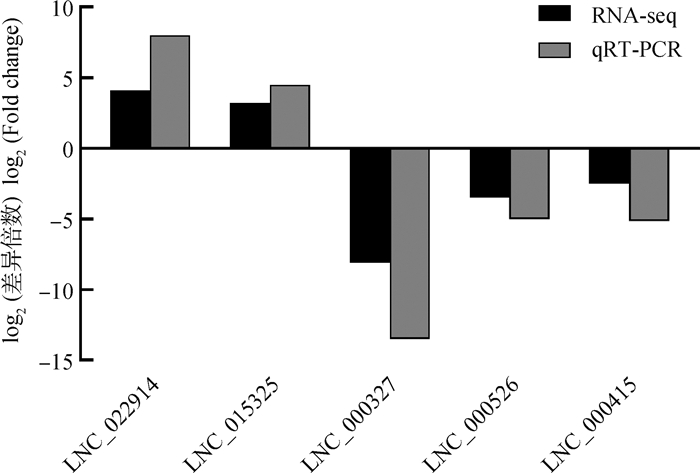
2.3 lncRNAs的差异表达分析及验证利用cuffdiff进行差异分析得到初步差异分析的结果。设置筛选差异基因的标准为log2(fold change) ≥2且q-value < 0.05,得到的差异基因结果如图 2所示,在OAF和OAL中获得1 379个差异表达的lncRNAs,其中1 158个表达上调,221个表达下调。随机挑选5个差异表达lncRNAs进行qRT-PCR,验证RNA测序数据的可靠性。验证结果与测序数据结果基本一致(图 3),表明测序数据结果可靠性高。
|
图 2 差异表达lncRNAs的火山图 Fig. 2 Volcano plot of differentially expressed lncRNAs |
|
图 3 差异表达lncRNAs的qRT-PCR验证 Fig. 3 Validation of the differentially expressed lncRNAs by qRT-PCR |
对1 379个差异表达的lncRNA的共定位靶基因进行生物功能分析,共显著富集到3个GO条目(P < 0.05),包括1个分子功能(molecular function,MF),2个生物学过程(biological process,BP),没有富集到细胞组分(cellular component,CC)。显著富集到的GO条目如表 4所示,在分子功能归类中,差异基因显著富集到谷胱甘肽转移酶活性(glutathione transferase activity)。在生物学过程中,差异基因显著富集到病毒过程的负调控(negative regulation of viral process)、病毒基因组复制的负调控(negative regulation of viral genome replication)。
|
|
表 4 lncRNA的共定位基因前3个GO条目 Table 4 The top 3 GO terms of lncRNA co-location genes |
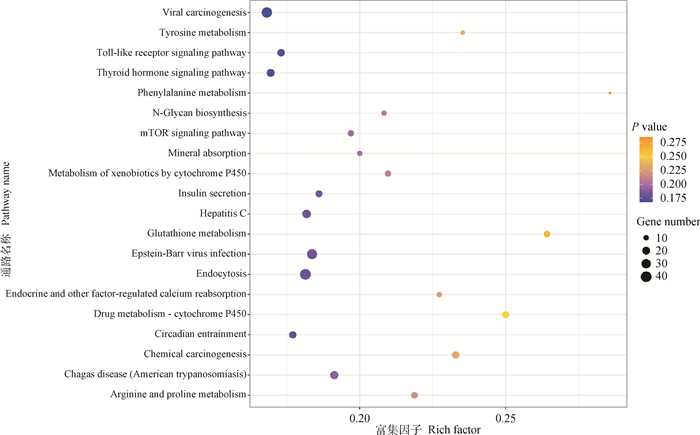
在KEGG富集分析中,差异表达lncRNA的共定位靶基因涉及265条信号通路,其中共显著富集到10条通路(P < 0.05),包括胞吞作用(endocytosis)、谷胱甘肽代谢(glutathione metabolism)、药物代谢-细胞色素P450(drug metabolism-cytochrome P450)、精氨酸和脯氨酸代谢(arginine and proline metabolism)、细胞色素P450代谢外源物质(metabolism of xenobiotics by cytochrome P450)等。P值前20的通路见图 4。
|
图 4 差异表达lncRNA共定位基因的KEGG分析 Fig. 4 KEGG analysis of differentially expressed lncRNA co-location genes |
对1 379个差异表达lncRNAs的共表达靶基因进行生物功能分析,共显著富集到482个GO条目(P < 0.05),包括63个分子功能,378个生物学过程,41个细胞组分。前10条显著富集到的GO条目如表 5所示,在分子功能归类中,差异基因显著富集到结合(binding)、蛋白结合(protein binding)等。在生物学过程中,差异基因显著富集到卵泡发育(ovarian follicle development)、排卵周期过程(ovulation cycle process)等。在细胞组分中,差异基因显著富集到质膜(plasma membrane part)、神经元投射(neuron projection)等。
|
|
表 5 lncRNA的共表达基因前10个GO条目 Table 5 The top 10 GO terms of lncRNA co-expression genes |
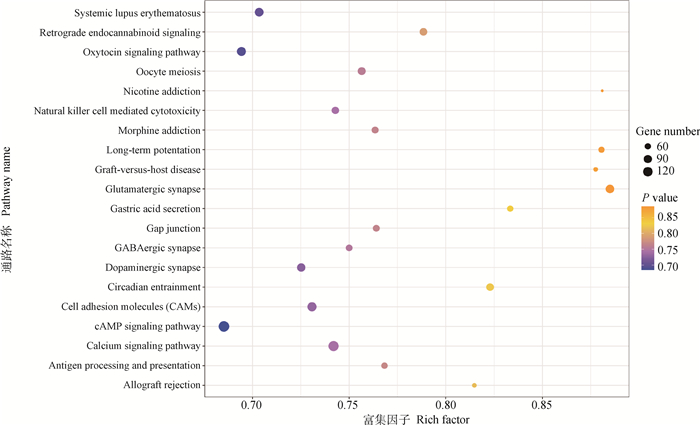
在KEGG富集分析中,差异表达lncRNA的共表达靶基因涉及275条信号通路,其中共显著富集到28条通路(P < 0.05),包括钙离子信号通路(calcium signaling pathway)、卵母细胞减数分裂(oocyte meiosis)、cAMP信号通路(cAMP signaling pathway)、催产素信号通路(oxytocin signaling pathway)、MAPK信号通路(MAPK signaling pathway)、甲状腺激素合成通路(thyroid hormone synthesis)、雌激素信号通路(estrogen signaling pathway)等。P值前20的通路见图 5。
|
图 5 差异表达lncRNA共表达基因的KEGG分析 Fig. 5 KEGG analysis of differentially expressed lncRNA co-expression genes |
本研究KEGG富集分析得到了可能与繁殖有关的通路基因(表 6)。这些通路包括钙离子信号通路、卵母细胞减数分裂、cAMP信号通路、催产素信号通路、MAPK信号通路、甲状腺激素合成通路、雌激素信号通路。
|
|
表 6 繁殖相关的候选信号通路及基因 Table 6 Signaling pathways and genes related to reproduction |
通过筛选KGEE富集到信号通路,筛选出5个显著差异基因,与多个不同lncRNA存在靶标关系(表 7)。其中多个lncRNAs靶向同一个差异基因,如LNC_003902、LNC_003906、LNC_003907均靶向PPP3CA等;也存在一个lncRNA同时具有多个靶基因,如ADCY5、ADCY1、CDC25A等都是LNC_ 012847的潜在目标,这些lncRNAs呈现出了多种方向的调控作用。
|
|
表 7 差异基因与lncRNAs的靶标关系 Table 7 Target relationship between differential genes and lncRNAs |
繁殖能力对绵羊的经济效益有重要影响。lncRNA因其在基因调控网络和广泛的生物学过程中的作用而受到广泛关注。研究发现,lncRNA涉及多种动物繁殖相关的过程,如妊娠[22]、性腺激素应答[23]、卵子成熟[24]和胎盘形成[25]等。本研究对绵羊不同发情周期卵巢转录组分析,选择不同发情周期的卵巢筛选出的差异基因显著富集到的通路及通路中的靶基因进行有关繁殖调控的讨论。
本研究在KEGG富集分析得到的多个与繁殖调控相关的候选通路中选择卵母细胞减数分裂、MAPK、甲状腺激素合成、雌激素信号通路,并对通路及其中的靶基因进行繁殖相关调控的讨论。
卵母细胞减数分裂被认为是形成单倍体配子所需的特殊细胞分裂形式,卵母细胞减数分裂的重新启动在卵泡期中也起到关键作用[26]。卵母细胞减数分裂期间染色体分离的精确调节对哺乳动物的繁殖至关重要。PPP3CA(protein phosphatase 3 catalytic subunit alpha)是蛋白磷酸酶3的催化亚基A,广泛存在于真核生物的多种细胞中[27]。研究发现,PPP3CA与山羊繁殖力性状有关[28],基因内缺失突变显著影响山羊产仔数[29]。在卵泡期显著上调的LNC_003902、LNC_003906、LNC_003907靶向的PPP3CA在此通路中显著富集。推测上述lncRNA可能通过调控PPP3CA影响绵羊产羔数。CDC23(cell division cycle 23)是小鼠卵母细胞减数分裂期间纺锤体组装和染色体分离的关键调节因子[30],这表明CDC23在减数分裂卵母细胞中具有独特的作用。LNC_020278、LNC_020280、LNC_020281、LNC_020279靶向的CDC23在此通路中显著富集。根据已知CDC23功能的研究,推测上述lncRNA可能在卵母颗粒细胞减数分裂中有重要作用。Liu等[31]在高、低繁殖力寒泊羊筛选的卵泡期差异表达lncRNA也显著富集到此通路,与本研究结果相似。
MAPK信号通路是是真核生物信号传递网络中的重要途径之一,MAPK被一系列细胞外的刺激信号激活并介导信号从细胞膜向细胞核传导,是调节细胞增殖、分化、凋亡和死亡等多种生理过程的关键信号通路[32-33]。MAPK信号通路被催乳素抑制,该通路的功能对正常卵泡发育至关重要[34]。MAPK1(mitogen-activated protein kinase 1)是MAP激酶家族的成员,在颗粒细胞中的磷酸化由LH受体激活引起并介导卵母细胞成熟[35-36]。本研究发现,在黄体期显著上调的LNC_011239靶向的MAPK1在此通路显著富集,根据靶基因的功能分析,推测LNC_011239可能在黄体期通过调控MAPK1影响卵母细胞成熟。
甲状腺激素合成通路已被确定对脊椎动物胚胎发生和胎儿成熟至关重要。此外,甲状腺素(T4)和三碘甲状腺原氨酸(T3)对正常发育、生长和代谢稳态至关重要[37]。有研究报道,大鼠在怀孕期间甲状腺激素缺乏,导致产仔数减少[38]。雌激素是由卵巢和胎盘分泌的一种类固醇类激素,在动物发情、分娩等生殖功能方面有重要作用[39],雌激素信号通路是指雌激素与细胞核或细胞膜中的雌激素受体(ER)结合后,作用于雌激素受体反应元件调节基因表达。当雌激素与受体结合后,可以维持雌性个体第二性征,可以刺激和维持黄体功能,在雌性动物繁殖周期中对卵泡的生长发育起重要作用。ADCY1(adenylate cyclase 1)和ADCY5(adenylate cyclase 5)是腺苷酸环化酶家族的成员。ADCY1和ADCY5被发现是影响山羊和奶牛繁殖力的候选基因[40-41]。在卵泡期显著上调的LNC_003902、LNC_003906、LNC_003907、LNC_012843、LNC_012847、LNC_021474、LNC_021475和LNC_012847靶向的ADCY1和ADCY5在这两条信号通路均有显著富集,提示LNC_012847可能与绵羊繁殖力存在一定相关性。
4 结论本研究采用高通量测序技术和生物信息学工具,对发情周期不同阶段绵羊卵巢的lncRNAs进行了鉴定,在OAF和OAL中获得1 379个差异表达的lncRNAs,其中1 158个表达上调,221个表达下调。共表达和共定位的GO注释和KEGG富集分析差异lncRNAs的靶基因富集到多个繁殖相关信号通路中,其中LNC_011239、LNC_012847、LNC_003902、LNC_003906、LNC_003907等靶向的MAPK1、ADCY1、ADCY5、PPP3CA和CDC23可能发挥关键的调控作用。此外,这些差异lncRNAs表达谱为揭示绵羊繁殖能力的分子机制提供了有价值的资源。绵羊繁殖相关的候选lncRNAs及鉴定的关键靶基因可能需要通过敲除、过表达等方法进一步研究,以验证lncRNA在绵羊卵巢中的特异功能。
| [1] |
甘春艳, 李军, 金海. 2021年我国肉羊产业发展概况、未来发展趋势及建议[J]. 中国畜牧杂志, 2022, 58(3): 258-263. GAN C Y, LI J, JIN H. Development situation, future development trend and suggestions of meat sheep industry in China in 2021[J]. Chinese Journal of Animal Science, 2022, 58(3): 258-263. DOI:10.19556/j.0258-7033.20220209-08 (in Chinese) |
| [2] |
周书杰, 张应利, 张传辉. 全国肉羊养殖成本与收益分析[J]. 合作经济与科技, 2022(3): 100-101. ZHOU S J, ZHANG Y L, ZHANG C H. Cost and benefit analysis of mutton sheep breeding in China[J]. Co-Operative Economy & Science, 2022(3): 100-101. (in Chinese) |
| [3] |
LAURENT G S, WAHLESTEDT C, KAPRANOV P. The Landscape of long noncoding RNA classification[J]. Trends Genet, 2015, 31(5): 239-251. DOI:10.1016/j.tig.2015.03.007 |
| [4] |
TUCK A C, TOLLERVEY D. A transcriptome-wide atlas of RNP composition reveals diverse classes of mRNAs and lncRNAs[J]. Cell, 2013, 154(5): 996-1009. DOI:10.1016/j.cell.2013.07.047 |
| [5] |
DERRIEN T, JOHNSON R, BUSSOTTI G, et al. The GENCODE v7 catalog of human long noncoding RNAs: analysis of their gene structure, evolution, and expression[J]. Genome Res, 2012, 22(9): 1775-1789. DOI:10.1101/gr.132159.111 |
| [6] |
KNOWLING S, MORRIS K V. Non-coding RNA and antisense RNA.Nature's trash or treasure?[J]. Biochimie, 2011, 93(11): 1922-1927. DOI:10.1016/j.biochi.2011.07.031 |
| [7] |
HOMBACH S, KRETZ M. Non-coding RNAs: classification, biology and functioning[J]. Adv Exp Med Biol, 2016, 937: 3-17. |
| [8] |
STATELLO L, GUO C J, CHEN L L, et al. Gene regulation by long non-coding RNAs and its biological functions[J]. Nat Rev Mol Cell Biol, 2021, 22(2): 96-118. DOI:10.1038/s41580-020-00315-9 |
| [9] |
MATTICK J S. Long noncoding RNAs in cell and developmental biology[J]. Semin Cell Dev Biol, 2011, 22(4): 327. DOI:10.1016/j.semcdb.2011.05.002 |
| [10] |
CHEN Y G, SATPATHY A T, CHANG H Y. Gene regulation in the immune system by long noncoding RNAs[J]. Nat Immunol, 2017, 18(9): 962-972. DOI:10.1038/ni.3771 |
| [11] |
李耀坤, 许香萍, 陆婷婷, 等. 影响山羊产羔数lncRNA的筛选及功能分析[J]. 畜牧兽医学报, 2020, 51(8): 1853-1865. LI Y K, XU X P, LU T T, et al. Screening and functional analysis of lncRNA affecting litter size in goats[J]. Acta Veterinaria et Zootechnica Sinica, 2020, 51(8): 1853-1865. (in Chinese) |
| [12] |
FENG X, LI F Z, WANG F, et al. Genome-wide differential expression profiling of mRNAs and lncRNAs associated with prolificacy in Hu sheep[J]. Biosci Rep, 2018, 38(2): BSR20171350. DOI:10.1042/BSR20171350 |
| [13] |
SU T, YU H L, LUO G, et al. The interaction of lncRNA XLOC-2222497, AKR1C1, and progesterone in porcine endometrium and pregnancy[J]. Int J Mol Sci, 2020, 21(9): 3232. DOI:10.3390/ijms21093232 |
| [14] |
CHEN S, GUO X F, HE X Y, et al. Transcriptome analysis reveals differentially expressed genes and long non-coding RNAs associated with fecundity in sheep hypothalamus with different FecB genotypes[J]. Front Cell Dev Biol, 2021, 9: 633747. DOI:10.3389/fcell.2021.633747 |
| [15] |
PERTEA M, KIM D, PERTEA G M, et al. Transcript-level expression analysis of RNA-seq experiments with HISAT, stringtie and ballgown[J]. Nat Protoc, 2016, 11(9): 1650-1667. DOI:10.1038/nprot.2016.095 |
| [16] |
PERTEA M, PERTEA G M, ANTONESCU C M, et al. StringTie enables improved reconstruction of a transcriptome from RNA-seq reads[J]. Nat Biotechnol, 2015, 33(3): 290-295. DOI:10.1038/nbt.3122 |
| [17] |
YOUNG M D, WAKEFIELD M J, SMYTH G K, et al. Gene ontology analysis for RNA-seq: accounting for selection bias[J]. Genome Biol, 2010, 11(2): R14. DOI:10.1186/gb-2010-11-2-r14 |
| [18] |
KANEHISA M, ARAKI M, GOTO S, et al. KEGG for linking genomes to life and the environment[J]. Nucleic Acids Res, 2008, 36: D480-D484. |
| [19] |
MAO X Z, CAI T, OLYARCHUK J G, et al. Automated genome annotation and pathway identification using the KEGG Orthology (KO) as a controlled vocabulary[J]. Bioinformatics, 2005, 21(19): 3787-3793. DOI:10.1093/bioinformatics/bti430 |
| [20] |
马骏杰, 汪亮, 刘娣, 等. 冷应激后民猪背脂中差异lncRNA的筛选与分析[J]. 畜牧兽医学报, 2021, 52(4): 932-942. MA J J, WANG L, LIU D, et al. Screening and analysis of differentially expressed lncRNA in Min pig back fat after cold stress[J]. Acta Veterinaria et Zootechnica Sinica, 2021, 52(4): 932-942. (in Chinese) |
| [21] |
王瑞雪, 刘佳森, 李蕴华, 等. 苏尼特羊不同生长时期肌肉组织lncRNA的差异表达分析[J]. 中国农业大学学报, 2021, 26(1): 51-61. WANG R X, LIU J S, LI Y H, et al. Differential expression analysis of lncRNA in muscle tissue of Sunite sheep at different growth stages[J]. Journal of China Agricultural University, 2021, 26(1): 51-61. (in Chinese) |
| [22] |
NAKAGAWA S, SHIMADA M, YANAKA K, et al. The lncRNA Neat1 is required for corpus luteum formation and the establishment of pregnancy in a subpopulation of mice[J]. Development, 2014, 141(23): 4618-4627. DOI:10.1242/dev.110544 |
| [23] |
LI W B, NOTANI D, MA Q, et al. Functional roles of enhancer RNAs for oestrogen-dependent transcriptional activation[J]. Nature, 2013, 498(7455): 516-520. DOI:10.1038/nature12210 |
| [24] |
LI J, CAO Y X, XU X F, et al. Increased new lncRNA-mRNA gene pair levels in human cumulus cells correlate with oocyte maturation and embryo development[J]. Reprod Sci, 2015, 22(8): 1008-1014. DOI:10.1177/1933719115570911 |
| [25] |
GAO W L, LIU M, YANG Y, et al. The imprinted H19 gene regulates human placental trophoblast cell proliferation via encoding miR-675 that targets Nodal Modulator 1 (NOMO1)[J]. RNA Biol, 2012, 9(7): 1002-1010. DOI:10.4161/rna.20807 |
| [26] |
JAFFE L A, EGBERT J R. Regulation of mammalian oocyte meiosis by intercellular communication within the ovarian follicle[J]. Annu Rev Physiol, 2017, 79: 237-260. DOI:10.1146/annurev-physiol-022516-034102 |
| [27] |
RUSNAK F, MERTZ P. Calcineurin: form and function[J]. Physiol Rev, 2000, 80(4): 1483-1521. DOI:10.1152/physrev.2000.80.4.1483 |
| [28] |
ISLAM R, LIU X X, GEBRESELASSIE G, et al. Genome-wide association analysis reveals the genetic locus for high reproduction trait in Chinese Arbas Cashmere goat[J]. Genes Genomics, 2020, 42(8): 893-899. DOI:10.1007/s13258-020-00937-5 |
| [29] |
BAI Y Y, LI J, ZHU H J, et al. Deletion mutation within the goat PPP3CA gene identified by GWAS significantly affects litter size[J]. Reprod Fertil Dev, 2021, 33(7): 476-483. DOI:10.1071/RD20337 |
| [30] |
ZHOU Q, LI J, YUE W, et al. Cell division cycle 23 is required for mouse oocyte meiotic maturation[J]. FASEB J, 2020, 34(7): 8990-9002. DOI:10.1096/fj.202000131R |
| [31] |
LIU A J, LIU M H, LI Y X, et al. Differential expression and prediction of function of lncRNAs in the ovaries of low and high fecundity Hanper sheep[J]. Reprod Domest Anim, 2021, 56(4): 604-620. DOI:10.1111/rda.13898 |
| [32] |
KANG H M, JEONG C B, LEE Y H, et al. Cross-reactivities of mammalian MAPKs antibodies in rotifer and copepod: Application in mechanistic studies in aquatic ecotoxicology[J]. Mar Pollut Bull, 2017, 124(2): 614-623. DOI:10.1016/j.marpolbul.2016.11.049 |
| [33] |
刘永刚, 李文哲, 苏建民. 哺乳动物卵母细胞成熟相关信号的调控机制研究进展[J]. 动物医学进展, 2019, 40(9): 93-97. LIU Y G, LI W Z, SU J M. Progress on regulation mechanisms of mammalian oocyte maturation signals[J]. Progress in Veterinary Medicine, 2019, 40(9): 93-97. DOI:10.3969/j.issn.1007-5038.2019.09.019 (in Chinese) |
| [34] |
DEVI Y S, SEIBOLD A M, SHEHU A, et al. Inhibition of MAPK by prolactin signaling through the short form of its receptor in the ovary and decidua: involvement of a novel phosphatase[J]. J Biol Chem, 2011, 286(9): 7609-7618. DOI:10.1074/jbc.M110.166603 |
| [35] |
SALVADOR L M, MAIZELS E, HALES D B, et al. Acute signaling by the LH receptor is independent of protein kinase C activation[J]. Endocrinology, 2002, 143(8): 2986-2994. DOI:10.1210/endo.143.8.8976 |
| [36] |
SELA-ABRAMOVICH S, CHOREV E, GALIANI D, et al. Mitogen-activated protein kinase mediates luteinizing hormone-induced breakdown of communication and oocyte maturation in rat ovarian follicles[J]. Endocrinology, 2005, 146(3): 1236-1244. DOI:10.1210/en.2004-1006 |
| [37] |
YOSHIMURA T, YASUO S, WATANABE M, et al. Light-induced hormone conversion of T4 to T3 regulates photoperiodic response of gonads in birds[J]. Nature, 2003, 426(6963): 178-181. DOI:10.1038/nature02117 |
| [38] |
IKEGAMI K, YOSHIMURA T. The hypothalamic-pituitary-thyroid axis and biological rhythms: The discovery of TSH's unexpected role using animal models[J]. Best Pract Res Clin Endocrinol Metab, 2017, 31(5): 475-485. DOI:10.1016/j.beem.2017.09.002 |
| [39] |
任巧玲, 马强, 白红杰, 等. 雌激素及其对动物生殖功能的影响[J]. 河南农业科学, 2006(4): 114-115. REN Q L, MA Q, BAI H J, et al. Estrogen and its effect on animal reproductive function[J]. Journal of Henan Agricultural Sciences, 2006(4): 114-115. DOI:10.3969/j.issn.1004-3268.2006.04.038 (in Chinese) |
| [40] |
CAI Z X, GULDBRANDTSEN B, LUND M S, et al. Prioritizing candidate genes for fertility in dairy cows using gene-based analysis, functional annotation and differential gene expression[J]. BMC Genomics, 2019, 20(1): 255. DOI:10.1186/s12864-019-5638-9 |
| [41] |
ISLAM R, LI Y F, LIU X X, et al. Genome-wide runs of homozygosity, effective population size, and detection of positive selection signatures in six chinese goat breeds[J]. Genes (Basel), 2019, 10(11): 938. DOI:10.3390/genes10110938 |
(编辑 郭云雁)



