2. 湘西州畜牧水产事务中心, 吉首 416000
2. Animal Husbandry and Aquatic Products Affairs Center of Xiangxi Autonomous Prefecture, Jishou 416000, China
五指山猪是一种海南省特有的小型猪,具有耐粗饲、瘦肉率高、肉质好、抗逆性强、遗传性能稳定等特征[1]。由于早期饲养方式以放养为主,五指山猪采食草茎、野果、甘薯、玉米、木薯等作物,形成了其耐粗饲的消化能力,同时,长期的户外活动塑造了五指山猪体格强健、野性强、善于跳跃的习性。此外,长期野外放养的方式也增强了五指山猪的抗逆性,使这个群体能够适应野外复杂的生态环境,抵御各种生物和非生物胁迫造成的危害。在不断的繁衍过程中,五指山猪保留并强化了适应不利生存环境的能力。
目前,五指山猪的研究内容大部分是关于表型测定,少数基因的检测和基因表达分析。例如,通过对比饲喂全价饲料和牧草混合饲料,发现后者提高了料重比,但是降低了日增重[2]。五指山猪和长白猪的背最长肌的蛋白组测序分析表明,两个品种调控骨骼肌分化过程的PI3K/AKT信号通路和影响脂肪沉积的PPAR信号通路有显著差异[3]。五指山猪背最长肌和半腱肌、背部和腹部皮下脂肪组织间的miRNA的表达有显著差异[4]。此外,由于五指山猪和人类在解剖学、生理结构、遗传学等方面具有高度相似性,是一种理想的人类疾病转化医学研究动物模型[5-6]。通过比较链唑霉素处理和正常饲喂的五指山猪,发现链唑霉素处理的五指山猪具有糖尿病类似的症状,例如血糖升高、眼睛出现白内障并发症、肾小球扩展等,说明五指山猪可以用来模拟人类糖尿病的病理特征[7]。
选择性清除是自然种群基因组中频繁且多样的特征,因此,检测一个群体基因组受选择的程度对于理解自然选择的适应机制很重要。种群发生基因突变后,经过自然选择的作用,被选择位点周围的单倍型多样性降低,并且这种单倍型与背景变异模式的偏差是可以检测到的[8-9]。韩国济州本地猪、巴克夏、约克夏猪的选择信号分析表明,韩国济州本地猪的遗传多态性显著低于另外两个品种,而且选择信号较强的基因组区域的功能主要集中在上皮细胞发育和神经元刺激通路[10]。通过分析10个不同地理位置藏猪的选择信号,发现肺组织发育,体型、毛色和嗅觉相关的基因是受到强烈选择的[11]。
然而,五指山猪抗逆性相关基因的进化特征尚不清楚。本研究通过比较30头五指山猪和29头杜洛克猪的全基因组重测序数据,提取SNPs,注释对应的基因结构和功能。采用选择性清除的方法,筛选五指山猪受到强烈选择的基因组区域,进行功能富集,分析该区域SNPs相关基因的作用和特征。
1 材料与方法 1.1 样本采集和数据搜集海南国家级五指山猪保种场是国内唯一的五指山猪原产地活体保种场,繁育各种类型、不同血缘、不同品系的五指山猪,保持种群的遗传多样性,从该场采集30头4月龄健康(10公,20母)五指山猪的耳组织,速冻,置于-20 ℃冰箱,用于提取DNA。从NCBI数据库下载29头杜洛克猪全基因组重测序数据(SRA:PRJNA378496)。
1.2 建库测序和数据过滤提取DNA和建库测序委托北京诺禾致源科技股份有限公司完成,建库采用PE150方法,测序平台是Illumina NovaSeq,平均每个样本测序10×。采用trimmomatic软件去掉原始数据adaptor和低质量序列[12],过滤后获得高质量序列,然后比对到猪参考基因组序列(Sscrofa 11.1),获得sam文件,进一步用samtools转化成bam文件。
杜洛克猪的重测序数据从NCBI下载(PRJNA378496),这批数据共29头猪耳组织样本,采自江苏省盐城市,测序平台是Illumina HiSeq 2000,下载的SRA文件首先用fastq-dump转化成fastq格式,再用trimmomatic软件过滤、比对,方法与五指山猪类似。
1.3 基因组选择区域分析bam文件进一步用samtools排序,picard软件去重(MarkDuplicates),再用gatk-4.2.2.0提取突变位点(HaplotypeCaller -ERC GVCF),每个样本获得一个gvcf文件,然后合并成一个群体gvcf文件。提取SNPs后过滤,参数指标包括:测序深度≥10×、Fisher检验值=0、比对率≥90%等。
进一步对过滤的SNPs位点采用snpEff注释结构和功能,统计五指山猪和杜洛克猪的SNPs位点在基因组上的分布特征,包括具体位点、区域(如promoter、exon、intron),然后用XP-CLR (the cross-population composite likelihood ratio test) 按照50 000 bp的窗口距离,计算染色体不同区域的选择程度[13]。结合SNPs注释结果,对选择分数高的区域进行GSEA功能富集,并分析富集通路基因的特征。可视化结果均用R包作图。
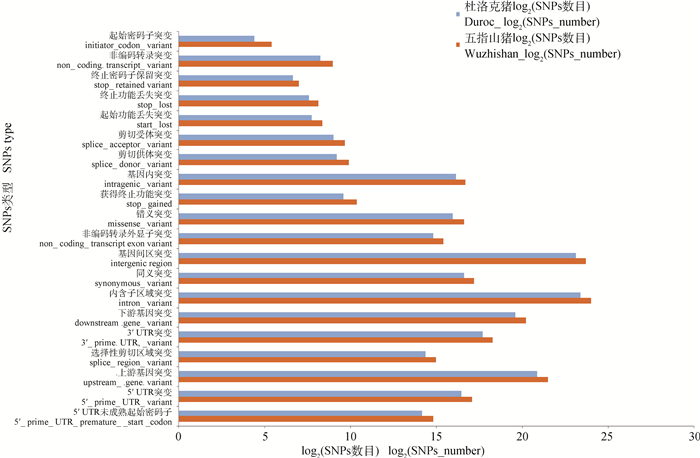
2 结果 2.1 突变位点的基因组结构特征平均每头五指山猪全基因组重测序的测序量为26G,软件trimmomatic去掉adaptor和低质量序列,过滤之后的数据约占原始数据的95%。过滤的数据比对到猪的参考基因组序列(Sscrofa 11.1),比对率约96%。五指山猪筛选到36 961 902个SNPs,内含子区域包含16 729 364个SNPs,基因上游(upstream_gene_variant)包含2 952 410个SNPs,5′端包含167 953个SNPs、基因下游(downstream_gene_variant)包含1 209 238个SNPs,3′端包含319 451个SNPs,编码区(stop_gained、start_lost、stop_lost、stop_retained_variant、initiator_codon_variant)包含2 073个SNPs,选择性剪接包含34 144个SNPs,错义突变包含99 638个SNPs(图 1)。杜洛克猪筛选到24 703 082个SNPs,内含子区域包含11 149 216个SNPs,基因上游(upstream_gene_variant)包含1 941 445个SNPs,5′端包含108 435个SNPs,基因下游(downstream_gene_ variant)包含799 363个SNPs,3′端包含211 625个SNPs,编码区包含1 292个SNPs,选择性剪接包含22 282个SNPs,错义突变包含62 995个SNPs(图 1)。由此可以看出,五指山猪的SNPs位点总数、调控区数目、编码区数目、选择性剪接区域、错义突变的数目均多于杜洛克猪。
|
蓝色和橙色柱形图分别表示杜洛克猪和五指山猪SNPs在染色体不同区域的分布。纵轴表示突变在染色体上的区域,横轴表示对染色体各个区域的SNPs数目取以2为底数的对数值 Blue and orange present the SNPs distribution of Duroc pig and Wuzhishan pig on different chromosomes, respectively. Vertical axis indicate the different chromosome regions, horizontal axis indicate log2(SNPs_number) 图 1 SNPs在染色体不同区域的分布 Fig. 1 The distribution of SNPs on different chromosomes |
猪包含18对常染色体,2条性染色体(X和Y)。第13条染色体SNPs数量最多,共有2 478 299个,其次是第6号染色体,有2 448 830个,Y染色体SNPs数量最少,仅有103 839个。五指山猪SNPs在基因组区域上分布主要集中在染色体两端,最密集的区域密度达到36 820个SNPs/Mb,最稀疏的区域密度只有1个SNPs/Mb,染色体中间区域范围的SNPs分布相对较少(图 2)。

|
绿色色块表示低密度SNPs区域,红色色块表示高密度SNPs区域 The green blocks mean low density SNPs region, and the red blocks mean high density SNPs region 图 2 五指山猪SNPs密度分布 Fig. 2 The SNPs density distribution of Wuzhishan pig |
根据五指山猪和杜洛克猪SNPs等位基因频率差异,采用XP-CLR计算五指山猪基因组不同区域的选择分数。除了Y染色体没有分值高于10的SNPs,18条常染色体和X染色体XP-CLR分值高于10的SNPs共有3 296 927个,分值高于60的SNPs共有44 970个,分值最高(125.252)的SNPs位点共1 102个,位于第4号染色体。第13号染色体XP-CLR分值≥60的SNPs共有10 991个,多于其它染色体(图 3),说明这段区域在进化过程中受到强烈选择,这段区域的基因功能对五指山猪适应生存环境具有重要作用。

|
横坐标轴表示18条常染色体和X染色体,纵坐标轴表示XP-CLR分数 The horizontal axis mean different chromosomes, and the vertical axis mean XP-CLR score 图 3 XP-CLR分值大于10的SNPs Fig. 3 The SNPs with XP-CLR score more than 10 |
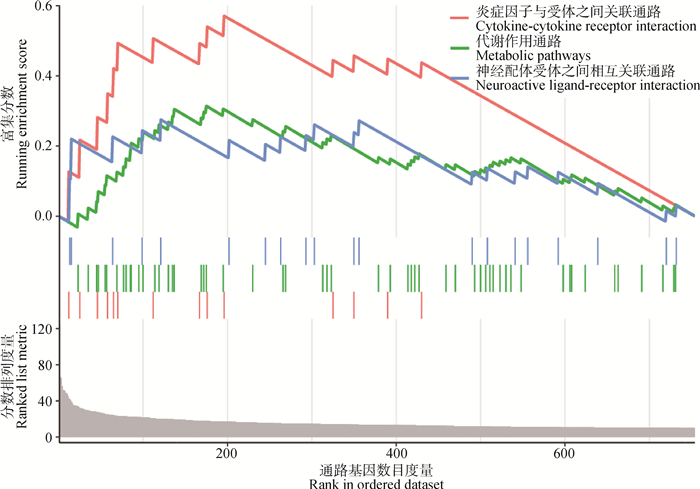
根据snpEff注释结果,提取XP-CLR分值大于10且属于基因功能区的SNPs位点对应的基因,并进行GSEA功能富集。富集的通路主要集中在3个方面:免疫应答、代谢反应、神经传导。其中免疫应答功能的炎症因子分泌通路上调,而且炎症反应通路(cytokine-cytokine receptor interaction,富集分数=0.57,P.adjust=0.01)上调的程度大于另外两条通路(metabolic pathways, 富集分数=0.31,P.adjust=0.03;neuroactive ligand-receptor interaction,富集分数=0.27,P.adjust=0.04)(图 4)。炎症因子通路共含有9个基因,分别是非典型趋化因子受体4(atypical chemokine receptor 4,ACKR4)、骨形态发生蛋白II型受体(bone morphogenetic protein receptor type 2,BMPR2)、白介素15(interleukin15,IL15)、白介素26(interleukin26,IL26)、转化生长因子-β受体2(transforming growth factor beta receptor 2,TGFBR2)、肿瘤坏死因子受体超家族11b(TNF receptor superfamily member 11b,TNFRSF11B)、肿瘤坏死因子超家族15(TNF superfamily member 15,TNFSF15)、肿瘤坏死因子超家族4(TNF superfamily member 4,TNFSF4)、肿瘤坏死因子超家族8(TNF superfamily member 8,TNFSF8)。脂类代谢筛选到3个关键基因,分别是胰岛素受体底物2(insulin receptor substrate 2,IRS2)、cGMP依赖蛋白激酶1(protein kinase cGMP-dependent 1,PRKG1)、腺苷酸环化酶5 (adenylate cyclase 5,ADCY5)。
|
图中红色线条表示炎症因子受体通路(炎症因子与受体之间关联通路),绿色线条表示代谢通路(代谢作用通路),蓝色线条表示神经组织受体通路(神经配体受体之间相互关联通路) The red line mean inflammatory factor pathway (cytokine-cytokine receptor interaction), the green line mean metabolic pathway (metabolic pathways), and the blue line mean nervous pathway (neuroactive ligand-receptor interaction) 图 4 XP-CLR分值大于10的SNPs位点注释的GSEA功能富集 Fig. 4 The GSEA enrichment of SNPs with XP-CLR score more than 10 |
免疫相关的9个基因的SNPs均在基因功能区域,其中基因ACKR4、BMPR2突变位点位于启动子区,基因TNFSF4突变位点位于5′端,基因TNFRSF11B、TNFSF15突变位点位于3′端(表 1,图 5A)。更重要的是,五指山猪基因TNFSF15与TNFSF8连锁不平衡程度r2>0.8,基因TNFSF15位点255341885与基因TNFSF8位点255455487几乎处于完全连锁状态(r2=1)(图 5B),因此基因TNFSF15和TNFSF8很可能发生连锁遗传。脂类代谢基因IRS2、ADCY5的突变位点分别位于启动子区和3′端,可能发挥调控基因表达的作用。9个炎症因子基因位点(ACKR4:A73775544G,BMPR2:A106043441C,IL15:C85672453T,IL26:C32573611T,TGFBR2:C16817427T,TNFRSF11B:T19878575A,TNFSF15:C255349468T,TNFSF4:C115548060T,TNFSF8:T255455487A)在五指山猪中都只检测到一种突变纯合基因型,而在杜洛克猪中至少检测到两种基因型(表 1)。3个脂类代谢基因位点(IRS2: G76647256A, PRKG1: C97602215A, ADCY5: A137133735G)在五指山猪中也只存在一种突变纯合基因型,而在杜洛克猪中至少检测到两种基因型(表 1)。12个基因的SNPs都是在五指山猪中突变纯合基因型频率达到100%,在杜洛克猪中野生纯合基因型比例最高,所占比例为48%~93%(14/29~27/29)(图 5A),说明这12个基因在在进化过程中受到强烈选择,对五指山猪免疫和脂类代谢具有重要作用。
|
|
表 1 五指山猪和杜洛克猪炎症因子、脂类代谢相关基因的等位基因 Table 1 The alleles of inflammatory factors and lipid metabolism related genes of Wuzhishan pig and Duroc pig |

|
A图的图例中,不同颜色表示突变位点在染色体上的位置和基因型类别。染色体不同功能区域hemo表示突变纯合子,hetero表示杂合子,白色方块代表野生纯合子。B. 基因TNFSF15和TNFSF8连锁不平衡的程度,红色由浅到深表示连锁不平衡程度越来越强 In the legend of Figure A, the different colors of the legend mean the genomic location of the SNPs and the genotypes. The hemo of chromosome functional regions mean variant homozygote, and hetero mean heterozygous, the white squares mean wild homozygote. B. The LD value between gene TNFSF15 and TNFSF8, the red color from the light to the dark mean the LD values of the SNPs have becoming stronger 图 5 五指山猪和杜洛克猪炎症因子和脂类代谢基因SNPs的基因型图和连锁不平衡分析 Fig. 5 The analysis of genotypes and linkage disequilibrium (LD) of inflammatory factors and lipid metabolism related genes of Wuzhishan pig and Duroc pig |
海南岛独特的地理环境为物种隔离创造了条件,限制了五指山猪的祖先与大陆的其它品种进行基因交流,减少了基因多态性,经过世代繁衍,逐渐形成了基因结构相对单一的品系[14]。从物种进化的角度分析,在几百年前甚至更早的时候,五指山猪的祖先为了生存,产生了能够适应恶劣环境的相关基因的突变位点,在自然选择的条件下,这些突变位点被保留并固定下来,基因组上的相关区域很少甚至不再发生变化,从而造就了五指山猪特有的遗传稳定性[15-16],并以纯合突变基因型的形式表现出来。
随着全基因组重测序越来越普遍,采用全基因组分析家畜遗传特征的方法也被广泛使用[17]。冯雪燕等[18]根据芯片数据分析海南猪驯化过程中受选择的情况,使用整合单倍型分数方法得到395个潜在受选择位点,91个候选基因显著富集在生长代谢、免疫应答和雌激素信号通路。李开军等[19]采用全基因组分析方法鉴别了苏淮猪中性洗涤纤维表观消化率相关候选基因位点。XP-CLR是一种基于选择性清除(selective sweeep)的跨群体复合似然比检验方法,通过计算XP-CLR分值判断等位基因受选择的程度,从而筛选出物种祖先突变位点在进化过程中保留的基因型。XP-CLR分值越高的代表该位点受选择程度越高,对应的基因功能越有利于物种在进化过程中生存[20]。本研究中,五指山猪受到选择的区域集中在第1、4、8、13号染色体,富集的功能以免疫应答为主,其次是代谢反应。9个炎症因子相关基因TGFBR2、IL26、IL15、BMPR2、ACKR4、TNFRSF11B、TNFSF15、TNFSF4、TNFSF8的SNPs位点在五指山猪中只存在一种突变纯合基因型,而在杜洛克猪中存在3种基因型,且野生纯合基因型占较大比例,表明两个品种炎症因子分泌功能可能有较大差异。尤其是启动子区发生突变的2个SNPs位点(ACKR4:A73775544G,BMPR2:A106043441C),和5′端和3′端发生突变的3个SNPs位点(TNFSF4:C115548060T,TNFRSF11B:T19878575A,TNFSF15:C255349468T),在基因转录过程中具有重要调控作用[21],能够通过调控基因的表达量影响五指山猪的免疫功能。TGFBR2具有促进免疫细胞移动、黏附,维持血管内皮细胞正常功能的作用[22-23]。IL26与易感自身免疫和过敏性疾病区域的基因高度同源,例如,在克罗恩疾病患者中检测到大量IL26表达[24-25]。IL15作为多种免疫细胞的趋化因子,能够激活T细胞[26]、抑制肿瘤细胞增殖和自身免疫疾病[27-28],减缓氧化应激反应[29]。虽然3个SNPs位点(TGFBR2:C16817427T,IL26:C32573611T和IL15:C85672453T)位于内含子区,但是也可能调控基因功能。BMPR2是维持血管内皮细胞正常功能的重要基因,控制内皮细胞的炎症反应[30]。TNF家族对T细胞、巨噬细胞均有调控作用,诱导细胞凋亡或生成炎症因子,在免疫反应中发挥关键作用[31]。更重要的是,五指山猪的TNFSF15与TNFSF8基因都位于1号染色体,连锁不平衡程度较高(图 5B),而且两个基因功能相似,能够诱导免疫细胞产生大量细胞因子[32-33]。研究表明,脂类代谢基因IRS2协调胰岛素将剩余的葡萄糖转化成脂肪并储存,作用是维持糖类和脂类代谢平衡[34],同时,IRS2也能通过调节PPAR的活性来介导脂类分解和糖脂代谢[35]。ADCY5基因的非编码区调控脂类代谢,当基因表达出现异常时,糖类脂类代谢也会发生紊乱[36]。因此,基因IRS2和ADCY5在脂类代谢和沉积过程中具有重要作用。
基因组的选择信号分析结果表明,五指山猪免疫和脂类代谢相关基因受到选择的突变位点以突变纯合基因型为主,而杜洛克猪大部分个体具有野生纯合基因型、杂交基因型、突变纯合基因型3种基因型,其中野生纯合基因型所占比例最多。这个特征和大部分中国本地猪类似。例如,河南本地猪(淮南、二花脸、确山)亲缘关系较近,纯合基因型位点多于外国品种(杜洛克猪、长白、大白),人工驯化痕迹少,遗传稳定性高[37]。梅山猪和杜洛克猪的全基因组重测序分析发现,约有4.02%的结构差异(structural variations)是梅山猪特有的,而且驯养的梅山猪X染色体上约有30M特异性选择信号区域,然而野生型梅山猪没有[38]。通过分析5个不同地区太湖猪群体的遗传多态性,结果表明,SNPs相关基因的差异主要集中在疾病抵抗力、脂肪组织代谢功能[39]。另外,亲缘关系较近的群体生活在不同地理位置也会造成遗传性状差异[40],江西东乡族、玉山两个地区的猪群体的遗传多样性比较相似,纯合基因型位点占较大比例,而萍乡猪的杂合基因型相对较多[41]。这些中国本地猪具有的纯合基因型是祖先基因组一段区域发生突变,在自然选择的作用下经过世代繁衍,同时伴随着一定程度的地理隔离,从而形成了能够帮助物种更好地适应环境的基因型,那么基因组上这段区域将趋于稳定遗传,很少发生突变,然后逐渐衍变成为了现在的纯合基因型。虽然五指山猪比杜洛克猪SNPs总数多,但是五指山猪突变的位点很可能以纯合基因型为主,也就是在历代繁衍中受到选择的区域,这些区域的基因早先发生的突变在漫长的进化过程中被固定下来,海南岛上相对封闭的环境加上一定程度的近亲繁殖,形成了现在的突变纯合基因型。受到选择的基因型的功能相对于其它基因型更有利于五指山猪适应海南岛的环境,例如位于基因调控区的突变位点ACKR4:A73775544G,BMPR2:A106043441C,TNFSF4:C115548060T,TNFRSF11B:T19878575A,TNFSF15:C255349468T,不仅通过调控基因表达影响免疫和脂类代谢方面的功能,同时也是品种鉴定的重要依据。
4 结论通过比较五指山猪和杜洛克猪2个群体共59个样本全基因组重测序数据,本研究筛选出了9个炎症因子基因和3个脂类代谢基因的功能区域共12个SNPs位点,其中5个炎症基因的SNPs均在上下游调控区域,可能在基因转录过程中具有重要调控作用。12个基因在五指山猪中只存在突变纯合基因型,而在杜洛克猪中存在3种基因型,以野生纯合基因型比例最高,说明五指山猪免疫和脂类代谢相关基因的受选择程度比杜洛克猪更强一些,为揭示五指山猪特色性状形成的分子机制提供参考。
| [1] |
曹婷, 周汉林, 于萍, 等. 五指山猪资源现状、保护及开发利用[J]. 家畜生态学报, 2017, 38(7): 79-83. CAO T, ZHOU H L, YU P, et al. The status quo, protection, development and utilization of Wuzhishan pig resources[J]. Acta Ecologae Animalis Domastici, 2017, 38(7): 79-83. DOI:10.3969/j.issn.1673-1182.2017.07.016 (in Chinese) |
| [2] |
郑宁, 陈萍, 夏云, 等. 饲料中添加牧草对育肥期五指山猪、杜陆猪生长性能的影响[J]. 上海畜牧兽医通讯, 2020(4): 22-23. ZHENG N, CHEN P, XIA Y, et al. Effects of feeding forage on the growth performance of Wuzhishan pig and Dulu pig during fattening period[J]. Shanghai Journal of Animal Husbandry and Veterinary Medicine, 2020(4): 22-23. DOI:10.14170/j.cnki.cn31-1278/s.2020.04.008 (in Chinese) |
| [3] |
孙瑞萍, 陈婷, 王峰, 等. 五指山猪和长白猪背最长肌蛋白组学研究[J]. 湖南农业大学学报(自然科学版), 2021, 47(5): 562-570. SUN R P, CHEN T, WANG F, et al. Proteomic analysis of longissimus dorsi muscle of Wuzhishan pigs and Landrace pigs[J]. Journal of Hunan Agricultural University (Natural Sciences), 2021, 47(5): 562-570. DOI:10.13331/j.cnki.jhau.2021.05.012 (in Chinese) |
| [4] |
晁哲, 吴望军, 王林杰, 等. 五指山猪肌肉和脂肪组织miRNA的鉴定与分析[J]. 畜牧与兽医, 2020, 52(9): 7-12. CHAO Z, WU W J, WANG L J, et al. Identification and analysis of miRNA in the muscle & adipose tissue of Wuzhishan pigs[J]. Animal Husbandry & Veterinary Medicine, 2020, 52(9): 7-12. (in Chinese) |
| [5] |
晁哲, 孙瑞萍, 刘海隆, 等. 五指山猪实验动物化研究进展及其应用前景[C]//2015年(第五届)药物毒理学年会论文集. 海口: 中国毒理学会, 2015: 304-305. CHAO Z, SUN R P, LIU H L, et al. Research progress and application prospects of experimental animalization of Wuzhishan pig[C]// Proceedings of 2015's (the 5th) Annual Meeting of Drug Toxicology. Haikou: 2015: 304-305. (in Chinese) |
| [6] |
VINEREAN H V, GAZDA L S, HALL R D, et al. Streptozotocin is responsible for the induction and progression of renal tumorigenesis in diabetic Wistar-Furth rats treated with insulin or transplanted with agarose encapsulated porcine islets[J]. Islets, 2011, 3(4): 196-203. DOI:10.4161/isl.3.4.16129 |
| [7] |
NIU M M, LIU Y Q, XIANG L, et al. Long-term case study of a Wuzhishan miniature pig with diabetes[J]. Anim Model Exp Med, 2020, 3(1): 22-31. DOI:10.1002/ame2.12098 |
| [8] |
HARRIS A M, DEGIORGIO M. A likelihood approach for uncovering selective sweep signatures from haplotype data[J]. Mol Biol Evol, 2020, 37(10): 3023-3046. DOI:10.1093/molbev/msaa115 |
| [9] |
OSMOND M M, COOP G. Genetic signatures of evolutionary rescue by a selective sweep[J]. Genetics, 2020, 215(3): 813-829. DOI:10.1534/genetics.120.303173 |
| [10] |
LEE Y S, SHIN D, WON K H, et al. Genome-wide scans for detecting the selection signature of the Jeju-island native pig in Korea[J]. Asian-Australas J Anim Sci, 2020, 33(4): 539-546. |
| [11] |
SHANG P, LI W T, TAN Z K, et al. Population genetic analysis of ten geographically isolated Tibetan pig populations[J]. Animals (Basel), 2020, 10(8): 1297. |
| [12] |
BOLGER A M, LOHSE M, USADEL B. Trimmomatic: a flexible trimmer for Illumina sequence data[J]. Bioinformatics, 2014, 30(15): 2114-2120. DOI:10.1093/bioinformatics/btu170 |
| [13] |
VATSIOU A I, BAZIN E, GAGGIOTTI O E. Detection of selective sweeps in structured populations: a comparison of recent methods[J]. Mol Ecol, 2016, 25(1): 89-103. DOI:10.1111/mec.13360 |
| [14] |
孙瑞萍, 魏立民, 晁哲, 等. 海南原产地五指山猪资源保护与开发利用[J]. 养猪, 2015(3): 73-75. SUN R P, WEI L M, CHAO Z, et al. Hainan Wuzhishan pig of origin resources protection and the development and utilization[J]. Swine Prod, 2015(3): 73-75. DOI:10.3969/j.issn.1002-1957.2015.03.037 (in Chinese) |
| [15] |
BARGHI N, SCHLÖTTERER C. Distinct patterns of selective sweep and polygenic adaptation in evolve and resequence studies[J]. Genome Biol Evol, 2020, 12(6): 890-904. DOI:10.1093/gbe/evaa073 |
| [16] |
STEPHAN W. Selective sweeps[J]. Genetics, 2019, 211(1): 5-13. DOI:10.1534/genetics.118.301319 |
| [17] |
袁泽湖, 葛玲, 李发弟, 等. 整合生物学先验信息的全基因组选择方法及其在家畜育种中的应用进展[J]. 畜牧兽医学报, 2021, 52(12): 3323-3334. YUAN Z H, GE L, LI F D, et al. The method of genomic selection by integrating biological prior information and its application in livestock breeding[J]. Acta Veterinaria et Zootechnica Sinica, 2021, 52(12): 3323-3334. DOI:10.11843/j.issn.0366-6964.2021.012.001 (in Chinese) |
| [18] |
冯雪燕, 刁淑琪, 刘玉强, 等. 基于SNP芯片的海南猪全基因组选择信号分析[J]. 畜牧兽医学报, 2022, 53(2): 349-359. FENG X Y, DIAO S Q, LIU Y Q, et al. Analysis of selection signatures for Hainan pigs across the whole genome based on SNP BeadChips[J]. Acta Veterinaria et Zootechnica Sinica, 2022, 53(2): 349-359. (in Chinese) |
| [19] |
李开军, 侯黎明, 蒲广, 等. 基于全基因组Fst和nSL分析鉴别苏淮猪中性洗涤纤维表观消化率相关候选基因位点[J]. 畜牧兽医学报, 2021, 52(7): 1809-1819. LI K J, HOU L M, PU G, et al. Identification of candidate gene loci related to apparent NDF digestibility of Suhuai pigs based on genome-wide Fst and nSL analyses[J]. Acta Veterinaria et Zootechnica Sinica, 2021, 52(7): 1809-1819. (in Chinese) |
| [20] |
ZHANG X F, QU K X, JIA P, et al. Assessing genomic diversity and productivity signatures in dianzhong cattle by whole-genome scanning[J]. Front Genet, 2021, 12: 719215. DOI:10.3389/fgene.2021.719215 |
| [21] |
GRENCEWICZ D J, ROMIGH T, THACKER S, et al. Redefining the PTEN promoter: identification of novel upstream transcription start regions[J]. Hum Mol Genet, 2021, 30(22): 2135-2148. DOI:10.1093/hmg/ddab175 |
| [22] |
KUMAR P, SMITH T, RAEMAN R, et al. Periostin promotes liver fibrogenesis by activating lysyl oxidase in hepatic stellate cells[J]. J Biol Chem, 2018, 293(33): 12781-12792. DOI:10.1074/jbc.RA117.001601 |
| [23] |
ZHU J, ANGELOV S, ALP YILDIRIM I A, et al. Loss of transforming growth factor beta signaling in aortic smooth muscle cells causes endothelial dysfunction and aortic hypercontractility[J]. Arterioscler Thromb Vasc Biol, 2021, 41(6): 1956-1971. DOI:10.1161/ATVBAHA.121.315878 |
| [24] |
MEDOVIC M V, JAKOVLJEVIC V L, ZIVKOVIC V I, et al. Psoriasis between autoimmunity and oxidative stress: changes induced by different therapeutic approaches[J]. Oxid Med Cell Longev, 2022, 2022: 2249834. |
| [25] |
NIESS J H, HRUZ P, KAYMAK T. The interleukin-20 cytokines in intestinal diseases[J]. Front Immunol, 2018, 9: 1373. DOI:10.3389/fimmu.2018.01373 |
| [26] |
ALIZADEH D, WONG R A, YANG X, et al. IL15 enhances CAR-T Cell antitumor activity by reducing mTORC1 activity and preserving their stem cell memory phenotype[J]. Cancer Immunol Res, 2019, 7(5): 759-772. DOI:10.1158/2326-6066.CIR-18-0466 |
| [27] |
GUO Y, LUAN L M, PATIL N K, et al. Immunobiology of the IL-15/IL-15Rα complex as an antitumor and antiviral agent[J]. Cytokine Growth Factor Rev, 2017, 38: 10-21. DOI:10.1016/j.cytogfr.2017.08.002 |
| [28] |
JIANG W, ZHANG C, TIAN Z G, et al. hIL-15-gene modified human natural killer cells (NKL-IL15) exhibit anti-human leukemia functions[J]. J Cancer Res Clin Oncol, 2018, 144(7): 1279-1288. DOI:10.1007/s00432-018-2654-0 |
| [29] |
NGUYEN V, AMERI K, HUYNH K, et al. Interleukin-15 modulates the response of cortical neurons to ischemia[J]. Mol Cell Neurosci, 2021, 115: 103658. DOI:10.1016/j.mcn.2021.103658 |
| [30] |
MIYAGAWA K, SHI M Y, CHEN P I, et al. Smooth muscle contact drives endothelial regeneration by BMPR2-notch1-mediated metabolic and epigenetic changes[J]. Circ Res, 2019, 124(2): 211-224. DOI:10.1161/CIRCRESAHA.118.313374 |
| [31] |
YE L P, HE S Q, MAO X L, et al. Effect of hepatic macrophage polarization and apoptosis on liver ischemia and reperfusion injury during liver transplantation[J]. Front Immunol, 2020, 11: 1193. DOI:10.3389/fimmu.2020.01193 |
| [32] |
SUN R, HEDL M, ABRAHAM C. TNFSF15 promotes antimicrobial pathways in human macrophages and these are modulated by TNFSF15 disease-risk variants[J]. Cell Mol Gastroenterol Hepatol, 2021, 11(1): 249-272. DOI:10.1016/j.jcmgh.2020.08.003 |
| [33] |
FAVA V M, SALES-MARQUES C, ALCAÏS A, et al. Age-dependent association of TNFSF15/TNFSF8 variants and leprosy type 1 reaction[J]. Front Immunol, 2017, 8: 155. |
| [34] |
BESSE-PATIN A, JEROMSON S, LEVESQUE-DAMPHOUSSE P, et al. PGC1A regulates the IRS1:IRS2 ratio during fasting to influence hepatic metabolism downstream of insulin[J]. Proc Natl Acad Sci U S A, 2019, 116(10): 4285-4290. DOI:10.1073/pnas.1815150116 |
| [35] |
MATSUSHITA M, AWAZAWA M, KOBAYASHI N, et al. An antisense transcript transcribed from Irs2 locus contributes to the pathogenesis of hepatic steatosis in insulin resistance[J]. Cell Chem Biol, 2022, 29(4): 680-689. DOI:10.1016/j.chembiol.2021.12.008 |
| [36] |
SINNOTT-ARMSTRONG N, SOUSA I S, LABER S, et al. A regulatory variant at 3q21.1 confers an increased pleiotropic risk for hyperglycemia and altered bone mineral density[J]. Cell Metab, 2021, 33(3): 615-628.E13. DOI:10.1016/j.cmet.2021.01.001 |
| [37] |
QIAO R, LI X, HAN X, et al. Population structure and genetic diversity of four Henan pig populations[J]. Anim Genet, 2019, 50(3): 262-265. DOI:10.1111/age.12775 |
| [38] |
ZHOU R, LI S T, YAO W Y, et al. The Meishan pig genome reveals structural variation-mediated gene expression and phenotypic divergence underlying Asian pig domestication[J]. Mol Ecol Resour, 2021, 21(6): 2077-2092. DOI:10.1111/1755-0998.13396 |
| [39] |
XIAO Q, ZHANG Z, SUN H, et al. Genetic variation and genetic structure of five Chinese indigenous pig populations in Jiangsu Province revealed by sequencing data[J]. Anim Genet, 2017, 48(5): 596-599. DOI:10.1111/age.12560 |
| [40] |
LIU Y C, MAO X W, KRAUSE J, et al. Insights into human history from the first decade of ancient human genomics[J]. Science, 2021, 373(6562): 1479-1484. DOI:10.1126/science.abi8202 |
| [41] |
WANG X, WANG C, HUANG M, et al. Genetic diversity, population structure and phylogenetic relationships of three indigenous pig breeds from Jiangxi Province, China, in a worldwide panel of pigs[J]. Anim Genet, 2018, 49(4): 275-283. DOI:10.1111/age.12687 |
(编辑 郭云雁)



