2. 贵州大学动物疫病研究所, 贵阳 550025;
3. 贵州省农业科学院畜牧兽医研究所, 贵阳 550025
2. Institute of Animal Diseases, Guizhou University, Guiyang 550025, China;
3. Institute of Animal Husbandry and Veterinary Medicine, Guizhou Academy of Agricultural Sciences, Guiyang 550025, China
STM sRNA GcvB是一类重要的RNA分子,其无法编码相应的蛋白质,而是通过与Hfq协同作用,转录后调控STM有关靶mRNA[1]。现已经验证确认受GcvB直接调控的靶mRNA在STM中近30个,其中,大部分靶mRNA与GcvB的结合位点也已阐明[2]。sRNA-Hfq -mRNA三联体在细菌中普遍存在,Hfq不仅能够维持sRNA在细菌中转录后的稳定性,而且可重塑sRNA与其靶mRNA的二级结构构象,使sRNA易于与其靶mRNA结合[3]。Hfq六聚体通常利用其近端面与sRNA富含U的序列结合并作用[4]。
STM Hfq具有维持GcvB在体内稳定性的作用,但Hfq与GcvB具体的作用结合位点尚未探究透彻。鉴于此,本研究将利用RNA二级结构预测软件分析GcvB二级结构并筛查Hfq结合偏好型的富U基序;利用λ-Red同源重组酶系统构建相应序列的突变或截短菌株;利用噬菌体转导技术构建相应的hfq或gcvB基因缺失菌株以及lacZ基因融合菌株;通过qRT-PCR检测不同重组菌株gcvB基因的转录水平;通过β-半乳糖苷酶试验检测不同重组菌株oppA基因的蛋白水平,最后分析Hfq与GcvB富U基序的作用关系,为阐明STM Hfq与GcvB的作用机理提供理论依据。
1 材料与方法 1.1 主要实验材料MA7455菌株(STM LT2/pKD46),MA10241菌株(STM LT2 ΔgcvB: :cat),MA10675菌株(STM LT2 Δhfq: :tetRA),P22噬菌体均由法国国家科学研究中心(CNRS)分子遗传学Bossi实验室惠赠。STM LT2 oppA: :lacZ菌株由本实验室构建。氯霉素、盐酸四环素、卡那霉素、dNTPs Mix及ONPG均购自北京索莱宝科技有限公司。AceQ Universal SYBR qPCR Master Mix和胶回收试剂盒均购自南京诺唯赞生物科技有限公司。NovoScript® Plus All-in-one 1 st Strand cDNA Synthesis SuperMix (gDNA Purge)购自近岸蛋白生物科技有限公司。
1.2 引物设计与合成登录NCBI网站(https://www.ncbi.nlm.nih.gov/),获取STM LT2株的全基因序列(GeneID:AE006468.2), 根据基因序列设计引物(表 1),引物由Primer select软件设计,并由上海生工生物工程有限公司合成。
|
|
表 1 PCR引物序列 Table 1 The primer sequence for PCR |
使用Mfold软件预测GcvB二级结构。登录网站(http://unafold.rna.albany.edu/?q=mfold),打开RNA Folding Form (Version 2.3 energies)选项,输入gcvB基因完整序列,其余参数按默认方案设置,导出并分析结果。筛查GcvB中Hfq结合偏好型富U基序。
1.4 GcvB富U基序突变或截短菌株的构建利用λ-Red同源重组酶系统,以P1/P2为引物,MA10241菌株总DNA为模板,PCR扩增后产物回收作为模板,P3/P10为引物,产物经纯化后与pKD46感受态细胞进行电转,在平板上培养后以P8/P9为引物鉴定其菌落,筛选出STM LT2 gcvB: : cat;以P4/P10为引物,PCR扩增用于替换gcvB基因内部为T的目的片段,分别以P5/P10、P6/P10、P7/P10和P8/P10为引物,PCR扩增用于截短gcvB基因T8的目的片段,用上述相同方法将鉴定正确的菌株分别命名为STM LT2 gcvB(130—134TTTTT>GGGGG): : cat、STM LT2 gcvB Δ(206): : cat、STM LT2 gcvB Δ(205—206): : cat、STM LT2 gcvB Δ(204—206): : cat和STM LT2 gcvB Δ(203—206): : cat;以STM LT2 gcvB Δ(204—206): : cat菌株总DNA为模板,P1/P2为引物,其产物为模板,P9/ P10为引物,以相同方法鉴定正确的菌株命名为STM LT2gcvB[Δ(203—206), (184—185): : CCCC, (192—193): : GGGG]: : cat。
1.5 P22噬菌体的转导将鼠伤寒沙门菌MA10675和STM LT2 oppA: : lacZ缺失菌株作为供体菌株,GcvB富U基序突变或截短菌株为受体菌,分别取受体菌100、50 μL稀释后供体菌borth至2.0 mL EP管中混匀置于37 ℃摇床孵育1 h后涂布筛选,以引物P10/P11、P6/P7分别对hfq基因的缺失、oppA-lacZ融合基因进行鉴定。
1.6 qRT-PCR反应根据R6950 Bacterial RNA Kit,提取鼠伤寒沙门菌LT2的RNA,以反转录得到的cDNA作为模板,P11/P12(gcvB基因)和P13/P14(16S rDNA基因)为引物,按照SYBR©Premix Ex TaqTM II(Tli RNaseH Plus)试剂盒说明书进行qPCR,以16S rDNA基因为内参基因,以STM LT2 gcvB: : cat菌株为对照。最终采用相对比较法即Ct法(Qr=2-ΔΔCt)计算分析数据[5]。
1.7 β-半乳糖苷酶活性的检测取50 μL待测菌株置于5 mL LB中,培养至OD600 nm=0.4后,取0.1 mL至新试管中,以Z buffer定容到1 mL。滴加3滴甲苯后在42 ℃水浴摇床中振荡2 h,将试管转于28 ℃水浴锅加入0.2 mL ONPG,记录加入时间(t0)。当试管中呈现出黄色时,加入0.5 mL Na2CO3终止反应,记录终止时间(tf)。将试管中终止所得液体离心后测其OD420nm,记录数值。通过公式计算β-半乳糖苷酶活性。
| $ \;\;\;\;\;\;\beta \text {-半乳糖苷酶活性 }=\left(1\;000 \times\left(\mathrm{OD}_{420 \mathrm{~nm}}\right) /\left[\left(t_f-\right.\right.\right.\\ \left.\left.t_0\right) \times \mathrm{OD}_{600 \mathrm{~nm}}\right] $ |
使用Mfold软件预测STM LT2 GcvB的二级结构。结果显示,GcvB碱基可退火形成6个茎环结构,查找到的两个Hfq结合偏好型基序,U5位于第4个与第5个茎环之间,U8位于GcvB 3′末端,且均处于单链状态。
2.2 GcvB U5基序突变菌株/U8基序截短菌株及其hfq基因缺失菌株的构建结果利用λ-Red同源重组酶系统和噬菌体转导技术构建相应的hfq基因缺失菌株。以STM LT2 gcvB: : cat菌株DNA为模板,利用λ-Red同源重组酶系统将gcvB基因T5(130—134 nt)突变为G5, 将gcvB基因T8 (199—207 nt)截短。PCR鉴定结果显示,所有重组菌株均出现与预期相符的918、1 964 bp目的条带,将PCR测序后进行峰图比对,STM LT2 gcvB基因T5(130—134 nt)已突变为G5,STM LT2 gcvB基因T8 (199—207 nt)已分别被截短突变为T7、T6、T5和T4。
2.3 gcvB T5基序突变对gcvB转录水平的影响经过qRT-PCR试验结果显示,与野生型菌株(WT)相比,T5基序突变后gcvB基因转录水平未产生明显变化,敲除hfq基因后gcvB基因转录水平下调52%,敲除hfq基因的基础上T5基序的突变使gcvB基因转录水平下调51%。以上结果表明,hfq与gcvB基因转录水平呈正相关,但gcvB T5基序与gcvB基因的转录水平无明显关联。
2.4 gcvB T8基序截短对GcvB转录水平的影响对gcvB T8基序截短菌株(T7、T6、T5和T4)及相应的hfq基因缺失菌株的gcvB转录水平进行检测。qRT-PCR检测结果显示,与WT相比,T7和T6菌株gcvB基因转录水平未产生明显变化,而T5和T4菌株分别下调40.5%和37.5%。敲除hfq基因的基础上截短T8基序,与WT相比,T7、T6、T5和T4菌株分别下调58.5%、57.8%、75.2%和77.8%(图 1)。以上结果表明,gcvB基因T8基序的截短对其转录水平产生了抑制作用,截短为T5和T4时作用最为明显。
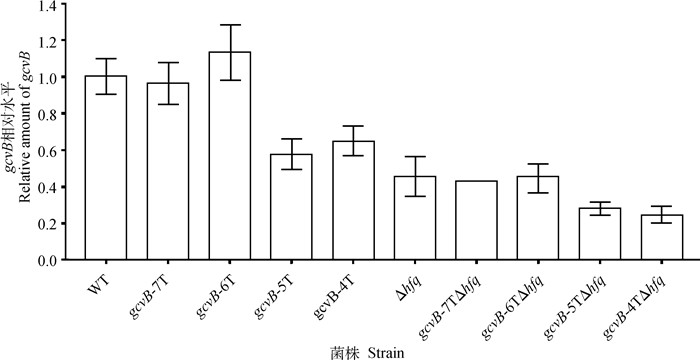
|
WT. STM LT2 gcvB: : cat; gcvB-7T. STM LT2 gcvB Δ(206): : cat; gcvB-6T. STM LT2 gcvB Δ(205—206): : cat; gcvB-5T. STM LT2 gcvB Δ(204—206): : cat; gcvB-4T. STM LT2 gcvB Δ(203—206): : cat; Δhfq. STM LT2 gcvB: : cat Δhfq: : tetRA; gcvB-7TΔhfq. STM LT2 gcvB Δ(206): : cat Δhfq: : tetRA; gcvB-6TΔhfq. STM LT2 gcvB Δ(205—206): : cat Δhfq: : tetRA; gcvB-5TΔhfq. STM LT2 gcvB Δ(204—206): : cat Δhfq: : tetRA; gcvB-4TΔhfq. STM LT2 gcvB Δ(203—206): : cat Δhfq: : tetRA 图 1 gcvB基因转录水平qRT-PCR检测结果 Fig. 1 The results of transcriptional expression level of gcvB gene by qRT-PCR |
利用λ-Red同源重组酶系统在gcvB基因185—186位碱基之间插入C4序列, 191—192位碱基之间插入G4序列,构建GcvB终止子茎延伸菌株,在此基础上,利用噬菌体转导技术构建hfq基因缺失菌株。PCR产物测序峰图比对表明目的片段已正确构建。
2.6 终止子茎延伸对GcvB转录水平的影响对STM LT2 gcvB Δ(203—206): : cat菌株GcvB终止子茎延伸菌株及其hfq基因缺失菌株的gcvB转录水平进行检测。qRT-PCR检测结果显示,与WT相比,gcvB-LS4T终止子茎延伸菌株gcvB基因转录水平略微下调19.6%,而在此基础上敲除hfq基因后,gcvB基因转录水平下调29%。与仅敲除hfq基因时相比,gcvB-LS4TgcvBΔhfq基因转录水平上调为原来的1.6倍(图 2)。以上结果表明,终止子茎的延伸与gcvB基因的转录水平呈正相关。
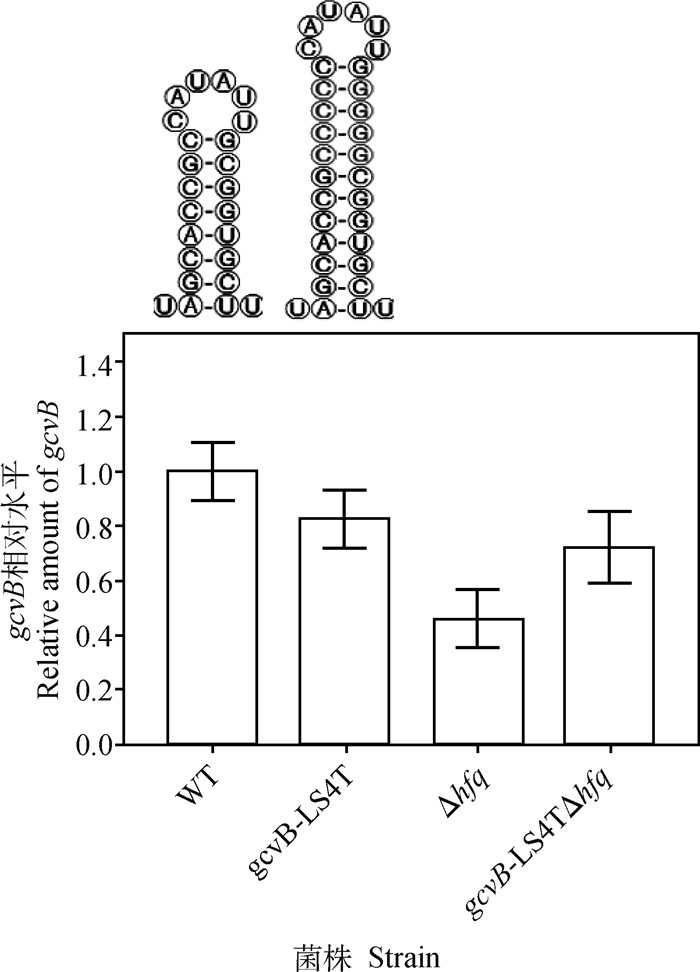
|
WT. STM LT2 gcvB: : cat; gcvB-LS4T. STM LT2 gcvB[Δ(203—206), (184—185): : CCCC, (192—193): : GGGG]: : cat; Δhfq. STM LT2 gcvB: : cat Δhfq: : tetRA; gcvB-LS4TΔhfq. STM LT2 gcvB[Δ(203—206), (184—185): : CCCC, (192—193): : GGGG]: : cat Δhfq: : tetRA 图 2 gcvB基因转录水平qRT-PCR检测结果 Fig. 2 The results of transcriptional expression level of gcvB gene by qRT-PCR |
以STM LT2 oppA: :lacZ菌株作为供体菌,STM LT2 gcvB: :cat、STM LT2 gcvB Δ(203—206): : cat和STM LT2 gcvB[Δ(203—206), (184—185): : CCCC, (192—193): : GGGG]: : cat分别为受体菌,运用噬菌体转导技术构建相应的oppA: : lacZ基因融合菌株。PCR鉴定结果显示,所有重组菌株均出现与预期相符的918、914、922、250 bp目的条带(图 3)。
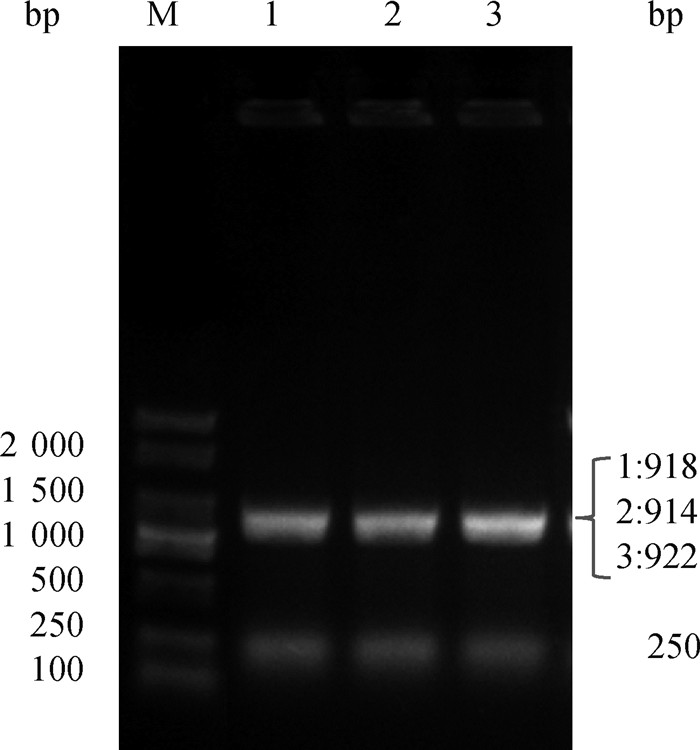
|
M. DL2000 marker; 1. STM LT2 gcvB: :cat oppA: :lacZ; 2. STM LT2 gcvB Δ(203—206): : cat oppA: :lacZ; 3. STM LT2 gcvB[Δ(203—206), (184—185): : CCCC, (192—193): : GGGG]: : cat oppA: :lacZ 图 3 oppA: :lacZ融合基因噬菌体转导试验PCR扩增片段电泳结果 Fig. 3 PCR electrophoresis results of phage transduction experiment of oppA: :lacZ fusion gene |
通过检测oppA: :lacZ基因融合菌株β-半乳糖苷酶活性,探究gcvB T8截短为T4以及延伸终止子茎对OppA蛋白表达的影响。结果显示,与WT相比,gcvB基因T8截短为T4后,oppA基因蛋白水平上调为原来的1.7倍。延伸该菌株的GcvB终止子茎后oppA基因蛋白水平上调为原来的1.7倍。以上结果表明,GcvB终止子茎的延伸虽恢复了因gcvB基因T8基序截短为T4时gcvB基因转录水平的下调,却未恢复下调OppA蛋白表达的能力。
3 讨论Sauer和Weichenrieder[6]通过分析尺寸排阻色谱法证实了大肠杆菌Hfq与sRNA RybB的3′末端U6具有很高的亲和力,由于大多数Hfq依赖型sRNA 3′末端都具有4~8个尿嘧啶,该研究揭示了一种重要的Hfq与sRNA作用模式。Otaka等[4]发现截短大肠杆菌sRNA SgrS 3′末端U8时,SgrS丧失了与Hfq结合并使ptsG mRNA沉默的能力,再次证实了sRNA 3′末端polyU对Hfq结合的重要性。本研究中,对沙门菌sRNA GcvB二级结构预测,考虑到U5处于GcvB内部且在茎环结构附近,缺失将严重引起二级结构的改变从而增加了变量,不利于试验分析GcvB与Hfq之间的作用关系,因此对U5进行了突变处理,对U8进行截短处理。先前的研究已表明,Hfq主要与SgrS 3′末端U8结合,从而增强了SgrS的稳定性。试验检测了突变的U5及截短的U8菌株gcvB基因的转录水平,结果显示,U5的突变并未引起gcvB基因转录水平的显著变化,而U8的截短导致gcvB基因转录水平的明显变化,当GcvB 3′末端尿嘧啶数量缩减为5个时,GcvB转录水平开始表现出明显的下调。经初步验证,U8基序对于Hfq维持GcvB的稳定性以及协助GcvB负调控OppA的表达是必要的。
由于怀疑GcvB U8基序的缩短破坏了gcvB的转录终止进程,本研究在GcvB U8基序截短为U4的基础上延伸了U8基序前的终止子茎,与未延伸终止子茎相比,GcvB转录水平上调,几乎恢复至野生型菌株的水平,在此基础上敲除hfq基因也并未使GcvB转录水平发生明显变化,仅略微下调。但结果仍不能充分说明GcvB U8基序截短为U4 GcvB转录水平下调的原因是破坏了gcvB的转录终止进程或是破坏了GcvB与Hfq结合的关键基序,研究推测两种方式均存在。而通过对oppA基因蛋白水平的检测,发现GcvB U4菌株和其终止子茎延伸菌株oppA基因蛋白水平均较野生型菌株上调,该结果提示尽管终止子茎的延伸恢复了GcvB转录水平,但其抑制oppA mRNA的翻译的能力已然丧失,而Hfq是GcvB发挥调控作用依赖的伴侣蛋白。结果进一步说明GcvB U8基序对于Hfq维持GcvB的稳定性是重要的。
4 结论U5基序与Hfq维持GcvB稳定性无明显关联。U8基序对Hfq协助GcvB转录后负调控oppA mRNA以及维持GcvB稳定性来说是重要的,推测其为Hfq与GcvB的作用结合位点。
| [1] |
ARGAMAN L, HERSHBERG R, VOGEL J, et al. Novel small RNA-encoding genes in the intergenic regions of Escherichia coli[J]. Curr Biol, 2001, 11(12): 941-950. DOI:10.1016/S0960-9822(01)00270-6 |
| [2] |
MIYAKOSHI M, CHAO Y J, VOGEL J. Cross talk between ABC transporter mRNAs via a target mRNA-derived sponge of the GcvB small RNA[J]. EMBO J, 2015, 34(11): 1478-1492. DOI:10.15252/embj.201490546 |
| [3] |
SANTIAGO-FRANGOS A, WOODSON S A. Hfq chaperone brings speed dating to bacterial sRNA[J]. Wiley Interdiscip Rev RNA, 2018, 9(4): e1475. |
| [4] |
OTAKA H, ISHIKAWA H, MORITA T, et al. PolyU tail of rho-independent terminator of bacterial small RNAs is essential for Hfq action[J]. Proc Natl Acad Sci U S A, 2011, 108(32): 13059-13064. DOI:10.1073/pnas.1107050108 |
| [5] |
SCHMITTGEN T D, LIVAK K J. Analyzing real-time PCR data by the comparative CT method[J]. Nat Protoc, 2008, 3(6): 1101-1108. DOI:10.1038/nprot.2008.73 |
| [6] |
SAUER E, WEICHENRIEDER O. Structural basis for RNA 3'-end recognition by Hfq[J]. Proc Natl Acad Sci U S A, 2011, 108(32): 13065-13070. DOI:10.1073/pnas.1103420108 |
(编辑 白永平)



