产气荚膜梭菌(Clostridium perfringens)是杆菌科、梭菌属的一种革兰阳性厌氧菌[1],可广泛存在于人类和动物肠道以及食物、河流和土壤等多种自然环境中[2]。作为一种常见的条件性致病菌,产气荚膜梭菌因能够导致多种疾病、造成较大畜牧经济损失和公共卫生安全危害而被广泛关注[3]。正常条件下,产气荚膜梭菌在人和动物肠道中存在但并不致病,但当外界环境突变时其能够迅速繁殖并产生20余种蛋白性毒素并侵袭机体,导致机体发生气性坏疽和多种胃肠道疾病[4]。根据对CPA、CPB、ETX、ITX、CPE和NetB 6种毒素的产生能力,产气荚膜梭菌被分为A~G 7个毒素型[5]。
D型产气荚膜梭菌能够引起山羊、绵羊和牛等多种动物的肠毒血症,造成腹泻和猝死等多种症状,对反刍动物的规模化饲养带来巨大经济损失[6]。D型产气荚膜梭菌产生的毒素ETX是由位于质粒上的毒素基因etx编码、分子量大小为33 ku的穿孔毒素[7]。ETX被认为是毒性最强的产气荚膜梭菌毒素,毒性仅次于肉毒毒素和破伤风毒素[8],能够作用于肠道、肾和肝多种器官,导致包括猝死在内的多种症状[9];并且ETX还能够和神经细胞结合,造成神经递质异常释放从而导致机体神经系统疾病[10]。此外,产气荚膜梭菌能够引起人类食源性感染[11],据相关机构统计,产气荚膜梭菌引起的食物中毒在美国和韩国食源性感染中排名第2位,在英国和法国则排在第3位[12]。
关中奶山羊是由萨能奶山羊和陕西本地山羊杂交后获得的新品种,是我国奶山羊中的优良品种。本试验对采集自腹泻关中奶山羊的肛门拭子进行了产气荚膜梭菌的分离鉴定并对分离到的5株D型产气荚膜梭菌进行了全基因组序列测定分析。本研究系我国首次对D型产气荚膜梭菌基因组进行研究,对产气荚膜梭菌疾病的防控及D型产气荚膜梭菌基因组的后续研究具有重要参考意义。
1 材料与方法 1.1 材料1.1.1 样品来源 肛门拭子样品于2021年下旬采集自陕西省富平县某规模化关中奶山羊养殖场6只精神沉郁、伴有不同程度腹泻的奶山羊。样品采集后置于运输培养基并保存于4 ℃采样箱,12 h内送回西北农林科技大学兽医传染病实验室进行细菌分离鉴定。
1.1.2 主要试剂仪器 细菌DNA提取试剂盒购自天根生化科技有限公司;2×Taq PCR StarMix为北京Genstar生物科技有限公司产品;胰胨-亚硫酸盐-环丝氨酸(TSC)培养基、厌氧肉肝汤培养基、CAMHB肉汤和普通营养琼脂购自青岛海博生物技术有限公司;厌氧培养箱为上海新苗医疗器械制造有限公司产品。
1.2 方法1.2.1 产气荚膜梭菌分离鉴定 将所采集的肛门拭子接种于5 mL厌氧肉肝汤培养基,37 ℃温箱静置培养18 h增菌。培养完成后,取1 mL菌液3 000 r·min-1离心2 min,并划线接种于TSC培养基,置于厌氧培养箱37 ℃培养24 h。挑取TSC培养基上黑色菌落,接种于5%绵羊血平板培养基,进行进一步分离纯化;挑取血平板上带有双重溶血环的单菌落,生理盐水稀释煮菌后,PCR扩增分离菌株16S rRNA序列,并送至西安擎科泽西生物技术有限公司序列测定。
1.2.2 分离菌株毒素分型 毒素基因cpa、cpb、etx、itx、cpe、netB的特异引物见表 1[5],由西安擎科泽西生物技术有限责任公司合成。水煮法提取菌株DNA,毒素基因cpa、cpb、etx、itx采用多重PCR, 50 μL体系:25 μL 2×Mix预混液,cpa、cpb、etx、itx毒素上下游引物各1 μL,DNA模板1 μL, 双蒸水补至50 μL。反应程序:94 ℃ 10 min;94 ℃ 30 s,51.6 ℃ 30 s,72 ℃ 30 s, 30个循环;72 ℃ 10 min。cpe、netB采用单重PCR,20 μL体系:10 μL 2×Mix预混液,上下游引物各1 μL,模板DNA 1 μL,双蒸水补至20 μL。反应程序:94 ℃ 10 min;94 ℃ 1 min,54 ℃ 1 min,72 ℃ 1 min, 30个循环;72 ℃ 10 min。
|
|
表 1 毒素基因PCR引物 Table 1 Primers used for PCR detection of toxin genes |
1.2.3 分离菌株全基因组序列测定及分析 将分离菌株接种于厌氧肉肝汤中过夜培养,无菌条件下取过夜培养菌液3 mL,25 ℃ 12 000 r·min-1离心2 min,按照细菌DNA提取试剂盒流程提取分离菌株DNA,并送至金唯智生物科技有限公司进行全基因组序列测定。分离菌株基因组序列注释在Rapid Annotations using Subsystem Technology (RAST) (http://rast.nmpdr.org/)上完成;MLST分型通过PubMLST (https://pubmlst.org/) 完成,耐药基因、毒素基因和插入序列分别通过ResFinder 4.1 (https://cge.food.dtu.dk/services/ResFinder/),ISfinder (https://www-is.biotoul.fr/),Virulence Factor Database (http://www.mgc.ac.cn/VFs/) 和本地blast进行检测。基因序列信息和基因环境的对比分析通过Snapgene 4.2.4和Easyfig 2.1实现。
2 结果 2.1 细菌分离鉴定及毒素分型分离菌株在37 ℃厌氧条件下培养24 h后,在TSC平板上为中心黑色边缘半透明中等大小菌落(图 1A),5%绵羊血平板培养基上为带有双重溶血环的半透明光滑较小菌落(图 1B)。PCR扩增分离菌株16S rRNA序列后比对显示,其与参考产气荚膜梭菌菌株ATCC13124的16S rRNA序列相似度均达99%以上,综合判定分离菌株均为产气荚膜梭菌。毒素分型显示,5株产气荚膜梭菌分离株均为D型产气荚膜梭菌(cpa+,etx+),根据菌株分离年份和毒素型将分离到的5株D型产气荚膜梭菌命名21-D-1、21-D-2、21-D-3、21-D-4和21-D-5;其中,菌株21-D-5中检测食源性致病毒素基因cpe。
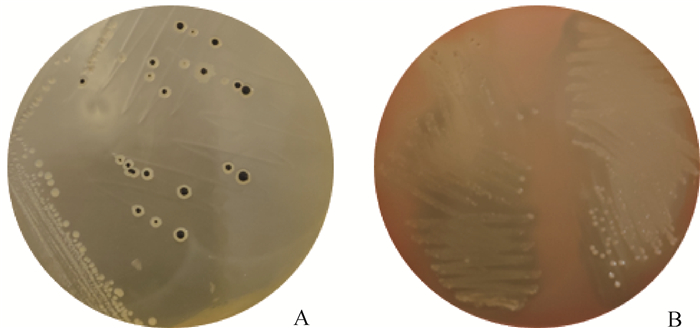
|
图 1 分离菌株21-D-1在TSC平板(A)和5%绵羊血平板(B)上菌落形态观察 Fig. 1 Colony morphology observation of isolate 21-D-5 on TSC agars (A) and 5% sheep blood agars (B) |
5株D型产气荚膜梭菌分离株基因组序列已上传至NCBI数据库(Bioproject:PRJNA811729);分离菌株的基因组大小为3 403 167~3 570 770 bp,平均每个基因组中含有3 298个基因编码区(CDS),且基因组序列GC含量稳定于28.0%~28.1%(表 2)。多位点序列分析(MLST)显示,21-D-1、21-D-2、21-D-3、21-D-4和21-D-5属于3个不同的ST型,21-D-1/21-D-2、21-D-3/21-D-4和21-D-5的等位基因组合分别为129-5-1-1-8-19-4-1、37-134-81-34-8-19-1-8和153-94-86-11-90-7-2-140。5株D型产气荚膜梭菌分离株中共检测到15种毒素基因,其中,cpa、etx、pfoA、colA、cloSI、nanH、nagK、nagI、nagJ、nanI、nanJ、cpb2和nagH在5株D型产气荚膜梭菌中均被检测到,nagL在21-D-1、21-D-2和21-D-5中检测到,而cpe仅在21-D-5中被检测到(表 3)。除毒素基因外,分离菌株的基因组序列中还检测到9种耐药基因,包括四环素类耐药基因tetA(P)和tetB(P)、氨基糖苷类耐药基因aac(6′)-aph(2″)、林可霉素抗性基因lnu(P);大环内酯类耐药基因erm(A)、erm(B)和erm(Q)、氟苯尼考耐药基因fexA和恶唑烷酮耐药基因optrA, 并且这9中耐药基因均在21-D-5的基因组中被检测到(表 4)。
|
|
表 2 D型产气荚膜梭菌基分离株基因组信息 Table 2 Genomic information of the newly sequenced C. perfringens type D isolates |
|
|
表 3 D型产气荚膜梭菌分离株毒素基因汇总 Table 3 Summary of the toxin genes detected among the C. perfringens type D isolates |
|
|
表 4 D型产气荚膜梭菌分离株耐药基因检测 Table 4 Antimicrobial resistant genes detected among the C. perfringens type D isolates |
选取NCBI数据库中B型产气荚膜梭菌(ATCC3626)和D型产气荚膜梭菌(NCTC8503)etx序列作为参考对本研究中D型产气荚膜梭菌毒素基因etx序列的保守性进行评估。结果显示(表 5),毒素基因etx序列在本研究新分离到的D型产气荚膜梭菌和参考序列之间保守性较强(99.8%~100%),仅在第51位核苷酸(由C-G变为G-C)和726位核苷酸(由G-C变为A-T)处存在差异,且不影响对应位置丝氨酸的编码。
|
|
表 5 分离菌株毒素基因etx序列与参考etx序列对比 Table 5 Comparisons between the etx sequences of newly obtained C. perfringens strains and reference etx sequences |
选取NCBI数据库中D型产气荚膜梭菌NCTC8503 (NCBI BioSample: SAMEA3879480), JGS1721 (NCBI BioSample: SAMN02436277), IQ1 (NCBI BioSample: SAMEA5818795)基因组序列与本研究新分离的D型产气荚膜梭菌基因组进行单核苷酸多态性(SNPs)分析。结果表明,21-D-1和21-D-2以及21-D-3和21-D-4之间的SNPs差异很小(< 25),而21-D-1/21-D-2,21-D-3/21-D-4与其他菌株之间的SNPs则在16 440~21 943不等。依照SNPs构建的进化树显示,21-D-1和21-D-2,21-D-3和21-D-4之间的进化关系最为相近,其次为21-D-5,最后为分离自其他国家的参考菌株NCTC8503、JGS1721和IQ1(图 2)。
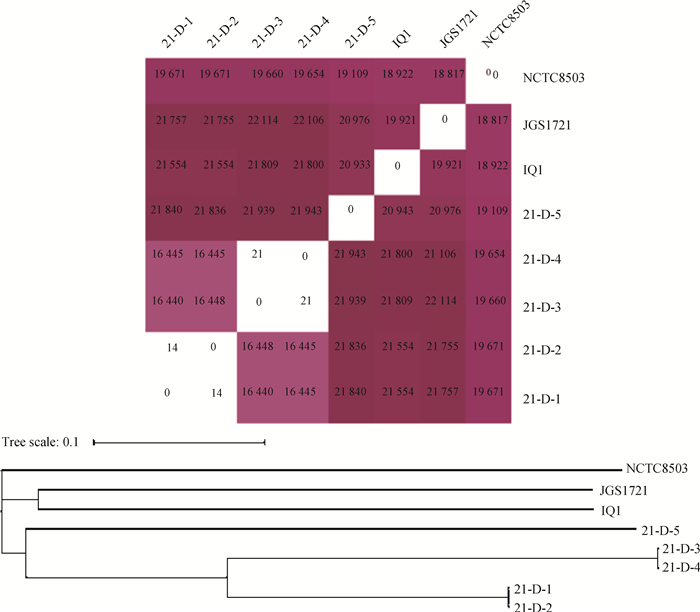
|
图 2 D型产气荚膜梭菌分离菌株及参考株之间SNPs差异及进化关系 Fig. 2 SNPs diversity and phylogenic analysis of the C. perfringens type D isolates and the reference C. perfringens isolates |
选取SNPs分析中使用的D型产气荚膜梭菌菌株与本研究分离到的D型产气荚膜梭菌对毒素基因etx的基因环境进行分析。结果显示,etx基因环境在菌株21-D-1、21-D-2、21-D-3、21-D-4、NCTC8503、JGS1721和IQ1中一致,均为移动元件-插入序列IS1151-etx-转座酶编码基因-插入序列ISCp1;但在分离株21-D-5中etx的下游额外检测到了插入序列IS1151、毒素基因cpe和一些假定蛋白编码基因hp;并且序列分析显示,分离株21-D-5中etx上下游插入序列IS1151与21-D-1~21-D-4中etx上游插入序列IS1151序列相似度较低,为92%~93%(图 3)。

|
图 3 D型产气荚膜梭菌菌株及参考D型产气荚膜梭菌菌株etx基因环境分析 Fig. 3 Analysis of the genetic environment of etx among the C. perfringens type D isolates and the reference C. perfringens isolates |
对2020年和2022年我国报道的携带有噁唑烷酮类耐药基因optrA的产气荚膜梭菌菌株(2C45,QHY-2)[13-14],与本研究菌株21-D-5进行optrA基因环境分析显示,optrA基因环境在产气荚膜梭菌菌株2C45、QHY-2和21-D-5之间高度相似,均为ISVlu1-IS1216-erm(A)-optrA-IS1216-fexA-ISVlu1(图 4);表明携带耐药基因erm(A)、optrA和fexA的DNA区段可能作为一个可移动元件在产气荚膜梭菌菌株间进行传播。

|
图 4 菌株21-D-5和参考产气荚膜梭菌菌株optrA基因环境 Fig. 4 Genetic environment of optrA among 21-D-5 and the reference genomes |
关中奶山羊为陕西关中地区的乳用型山羊,具有适应性广、耐粗饲、繁殖率高、适于放牧或舍饲、乳用性能好等优点。D型产气荚膜梭菌与多种动物的胃肠道疾病密切相关,尤其是绵羊和山羊的肠毒血症。本研究自6份腹泻关中奶山羊肛拭子样品中分离到5株D型产气荚膜梭菌,表明该场区关中奶山羊正在遭到D型产气荚膜梭菌侵袭。结合产气荚膜梭菌分离情况和羊伴有精神沉郁和腹泻症状、可初步判定此次奶山羊腹泻与D型产气荚膜梭菌相关;但由于未检测粪便中是否含有毒素ETX,无法确证关中奶山羊的腹泻是由分离到的D型产气荚膜梭菌菌株引起。
全基因组序列测定分析能够从基因水平阐述病原菌毒性和耐药性等特征,对细菌性疾病的精准防控和治疗具有参考意义。全基因组序列测定显示分离的D型产气荚膜梭菌基因组大小、编码基因数量和GC含量等特征稳定;并且分离株21-D-1/21-D-2和21-D-3/21-D-4基因组之间SNPs差异极小,表明分离株21-D-1/21-D-2和21-D-3/21-D-4可能分别属于两株不同的产气荚膜梭菌菌株、D型产气荚膜梭菌菌株可能在关中奶山羊之间具有一定程度的传播。
不同菌株间etx序列及基因环境比对显示,该基因在不同D型产气荚膜梭菌中的核苷酸序列相似度极高、拥有完全一致的氨基酸编码序列;且在不同D型产气荚膜梭菌中的基因环境基本一致,一定程度表明不同D型产气荚膜梭菌菌株产生的ETX毒性相近。插入序列(ISs)位于复合型转座子的末端,能够将基因组片段从供体细菌的基因组切割并将其连接到受体细菌的基因组当中,从而造成包括耐药基因和毒素基因在内的多种基因的传播[15]。本研究在毒素基因etx的上下游分别发现插入序列IS1151和ISCp1,表明etx可能通过IS1151和ISCp1的介导进行转移。除毒素基因etx外,分离株21-D-5中还检测到食源性致病毒素基因cpe,基因环境分析显示,其位于毒素基因etx下游,表明在菌株21-D-5中毒素基因etx和cpe能够位于同一毒素质粒。
动物源细菌耐药性的增强不仅给畜牧经济效益带来了较大的危害,对人类健康也具有一定威胁[16]。本研究中,耐药基因optrA和其他多种耐药基因均在产气荚膜梭菌分离株21-D-5中被检测到,耐药基因optrA基因通过编码一种ATP结合蛋白导致噁唑烷酮类抗生素的耐药性[14]。作为一类新型非兽用抗生素,噁唑烷酮类耐药基因在关中奶山羊源产气荚膜梭菌中的出现一定程度揭示了耐药基因optrA在人和动物源细菌之间的传播。通过对optrA基因环境的进一步分析,插入序列IS1216位于erm(A)-optrA的两侧,且插入序列ISVlu1位于erm(A)-optrA-fexA的两侧;进一步佐证了耐药基因optrA的传播,并揭示了optrA在不同菌株间可能以IS1216-erm(A)-optrA-IS1216和ISVlu1-IS1216-erm(A)-optrA-IS1216-fexA-ISVlu1这两种形式进行转移。合理使用抗生素是减少耐药菌数量、防止相应耐药基因传播的重要方法,但多种耐药基因的共同传播会造成多重耐药,对抗生素使用带来挑战。
本研究为国内首次对D型产气荚膜梭菌基因组进行序列测定分析,在描述D型产气荚膜梭菌基因组特征的基础上,通过对相应毒素基因及耐药基因的序列分析在基因水平揭示了其特征和传播的可能性。除此之外,该研究发现了同时携带肠毒血症关键毒素基因etx、人类食源性感染关键毒素基因cpe和多种耐药基因的D型产气荚膜梭菌菌株21-D-5。毒素基因cpe在引起人类食物中毒的产气荚膜梭菌菌株中普遍存在,达96.3%(105/109),与人类食源性疾病密切相关[17]。像21-D-5这种携带有多种重要毒素和耐药基因的产气荚膜梭菌菌株,其流行无疑会对公共卫生安全造成较大的威胁;在畜牧生产中应规范对抗生素的使用、加强饲养管理及环境消杀,以防止产气荚膜梭菌耐药性的增强及其在动物和人类之间的传播致病。
4 结论自腹泻奶山羊肛门拭子分离到5株D型产气荚膜梭菌(21-D-1~21-D-5),全基因组序列测定显示,5株菌基因组大小、GC含量和基因数量稳定;单核苷酸多态性(SNPs)分析显示,21-D-1和21-D-2以及21-D-3和21-D-4大概率属于相同的产气荚膜梭菌菌株。毒素基因检测表明,其均为携带etx的D型产气荚膜梭菌,且发现21-D-5同时携带肠毒血症关键毒素基因etx、人类食源性感染关键毒素基因cpe和多种耐药基因。
| [1] |
TIMBERMONT L, HAESEBROUCK F, DUCATELLE R, et al. Necrotic enteritis in broilers: an updated review on the pathogenesis[J]. Avian Pathol, 2011, 40(4): 341-347. DOI:10.1080/03079457.2011.590967 |
| [2] |
GOHARI I M, NAVARRO M A, LI J H, et al. Pathogenicity and virulence of Clostridium perfringens[J]. Virulence, 2021, 12(1): 723-753. DOI:10.1080/21505594.2021.1886777 |
| [3] |
吴克, 赵学亮, 冯航, 等. 甘肃省牛和羊源产气荚膜梭菌耐药性分析[J]. 动物医学进展, 2022, 43(1): 141-144. WU K, ZHAO X L, FENG H, et al. Antimicrobial susceptibility test of Clostridium perfringens from sheep and cattle in Gansu province[J]. Progress in Veterinary Medicine, 2022, 43(1): 141-144. DOI:10.3969/j.issn.1007-5038.2022.01.025 (in Chinese) |
| [4] |
KIU R, HALL L J. An update on the human and animal enteric pathogen Clostridium perfringens[J]. Emerg Microbes Infect, 2018, 7(1): 141. |
| [5] |
ROOD J I, ADAMS V, LACEY J, et al. Expansion of the Clostridium perfringens toxin-based typing scheme[J]. Anaerobe, 2018, 53: 5-10. DOI:10.1016/j.anaerobe.2018.04.011 |
| [6] |
LAYANA J E, MIYAKAWA M E F, UZAL F A. Evaluation of different fluids for detection of Clostridium perfringens type D epsilon toxin in sheep with experimental enterotoxemia[J]. Anaerobe, 2006, 12(4): 204-206. DOI:10.1016/j.anaerobe.2006.05.001 |
| [7] |
MIYATA S, MATSUSHITA O, MINAMI J, et al. Cleavage of a C-terminal peptide is essential for heptamerization of Clostridium perfringens ε-toxin in the synaptosomal membrane[J]. J Biol Chem, 2001, 276(17): 13778-13783. DOI:10.1074/jbc.M011527200 |
| [8] |
TAMAI E, ISHIDA T, MIYATA S, et al. Accumulation of Clostridium perfringens epsilon-toxin in the mouse kidney and its possible biological significance[J]. Infect Immun, 2003, 71(9): 5371-5375. DOI:10.1128/IAI.71.9.5371-5375.2003 |
| [9] |
POPOFF M R. Epsilon toxin: a fascinating pore-forming toxin[J]. FEBS J, 2011, 278(23): 4602-4615. DOI:10.1111/j.1742-4658.2011.08145.x |
| [10] |
UZAL F A. Diagnosis of Clostridium perfringens intestinal infections in sheep and goats[J]. Anaerobe, 2004, 10(2): 135-143. DOI:10.1016/j.anaerobe.2003.08.005 |
| [11] |
BRYNESTAD S, GRANUM P E. Clostridium perfringens and foodborne infections[J]. Int J Food Microbiol, 2002, 74(3): 195-202. DOI:10.1016/S0168-1605(01)00680-8 |
| [12] |
HAVELAAR A H, HAAGSMA J A, MANGEN M J J, et al. Disease burden of foodborne pathogens in the Netherlands, 2009[J]. Int J Food Microbiol, 2012, 156(3): 231-238. DOI:10.1016/j.ijfoodmicro.2012.03.029 |
| [13] |
WU K, FENG H, MA J G. Prevalence, toxin-typing and antimicrobial susceptibility of Clostridium perfringens in sheep with different feeding modes from Gansu and Qinghai provinces, China[J]. Anaerobe, 2022, 73: 102516. DOI:10.1016/j.anaerobe.2022.102516 |
| [14] |
ZHOU Y Q, LI J Y, SCHWARZ S, et al. Mobile oxazolidinone/phenicol resistance gene optrA in chicken Clostridium perfringens[J]. J Antimicrob Chemother, 2020, 75(10): 3067-3069. DOI:10.1093/jac/dkaa236 |
| [15] |
VAN OPIJNEN T, CAMILLI A. Transposon insertion sequencing: a new tool for systems-level analysis of microorganisms[J]. Nat Rev Microbiol, 2013, 11(7): 435-442. DOI:10.1038/nrmicro3033 |
| [16] |
张仕泓, 王少林. 动物源产气荚膜梭菌耐药性研究进展[J]. 畜牧兽医学报, 2021, 52(10): 2762-2771. ZHANG S H, WANG S L. Research progress on antimicrobial resistance of Clostridium perfringens of animal origins[J]. Acta Veterinaria et Zootechnica Sinica, 2021, 52(10): 2762-2771. DOI:10.11843/j.issn.0366-6964.2021.010.007 (in Chinese) |
| [17] |
KIU R, CAIM S, PAINSET A, et al. Phylogenomic analysis of gastroenteritis-associated Clostridium perfringens in England and Wales over a 7-year period indicates distribution of clonal toxigenic strains in multiple outbreaks and extensive involvement of enterotoxin-encoding (CPE) plasmids[J]. Microb Genom, 2019, 5(10): e000297. |
(编辑 白永平)



