2. 西南民族大学畜牧兽医学院, 成都 610041
2. College of Animal and Veterinary Sciences, Southwest Minzu University, Chengdu 610041, China
瘤胃是反刍动物特有的消化器官,从出生到成年,瘤胃结构与功能会发生巨大的变化。新生反刍动物摄食液体乳时,经食管沟直接进入皱胃消化,利用葡萄糖为机体供能[1-2];成年反刍动物瘤胃是营养物质消化吸收的主要场所,因此瘤胃充分发育且功能健全是维持机体健康和发挥生产性能的重要前提。瘤胃的发育对其进入反刍阶段极其关键,发育完全的瘤胃可为反刍动物提供储存食物空间,其发酵产生的挥发性脂肪酸(volatile fatty acid,VFA)可替代葡萄糖供能,VFA代谢可为机体提供70%~80%能量[3]。瘤胃上皮对不同VFA的吸收速率不一样,其中丁酸的吸收速率最高,丙酸次之,乙酸最小;约95%的丁酸在瘤胃上皮细胞内被代谢掉,主要为瘤胃壁的生长发育提供能量[4],丙酸与乙酸代谢主要为整个机体发育提供能量[2]。
VFA的代谢终产物为酮体与胆固醇,过多酮体转运到血液中会引发酮病[5],而细胞内胆固醇及其代谢物(类异戊二烯)积累过多时会增大膜通透性,进而引发炎症反应[6]。瘤胃VFA累积和生态失调会引发亚急性瘤胃酸中毒(subacute ruminal acidosis,SARA)[7]。因此,瘤胃VFA代谢与瘤胃及机体健康息息相关;瘤胃微生物释放脂多糖(lipopolysaccharides,LPS)和组胺等内毒素可诱发疾病,损害机体健康[8];SARA使奶牛的牛乳磷脂、乳清酸等营养成分降低,导致乳品质下降[9];瘤胃内的毒素若进入血液循环可抑制奶牛卵泡发育、成熟和排卵,甚至直接抑制胚胎的发育,影响其繁殖性能[10]。上述研究表明,瘤胃的健康与机体健康、产奶性能和繁殖性能密切相关。
转录组测序被广泛应用于瘤胃发育的分子机制研究。Kong等[11]对相同年龄段不同采食量肉牛瘤胃进行转录组测序分析,筛选出的差异表达基因和通路主要与免疫功能、炎症、凋亡、细胞生长/增殖、营养转运和代谢途径相关。Baldwin等[12]对14、42、56和70 d断奶前后的荷斯坦奶牛瘤胃进行转录组研究,发现断奶前后,荷斯坦奶牛瘤胃上皮从利用葡萄糖过渡到利用VFA为机体供能。Wang等[13]研究发现,日粮的营养素和颗粒大小会影响参与瘤胃上皮细胞增殖/凋亡过程和补体复合体的基因表达。崔占鸿[14]通过转录组研究相同年龄段不同饲养方式下牦牛瘤胃发育情况,发现放牧牦牛瘤胃上皮乳头发育缓慢和瘤胃炎症基因表达量较高;代乳+补饲组有利于免疫细胞分化和改善免疫功能,有助于牦牛牛犊瘤胃健康发育。Li等[15]通过转录组研究哺乳与断奶期奶牛瘤胃酸中毒对瘤胃发育的影响,富集到细胞信号转导和形态发生等通路,表明瘤胃酸中毒会影响瘤胃上皮发育。Mackey[16]通过转录组研究发现,在SARA奶牛瘤胃中同源细胞黏附的分子途径上调,这表明瘤胃黏膜组织中存在结构性反应,以对抗炎症反应导致的通透性屏障减弱。
牦牛(Bos grunniens)是一种大型反刍家畜,全球95%的牦牛分布在我国青海、西藏、四川、甘肃、云南及新疆等地。牦牛作为一种全能经济家畜,为当地农牧民提供肉、皮、毛、乳、牛粪等生活和生产资料[17]。牛属动物瘤胃转录组研究主要集中在肉牛和奶牛,牦牛相关研究较少。不同年龄段牦牛瘤胃转录组研究鲜有报道。鉴于此,本研究依据瘤胃组织形态发育的3个阶段:非反刍阶段(0~3周)、过渡阶段(3~8周)和反刍阶段(8周后)[18],选取1日龄、20日龄、60日龄、15月龄和3岁龄的放牧牦牛作为研究对象,对瘤胃组织进行转录组分析,结合组织形态学鉴定对不同发育阶段牦牛瘤胃进行研究,以期为提高牦牛的生产性能提供理论参考与新思路。
1 材料与方法 1.1 样品采集与处理四川省阿坝藏族羌族自治州红原县牦牛培育方式多采用自然放牧,即出生后随母哺乳,并自由采食牧草与饮水。本试验选取非反刍阶段(1日龄与20日龄)、过渡阶段(60日龄)和反刍阶段(15月龄与3岁龄)的健康牦牛作为研究对象,共5个年龄组,每组3头。用灭菌剪刀取瘤胃1 cm×1 cm上皮及皮下组织块,DEPC水冲掉附着的杂质后迅速置于液氮中保存;取1.5 cm×1.5 cm瘤胃上皮及皮下组织块放入4%多聚甲醛固定液中固定,用于瘤胃组织形态观察。
1.2 瘤胃指数测量准确测量样本体重,屠宰后取出复胃,将瘤胃、瓣胃、网胃和皱胃上相连部位结扎并将4个胃室分开,解剖发现刚出生的1日龄组瘤胃无固体内容物;20日龄组瘤胃中有极少量牧草和大量奶块;60日龄组瘤胃中有部分牧草和大量奶块;15月龄与3岁组瘤胃中只有牧草。清空胃室内容物,准确测量瘤胃重量并记录。瘤胃指数为瘤胃重(g)/体重(kg),利用SPSS23.0软件对瘤胃重量和瘤胃指数进行分析,采用单因素方差分析(ANOVA),差异显著性使用Duncan’s法进行多重比较(LSD),定义P<0.05为差异显著,P<0.01为差异极显著,数据以“平均数±标准差(Mean±SD)”表示。
1.3 瘤胃组织切片制作及指标测量取4%多聚甲醛固定48 h的瘤胃组织,进行脱水、透明、浸蜡和包埋处理,在切片机上切成厚约5 μm的切片,进一步粘片、脱蜡至水和苏木素伊红(hematoxylin eosin,H.E)染色5 min,最后用中性树胶固定,用显微镜对染色切片进行观察并拍照记录。用DM750型光学显微镜在40倍下观测瘤胃的乳头数量与肌层厚度,采用case viewer图像分析软件进行测量。
1.4 RNA提取与cDNA文库构建使用mirVanaTM miRNA ISOlation Kit(ambion- 1561)试剂盒提取15个牦牛瘤胃组织总RNA,使用nanodrop-2000核酸蛋白测定仪(thermo fisher scientific)测定总RNA的浓度、纯度(OD260 nm/OD280 nm)和RIN值。RNA提取后经1%琼脂糖凝胶电泳检测,质检合格的RNA放入-80 ℃超低温冰箱中保存备用。
使用I llmina®Tru SeqTM RNA样品制备试剂盒构建cDNA文库。主要步骤包括:oligo(dT)的磁珠富集mRNA并将mRNA打断成短片段。以打断后的mRNA为模板,用六碱基随机引物合成一链cDNA,合成双链cDNA后使用试剂盒纯化双链cDNA;末端修复纯化的双链cDNA、加A尾并连接测序接头,然后进行片段大小选择,最后进行PCR扩增;构建好的文库用agilent 2100 bioanalyzer检测文库大小、浓度及质量,得到合格cDNA文库。
1.5 转录组测序与数据处理利用Illumina二代高通测序平台(Illumina HiSeq X ten),采用PE150测序方式对文库进行测序,获得原始序列信息。利用trimmomatic软件[19]对原始序列数据(raw reads)进行质控,去除带接头(adaptor)和低质量的reads,过滤掉低质量碱基以及N碱基得到有效数据(clean reads),统计clean reads的Q30、GC含量。最后用Hisat2[20](版本:2.2.1,http://ccb.jhu.edu/software/hisat2)将clean reads与牦牛参考基因组(GCF_000298355.1)进行序列比对。
1.6 主成分分析使用HTSeq-count[21]软件获取比对到蛋白编码基因上的reads数,cufflinks[22]软件计算蛋白编码基因的表达量FPKM值,用R语言绘制主成分分析(principal component analysis,PCA)二维图。
1.7 差异表达基因筛选使用HTSeq-count[21]软件获取每个样本中比对到蛋白编码基因上的reads数,使用DESeq (2012) R package[23]的estimateSizeFactors函数对其进行标准化处理,使用nbinomTest函数计算差异显著性(P-value)和差异倍数(Fold Change),将P < 0.05,log2(Fold Change)>2作为筛选差异表达基因(DEGs)的标准。
1.8 差异表达基因GO与KEGG富集分析以1日龄样本为参照,利用GOSeq对20日龄、60日龄、15月龄和3岁组牦牛瘤胃DEGs进行GO富集分析,基于GO数据库功能,将4个年龄段的DEGs按照生物过程(biological process,BP)、细胞组分(cellular component,CC)和分子功能(molecular function,MF)进行分类,P<0.05为标准筛选显著富集的GO条目和KEGG[24]信号通路。在显著性通路中筛选与放牧牦牛瘤胃发育相关的基因。
1.9 实时荧光定量PCR验证选取关键差异表达基因,利用Primer BLAST网站设计引物(表 1)。将样品测序返回的RNA通过TaKaRa逆转录试剂盒反转为cDNA,以gapdh 作为内参基因,通过Quant Studio 5仪器进行实时荧光定量PCR,采用2-△△ct法分析数据,验证测序结果。
|
|
表 1 引物信息 Table 1 Primer sequence information |
1、20和60日龄牦牛的瘤胃重量与瘤胃指数差异不显著(P>0.05),15月龄与3岁牦牛的瘤胃重量与瘤胃指数显著增加(P<0.05,图 1)。瘤胃重量与指数增长趋势一致,非反刍阶段至过渡阶段几乎无变化,反刍阶段显著增长。
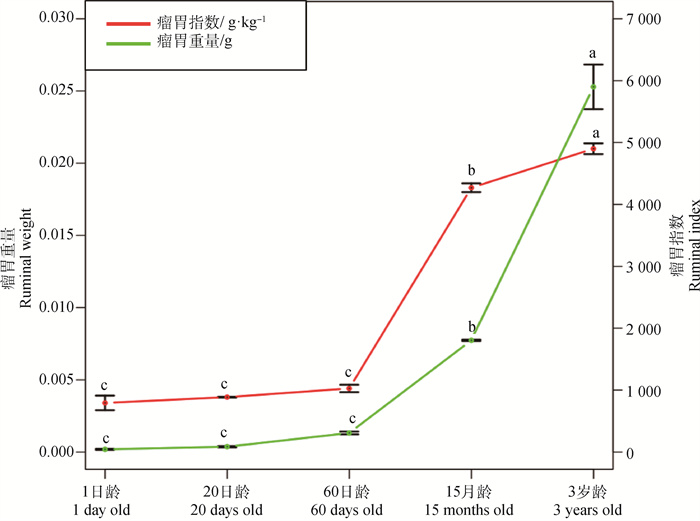
|
横坐标表示年龄,N=3/组;纵坐标(左)表示瘤胃重量,纵坐标(右)表示瘤胃指数 The abscissa represents the age, N=3/group, the ordinate on the left side represents the ruminal weight, and the right side represents the ruminal index 图 1 不同年龄组放牧牦牛瘤胃重量与指数分析 Fig. 1 Analysis of ruminal weight and index of grazing yak at different ages |
组织切片H.E染色结果显示(图 2),1日龄和20日龄,瘤胃肌纤维排列密集,乳头排列紧密,相较于1日龄,20日龄的瘤胃高度和宽度有所增加,但不明显;60日龄的瘤胃肌纤维出现间隔,乳头高度和宽度明显增加;15月龄和3岁牦牛的瘤胃的肌纤维间隙明显,乳头高度与宽度进一步增加。统计结果(表 2)显示,牦牛瘤胃的肌层厚度随着日龄的增加显著增加(P < 0.05);1日龄和20日龄牦牛的瘤胃乳头高度和宽度均差异不显著(P>0.05),20日龄后,瘤胃乳头高度和宽度随着日龄的增加显著增加(P < 0.05)。切片形态变化与数据统计结果基本一致。

|
A-E. 1日龄、20日龄、60日龄、15月龄和3岁龄 A-E. 1 day old, 20 days old, 60 days old, 15 months old and 3 years old, respectively 图 2 不同年龄组放牧牦牛瘤胃上皮H.E染色切片 Fig. 2 H.E staining sections of rumen epithelium of grazing yaks at different ages |
|
|
表 2 不同年龄放牧牦牛瘤胃上皮的形态结构(Mean±SD) Table 2 The morphological structure of rumen epithelium of grazing yaks in different ages (Mean±SD) |
总RNA浓度均大于100 μg·μL-1,RIN值大于7,纯度(OD260 nm/OD280 nm)均在2.0~2.2之间,28S/18S核糖体RNA条带清晰且≥0.7,表明提取的RNA完整性较好,RNA质量检验合格,可用于后续试验分析。Agilent 2100 bioanalyzer检测文库的片段大小符合预期;文库有效浓度大于4 nmol·L-1,结果表明高质量cDNA文库构建成功。
15个瘤胃组织样本得到720.41 Mb的clean reads,其中Q30百分比均在90%以上,GC含量在50%左右,比对到参考基因组的reads与clean reads的比率均在90%左右,表明测序质量高,数据准确可靠,所选参考基因组的组装满足分析要求。
2.4 瘤胃基因表达水平分析PCA图结果显示(图 3),1日龄、20日龄、60日龄、15月龄和3岁牦牛的瘤胃基因表达水平组间独立分散,形成非反刍阶段(1日龄和20日龄)、过渡阶段(60日龄)和反刍阶段(15月龄和3岁)3个独立子群。
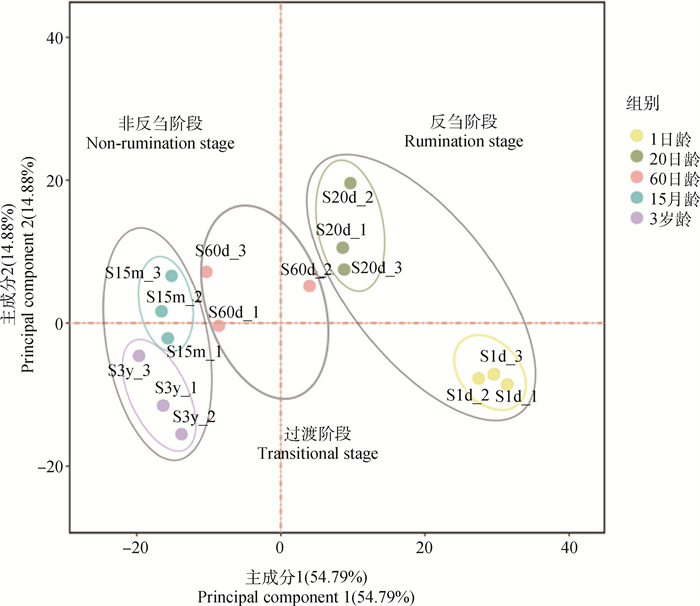
|
图 3 不同年龄放牧牦牛瘤胃PCA图 Fig. 3 PCA diagram of rumen of grazing yaks at different ages |
以1日龄组为对照,在20日龄、60日龄、15月龄和3岁组中分别鉴定到1 462、2 146、3 454和3 003个DEGs,去掉重复后共获4 635个DEGs(图 4A);20日龄、60日龄、15月龄和3岁组特有DEGs分别为163、298、688和511个,4个年龄组共有基因782个,包括SLC26A3、PPARG、HMGCS2等基因。差异基因聚类分析结果显示,20与60日龄组差异基因表达谱相近,15月龄与3岁组差异基因表达谱相近,提示20日龄与60日龄组发育情况相近,15月龄与3岁组发育情况相近(图 4B)。
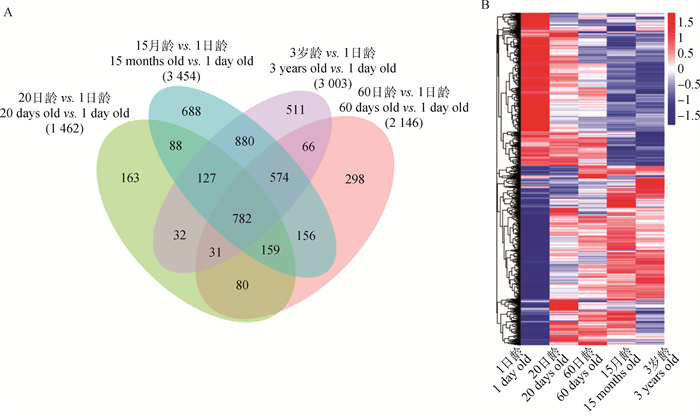
|
A.不同年龄放牧牦牛瘤胃差异表达基因韦恩图;B. 不同年龄放牧牦牛瘤胃差异表达基因表达量层次类聚图 A. The Venn map of differentially expressed genes in rumen of grazing yaks at different ages; B. Hierarchical clustering of differentially expressed genes in rumen of grazing yaks at different ages 图 4 差异表达基因韦恩图与热图 Fig. 4 Venn map and heat map of differentially expressed genes |
对4个年龄组的DEGs进行GO富集分析,在20日龄、60日龄、15月龄和3岁组中分别有2 501、2 891、3 565和3 219条显著条目。按P值排序筛选BP、CC和MF三大类的前10条显著性GO条目(图 5A、B、C)。结果表明,不同年龄组的前10条显著性GO条目中有共有的,也有各年龄阶段特有的。如在生物过程中,20日龄和15月龄富集到胆固醇生物合成显著性条目;60日龄组富集到脂肪酸代谢显著性条目;15月龄和3岁组富集到脂肪酸β-氧化显著性条目。
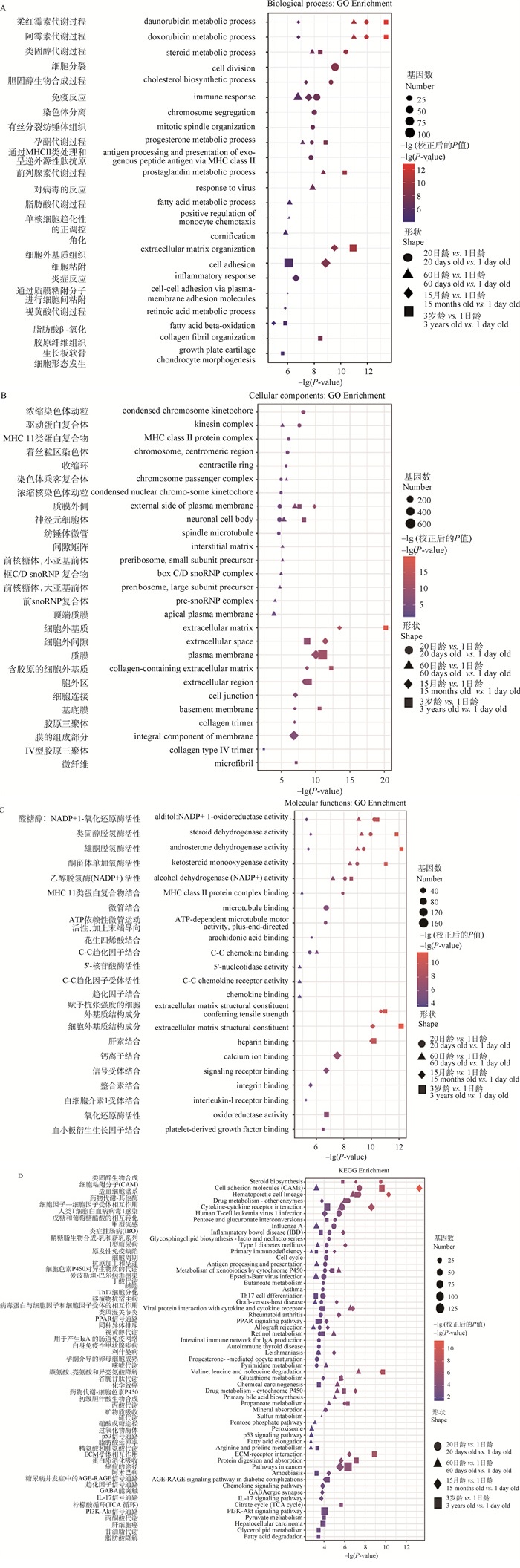
|
A-C.分别指4个年龄组DEGs在BP、CC、MF三大类中前10个GO条目;D. 4个年龄组DEGs富集的前30条KEGG通路。气泡大小表示DEGs的数量,气泡颜色表示错误发现率(false discovery rate, FDR P-value),横轴为-lg(P-value)值,纵轴显示GO条目或者KEGG通路 A-C. Indicates the top 10 GO items in BP, CC and MF categories of DEGs in 4 age groups; D. The top 30 KEGG pathways enriched by DEGs in the 4 age groups. The size of the bubble indicates the number of DEGs, the color the bubble indicates the false discovery rate (FDR P-value), the abscissa indicates the -lg(P-value), and ordinate indicates the GO items or KEGG pathways 图 5 差异表达基因的GO与KEGG富集气泡图 Fig. 5 Bubble charts of GO and KEGG enrichment of differentially expressed genes |
分别对4个年龄组的差异表达基因进行KEGG功能富集分析,在20日龄、60日龄、15月龄和3岁组中分别有86、89、97和99条显著通路。按P值排序筛选4个年龄组的前30条显著性KEGG通路(图 5D)。结果显示,每个年龄组前30条显著通路同样存在共有的,也有各年龄阶段特有。如20日龄组富集到丁酸代谢通路;60日龄、15月龄、3岁组富集到丙酸代谢通路;4个年龄组共有的P450对外源性物质的代谢和细胞因子-细胞因子受体相互作用途径。
2.7 瘤胃发育候选基因筛选本研究富集12条到参与瘤胃发育的通路,筛选到36个候选基因(表 3)。其中HMGCS2、SLC26A3、PPARG和PPARD与瘤胃营养吸收和代谢相关的重要候选基因,CCL5研究相对较少。
|
|
表 3 参与瘤胃VFA吸收与代谢的候选通路与基因 Table 3 The candidate pathways and genes involved in ruminal VFA absorption and metabolism |
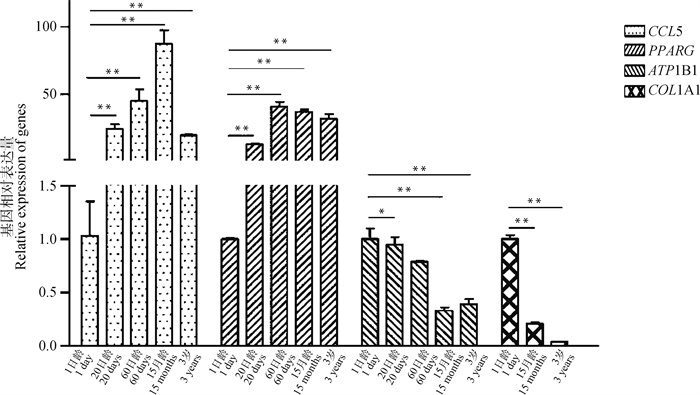
验证转录组测序数据结果显示(图 6),以1日龄为对照,CCL5和PPARG基因在20日龄、60日龄、15月龄和3岁组显著上调;ATP1B1在20日龄、60日龄、15月龄和3岁组显著下调;COL1A1在15月龄和3岁组显著下调。上述所挑选的差异表达基因的RT-qPCR变化规律与转录组测序数据表达规律一致,进一步表明所分析的数据准确可信。
|
图 6 差异表达基因的实时荧光定量PCR验证 Fig. 6 RT-qPCR verification of DEGs |
牦牛瘤胃发育与其机体健康和生产性能的发挥紧密相关,引起了广泛的关注,但目前牦牛瘤胃发育机理尚不清楚,还需进一步研究相关分子机制,为牦牛生产提供理论参考和新思路。瘤胃发育涉及组织形态以及代谢等多个层面的变化,研究表明瘤胃上皮乳头高度与宽度、肌层厚度是衡量瘤胃发育程度的重要指标[14],上皮乳头可以增加瘤胃上皮的吸收表面积[25];瘤胃肌层厚度的增加,在瘤胃容积和重量的扩增、蠕动性增强方面发挥积极作用[26]。本研究中,随着年龄增长,牦牛瘤胃重量与指数增长速率增加,提示随着年龄增加瘤胃发挥更重要的作用。20日龄、60日龄、15月龄和3岁组牦牛瘤胃中均有内容物;组织切片结果显示,乳头与肌层发育良好,肌层厚度显著增加(P < 0.05),20日龄后瘤胃乳头高度与宽度随着年龄增加而显著增加(P < 0.05),肌层厚度发育较乳头发育更早,而张科[27]研究表明,28日龄绒山羊瘤胃乳头发育比肌层厚度发育更早。原因在于牦牛以放牧方式培育,开食较早,在20日龄时已经开食牧草,因此牦牛瘤胃肌层厚度发育更早;另外可能由于物种特异性,牦牛瘤胃乳头发育更晚。
研究表明,粗饲料对瘤胃肌层厚度起到物理刺激作用[28],牧草进入瘤胃,经瘤胃微生物发酵产生VFA,可刺激瘤胃乳头发育[29]。本研究中GO富集分析显示,20日龄与15月龄DEGs富集到胆固醇生物合成通路,该通路参与瘤胃上皮细胞胆固醇平衡,而瘤胃上皮细胞胆固醇浓度与VFA代谢相关[30],推测以放牧方式培育的牦牛在20日龄时可能已经开始采食牧草,并经瘤胃微生物发酵产生VFA,此时瘤胃可能己经开始进行VFA吸收与代谢。60日龄DEGs富集到脂肪酸代谢,15月龄和3岁DEGs富集到β脂肪酸-氧化,研究表明这些通路与瘤胃反刍阶段转变为以VFA氧化供能相关[12],提示60日龄时牦牛瘤胃可能转变为以VFA代谢供能。本研究中KEGG富集结果显示,20日龄的DEGs富集到丁酸代谢通路与PPAR信号通路,已有研究报道丁酸可促进瘤胃上皮细胞的有丝分裂与增殖,进而促进瘤胃乳头生长发育[31],PPAR信号通路可能通过调控VFA代谢影响瘤胃发育[32],进一步说明20日龄牦牛瘤胃开始进行VFA代谢,以促进瘤胃快速发育,与GO分析结果一致。60日龄、15月龄和3岁组DEGs富集到丙酸代谢通路,3岁组DEGs富集到丙酮酸代谢通路,丙酸是反刍动物瘤胃中主要的VFA之一,最后转化为糖类为机体供能[33],丙酮酸是糖、蛋白质、脂肪三大营养物质代谢的重要枢纽[34],因此推测丙酸代谢通路和丙酮酸代谢通路可能参与调控60日龄牦牛瘤胃发育;60日龄和15月龄组DEGs富集到矿物质吸收通路,矿物质是细胞的重要组成部分,参与营养吸收代谢,与VFA吸收相关[32]。上述结果表明,20日龄放牧牦牛瘤胃可能开始VFA代谢,通过VFA吸收与代谢相关通路调控瘤胃快速发育。
GO富集分析显示,20日龄、60日龄和15月龄组DEGs富集到免疫反应生物过程,15月龄和3岁组DEGs富集到与LPS刺激瘤胃上皮细胞迁移相关的细胞粘连生物过程[35]。KEGG富集结果显示,20日龄、60日龄、15月龄和3岁组DEGs均显著富集到细胞因子-细胞因子受体相互作用通路,3岁组DEGs富集到PI3K-Akt信号通路,与LPS刺激瘤胃上皮细胞迁移相关[35];20日龄、15月龄和3岁组DEGs富集到Th17细胞分化通路,该通路与免疫细胞分化与改善免疫功能相关[14]。20日龄、60日龄、15月龄和3岁组DEGs显著富集到细胞色素P450外源性物质的代谢途径,20日龄、15月龄和3岁组DEGs显著富集到类固醇生物合成途径,均致力于消除瘤胃上皮细胞内的饮食副产物、外源性物质和环境污染物,表明上皮可能受到来自内腔的大量异源抗原的侵袭[36];15月龄和3岁组DEGs富集到ECM受体相互作用途径,分布在细胞间的胶原蛋白、丝蛋白和纤维连接蛋白等生物大分子,维持瘤胃上皮的完整性和屏障功能[37]。提示可能由于高原环境污染,牦牛瘤胃上皮受到侵袭;而20日龄牦牛瘤胃开始通过调控免疫和上皮维护相关通路促进瘤胃健康发育。
在上述通路中筛选到HMGCS2、SLC26A3、PPARG、PPARD和CCL5等与瘤胃营养吸收、代谢和免疫相关的候选基因。3-羟基3-甲基戊二酰辅酶A合成酶2(3-hydroxy-3-methylglutaryl-CoA synthase 2,HMGCS2)是瘤胃上皮细胞酮体合成的限速酶,调控VFA代谢[38-39],其表达受过氧化物酶体增殖物激活受体(peroxisome proliferators-activated receptors,PPARs)的调控[40]。溶质载体家族26成员3(solute carrier family 26 member 3,SLC26A3)编码的蛋白质是一种跨膜糖蛋白,通过细胞膜运输氯离子以交换碳酸氢根离子,参与VFA/HCO3交换,对瘤胃VFA吸收起作用[41]。过氧化物酶体增殖物激活受体γ(PPARG)和过氧化物酶体增殖物激活受体δ(PPARD)均属于PPARs家族,PPARG调控瘤胃内细胞分化,影响瘤胃发育[42];PPARD在瘤胃上皮细胞的生长和分化中起作用,是瘤胃上皮发育的重要调控因子[43]。C-C基序趋化因子配体5(C-C motif chemokine ligand 5,CCL5)是CC亚家族趋化因子基因,具有调节正常T细胞表达和分泌的功能,促进炎性细胞因子的释放,参与免疫调节和炎症过程[44]。研究表明,CCL5分布在瘤胃上皮的基底和棘突层,CCL5可能是瘤胃免疫系统关键角色之一,在瘤胃内环境稳定中发挥重要作用[45]。
4 结论本研究中,牦牛瘤胃形态结构发育较早,20日龄是其发育的重要临界点。牦牛瘤胃肌层厚度发育相较于瘤胃乳头更早,肌层厚度从20日龄快速增加,瘤胃乳头从20日龄后快速发育;20日龄开始VFA代谢,促进瘤胃快速健康地发育。本研究为牦牛瘤胃发育机理研究提供理论参考,同时为牦牛生产提供新思路。但与瘤胃营养吸收、代谢和免疫相关的候选基因HMGCS2、SLC26A3、PPARG、PPARD和CCL5等未进行验证,后期可以在细胞水平进行基因的功能验证,以及结合多组学研究,探究牦牛瘤胃发育的调控网络。
| [1] |
BALDWIN VI R L, MCLEOD K R, KLOTZ J L, et al. Rumen development, intestinal growth and hepatic metabolism in the pre- and postweaning ruminant[J]. J Dairy Sci, 2004, 87 Suppl 1: E55-E65. |
| [2] |
PARSONS S D, STEELE M A, LESLIE K E, et al. Effect of a milk byproduct-based calf starter feed on dairy calf nutrient consumption, rumen development, and performance when fed different milk levels[J]. J Dairy Sci, 2022, 105(1): 281-300. DOI:10.3168/jds.2021-21018 |
| [3] |
田发益, 武俊喜. 放牧与舍饲对彭波半细毛羊瘤胃细菌群落的生物学信息影响[J]. 畜牧兽医学报, 2019, 50(11): 2252-2263. TIAN F Y, WU J X. Effects of grazing and barn feeding on biological information of rumen bacterial communities in Pengbo semi-fine wool sheep[J]. Acta Veterinaria et Zootechnica Sinica, 2019, 50(11): 2252-2263. DOI:10.11843/j.issn.0366-6964.2019.11.009 (in Chinese) |
| [4] |
SEHESTED J, DIERNÆS L, MØLLER P D, et al. Ruminal transport and metabolism of short-chain fatty acids (SCFA) in vitro: effect of SCFA chain length and pH[J]. Comp Biochem Physiol Part A: Mol Integr, 1999, 123(4): 359-368. DOI:10.1016/S1095-6433(99)00074-4 |
| [5] |
孙玲伟, 包凯, 李影, 等. 奶牛临床和亚临床酮病的血浆代谢组学研究[J]. 中国农业科学, 2014, 47(8): 1588-1599. SUN L W, BAO K, LI Y, et al. Plasma metabolomics study of dairy cows with clinical and subclinical ketosis[J]. Scientia Agricultura Sinica, 2014, 47(8): 1588-1599. DOI:10.3864/j.issn.0578-1752.2014.08.015 (in Chinese) |
| [6] |
ONTSOUKA E C, ALBRECHT C. Cholesterol transport and regulation in the mammary gland[J]. J Mammary Gland Biol Neoplasia, 2014, 19(1): 43-58. DOI:10.1007/s10911-014-9316-x |
| [7] |
韩郭皓, 高晓莎, 段晋伟, 等. 酵母对亚急性瘤胃酸中毒绵羊缓解效果及作用机制研究[J]. 畜牧兽医学报, 2022, 53(5): 1475-1485. HAN G H, GAO X S, DUAN J W, et al. Study on relieving effect and mechanism of yeast on subacute ruminal acidosis in sheep[J]. Acta Veterinaria et Zootechnica Sinica, 2022, 53(5): 1475-1485. (in Chinese) |
| [8] |
胡新旭, 卞巧, 张善鹏, 等. 奶牛瘤胃健康、机体健康和繁殖性能的关系[J]. 湖南饲料, 2021(3): 31-34. HU X X, BIAN Q, ZHANG S P, et al. The relationship between rumen health, body health and reproductive performance in dairy cows[J]. Hunan Feed, 2021(3): 4. (in Chinese) |
| [9] |
张涛, 牟英玉, 亓王盼, 等. 亚急性瘤胃酸中毒耐受性不同的奶牛血浆和乳中脂肪酸及代谢物组成分析[J]. 草业学报, 2021, 30(7): 101-110. ZHANG T, MU Y Y, QI W P, et al. Analysis of plasma and milk fatty acid and metabolite composition in lactating dairy cows with differing tolerance to subacute ruminal acidosis[J]. Acta Prataculturae Sinica, 2021, 30(7): 101-110. (in Chinese) |
| [10] |
施振旦, 李孝伟, 田允波, 等. 奶牛繁殖杀手——内毒素[J]. 中国奶牛, 2007(3): 29-32. SHI Z D, LI X W, TIAN Y B, et al. Slayer of dairy cow's reproductive performance: endotoxin[J]. China Dairy Cattle, 2007(3): 29-32. (in Chinese) |
| [11] |
KONG R S G, LIANG G X, CHEN Y H, et al. Transcriptome profiling of the rumen epithelium of beef cattle differing in residual feed intake[J]. BMC Genomics, 2016, 17: 592. DOI:10.1186/s12864-016-2935-4 |
| [12] |
BALDWIN VI R L, LIU M, CONNOR E E, et al. Transcriptional reprogramming in rumen epithelium during the developmental transition of pre-ruminant to the ruminant in cattle[J]. Animals, 2021, 11(10): 2870. DOI:10.3390/ani11102870 |
| [13] |
WANG B, WANG D M, WU X H, et al. Effects of dietary physical or nutritional factors on morphology of rumen papillae and transcriptome changes in lactating dairy cows based on three different forage-based diets[J]. BMC Genomics, 2017, 18(1): 353. DOI:10.1186/s12864-017-3726-2 |
| [14] |
崔占鸿. 牦牛犊牛培育方式对生长和消化道发育的影响[D]. 杨凌: 西北农林科技大学, 2020. CUI Z H. Effects of rearing patterns on growth and digstive tract development in yak calves[D]. Yangling: Northwest A&F University, 2020. (in Chinese) |
| [15] |
LI W L, GELSINGER S, EDWARDS A, et al. Transcriptome analysis of rumen epithelium and meta-transcriptome analysis of rumen epimural microbial community in young calves with feed induced acidosis[J]. Sci Rep, 2019, 9(1): 4744. DOI:10.1038/s41598-019-40375-2 |
| [16] |
MACKEY E. Effects of ruminal acidosis on rumen papillae transcriptome[D]. Newark: University of Delaware, 2013.
|
| [17] |
吕小康, 王杰, 刁其玉, 等. 瘤胃上皮细胞增殖和物质转运分子机制的研究进展[J]. 动物营养学报, 2017, 29(8): 2657-2664. LYU X K, WANG J, DIAO Q Y, et al. Molecular mechanisms of ruminal epithelial cell proliferation and substance transportation[J]. Chinese Journal of Animal Nutrition, 2017, 29(8): 2657-2664. (in Chinese) |
| [18] |
张剑搏, 丁考仁青, 梁泽毅, 等. 早期营养干预对幼龄反刍动物瘤胃微生物区系发育的影响[J]. 草业学报, 2021, 30(2): 199-211. ZHANG J B, DING K R Q, LIANG Z Y, et al. Effect of early nutrition intervention on rumen microflora development in young ruminants[J]. Acta Prataculturae Sinica, 2021, 30(2): 199-211. (in Chinese) |
| [19] |
BOLGER A M, LOHSE M, USADEL B. Trimmomatic: a flexible trimmer for Illumina sequence data[J]. Bioinformatics, 2014, 30(15): 2114-2120. DOI:10.1093/bioinformatics/btu170 |
| [20] |
KIM D, LANGMEAD B, SALZBERG S L. HISAT: a fast spliced aligner with low memory requirements[J]. Nat Methods, 2015, 12(4): 357-360. DOI:10.1038/nmeth.3317 |
| [21] |
ANDERS S, PYL P T, HUBER W. HTSeq--a Python framework to work with high-throughput sequencing data[J]. Bioinformatics, 2015, 31(2): 166-169. DOI:10.1093/bioinformatics/btu638 |
| [22] |
TRAPNELL C, WILLIAMS B A, PERTEA G, et al. Transcript assembly and quantification by RNA-Seq reveals unannotated transcripts and isoform switching during cell differentiation[J]. Nat Biotechnol, 2010, 28(5): 511-555. DOI:10.1038/nbt.1621 |
| [23] |
ANDERS S, HUBER W. Differential expression of RNA-Seq data at the gene level-the DESeq package[R]. Heidelberg, Germany: European Molecular Biology Laboratory (EMBL), 2012.
|
| [24] |
KANEHISA M, ARAKI M, GOTO S, et al. KEGG for linking genomes to life and the environment[J]. Nucleic Acids Res, 2008, 36: D480-D484. |
| [25] |
GRAHAM C, SIMMONS N L. Functional organization of the bovine rumen epithelium[J]. Am J Physiol Regul Integr Comp Physiol, 2005, 288(1): R173-R181. |
| [26] |
王立斌. 在饲喂开食料的基础上补饲苜蓿对犊牛胃肠道发育的影响[D]. 北京: 中国农业大学, 2013. WANG L B. Influence of supplementary feeding of alfalfa on the gastrointestinal tract development of calves feeding on milk and starter[D]. Beijing: China Agricultural University, 2013. (in Chinese) |
| [27] |
张科. 陕北白绒山羊0~56日龄羔羊胃肠道发育及瘤胃微生物区系研究[D]. 杨凌: 西北农林科技大学, 2017. ZHANG K. Researchon Gi development and rumen microbiome from 0 to 56-day-old at cashmere goat[D]. Yangling: Northwest A and F University, 2017. (in Chinese) |
| [28] |
NOROUZIAN M A, VALIZADEH R. Effect of forage inclusion and particle size in diets of neonatal lambs on performance and rumen development[J]. J Anim Physiol Anim Nutr, 2014, 98(6): 1095-1101. |
| [29] |
RAGIONIERI L, CACCHIOLI A, RAVANETTI F, et al. Effect of the supplementation with a blend containing short and medium chain fatty acid monoglycerides in milk replacer on rumen papillae development in weaning calves[J]. Ann Anat-Anat Anz, 2016, 207: 97-108. |
| [30] |
STEELE M A, VANDERVOORT G, ALZAHAL O, et al. Rumen epithelial adaptation to high-grain diets involves the coordinated regulation of genes involved in cholesterol homeostasis[J]. Physiol Genomics, 2011, 43(6): 308-316. |
| [31] |
MALHI M, GUI H B, YAO L, et al. Increased papillae growth and enhanced short-chain fatty acid absorption in the rumen of goats are associated with transient increases in cyclin D1 expression after ruminal butyrate infusion[J]. J Dairy Sci, 2013, 96(12): 7603-7616. |
| [32] |
SUN D M, YIN Y Y, GUO C Z, et al. Transcriptomic analysis reveals the molecular mechanisms of rumen wall morphological and functional development induced by different solid diet introduction in a lamb model[J]. J Anim Sci Biotechnol, 2021, 12(1): 33. |
| [33] |
LIN L M, XIE F, SUN D M, et al. Ruminal microbiome-host crosstalk stimulates the development of the ruminal epithelium in a lamb model[J]. Microbiome, 2019, 7(1): 83. |
| [34] |
SCHORMANN N, HAYDEN K L, LEE P, et al. An overview of structure, function, and regulation of pyruvate kinases[J]. Protein Sci, 2019, 28(10): 1771-1784. |
| [35] |
YANG C L, ZHU B N, YE S J, et al. Isomer-specific effects of cis-9, trans-11-and trans-10, cis-12-CLA on immune regulation in ruminal epithelial cells[J]. Animals, 2021, 11(4): 1169. |
| [36] |
HU R, ZOU H W, WANG Z S, et al. Nutritional interventions improved rumen functions and promoted compensatory growth of growth-retarded yaks as revealed by integrated transcripts and microbiome analyses[J]. Front Microbiol, 2019, 10: 318. |
| [37] |
MCBRIDE B W, ALZAHAL O, STEELE M A, et al. Adaptation to high grain diets proceeds through minimal immune system stimulation and differences in extracellular matrix protein expression in a model of subacute ruminal acidosis in non-lactating dairy cows[J]. Am J Anim Vet Sci, 2012, 7(2): 84-91. |
| [38] |
高景, 齐智利. 瘤胃上皮短链脂肪酸的吸收和代谢[J]. 动物营养学报, 2018, 30(4): 1271-1278. GAO J, QI Z L. Absorption and metabolism of short chain fatty acids in ruminal epithelium[J]. Chinese Journal of Animal Nutrition, 2018, 30(4): 1271-1278. (in Chinese) |
| [39] |
庄一民, 刁其玉, 张乃锋. 幼龄反刍动物瘤胃上皮细胞β-羟基丁酸代谢与调控机制[J]. 畜牧兽医学报, 2020, 51(4): 660-669. ZHUANG Y M, DIAO Q Y, ZHANG N F. Metabolism and regulation mechanism of beta-hydroxybutyric acid in ruminal epithelium cells of young ruminants[J]. Acta Veterinaria et Zootechnica Sinica, 2020, 51(4): 660-669. (in Chinese) |
| [40] |
NAEEM A, DRACKLEY J K, STAMEY J, et al. Role of metabolic and cellular proliferation genes in ruminal development in response to enhanced plane of nutrition in neonatal Holstein calves[J]. J Dairy Sci, 2012, 95(4): 1807-1820. |
| [41] |
GÁLFI P, KUTAS F, NEOGRÁDY S. Immunohistochemical detection of bovine ruminal carbonic anhydrase isoenzyme[J]. Histochemistry, 1982, 74(4): 577-584. |
| [42] |
XUE M M, WANG K J, WANG A S, et al. MicroRNA sequencing reveals the effect of different levels of non-fibrous carbohydrate/neutral detergent fiber on rumen development in calves[J]. Animals, 2019, 9(8): 496. |
| [43] |
CONNOR E E, BALDWIN VI R L, WALKER M P, et al. Transcriptional regulators transforming growth factor-β1 and estrogen-related receptor-α identified as putative mediators of calf rumen epithelial tissue development and function during weaning[J]. J Dairy Sci, 2014, 97(7): 4193-4207. |
| [44] |
CHAN O, BURKE D J, GAO D F, et al. The chemokine CCL5 regulates glucose uptake and AMP kinase signaling in activated T cells to facilitate chemotaxis[J]. J Biol Chem, 2012, 287(35): 29406-29416. |
| [45] |
ROH S. The control of homeostasis in rumen epithelium of Japanese Black cattle[J]. J Anim Sci, 2018, 97(S3): 1. |
(编辑 郭云雁)



