2. 奥胡斯大学遗传与基因组中心,切勒 8830;
3. 北京奶牛中心,北京 100192
2. Center for Quantitative Genetics and Genomics, Aarhus University, Tjele 8830, Denmark;
3. Beijing Dairy Cattle Center, Beijing 100192, China
近年来,生长性状在奶牛遗传育种中受到新的关注[1]。在过去,人们对生长性状的研究主要集中在肉牛上,对奶牛生长性状认识不够充分,其测定目的多以辅助提高生产性状,改善牧场管理为主。但随着奶牛遗传育种工作的深入以及消费者需求的市场导向,人们逐渐意识到奶牛的生长对经济效益产生着诸多重要影响。首先,体重的变化可以直观地反映奶牛维持生存或生产的饲养需求[2]。能量负平衡会影响健康和繁殖,以及泌乳牛的生产性能。通过体重变化可以建立能量平衡模型[3],且与干物质采食量、甲烷排放等决定能量负平衡的因素相比,体重性状在牛场条件下更容易测量;其次,体重对饲料转化率有着重要影响。从经济利益最大化的角度来考虑,饲养奶牛时应尽可能投入最低的饲料成本,但使其具有最佳的产犊年龄,发挥最大的泌乳潜力。体重已被建议纳入育种目标或选择指数中。Pryce等[4]考虑生长性状与生产性状的最佳组合,将其应用到选择指数中,从而达到控制成本和提高饲料效率的目的。Spurlock等[5]评估了干物质采食量、体重、体况评分、能量校正乳和总饲料转化效率的遗传参数,建议将这些性状共同选择。以放牧为主要饲养模式的澳大利亚和新西兰已经将体重直接加入到选择指数中。体重性状在澳大利亚平衡性能指数(balanced performance index,BPI)、体型权重指数(type weighted index,TWI)和健康权重指数(health weighted index,HWI)中的权重分别为-2.50、-2.50和-5.44[2]。
遗传参数的准确估计对开展遗传育种至关重要。早年的报道中,研究者收集荷斯坦牛体重性状,目的在于探究奶牛生长发育规律,制定合理的饲养标准,样本数往往较小[6-7]。为了解荷斯坦牛生长和其它重要性状之间的遗传关系,关于体重的遗传分析,目标性状大多为初生重和成年体重[8-9]。对于后备牛,大部分研究仅关注断奶、配种、产犊阶段的体重[10-11],缺少对各月龄体重的连续称量。Koenen和Groen[12]指出,后备牛体重之间的遗传变异可以反映出个体成年体重和未发育完全时特定时间段发育成熟度两个方面的信息。随着电子自动体重秤、挤奶机器人等设备的出现,生长性状的连续测定更加便利,为荷斯坦牛生长性状基因组选择的实现提供可能。Brotherstone等[13]收集了奶牛从出生至1 000日龄的体重数据,平均每头奶牛有35次测定记录。估计产犊前体重遗传力范围为0.41~0.59,产犊后体重遗传力范围为0.74~0.82。Yin和Konig[3]对荷斯坦牛0~24月龄体重进行遗传分析,直接遗传力范围为0.29~0.83。但国内尚未见相关报道。
估计遗传参数时,可通过系谱信息构建亲缘关系矩阵,从而明确个体间的遗传联系。但在实际应用中,系谱记录存在缺失或错误的情况。Banos等[14]指出,各国家奶牛系谱错误率平均为11%。英国、荷兰、丹麦、爱尔兰、新西兰和以色列奶牛系谱错误率分别约为10%、12%、5%~15%、7%~20%、4%~15%和10.8%[15-17]。在我国,规范化的系谱登记起步较晚,牧场管理尚不完善,奶牛系谱记录存在更高的错误率。杨湛澄等[18]统计分析了中国24个省市2 374头中国荷斯坦良补公牛6代系谱的完整性,系谱缺失率为15%。因此,仅基于系谱信息进行构建的亲缘关系矩阵并不准确。系谱错误率的增加,会造成遗传力的估计值偏低。这对奶牛群体的遗传改良存在较大的负面影响。随着分子遗传学的发展,个体间亲缘关系矩阵也可以利用基因组遗传信息进行构建。但由于金钱、人力等限制,较大育种群体中无法对全部个体进行基因型测定。在此基础上,Misztal等[19]提出利用系谱信息和基因组信息合并构建亲缘关系矩阵进行遗传估计。本研究收集荷斯坦牛0~12月龄各月龄体重,基于系谱信息(linear mixed model with pedigree relationship matrix, LM_A)和系谱-基因组信息构建亲缘关系矩阵(linear mixed model with genotype-pedigree joint relationship matrix, LM_H),结合不同模型,拟确定一套较为全面、准确的遗传参数,为建立荷斯坦牛生长性状基因组选择体系提供理论参考依据。
1 材料与方法 1.1 表型数据本研究以山东和河南地区两个规模化牧场的荷斯坦牛为试验群体,使用Tru-Test Model ID5000称重指示器和Datamars SA电子耳标(型号:RAL-1016),于2019—2020年连续测定2 173头荷斯坦牛25 216条体重数据。此外,收集试验牛场2014—2020年7 122头荷斯坦母牛的出生记录和产犊记录。两个数据集共计32 338条体重数据。对原始数据进行质控,具体质控步骤如下:1)采用稳健回归分析法(robust regression followed by outlier identification, ROUT)剔除异常值[20];2)保留0、2~12月龄体重数据;3)同一个体相同月龄如存在重复测定,剔除除首次测定记录外的其它记录;4)剔除系谱和基因组信息均缺失的个体。最终,获得7 118头荷斯坦牛初生重记录,2 173头荷斯坦牛20 714条2~12月龄体重记录。
1.2 系谱数据综合利用牛场管理软件阿菲金数据库、北京奶牛中心数据库和加拿大奶业网数据库,追溯3代以内系谱信息。用于遗传评估的系谱共包括16 567头荷斯坦牛。其中,母牛14 643头,公牛1 924头。具有表型的7 118头荷斯坦牛为5 706头母牛的后代,即每头母牛平均有1.2个女儿。
1.3 基因型数据本试验通过颈静脉采血,采集约6 mL抗凝血液,对其进行基因型检测。采用Illumina公司150 K Bovine BeadChip (Illumina Inc., San Diego,CA)进行基因分型。试验共收集1 002头荷斯坦牛的基因型数据,共包含139 376个SNPs。对缺失基因型使用BEAGLE软件[21]进行填充。并根据以下质控标准进行剔除:1)个体基因分型检出率小于90%;2)单个SNP基因型检出率小于90%;3)最小等位基因频率(minor allele frequency,MAF)小于0.01;4)性染色体上的SNP。经过质控之后,共有971头荷斯坦牛114 658个SNPs用于亲缘关系矩阵构建。
1.4 遗传分析本研究采用不同模型对荷斯坦牛初生重、2~12月龄体重性状进行遗传参数估计。用于评估初生重的单性状动物模型为:
| $ \begin{gathered} y_{i j k l m}=\mu+F_i+B Y S_j+D P_k+g_l+ \\ \ \ m_m+m p e_m+e_{i j k l m} \end{gathered} $ | (模型1) |
其中,yijklm为初生重的观察值;μ为群体均值;Fi为场效应;BYSj为出生年季效应;DPk为母亲胎次效应;gl为随机加性遗传效应;mm为母体遗传效应;mpem为母体永久环境效应;eijklm为随机残差效应。
用于估计2~12月龄体重的单性状动物模型如下:
| $ \begin{gathered} y_{i j k l}=\mu+F_i+B Y S_j+D P_k+ \\ \beta_1 A g e+\beta_2 B W+g_l+e_{i j k l} \end{gathered} $ | (模型2) |
| $ \begin{gathered} y_{i j k l}=\mu+F_i+B Y S_j+D P_k+\beta_1 A g e+ \\ g_l+e_{i j k l} \end{gathered} $ | (模型3) |
其中,yijkl为2~12月龄体重的观察值;μ为群体均值;BW为初生重,Age为测定日龄,β1和β2为回归系数;其余效应与模型1中含义相同。对于2~12月龄体重数据,几乎所有个体均没有相同的母亲,因此模型2和3未加入母性效应的因素,预计母体效应将包括在随机残余效应中。
选择同时具有初生重和2~12月龄体重(各月龄至少有1次记录)的个体,通过双性状动物模型估计初生重和2~12月龄体重的遗传相关。双性状动物模型的矩阵形式如下:
| $ \left[\begin{array}{l} y_1 \\ y_2 \end{array}\right]=\left[\begin{array}{cc} \boldsymbol{X}_1 & 0 \\ 0 & \boldsymbol{X}_2 \end{array}\right]\left[\begin{array}{l} b_1 \\ b_2 \end{array}\right]+\left[\begin{array}{cc} \boldsymbol{Z}_1 & 0 \\ 0 & \boldsymbol{Z}_2 \end{array}\right]\left[\begin{array}{l} g_1 \\ g_2 \end{array}\right]+\left[\begin{array}{l} e_1 \\ e_2 \end{array}\right] $ | (模型4) |
其中,y1和y2分别为初生重和某一月龄(2~12)体重的观察值;b1和b2为性状1和性状2的固定效应向量,初生重的固定效应与模型1一致,2~12月龄体重的固定效应与模型3一致;g1和g2为性状1和性状2的随机加性遗传效应向量;e1和e2为性状1和性状2的随机残差效应向量;X1、X2、Z1和Z2为相应的关联矩阵。
模型1~4分别采用基于系谱信息的LM_A方法和基于系谱及基因组信息的LM_H方法。在LM_A中,g~N(0, Aσg2),A为基于系谱信息构建的亲缘关系矩阵,σg2为遗传方差。在LM_H中,g~N(0, Hσg2),H为基于系谱信息(A矩阵)和基因组信息(G矩阵)构建的亲缘关系矩阵,其逆矩阵公式如下:
| $ \boldsymbol{H}^{-1}=\boldsymbol{A}^{-1}+\left[\begin{array}{cc} 0 & 0 \\ 0 & \boldsymbol{G}^{-1}-\boldsymbol{A}_{22} \end{array}\right] $ |
其中,H-1、A-1、G-1分别为 H、A、G的逆矩阵;A22为A矩阵中具有基因组信息个体所组成的子矩阵。对 G 矩阵进行加权,采用Gω代替 G 矩阵,
| $ \boldsymbol{G}_\omega=(1-\omega) \boldsymbol{G}+\omega \boldsymbol{A}_{22} $ |
其中,ω为基因组关系矩阵和系谱关系矩阵的权重比,表示遗传关系未被SNPs解释的比例,本研究中设定ω=0.05。
本试验使用BLUPF90软件[22],采用平均信息-约束最大似然法(average information restricted maximum likelihood, AI-REML)估计方差组分,并计算初生重、2~12月龄体重各性状的遗传力及其标准误,以及初生重与2~12月龄体重的遗传相关及其标准误。初生重直接加性遗传效应和母体遗传效应的遗传相关(rgm)、直接遗传力(hd2)、母体遗传力(hm2)和总遗传力(ht2)分别定义为:
| $ r_{g m}=\frac{\sigma_{g m}}{\sqrt{\sigma_g^2 \times \sigma_m^2}} $ |
| $ h_d^2=\frac{\sigma_g^2}{\sigma_g^2+\sigma_m^2+\sigma_{g m}+\sigma_{p_m}^2+\sigma_e^2} $ |
| $ h_m^2=\frac{\sigma_m^2}{\sigma_g^2+\sigma_m^2+\sigma_{g m}+\sigma_{p_m}^2+\sigma_e^2} $ |
| $ h_t^2=\frac{\sigma_g^2+\sigma_m^2+2 \sigma_{g m}}{\sigma_g^2+\sigma_m^2+\sigma_{g m}+\sigma_{p_m}^2+\sigma_e^2} $ |
其中,σm2为母体遗传方差;σgm为直接加性遗传效应和母体遗传效应的协方差;σpm2为母体永久环境方差。
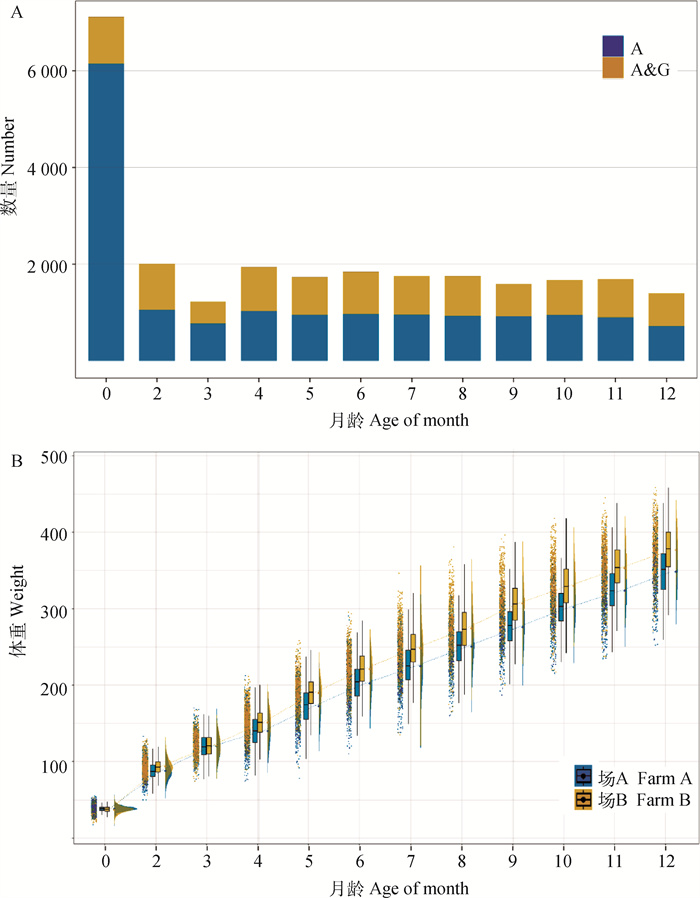
2 结果 2.1 荷斯坦牛体重性状描述性统计图 1展示了两个试验场荷斯坦牛各月龄体重性状的分布情况和个体数量。2~12月龄具有表型的个体数为1 215~2 004,其中3月龄和12月龄个体数较少,其余月龄的个体数均大于1 500。具有9次以上测定记录的个体有1 681头,占试验群体的77%,数据完整性较好。对于初生重,具有基因型信息的个体占具有表型的个体的13.61%;对于2~12月龄体重,具有基因型信息的个体占具有表型的个体的36.71%~48.31%,均值为45.16%。其中,3月龄比例最低,12月龄比例最高。
|
A. 各月龄具有表型的荷斯坦牛个体数。其中,A代表仅具有系谱信息的个体;A & G代表同时具有系谱和基因型信息的个体;B. 荷斯坦牛各月龄体重表型分布情况 A. The number of animals with only pedigree information (A) and with pedigree and genotype information (A & G) per growth month; B. Description of body weight traits in Holstein cattle 图 1 荷斯坦牛各月龄体重描述性统计 Fig. 1 Descriptive statistics of body weight traits in Holstein cattle at different months |
本试验中,荷斯坦牛初生重均值为37.92±3.88 kg,断奶重(2月龄)均值为90.65±11.52 kg,6月龄均值为211.44±27.66 kg,12月龄均值为362.57±35.78 kg。12月龄前,随着年龄的增长荷斯坦牛体重不断增加,生长速度未有变缓的趋势。从图 1B可以看出,两个试验场间体重存在显著差异(P < 0.05)。场A荷斯坦牛初生重均值高于场B荷斯坦牛1.04 kg,但2月龄后场A荷斯坦牛各月龄体重均低于场B,4月龄后体重差值随月龄的增加逐渐增大,12月龄时两个试验场的差值达到28.38 kg。
2.2 荷斯坦牛初生重遗传参数估计利用LM_A和LM_H方法估计荷斯坦牛初生重方差组分和遗传力结果如表 1所示。结果显示,初生重为中等遗传力性状,LM_A和LM_H方法估计直接遗传力分别为0.30和0.32,母体遗传力分别为0.08和0.09。个体直接遗传效应和母体遗传效应之间存在中高负遗传相关,分别为-0.65和-0.64。两种方法估计遗传参数结果相似,但LM_H的AIC值显著小于LM_A(ΔAIC>10)[23]。
|
|
表 1 荷斯坦牛初生重方差组分及遗传参数估计结果 Table 1 Variance components and genetic parameters of calf birth weight in Holstein cattle |
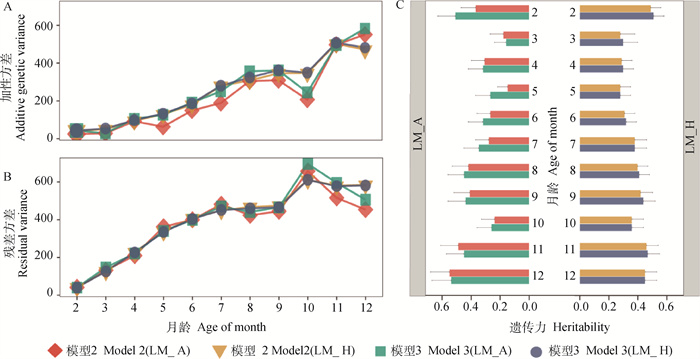
荷斯坦牛2~12月龄体重各模型的AIC值如表 2所示。由于各模型中采用了不同的数据集和固定效应,无法应用AIC值对模型2、3和4直接进行比较,但可用来比较相同模型内LM_A和LM_H两种方法的模型拟合优度。对于各模型,LM_A的AIC值均大于LM_H,差值范围为11.24~237.31。荷斯坦牛2~12月龄体重方差组分和遗传力结果如图 2所示。随着月龄增加,加性方差和残差方差整体呈现上升趋势,但在10月龄存在明显波动,表现为基于LM_A方法的加性方差相较于9月龄呈小幅下降,考虑初生重作为协变量的单性状动物模型(模型2)和未考虑初生重效应的单性状动物模型(模型3)分别下降102.53和116.02;残差方差从7月龄后增速放缓,但在10月龄增幅较大,相较于9月龄,基于LM_A方法两种模型分别增加212.21和230.09,基于LM_H方法分别增加135.69和145.24。利用初生重作为协变量的单性状动物模型(模型2)进行估计时,除12月龄体重外,LM_A方法估计的加性方差均小于LM_H方法。其中,10月龄体重两种方法估计的加性方差差值最大,为140.57。同一月龄下,两种方法估计的残差方差差值范围为0.37~131.04,差异小于加性方差:采用未考虑初生重效应的单性状动物模型(模型3)估计9月龄体重时残差方差相差最小,考虑初生重作为协变量的单性状动物模型(模型2)估计12月龄体重时残差方差相差最大。对于LM_A方法,2种模型估计加性方差的整体趋势较为一致,除12月龄外,其余月龄模型3的加性方差均大于模型2。2种模型间残差方差差值较小,均值为23.72。相较于其它月龄,11、12月龄2种模型之间差值较大。
|
|
表 2 荷斯坦牛2~12月龄体重性状各评估模型的赤池信息量准则值 Table 2 Model comparison based on values of Akaike information criterion (AIC) for body weights from month 2 to 12 in Holstein cattle |
|
图 2 荷斯坦牛2~12月龄体重性状加性方差组分(A)、残差方差组分(B)和遗传力(C)估计结果 Fig. 2 Additive genetic variance (A), residual variance (B) and heritability (C) of monthly body weights from month 2 to 12 in Holstein cattle |
荷斯坦牛2~12月龄体重为中高遗传力性状。基于LM_A和LM_H方法,考虑初生重作为协变量的单性状动物模型(模型2)估计遗传力的范围分别为0.15~0.55和0.28~0.49,未考虑初生重效应的单性状动物模型(Model 3)估计遗传力的范围分别为0.16~0.54和0.28~0.51。结合图 2C可以看出,LM_H方法估计各月龄体重遗传力的范围更集中,且2种模型的估计结果基本一致。3~6月龄体重遗传力约为0.3,7~10、12月龄体重遗传力约为0.4,2、11月龄遗传力约为0.5。而通过LM_A方法获得的估计遗传力,2种模型间差异较大,2月龄体重估计遗传力分别为0.37和0.51,5月龄体重估计遗传力分别为0.15和0.27。3和10月龄体重遗传力低于其它月龄,且低于相应LM_H方法的估计值。从遗传力标准误来看,LM_H方法(0.07~0.08)小于LM_A方法(0.10~0.13)。
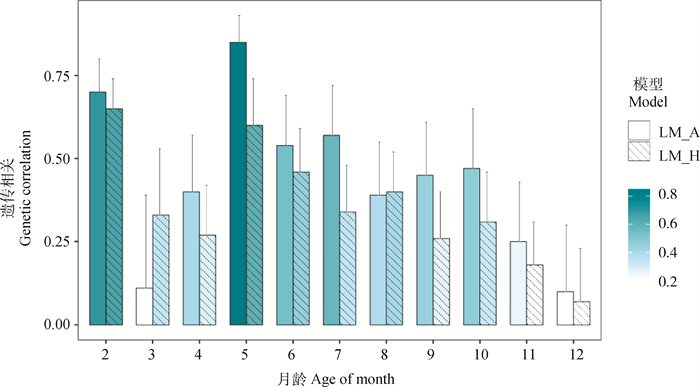
2.4 荷斯坦牛初生重与2~12月龄体重遗传相关图 3展示了荷斯坦牛初生重与其它月龄体重的遗传相关。初生重与2、5月龄为中高遗传相关,遗传相关系数均大于0.6。LM_A方法估计初生重与3月龄体重遗传相关较低,但标准误较大,为0.11±0.28。5月龄后,随着荷斯坦牛月龄的增长,遗传相关呈下降趋势。基于LM_A方法,初生重与5月龄体重遗传相关最高,为0.85±0.08,与12月龄体重遗传相关最低,为0.10±0.20;基于LM_H方法,初生重与2月龄体重遗传相关最高,为0.65±0.09,与12月龄体重遗传相关最低,为0.07±0.16。除3月龄(0.11±0.28 vs. 0.33±0.20)和8月龄(0.39±0.16 vs. 0.40±0.12)外,LM_A方法估计的初生重与其它月龄体重的遗传相关均大于LM_H方法,差值范围为0.03~0.25。其中,5和7月龄相差较大。
|
图 3 荷斯坦牛初生重与2~12月龄体重遗传相关 Fig. 3 Genetic correlations between calf birth weights and monthly body weights from month 2 to 12 in Holstein cattle |
本研究连续测定了荷斯坦牛0~12月龄体重数据,体重均值范围为37.92~362.57 kg。于志等[24]于2012—2013年在河北地区跟踪测量了150头中国荷斯坦牛的体重,初生至12月龄的平均体重范围为43.05~345.26 kg。除初生重较大外,其余各月龄的体重均小于本试验群体,相较于以往,荷斯坦牛群体生长速度更快,达到配种体重时的年龄更小。
基于系谱和基因组信息,利用不同模型对体重性状的遗传参数进行估计。相较于其它月龄体重,荷斯坦牛初生重遗传分析的相关报道较多,直接遗传力范围为0.22~0.58[3, 11, 13, 25-26]。Coffey等[11]采用多性状模型对486头荷斯坦牛进行估计,直接遗传力为0.53±0.12,个体数较少,且模型中未考虑母体效应,可能会造成遗传力高估[27]。在对初生重进行遗传分析时,前人大多在模型中加入母体效应。Everett和Magee[26]估计荷斯坦牛初生重的直接遗传力为0.22,母体遗传力为0.04,低于本研究的估计值;Johanson[25]采用贝叶斯多性状模型进行估计,直接遗传力为0.26,母体遗传力为0.08,与本研究结果相似;Yin和Konig[3]对来自53个牧场57 868头荷斯坦牛的初生重进行遗传参数估计,直接遗传力为0.47±0.02,母体遗传力为0.19±0.01,总遗传力为0.39±0.01,直接遗传和母体遗传效应的遗传相关为-0.39±0.02。直接遗传和母体遗传效应的遗传相关估计准确性与具有表型的母亲个体数有关[28]。本研究中,7 118头个体是5 706头母牛的后代,其中,具有表型的母亲为1 755头,比例为30.76%。Yin和Konig[3]的研究中具有表型的母亲比例为31.57%,略高于本研究。LM_A和LM_H方法估计初生重遗传参数结果基本一致,这可能是由于该性状基因型个体数量占系谱总个体数比例较少,仅为5.86%。
研究表明,畜禽的初生重对后期生长性状存在一定影响[29-30]。因此,本研究对2~12月龄体重进行分析时,将初生重作为固定效应加入到单性状动物模型Model 2中,校正初生重效应后估计2~12月龄体重的遗传参数,同时估计未校正初生重效应的2~12月龄体重的遗传参数,以期获得不同定义的体重遗传参数。从方差组分和遗传力的结果上看,2种模型的差异主要体现在LM_A方法上。基于LM_A方法利用Model 2获得的2、5~10月龄加性方差低于其它模型,遗传力也相应较低。其中,2和5月龄体重差异尤为明显。结合遗传相关的结果,可以发现2和5月龄体重与初生重呈现中高等遗传相关。因此,推测这可能是由于系谱信息存在错误以及初生重的影响共同导致的。对比LM_A和LM_H两种方法的估计结果,LM_H方法更稳定,AIC值更小,同一性状2种模型间结果更接近,遗传参数标准误较小,但方差组分和遗传力高低无明显规律。Watanabe等[31]同样采用这两种方法对内洛牛体重进行遗传参数估计,初生重遗传力无明显差异,通过LM_H方法估计8、12、15月龄体重获得的加性方差和遗传力更高。Loberg等[32]提出,利用系谱信息和基因型信息估计加性方差,两者之间的差距会随着性状遗传力的增大或样本数量的增加逐渐缩小。在实际应用中,仅利用系谱信息可能无法准确识别个体之间的亲缘关系,实际系谱数据可能存在信息不全或记录错误等情况。由于孟德尔抽样[33],利用基因组信息构建亲缘关系矩阵,可以使个体之间的关系更加全面准确,因此,将系谱信息和基因组信息结合构建亲缘关系矩阵,既可避免样本量的损失,还可根据准确的基因组信息提高遗传估计的准确性[34]。
在前人报道中,以月为间隔对一定群体规模荷斯坦后备母牛的体重进行连续称量并估计遗传参数的分析较少,目标性状大多聚焦于断奶重、周岁重和首次产犊体重[10-11]。Groen和Vos[35]对631头荷斯坦牛10、20、30、40和50周龄(约12.5月龄)体重进行分析,遗传力范围为0.49~0.62,高于本研究估计结果(0.3~0.5)。本试验中,3月龄体重加性方差无明显变化,残差方差增加,遗传力下降,10月龄体重加性方差减小,残差方差增加,遗传力下降。这可能是由于数据量、饲养管理等环境因素变化造成的。与其它月龄相比,3月龄数据量较低。本研究中的试验群体在断奶后7 d(67日龄)从犊牛岛转入断奶牛舍,3月龄进行首次口蹄疫免疫,10月龄时育成牛会进行一次转群。免疫、转群和饲养环境变化等因素使环境方差增大,因此估计遗传力降低。从整体趋势上看,各月龄体重与初生重的遗传相关系数随着时间间隔的增加而减小,这与其它研究结果相一致[3],初生重与断奶重之间的遗传相关为0.73,与首次产犊体重的遗传相关为0.30[11],50 d和900 d体重的遗传相关仅为0.14[13]。在Yin和Konig[3]的研究中,初生重和24月龄体重甚至为负遗传相关,为-0.20。这也间接表明不同阶段生长性状的遗传机制可能并不相同。
4 结论荷斯坦牛0~12月龄体重性状为中高遗传力性状(0.3~0.5),2~12月龄体重与初生重的遗传相关随着时间间隔的增加呈降低趋势,不同生长阶段的体重性状遗传机制可能不尽相同。在个体间亲缘关系较为准确的情况下,2~12月龄体重的实际遗传力与将初生重作为协变量校正初生重影响后的遗传力基本一致(差值为0.00~0.02)。在实际应用中,基于系谱-基因型信息构建亲缘关系矩阵有利于获得更加稳定、准确的遗传参数。
致谢: 感谢首农畜牧山东分公司、首农畜牧河南分公司在数据收集中提供的大力支持;感谢课题组成员安涛、王凯在数据测定中提供的帮助。
| [1] |
YIN T, KÖNIG S. Genomic predictions of growth curves in Holstein dairy cattle based on parameter estimates from nonlinear models combined with different kernel functions[J]. J Dairy Sci, 2020, 103(8): 7222-7237. DOI:10.3168/jds.2019-18010 |
| [2] |
BYRNE T J, SANTOS B F S, AMER P R, et al. New breeding objectives and selection indices for the Australian dairy industry[J]. J Dairy Sci, 2016, 99(10): 8146-8167. DOI:10.3168/jds.2015-10747 |
| [3] |
YIN T, KONIG S. Genetic parameters for body weight from birth to calving and associations between weights with test-day, health, and female fertility traits[J]. J Dairy Sci, 2018, 101(3): 2158-2170. DOI:10.3168/jds.2017-13835 |
| [4] |
PRYCE J E, GONZALEZ-RECIO O, NIEUWHOF G, et al. Hot topic: definition and implementation of a breeding value for feed efficiency in dairy cows[J]. J Dairy Sci, 2015, 98(10): 7340-7350. DOI:10.3168/jds.2015-9621 |
| [5] |
SPURLOCK D M, DEKKERS J C M, FERNANDO R, et al. Genetic parameters for energy balance, feed efficiency, and related traits in Holstein cattle[J]. J Dairy Sci, 2012, 95(9): 5393-5402. DOI:10.3168/jds.2012-5407 |
| [6] |
HEINRICHS A J, HARGROVE G L. Standards of weight and height for Holstein heifers[J]. J Dairy Sci, 1987, 70(3): 653-660. DOI:10.3168/jds.S0022-0302(87)80055-3 |
| [7] |
HEINRICHS A J, LOSINGER W C. Growth of Holstein dairy heifers in the United States[J]. J Anim Sci, 1998, 76(5): 1254-1260. DOI:10.2527/1998.7651254x |
| [8] |
ABDALLAH J M, MCDANIEL B T. Genetic parameters and trends of milk, fat, days Open, and body weight after calving in North Carolina experimental herds[J]. J Dairy Sci, 2000, 83(6): 1364-1370. DOI:10.3168/jds.S0022-0302(00)75004-1 |
| [9] |
VAN ELZAKKER P J M, VAN ARENDONK J A M. Feed intake, body weight and milk production: genetic analysis of different measurements in lactating dairy heifers[J]. Livest Prod Sci, 1993, 37(1): 37-51. |
| [10] |
DEMEKE S, NESER F W C, SCHOEMAN S J. Variance components and genetic parameters for early growth traits in a mixed population of purebred Bos indicus and crossbred cattle[J]. Livest Prod Sci, 2003, 84(1): 11-21. DOI:10.1016/S0301-6226(03)00074-5 |
| [11] |
COFFEY M P, HICKEY J, BROTHERSTONE S. Genetic aspects of growth of Holstein-Friesian dairy cows from birth to maturity[J]. J Dairy Sci, 2006, 89(1): 322-329. DOI:10.3168/jds.S0022-0302(06)72097-5 |
| [12] |
KOENEN E P C, GROEN A F. Genetic analysis of growth patterns of black and white dairy heifers[J]. J Dairy Sci, 1996, 79(3): 495-501. DOI:10.3168/jds.S0022-0302(96)76391-9 |
| [13] |
BROTHERSTONE S, COFFEY M P, BANOS G. Genetic parameters of growth in dairy cattle and associations between growth and health traits[J]. J Dairy Sci, 2007, 90(1): 444-450. DOI:10.3168/jds.S0022-0302(07)72646-2 |
| [14] |
BANOS G, WIGGANS G R, POWELL R L. Impact of paternity errors in cow identification on genetic evaluations and international comparisons[J]. J Dairy Sci, 2001, 84(11): 2523-2529. DOI:10.3168/jds.S0022-0302(01)74703-0 |
| [15] |
VISSCHER P M, WOOLLIAMS J A, SMITH D, et al. Estimation of pedigree errors in the UK dairy population using microsatellite markers and the impact on selection[J]. J Dairy Sci, 2002, 85(9): 2368-2375. DOI:10.3168/jds.S0022-0302(02)74317-8 |
| [16] |
周磊, 刘林, 初芹, 等. 奶牛亲子鉴定应用的标记和方法研究进展[J]. 中国奶牛, 2011(2): 26-29. ZHOU L, LIU L, CHU Q, et al. Progress on label and method of paternity test in dairy cattle[J]. China Dairy Cattle, 2011(2): 26-29. DOI:10.3969/j.issn.1004-4264.2011.02.008 (in Chinese) |
| [17] |
WELLER J I, FELDMESSER E, GOLIK M, et al. Factors affecting incorrect paternity assignment in the Israeli Holstein population[J]. J Dairy Sci, 2004, 87(8): 2627-2640. DOI:10.3168/jds.S0022-0302(04)73389-5 |
| [18] |
杨湛澄, 闫青霞, 黄河天, 等. 中国荷斯坦公牛系谱完整性研究[J]. 中国奶牛, 2017(2): 22-25. YANG Z C, YAN Q X, HUANG H T, et al. Evalutation of pedigree completeness in Chinese Holstein bulls[J]. China Dairy Cattle, 2017(2): 22-25. DOI:10.19305/j.cnki.11-3009/s.2017.02.006 (in Chinese) |
| [19] |
MISZTAL I, AGUILAR I, LEGARRA A, et al. Choice of parameters for single-step genomic evaluation for type[J]. J Dairy Sci, 2010, 93(S1): 166. |
| [20] |
MOTULSKY H J, BROWN R E. Detecting outliers when fitting data with nonlinear regression-a new method based on robust nonlinear regression and the false discovery rate[J]. BMC Bioinformatics, 2006, 7: 123. DOI:10.1186/1471-2105-7-123 |
| [21] |
BROWNING B L, BROWNING S R. A unified approach to genotype imputation and haplotype-phase inference for large data sets of trios and unrelated individuals[J]. Am J Hum Genet, 2009, 84(2): 210-223. DOI:10.1016/j.ajhg.2009.01.005 |
| [22] |
MISZTAL I, TSURUTA S, LOURENCO D, et al. Manual for BLUPF90 family of programs[EB/OL]. (2014-05-12). http://nce.ads.uga.edu/wiki/lib/exe/fetch.php?media=blupf90_all2.pdf.
|
| [23] |
BURNHAM K P, ANDERSON D R. Multimodel inference: understanding AIC and BIC in model selection[J]. Sociol Method Res, 2004, 33(2): 261-304. DOI:10.1177/0049124104268644 |
| [24] |
于志, 高艳霞, 曹玉凤, 等. 中国荷斯坦犊牛和育成牛生长发育规律的研究[J]. 中国畜牧兽医, 2014, 41(6): 121-125. YU Z, GAO Y X, CAO Y F, et al. Study on growth and development pattern of Chinese Holstein calf and heifer[J]. China Animal Husbandry & Veterinary Medicine, 2014, 41(6): 121-125. (in Chinese) |
| [25] |
JOHANSON J M, BERGER P J, TSURUTA S, et al. A Bayesian threshold-linear model evaluation of perinatal mortality, dystocia, birth weight, and gestation length in a Holstein herd[J]. J Dairy Sci, 2011, 94(1): 450-460. DOI:10.3168/jds.2009-2992 |
| [26] |
EVERETT R W, MAGEE W T. Maternal ability and genetic ability of birth weight and gestation length[J]. J Dairy Sci, 1965, 48(7): 957-961. DOI:10.3168/jds.S0022-0302(65)88368-0 |
| [27] |
CLÉMENT V, BIBÉ B, VERRIER É, et al. Simulation analysis to test the influence of model adequacy and data structure on the estimation of genetic parameters for traits with direct and maternal effects[J]. Genet Sel Evol, 2001, 33(4): 369-395. DOI:10.1186/1297-9686-33-4-369 |
| [28] |
HEYDARPOUR M, SCHAEFFER L R, YAZDI M H. Influence of population structure on estimates of direct and maternal parameters[J]. J Anim Breed Genet, 2008, 125(2): 89-99. DOI:10.1111/j.1439-0388.2007.00703.x |
| [29] |
BALDI F, ALBUQUERQUE L G, ALENCAR M M. Random regression models on Legendre polynomials to estimate genetic parameters for weights from birth to adult age in Canchim cattle[J]. J Anim Breed Genet, 2010, 127(4): 289-299. DOI:10.1111/j.1439-0388.2010.00853.x |
| [30] |
周身娉, 付帝生, 杨明, 等. 仔猪初生重对育肥期生长性状及采食性状的影响[J]. 中国畜牧杂志, 2019, 55(1): 41-45. ZHOU S P, FU D S, YANG M, et al. Effect of Duroc birth weight on their growth and feeding traits in fattening stage[J]. Chinese Journal of Animal Science, 2019, 55(1): 41-45. (in Chinese) |
| [31] |
WATANABE R N, DO NASCIMENTO G B, BERNARDES P A, et al. Inclusion of genomic information in estimation of genetic parameters for body weights and visual scores in Nelore cattle[J]. Rev Bras Zootec, 2021, 50: e20200077. DOI:10.37496/rbz5020200077 |
| [32] |
LOBERG A, DVRR J W, FIKSE W F, et al. Estimates of genetic variance and variance of predicted genetic merits using pedigree or genomic relationship matrices in six Brown Swiss cattle populations for different traits[J]. J Anim Breed Genet, 2015, 132(5): 376-385. DOI:10.1111/jbg.12142 |
| [33] |
SCHROOTEN I C. Genomic selection in dairy cattle[J]. Wur Wageningen Ur, 2011, 95(4): 2097-2107. |
| [34] |
VEERKAMP R F, MULDER H A, THOMPSON R, et al. Genomic and pedigree-based genetic parameters for scarcely recorded traits when some animals are genotyped[J]. J Dairy Sci, 2011, 94(8): 4189-4197. |
| [35] |
GROEN A F, VOS H. Genetic parameters for body weight and growth in Dutch Black and White replacement stock[J]. Livest Prod Sci, 1995, 41(3): 201-206. |
(编辑 郭云雁)



