2. 阿勒泰地区布尔津县禾木哈纳斯蒙古民族乡农业(畜牧业)发展服务中心,阿勒泰 836500
2. Altay area Buerjin County Hemuhanasi Mongolian Nationality Township Agriculture (Animal Husbandry) Development Service Center, Altay 836500, China
马属动物属草食单胃动物,其胃部虽然不能像反刍动物的瘤胃一样对食物进行发酵,但是其肠道的消化功能十分强大,里面包含了极其丰富和高度复杂的厌氧微生物群体[1]。这些肠道菌群通过影响个体的细胞增殖、神经信号等[2]对机体的新陈代谢、生理、营养和免疫产生影响[3-4]。因此,肠道菌群的适应性变化是马匹适应生存环境变化和食性组成变化的途径之一。
哈萨克马是新疆数量最多的马品种,大多数分布于阿勒泰地区,这里冬季气温一般为-30 ℃左右[5-6]。山区牧民饲养的哈萨克马能够很好地适应逐水草而居,自然放牧的生存环境。冬季时期用于骑乘或挤奶时,哈萨克马会被圈养。而那些冬季暂无用途的马匹,则被放于山中,在积雪20~30 cm厚的草场上刨雪觅食,吞雪解渴[7-8]。
有研究发现,不同饲养模式下蒙古马肠道菌群组成结构存在整体性差异[9]。目前,对于蒙古马肠道菌群已有较为系统的研究[10-12]。李晓斌等[13-14]研究了新疆伊犁马马驹及新疆本地自繁的纯血马母马与马驹的肠道菌群特点,发现肠道菌群在生长发育过程中呈现阶段性变化,且断奶前马驹与母马粪便菌群多样性和相似性均有一定差异。de Fombelle等[15]研究骟马的前肠道消化率时发现,与增加淀粉类谷物饲料相比,饲喂高纤维饲料能够促进胃肠道蠕动,显著缩短饲料在肠道的停留时间,从而增加肠道消化能力,这与其他研究[16-17]得出的结果一致。在两种不同饮食下,饲喂高纤维饲料的骟马肠道中纤维素降解菌浓度均高于饲喂高谷物饲料的骟马[18]。马属动物被喂养不同的饲料后,其肠道菌群明显不同,饲喂大麦的马匹与饲喂干草的马匹相比,其盲肠和结肠中的厌氧菌(如乳酸杆菌、链球菌等)显著增加,且肠道内纤维素分解菌的群落结构也不相同[19]。
哈萨克马作为我国重要的种质资源,对于其肠道菌群的研究鲜见报道,对于不同饲养模式对哈萨克马肠道菌群组成产生怎样的影响尚不清楚。因此,本研究选取了圈养和自由放牧的哈萨克马作为研究对象,旨在比较不同饲养模式下哈萨克马肠道菌群组成差异,从而探讨自由放牧和圈养因素对肠道菌群结构的影响,为进一步研究哈萨克马肠道菌群奠定基础,也为后期探究马属动物肠道菌群组成差异及功能方面提供理论依据。
1 材料与方法 1.1 试验材料选取圈养和自由放牧条件下成年哈萨克马各5匹为试验材料,编号分别为JY1~JY5和FM1~FM5。圈养组马匹以定时投喂干草和补饲一定精料的半放牧半舍饲方式饲养。自由放牧组马匹主要采食积雪下的干草,不投喂精料。圈养组马匹采食的干草为牧民夏季在山中收割而得,与放牧组采食的干草种类相同。在采集圈养组粪便时,圈养马匹的牧民较少,样本有限,因此圈养组样本有骟马粪便和母马粪便,骟马为牧民骑乘马匹,母马为牧民预备挤奶用马匹。放牧组马匹全为骟马。由于肠道菌群受性别影响非常有限[20-21],故在本试验中未将性别作为固定因素。在1月份无菌采集新鲜粪便,放于5 mL无菌无酶冻存管中,并立即投入液氮中低温保存。
1.2 总DNA的提取、纯化和构建文库提取粪便总DNA、使用通用引物(F: ACTCTACGGAGGCAGCA;R: GGACTACHVGGGTWTCTAAT)对16S rRNA基因的V3-V4区进行PCR扩增、构建文库后经NanoDrop定量和文库检测合格后,使用Illumina MiSeq PE250进行上机测序。以上步骤均由上海派森诺生物科技股份有限公司协助完成。
1.3 数据处理及分析根据上海派森诺生物科技股份有限公司给出的原始序列、有效序列等信息,使用DADA2分析进行序列质控(去引物、质量过滤、去噪、拼接和去嵌合体)。利用派森诺基因云分析平台(https://www.genescloud.cn)进行OTU聚类、物种注释、α多样性、β多样性以及各分类水平物种组成分析。使用Origin 2021、GraphPad Prism 8.0软件绘制堆积柱状图、条形图。使用IBM SPSS Statistics 26.0软件做独立样本t检验,其中,P<0.05具有统计学意义。
2 结果 2.1 测序结果和α多样性分析对哈萨克马的10个粪便样本进行测序,共获得高质量序列数735 234条,根据97%的序列一致性,共得到29 955个操作分类单元(operational taxonomic units,OTUs)。对两种饲养模式的哈萨克马肠道菌群进行α多样性检验(表 1),10个样本的测序覆盖率平均为94%以上,说明本试验中获得的序列代表了微生物界94%以上的细菌种系型,测序覆盖率好,每个样本中均观察到较丰富的物种。但放牧组和圈养组菌群丰度差异不显著(Chao1指数:6 538.38 vs. 6 485.338;Observed_species:4 655.04 vs. 4 683.24;Faith_pd:276.381 4 vs. 301.457 4;P>0.05)。放牧组菌群多样性显著高于圈养组(Shannon指数:10.853 48 vs. 10.194 9,P < 0.05;Simpson指数:0.998 7 vs. 0.984 0,P<0.01)。
|
|
表 1 测序结果及α多样性分析 Table 1 Sequencing result and α diversity analysis |
两组10匹马的粪便共鉴定出21个门,45个纲,70个目,126个科,226个属,286个种。圈养组与放牧组哈萨克马肠道菌群中丰度最高的5个菌门为厚壁菌门(Firmicutes,78.08% vs. 76.10%)、拟杆菌门(Bacteroidetes,15.72% vs. 17.16%)、疣微菌门(Verrucomicrobia,2.48% vs. 2.91%)、变形菌门(Proteobacteria,0.69% vs. 0.91%)和TM7菌门(0.69% vs. 0.56%),如图 1A。其中前三个优势菌门占据了两组96%的序列,未鉴定出的细菌序列占所有序列的0.82%。

|
图 1 门水平(A)和科水平(B)上Top10物种组成丰度堆积柱状图 Fig. 1 Accumulation histogram of Top 10 species composition abundance at phylum(A) and family(B) level |
科分类水平的分析显示(图 1B),在放牧组中优势菌科为瘤胃球菌科(Ruminococcaceae,30.44%)、毛螺菌科(Lachnospiraceae,21.96%)和艰难杆菌科([Mogibacteriaceae],4.86%)。而在圈养组的优势菌科为瘤胃球菌科(28.73%)、粪便链球菌科(Streptococcaceae,15.08%)和毛螺菌科(14.51%)。在两组中丰度占比最高的瘤胃球菌科是马匹发酵纤维素的基础。在圈养组中发现粪便链球菌科的显著上升(P < 0.05)和毛螺菌科占比下降。
圈养组与放牧组Top10的菌属为瘤胃球菌属(Ruminococcus,1.15% vs. 4.27%)、罗氏菌属(Roseburia,0.32% vs. 1.51%)、粪球菌属(Coprococcus,0.67% vs. 1.45%)、普雷沃氏菌属(Prevotella,0.21% vs. 0.80%)、考拉杆菌属(Phascolarctobacterium,0.50% vs. 0.95%)、艰难杆菌属(Mogibacterium,0.63% vs. 0.68%)、梭状芽孢菌属(Clostridium,1.46% vs. 1.41%)、颤螺菌属(Oscillospira,3.27% vs. 3.03%)、BF311(0.68% vs. 0.37%)、粪便链球菌属(Streptococcus,15.43% vs. 0.38%)。根据菌属差异显著分析显示(图 2),放牧组中瘤胃球菌属极显著高于圈养组(P<0.01),罗氏菌属、粪球菌属和普雷沃氏菌属显著高于圈养组(P<0.05);圈养组中粪便链球菌属显著高于放牧组(P < 0.05)。

|
图 2 属水平Top10物种差异显著分析 Fig. 2 Significant difference analysis of Top 10 species at genus level |
基于加权Unifrac指数,绘制个体的分布特征图(图 3)。非量度多维尺度(NMDS)分析中,应力值(stress)为0.074 9,一般认为该值小于0.2时,排序结果较为可靠。圈养组和放牧组的样本分别分布在主坐标NMDS1的左侧和右侧,表明圈养组和放牧组组间群落差异较大。在主坐标NMDS2上,圈养组和放牧组组内各样本之间较为分散,说明组内各样本肠道菌群组成也存在一定差异。
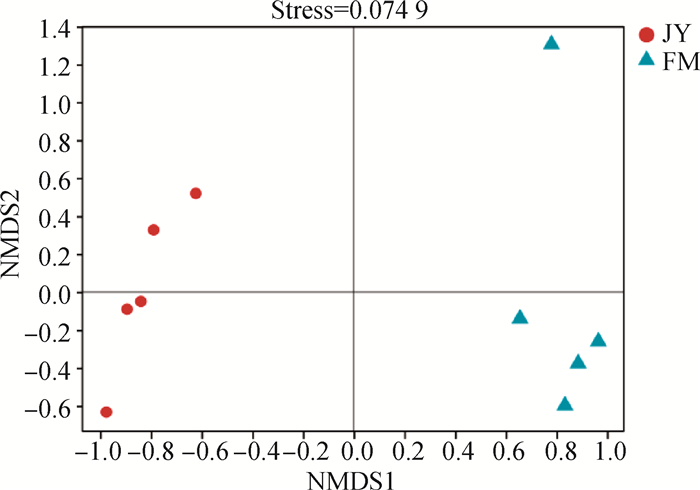
|
图 3 基于加权Unifrac指数的NMDS分析 Fig. 3 NMDS analysis based on weighted UniFrac index |
LDA Effect Size(LEfSe)分析是为了寻找在组内样本中都存在、但组与组之间具有统计学差异的物种,即标志物种。在LDA值(lg)>3的阈值下,圈养组与放牧组中分别发现了12和24个标志物种(图 4)。数据显示,圈养组中的标志物种有:粪便链球菌(Streptococcus)、乳酸杆菌(Lactobacillus)、WCHB1_25和韦荣氏球菌科的RFN20。放牧组中的标志物种主要以瘤胃球菌(Ruminococcus)、梭菌(Clostridium)、罗氏菌(Roseburia)、粪球菌(Coprococcus)、普雷沃氏菌(Prevotella)、假丁酸弧菌(Pseudo dutyrivibrio)和甲基杆菌(Methylobacterium)为主。
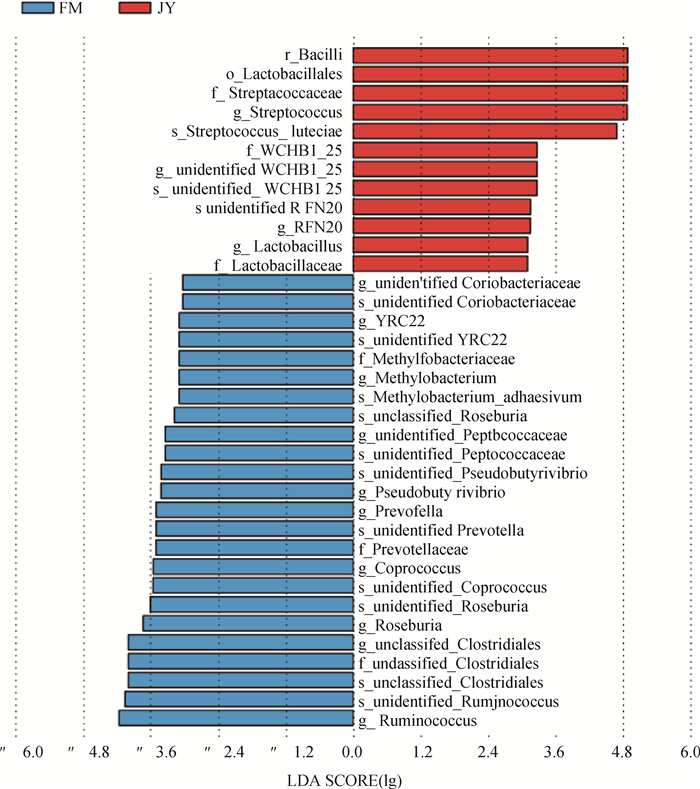
|
图 4 LEfSe分析 Fig. 4 LEfSe analysis |
基于序列功能预测分析,哈萨克马肠道菌群中共预测到175个直系同源分类(KOs),并将其注释到KEGG第一功能途径(图 5)。大多数功能序列被注释为代谢(77.44%),其次是遗传信息处理(14.59%)、细胞过程(5.06%)、环境信息处理(2.26%),生物系统和人类疾病占比最低,分别为0.46%和0.20%。

|
图 5 一级KEGG功能途径组成柱状图 Fig. 5 Histogram of functional pathway composition of primary KEGG |
进一步注释到次级KEGG功能途径(图 6)。放牧组与圈养组中“碳水化合物代谢”(13.68% vs. 14.018%)是“代谢”中最大的类别,其次是氨基酸代谢(12.933% vs. 13.04%)、辅助因子和维生素的代谢(12.128% vs. 11.992%)、萜类化合物和聚酮类化合物的代谢(10.51% vs. 9.779%)、其他氨基酸代谢(7.087% vs.7.312%)、脂质代谢(5.353% vs. 5.435%)以及能量代谢(5.426% vs. 5.095%)等。根据结果显示,放牧组与圈养组在次级功能途径上有4个功能途径存在显著差异(P < 0.05):传染病、能量代谢、萜类化合物和聚酮化合物的代谢、核苷酸代谢。其中传染病和核苷酸代谢在圈养组中显著富集,能量代谢和萜类化合物和聚酮化合物的代谢在放牧组中显著富集。
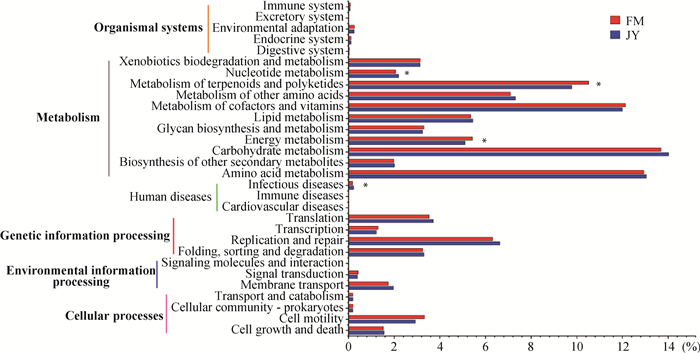
|
图 6 次级KEGG功能途径条形图 Fig. 6 Bar chart of secondary KEGG functional pathway |
由于遗传背景、生存环境及食性组成不同,每个个体的肠道菌群都是独一无二的。但同一地区遗传背景和生存环境又具有一定的相似性[22],故马匹中肠道菌群组成呈现一定的规律性。哈萨克马作为我国重要的马品种资源,数量占新疆马匹数的1/2、全国马匹数的1/10,对于其研究报道多见于生产性能和开发利用方面[7-8, 23-24],在肠道菌群方面研究较少,尤其是饲养模式对哈萨克马肠道菌群的组成结构方面有何影响鲜有报道。
3.1 不同饲养模式下哈萨克马肠道菌群多样性本研究在10个哈萨克马粪便样本中共获得高质量序列数735 234条,Dougal等[25]在10匹骟马的粪便样本中获得559 623条高质量序列,说明不同马匹的肠道菌群序列量存在较大差异。邢振存[9]在对比干草舍饲与青草放牧两种不同饲养条件下蒙古马肠道菌群结构时发现,干草舍饲的马匹肠道真菌群落多样性更高,但均匀度较低。本试验结果显示,放牧组物种多样性显著高于圈养组,这说明只采食干草的放牧组比补喂精料的圈养组肠道菌群组成更为多样。Julliand等[26]也得出不同饲喂条件下纤维源对肠道菌群具有较大影响的结论。Willing等[27]在研究骟马肠道菌群组成时发现,与投喂草料(猫尾草/干羊茅)的马匹相比,投喂草料(50%干物质(猫尾草/干羊茅))及精料(35.8%淀粉+41%燕麦+7%豆粕+1.35%麦麸))的马匹肠道菌群多样性偏低。这与本研究得出的结果相一致。
3.2 不同饲养模式下哈萨克马肠道菌群组成及变化有学者在研究阿拉伯马的粪便菌群时发现,分布最多的是厚壁菌门(Firmicutes,占细菌总数的43.7%),疣微菌门(Verrucomicrobia,4.1%),变形菌门(Proteobacteria,3.8%)和拟杆菌门(Bacteroidetes,3.7%)[28]。本试验中,圈养组与放牧组哈萨克马肠道菌群在门水平上由厚壁菌门(78.08% vs. 76.10%)、拟杆菌门(15.72% vs. 17.16%)和疣微菌门(2.48% vs. 2.91%)等组成。不同马品种在肠道菌群组成上可能不同。厚壁菌门的成员在代谢、消化吸收蛋白质和其他营养物质等功能中扮演着重要的作用[29-31],拟杆菌门是草食动物分解纤维素的主要菌群[32]。本研究中,厚壁菌门和拟杆菌门在圈养组与放牧组肠道中占比相近,可能是两种饲养条件下,宿主肠道菌群为应对能量和体温胁迫而做出的适应性波动。
在本试验中,瘤胃球菌是圈养组与放牧组的优势菌科和优势菌属,有研究发现,瘤胃球菌的数量与纤维素消化利用存在密切关系[33-36],在放牧组中还存在丰度较高的毛螺菌科、艰难杆菌科、罗氏菌属、粪球菌属和普雷沃氏菌属等,这些软化、发酵、降解纤维素的菌[37-39]是放牧组马匹能够充分利用干草的微生物基础。与放牧组相比,圈养组中粪便链球菌科显著增加。不仅如此,圈养组中存在丰度较高的乳酸杆菌。有研究表明,粪便链球菌和乳酸杆菌的主要功能是参与淀粉类碳水化合物的水解[35, 40],圈养组马匹乳酸杆菌和链球菌相对丰度的增加,可能与饲粮中添加了淀粉类谷物有关,这与其他一些体外和体内研究得出的结果一致[41-42]。
3.3 不同饲养模式下哈萨克马肠道菌群标志物种及菌群功能放牧组中的标志物种(瘤胃球菌、梭菌、罗氏菌、粪球菌、普雷沃氏菌、假丁酸弧菌、甲基杆菌等)均与降解/发酵纤维素有关[33-39]。Baniel等[43]在分析食性变化对狮尾狒(灵长类中唯一以草为食的种类)肠道菌群的影响时发现,以牧草为食的狮尾狒肠道中以纤维素分解/发酵细菌(梭菌、粪球菌、普雷沃氏菌科和瘤胃球菌科的两个菌属)为主。圈养组中的标志物种为粪便链球菌、乳酸杆菌、WCHB1_25和韦荣氏球菌科的RFN20等。Arnold[44]在饲喂谷物类淀粉的马匹中发现,在消化道前段没有被消化的玉米、燕麦、大麦和高粱等谷物淀粉到达后肠后,被马后肠道的链球菌和乳酸杆菌等转化为乳酸[45],乳酸继续被韦荣氏球菌等转化为丙酸[46],丙酸被盲肠壁吸收后为机体提供能量。这些研究结果可以为筛选不同饲养模式下标志物种提供一定的理论依据。
Baniel等[43]得出当狮尾狒处于温度较低的旱季(以干草等为食)时,菌群功能变化倾向于能量、氨基酸和脂质等代谢途径。本研究结果显示,圈养组和放牧组的马匹菌群功能倾向于碳水化合物代谢、氨基酸代谢、脂质代谢以及能量代谢等。在放牧组中能量代谢和萜类化合物和聚酮化合物的代谢显著富集,放牧组马匹在野外需要应对食物匮乏和风雪天气,因此放牧组有着更高的能量需求,这些功能途径的增加可以使放牧组马匹在具有挑战性的寒冷季节维持能量和温度调节。传染病这一功能途径在圈养组中显著富集,使马匹有患肠道疾病的可能,这很有可能与圈养组马匹补喂了谷物饲料有关[47],摄入的浓缩物、谷物以及淀粉,会引起肠道疼痛,增加肠道疾病的风险[48-49]。
4 结论本研究通过16S rRNA基因测序分析,对冬季不同饲养模式下的哈萨克马肠道菌群进行研究,发现哈萨克马在放牧与圈养条件下,肠道菌群的多样性及组成结构存在整体性差异。哈萨克马的肠道菌群是可塑的,其可以对饲养模式等变化做出快速的波动性反应。这些研究结果有助于研究人员进一步理解哈萨克马肠道菌群多样性与饲养模式之间的关系。
| [1] |
赵红艳, 孙泽威. 马属动物肠道微生物的研究进展[J]. 经济动物学报, 2014, 18(1): 59-62. ZHAO H Y, SUN Z W. Advances on microorganisms in intestine of equine[J]. Journal of Economic Animal, 2014, 18(1): 59-62. (in Chinese) |
| [2] |
MAYNARD C L, ELSON C O, HATTON R D, et al. Reciprocal interactions of the intestinal microbiota and immune system[J]. Nature, 2012, 489(7415): 231-241. DOI:10.1038/nature11551 |
| [3] |
HUANG W C, CHEN Y H, CHUANG H L, et al. Investigation of the effects of microbiota on exercise physiological adaption, performance, and energy utilization using a gnotobiotic animal model[J]. Front Microbiol, 2019, 10: 1906. DOI:10.3389/fmicb.2019.01906 |
| [4] |
赵晗旭. 不同野生动物肠道微生物多样性分析及功能初步研究[D]. 长春: 吉林农业大学, 2015. ZHAO H X. Study on the diversity and function of intestinal bacterial flora from different wild animals[D]. Changchun: Jilin Agricultural University, 2015. (in Chinese) |
| [5] |
青河县人民政府. 青河简介[EB/OL]. [2021-06-15]. http://www.xjqh.gov.cn/zjqh/003001/20181003/60ad72ab-48ec-46c1-86a4-479dbfd2ac97.html. Qinghe County People's Government. Introduction to Qinghe[EB/OL]. [2021-06-15]. http://www.xjqh.gov.cn/zjqh/003001/20181003/60ad72ab-48ec-46c1-86a4-479dbfd2ac97.html. (in Chinese) |
| [6] |
喀纳斯景区管理委员会. 新疆喀纳斯湖风景名胜区总体规划(2020-2035)[EB/OL]. [2020-07-10]. http://www.kns.gov.cn/govxxgk/001/2021-07-10/5213dbe4-fc5c-42ee-8bd1-4b1bbfb2ea4c.html. Kanas Scenic Spot Management Committee. Overall planning of Kanas lake scenic resort in Xinjiang (2020-2035)[EB/OL]. [2020-07-10]. http://www.kns.gov.cn/govxxgk/001/2021-07-10/5213dbe4-fc5c-42ee-8bd1-4b1bbfb2ea4c.html. (in Chinese) |
| [7] |
丁丽媛, 杜拉提·卡衣马尔旦, ИСХАН К, 等. 哈萨克马遗传资源特征研究[J]. 黑龙江畜牧兽医, 2018(3): 91-94. DING L Y, DULATI K, ИСХАН К, et al. Study on the characteristics of genetic resources of Kazakh horse[J]. Heilongjiang Animal Science and Veterinary Medicine, 2018(3): 91-94. (in Chinese) |
| [8] |
赵天佐. 哈萨克马[J]. 新疆农业科学, 1991(5): 222-223. ZHAO T Z. Kazakh horses[J]. Xinjiang Agricultural Sciences, 1991(5): 222-223. (in Chinese) |
| [9] |
邢振存. 蒙古马和纯血马肠道真菌多样性初步研究及分析[D]. 呼和浩特: 内蒙古农业大学, 2016. XING Z C. Mongolian horse and thoroughbred intestinal fungal diversity preliminary research and analysis[D]. Hohhot: Inner Mongolia Agricultural University, 2016. (in Chinese) |
| [10] |
伊如格勒图. 蒙古马肠道细菌多样性的研究[D]. 呼和浩特: 内蒙古农业大学, 2015. YIRUGELETU. Analysis of intestinal bacteria diversity in Mongolian horse[D]. Hohhot: Inner Mongolia Agricultural University, 2015. (in Chinese) |
| [11] |
苏少锋. 蒙古马胃肠道细菌群落组成及纤维素分解菌的研究[D]. 呼和浩特: 内蒙古农业大学, 2019. SU S F. Characterization of gastrointestinal bacterial community and cellulose-decomposing bacteria in Mongolian horses[D]. Hohhot: Inner Mongolia Agricultural University, 2019. (in Chinese) |
| [12] |
白娜. 内蒙古锡林郭勒牧区健康家畜肠道菌群多样性研究[D]. 呼和浩特: 内蒙古农业大学, 2014. BAI N. Diversity of intestinal microbiota of healthy pastoral livestock in Inner Mongolia Xilin Gol[D]. Hohhot: Inner Mongolia Agricultural University, 2014. (in Chinese) |
| [13] |
李晓斌, 赵国栋, 刘振, 等. 3~6月龄伊犁马肠道微生物群落多样性的研究[J]. 动物营养学报, 2017, 29(5): 1535-1544. LI X B, ZHAO G D, LIU Z, et al. A study on intestinal microbiota diversity of 3- to 6-month-old Yili horses[J]. Chinese Journal of Animal Nutrition, 2017, 29(5): 1535-1544. (in Chinese) |
| [14] |
李晓斌, 李海, 臧长江, 等. 断奶前马驹与母马粪便菌群多样性分析[J]. 中国畜牧兽医, 2020, 47(4): 1050-1057. LI X B, LI H, ZANG C J, et al. Diversity analysis of fecal flora on pre-weaning foal and mare[J]. China Animal Husbandry & Veterinary Medicine, 2020, 47(4): 1050-1057. (in Chinese) |
| [15] |
DE FOMBELLE A, VEIGA L, DROGOUL C, et al. Effect of diet composition and feeding pattern on the prececal digestibility of starches from diverse botanical origins measured with the mobile nylon bag technique in horses[J]. J Anim Sci, 2004, 82(12): 3625-3634. DOI:10.2527/2004.82123625x |
| [16] |
HALE C, HEMMINGS A, ADJOU K. Effets de la levure vivante Actisaf Sc47 mélangée à l'aliment sur le comportement du cheval[J]. La Semaine Vétérinaire, 2014, 1576. |
| [17] |
YODER M, MILLER E, ROOK J, et al. Fiber level and form: Effects on digestibility, digesta flow and incidence of gastrointestinal disorders[C]//Proceedings of the Fifteenth Equine Nutrition & Physiology Symposium. Savoy: Equine Nutrition & Physiology Society, 1997: 122-127.
|
| [18] |
DE FOMBELLE A, VARLOUD M, GOACHET A G, et al. Characterization of the microbial and biochemical profile of the different segments of the digestive tract in horses given two distinct diets[J]. Anim Sci, 2003, 77(2): 293-304. DOI:10.1017/S1357729800059038 |
| [19] |
DE FOMBELLE A, JULLIAND V, DROGOUL C, et al. Feeding and microbial disorders in horses: 1-effects of an abrupt incorporation of two levels of barley in a hay diet on microbial profile and activities[J]. J Equine Vet Sci, 2001, 21(9): 439-445. DOI:10.1016/S0737-0806(01)70018-4 |
| [20] |
ANTWIS R E, LEA J M D, UNWIN B, et al. Gut microbiome composition is associated with spatial structuring and social interactions in semi-feral Welsh Mountain ponies[J]. Microbiome, 2018, 6(1): 207. DOI:10.1186/s40168-018-0593-2 |
| [21] |
REN T T, GRIENEISEN L E, ALBERTS S C, et al. Development, diet and dynamism: Longitudinal and cross-sectional predictors of gut microbial communities in wild baboons[J]. Environ Microbiol, 2016, 18(5): 1312-1325. DOI:10.1111/1462-2920.12852 |
| [22] |
李莉莉, 于广利. 肠道微生物组: 见微知著的第三次医学革命[M]. 北京: 科学出版社, 2021. LI L L, YU G L. The gut microbiome: see the third revolution in medicine[M]. Beijing: Science Press, 2021. (in Chinese) |
| [23] |
丁丽媛. 哈萨克马表型及微卫星遗传多样性研究[D]. 阿拉尔: 塔里木大学, 2017. DING L Y. Study on the diversity of Kazakh horses phenotype and microsatellite inheritance[D]. Alaer: Tarim University, 2017. (in Chinese) |
| [24] |
周军. 哈萨克马新品系(品种)培育及产品开发利用[J]. 山东畜牧兽医, 2020, 41(12): 24-25. ZHOU J. Breeding and product development and utilization of new Kazakh horse strains(varieties)[J]. Shandong Journal of Animal Science and Veterinary Medicine, 2020, 41(12): 24-25. (in Chinese) |
| [25] |
DOUGAL K, DA LA FUENTE G, HARRIS P A, et al. Identification of a core bacterial community within the large intestine of the horse[J]. PLoS One, 2013, 8(10): e77660. DOI:10.1371/journal.pone.0077660 |
| [26] |
JULLIAND V, DE FOMBELLE A, DROGOUL C, et al. Feeding and microbial disorders in horses: Part 3-effects of three hay: Grain ratios on microbial profile and activities[J]. J Equine Vet Sci, 2001, 21(11): 543-546. DOI:10.1016/S0737-0806(01)70159-1 |
| [27] |
WILLING B, VÖRÖS A, ROOS S, et al. Changes in faecal bacteria associated with concentrate and forage-only diets fed to horses in training[J]. Equine Vet J, 2009, 41(9): 908-914. |
| [28] |
SHEPHERD M L, SWECKER W S JR, JENSEN R V, et al. Characterization of the fecal bacteria communities of forage-fed horses by pyrosequencing of 16S rRNA V4 gene amplicons[J]. FEMS Microbiol Lett, 2012, 326(1): 62-68. DOI:10.1111/j.1574-6968.2011.02434.x |
| [29] |
BRULC J M, ANTONOPOULOS D A, MILLER M E B, et al. Gene-centric metagenomics of the fiber-adherent bovine rumen microbiome reveals forage specific glycoside hydrolases[J]. Proc Natl Acad Sci USA, 2009, 106(6): 1948-1953. |
| [30] |
BERNINI L J, SIMÃO A N C, ALFIERI D F, et al. Beneficial effects of Bifidobacterium lactis on lipid profile and cytokines in patients with metabolic syndrome: A randomized trial.Effects of probiotics on metabolic syndrome[J]. Nutrition, 2016, 32(6): 716-719. |
| [31] |
BERRY D. The emerging view of Firmicutes as key fibre degraders in the human gut[J]. Environ Microbiol, 2016, 18(7): 2081-2083. |
| [32] |
SPENCE C, WELLS W G, SMITH C J, et al. Characterization of the primary starch utilization operon in the obligate anaerobe Bacteroides fragilis: regulation by carbon source and oxygen[J]. J Bacteriol, 2006, 188(13): 4663-4672. |
| [33] |
WOOD T M. Cellulase of Ruminococcus albus[J]. Methods Enzymol, 1988, 160: 216-221. |
| [34] |
EZAKI T. Ruminococcus[M]. New York: John Wiley & Sons Ltd., 2015.
|
| [35] |
PATRA A K, YU Z T. Essential oils affect populations of some rumen bacteria in vitro as revealed by microarray (RumenBactArray) analysis[J]. Front Microbiol, 2015, 6: 297. |
| [36] |
ZHAO X H, CHEN Z D, ZHOU S, et al. Effects of daidzein on performance, serum metabolites, nutrient digestibility, and fecal bacterial community in bull calves[J]. Anim Feed Sci Technol, 2017, 225: 87-96. |
| [37] |
SHINKAI T, UEKI T, KOBAYASHI Y. Detection and identification of rumen bacteria constituting a fibrolytic consortium dominated by Fibrobacter succinogenes[J]. Anim Sci J, 2010, 81(1): 72-79. |
| [38] |
杨明, 袁悦, 李宪臻, 等. 不同环境中纤维素降解菌群多样性差异分析[J]. 江西农业大学学报, 2020, 42(1): 174-186. YANG M, YUAN Y, LI X Z, et al. Differential analysis of diversity in cellulose-decomposing bacterial communities from diverse environments[J]. Acta Agriculturae Universitatis Jiangxiensis, 2020, 42(1): 174-186. (in Chinese) |
| [39] |
PEDERSEN H K, GUDMUNDSDOTTIR V, NIELSEN H B, et al. Human gut microbes impact host serum metabolome and insulin sensitivity[J]. Nature, 2016, 535(7612): 376-381. |
| [40] |
SUN B H, WANG X, BERNSTEIN S, et al. Marked variation between winter and spring gut microbiota in free-ranging Tibetan Macaques (Macaca thibetana)[J]. Sci Rep, 2016, 6: 26035. |
| [41] |
李倩, 黄新新, 张文杰, 等. 不同谷物淀粉对断奶马驹盲肠菌群多样性的影响[J]. 动物营养学报, 2022, 34(1): 488-499. LI Q, HUANG X X, ZHANG W J, et al. Effects of cereal starches on cecal microflora diversity of weaned foals[J]. Chinese Journal of Animal Nutrition, 2022, 34(1): 488-499. (in Chinese) |
| [42] |
VARLOUD M, FONTY G, ROUSSEL A, et al. Postprandial kinetics of some biotic and abiotic characteristics of the gastric ecosystem of horses fed a pelleted concentrate meal[J]. J Anim Sci, 2007, 85(10): 2508-2516. |
| [43] |
BANIEL A, AMATO K R, BEEHNER J C, et al. Seasonal shifts in the gut microbiome indicate plastic responses to diet in wild geladas[J]. Microbiome, 2021, 9(1): 26. |
| [44] |
ARNOLD F F. Prececal, postileal and total tract starch digestion in ponies fed corn, oats, barley or sorghum grain[D]. Texas: Texas A & M University, 1982.
|
| [45] |
MEDINA B, GIRARD I D, JACOTOT E, et al. Effect of a preparation of Saccharomyces cerevisiae on microbial profiles and fermentation patterns in the large intestine of horses fed a high fiber or a high starch diet[J]. J Anim Sci, 2002, 80(10): 2600-2609. |
| [46] |
HARLOW B E, LAWRENCE L M, FLYTHE M D. Diarrhea-associated pathogens, Lactobacilli and cellulolytic bacteria in equine feces: Responses to antibiotic challenge[J]. Vet Microbiol, 2013, 166(1-2): 225-232. |
| [47] |
赵芳芳, 王洪荣, 张营, 等. 谷物作为反刍动物能量饲料的营养特性及高谷物饲粮对消化道健康的影响[J]. 动物营养学报, 2020, 32(6): 2476-2482. ZHAO F F, WANG H R, ZHANG Y, et al. Nutritional characteristics of grain as energy feed for ruminants and effects of high-grain diet on gastrointestinal tract health[J]. Chinese Journal of Animal Nutrition, 2020, 32(6): 2476-2482. (in Chinese) |
| [48] |
KAYA G, SOMMERFELD-STUR I, IBEN C, et al. Risk factors of colic in horses in Austria[J]. J Anim Physiol Anim Nutr, 2009, 93(3): 339-349. |
| [49] |
GONÇALVES S, JULLIAND V, LEBLOND A. Risk factors associated with colic in horses[J]. Vet Res, 2002, 33(6): 641-652. |
(编辑 范子娟)



