乳脂是牛奶中主要营养成分之一,也是影响乳品质的重要指标,它含有人体所必需的脂肪酸及多种脂溶性维生素,是一种优质的油脂资源,为人体提供大量的营养物质与能量[1]。在生产中,乳脂主要用于制作黄油和酸奶[2]。乳脂合成是一个复杂的生物学过程,在反刍动物乳腺中,乳脂合成的1/2是来自奶牛每天的饲喂饮食,其余是由乳腺细胞自身合成[3]。在合成与分泌过程中,不同种类与数量的脂肪酸会形成不同分子量和饱和度的甘油三酯[4],而用于甘油三酯合成的脂肪酸的来源主要有两个途径:一种是乳腺上皮细胞的从头合成,另一种是从血液中吸收而来[5]。乳脂率的高低受到多种因素的影响,如遗传、饲养管理和营养等,其中参与乳脂合成的酶和转录因子的表达对乳脂合成具有极其重要的作用[6]。此外,许多重要基因也参与到脂质合成中,并形成一个复杂的基因调控网络[7]。随着分子生物学技术的发展,对乳脂合成的深入研究已经揭示了脂肪酸的从头合成、血液中脂肪酸的摄取、转运与去饱和、脂滴的形成、甘油三酯的合成与运输等过程中关键基因及信号通路的调控作用[8-9]。如今,牛奶中乳脂的含量不仅是乳品核心竞争力的重要指标,也是奶牛育种的目标性状,如何提高奶牛乳脂含量,一直是研究人员关注的热点[10]。
自从2006年完成牛的全基因组测序工作至今,国内外许多学者在与奶牛泌乳相关的转录组学、蛋白组学和代谢组学等方面开展了众多研究并取得了重要成果[11]。转录组测序(RNA-seq)是利用二代高通量测序技术更全面、快速地获取某一物种特定器官、组织或细胞在某一状态下的几乎所有的转录本,包括mRNA和一些非编码RNA[12]。自从发现RNA是基因组和蛋白质组之间的关键中间体以来,转录本鉴定和基因表达的量化一直是分子生物学的核心活动。目前,高通量测序技术已广泛应用于各种家畜组织差异表达基因的分析,例如在牛的胚胎[13]、体细胞[14]以及猪[15]、羊[16]等各种动物组织。同时,转录组技术已广泛应用于奶牛乳腺组织的研究,Dai等[17]使用RNA-seq转录组学分析鉴定了来自泌乳期和非泌乳期奶牛乳腺组织中的差异表达基因,Gao等[18]和Lin等[19]也利用转录组测序技术对奶牛乳腺组织进行了转录组分析。但有关于影响奶牛高、低乳脂率的泌乳机制方面的转录组研究相对较少。因此,通过RNA-seq阐明牛乳腺转录组对于挖掘影响奶牛乳脂相关的候选基因至关重要。
本研究对课题组前期基于高、低乳脂率(MFP)奶牛乳腺上皮细胞(BMECs)Illumina PE150测序结果中的mRNA表达谱数据进行综合分析,探索与MFP相关的标志性基因和重要功能富集途径,为理解奶牛乳脂合成过程中复杂的生物学过程和机制提供理论依据。
1 材料与方法 1.1 数据来源本研究中14 561条mRNA表达谱数据来源于课题组前期基于高、低MFP BMECs Illumina PE150的测序结果[20]。试验样本来自宁夏农垦贺兰山奶业茂盛牧场,试验牛饲喂相同的平衡全混合日粮(表 1)。选取饲养背景一致,月龄相近,处于泌乳中后期的荷斯坦奶牛245头。采集每头牛早、中、晚班次的乳样进行奶牛生产性能测定(DHI),筛选体细胞数在100 000个·mL-1以内且MFP具有极端差异的荷斯坦奶牛各4头(表 2)。无菌采集新鲜乳液分离BMECs[21],待细胞鉴定完成[22]后由北京诺禾致源科技股份有限公司进行细胞总RNA提取、文库构建和转录组测序[20]。
|
|
表 1 日粮成分及营养成分(以干物质为基础)百分比 Table 1 Ingredient and nutrient composition in diet (dry matter basis) |
|
|
表 2 荷斯坦牛高低乳脂率和SCC Table 2 High and low MFP and SCC of Holstein cattle |
基于课题组前期14 561条mRNA表达谱数据,将P < 0.05和|log2FoldChange|≥1.5设置为差异表达基因筛选的阈值,使用在线网站KOBAS(http://kobas.cbi.pku.edu.cn)数据库对所有差异表达基因进行GO功能注释和KEGG通路富集分析,再以P < 0.05为显著性阈值筛选显著富集的GO条目和KEGG通路。利用北京诺禾致源科技股份有限公司提供的在线售后工具平台(https://magic.novogene.com/customer/main#/home/2d9dc26d1e059b931b9ac5364)对mRNA表达谱数据进行相关性分析、表达量热图聚类、火山图分析等。使用String数据库[23]进行差异表达基因的蛋白互作网络分析。
1.3 细胞及组织总RNA提取、质量检测及cDNA合成使用课题组前期保存的高、低乳脂组奶牛BMECs的cDNAs验证转录组数据的准确性。奶牛的小肠、肝、乳腺、心、子宫、肾、卵巢组织均为课题组前期采集(3头泌乳中期的荷斯坦奶牛)。按照Trizol试剂盒(TaKaRa,大连)使用说明提取奶牛不同组织总RNA[24]。使用紫外可见光分光光度计(Eppendorf BioSpectrometer basic)进行浓度和纯度检测,总RNA完整性利用1%琼脂糖凝胶电泳进行检测。挑取完整性及纯度较好的RNA利用诺唯赞生物公司反转录试剂盒将其反转录为cDNAs,反转录完成后置于-20 ℃冰箱保存备用。
1.4 差异表达基因实时荧光定量PCR验证1.4.1 引物设计与合成 随机选择8个差异表达基因(LIPG、DGKI、FABP4、PLA2G16、PRKCA、ARAF、PDE4D、KCNMA1)及筛选出的脂代谢相关关键候选差异表达基因(ID2、PRKAA2、FABP4、ADCY5),以牛的GAPDH基因为内参进行qRT-PCR。根据NCBI上查找到的基因序列,运用Primer5.0设计荧光定量引物(表 3),由陕西中科羽瞳生物公司代为合成。
|
|
表 3 引物序列及退火温度 Table 3 Primer sequence and annealing temperature |
1.4.2 荧光定量PCR 采用实时荧光定量PCR验证基因的表达水平。每个样本设置3个技术重复,按照艾科瑞生物公司的产品SYBR Green Pro Taq HS预混型qPCR试剂盒进行检测,qRT-PCR反应体系:ddH2O 8 μL,2×SYBR Green PCR Master Mix 10 μL,cDNA 1 μL,上、下游引物各0.5 μL。qRT-PCR反应程序:95 ℃预变性30 s;95 ℃变性5 s,退火30 s,60 ℃延伸5 s,40个循环。
1.5 数据分析用Bio-Rad CFX Manager3.1软件收集荧光定量结果的数据,通过Microsoft Excel 2021对数据进行整理。不同组织基因的相对表达水平采用2-ΔΔCt法进行分析,结果以“平均值±标准差”来表示。最后利用SAS8.2软件进行统计分析,P < 0.05认为具有生物统计学意义。
2 结果 2.1 样本相关性分析及差异表达基因筛选和热图聚类样品间基因表达水平相关性是检验试验可靠性和样本选择与否的重要指标。本研究根据各样本所有基因的FPKM值,计算组内及组间样本的相关性系数,并绘制成热图(图 1A),图中可直观看出组内相关系数均在0.90以上,表明样品之间表达模式的相似度较高,同时组间样本也存在差异。本研究以|log2FoldChange|≥1.5、P < 0.05作为差异表达基因的筛选标准,结果如图 1C所示,高乳脂率组和低乳脂率组中共有578个差异表达mRNA,其中高乳脂组(试验组)有332个基因显著上调(低乳脂组为对照组),246个基因显著下调。聚类热图是差异表达基因的另一种展示方式,如图 1B所示,表达模式相近的基因被聚在一起,这些基因可能具有共同的功能或参与到共同的代谢途径。
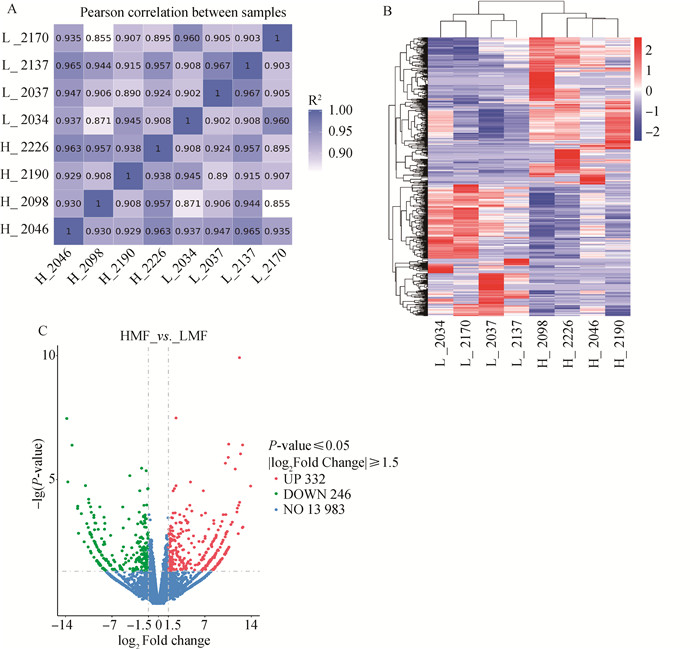
|
A.各样本相关性分析;B. 差异表达基因聚类热图;C. 差异表达基因火山图(HMF_VS_LMF) A. Pearson correlation between samples; B. Heat map of clustering for differentially expressed genes; C. Differentially expressed genes volcano map(HMF_VS_LMF) 图 1 各样本相关性分析及差异表达基因分析 Fig. 1 Pearson correlation between samples and analysis of differentially expressed genes |
对578个差异表达基因进行GO和KEGG富集分析,结果共显著富集到366个GO条目,包含224个生物学过程(BP),58个细胞组分(CC)及84个分子功能(MF)。图 2A中列出了显著富集到与脂代谢相关的前30个GO条目,其中与乳脂代谢关系最为密切的条目是中长链脂肪酸的运输、乳腺肺泡发育、花生四烯酸的结合和磷脂酰肌醇-4, 5-二磷酸结合。利用在线数据库KOBAS(http://kobas.cbi.pku.edu.cn)对差异表达基因进行KEGG富集分析,结果发现有47个信号通路被显著富集(P < 0.05),图 2B中列出了与脂代谢相关的前15的信号通路,其中与脂代谢相关的代谢通路为Apelin信号通路、Hippo信号通路、脂肪细胞中脂解的调节及MAPK信号通路等。之后结合GO和KEGG富集分析结果,最终筛选出可能调控乳脂代谢的4个候选差异表达基因,即ID2、PRKAA2、FABP4和ADCY5。其中ADCY5、PRKAA2基因表达下调,ID2、FABP4基因表达上调。值得关注的是,PRKAA2和FABP4基因显著富集至脂代谢相关BP和MF,同时富集在脂代谢相关KEGG通路中(图 3A、B),提示其参与调控乳脂代谢的可能性更大(表 4)。
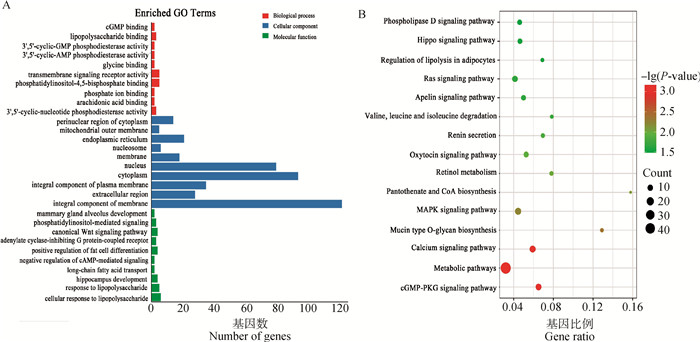
|
A.与脂代谢相关的差异表达基因富集分析中前30个 GO 条目;B.与脂代谢相关的差异表达基因富集分析中前15条 KEGG 通路图 A. The top 30 GO terms in the enrichment analysis of differentially expressed genes in lipid metabolism; B. The top 15 KEGG pathways in the enrichment analysis of differentially expressed genes in lipid metabolism 图 2 差异表达基因GO及KEGG代谢通路富集分析 Fig. 2 Enrichment analysis of GO and KEGG metabolic pathways of differentially expressed genes |

|
A. Apelin 信号通路;B.调节脂肪细胞的脂分解信号通路 A. Apelin signaling pathway; B. Regulation of lipolysis in adipocytes 图 3 脂代谢相关通路 Fig. 3 Lipid metabolism related pathways |
|
|
表 4 与脂代谢相关的差异表达基因汇总 Table 4 Summary of differential genes associated with lipid metabolism |
对筛选出可能调控乳脂代谢的差异表达基因ID2、PRKAA2、FABP4、ADCY5分别进行蛋白互作分析,结果如图 4所示,蛋白互作网络图中各有10个节点,置信度为0.7(高等),线的粗细表示了数据支撑的强度。从图中可以看出,ID2、PRKAA2、FABP4基因与多个脂代谢相关基因存在较强的互作关系,在调控乳脂代谢方面它们可能共同扮演着重要的角色,ADCY5基因与调控乳脂代谢方面的基因互作较少,数据支撑相对较少,但仍然具有一定的参考价值。
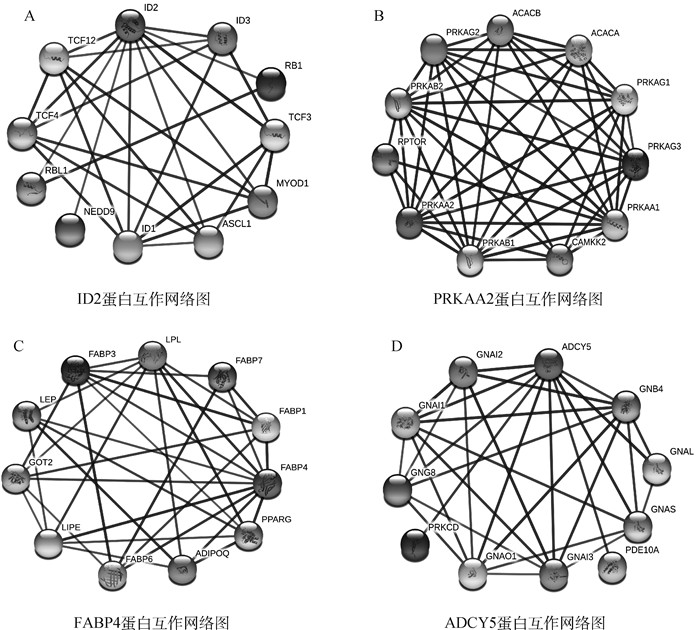
|
图 4 差异表达基因蛋白网络互作 Fig. 4 Differentially expressed genes protein network interaction |
各组织样本总RNA的提取质量会影响到后续实时荧光定量的结果,经1%的琼脂糖凝胶电泳检测后,如图 5所示,1~7分别是奶牛的小肠、肝、乳腺、心、子宫、肾、卵巢7个组织RNA凝胶电泳检测,可以清晰看到两条较明显的条带分别为28S、18S,并无明显降解,表明样品提取的总RNA的完整性较好。使用核酸浓度检测仪检测总RNA的OD260nm/OD280nm值均在1.8~2.0之间,说明RNA纯度较高可满足后续试验的要求。
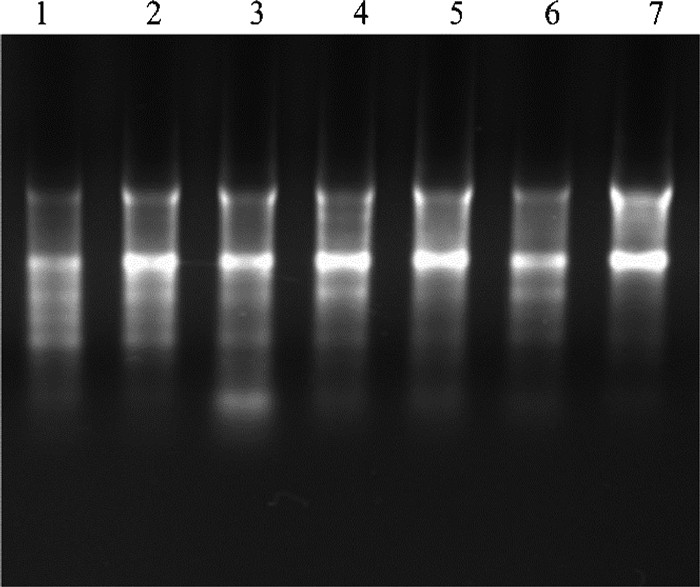
|
1~7. 奶牛小肠、肝、乳腺、心、子宫、肾、卵巢不同组织的RNA条带 1-7. RNA bands extracted from small intestine, liver, breast, heart, uterus, kidney and ovary of dairy cows, respectively 图 5 不同组织RNA提取电泳图 Fig. 5 Electrophoresis of RNA extracted from different tissues |
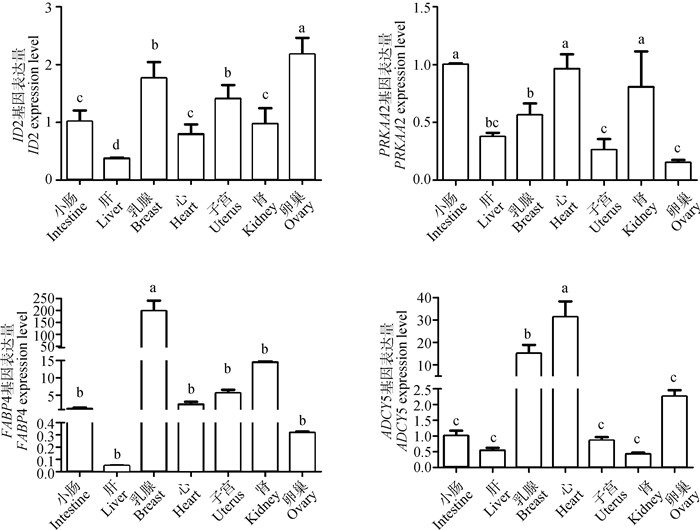
对ID2、PRKAA2、FABP4、ADCY5基因分别进行不同组织的qRT-PCR扩增,定量结果如图 6所示,所有基因的扩增曲线均呈现S型且具有指数增长期和平台期,均在15~30个循环之间起峰,表明扩增效率较高。基因的熔解曲线均为单峰,且起峰迅速形状狭窄,说明不存在非特异性片段,扩增特异性较好。qRT-PCR结果发现,以小肠作为参照,ID2基因在7个组织中都有表达,表达水平从高到低依次为卵巢、乳腺、子宫、小肠、肾、心及肝。其中,在卵巢组织中表达水平显著高于其他组织,在肝中的表达水平最低。PRKAA2基因在小肠中表达水平最高,与心和肾差异不显著,与其余组织表达水平差异显著(P < 0.05),FABP4在乳腺组织中表达水平最高,与其他组织差异显著(P < 0.05),其他组织之间的表达量差异不显著。ADCY5基因在心中表达水平最高,乳腺组织次之,与其余组织之间表达水平差异显著(P < 0.05, 图 7)。

|
图 6 Real-time PCR的熔解曲线和扩增曲线 Fig. 6 Melting curves and amplification curves of real-time PCR |
|
肩标不同字母表示差异显著(P < 0.05),相同小写字母表示差异不显著(P>0.05) Different letters on the shoulders means significant difference between the treatments(P < 0.05), same letter on the shoulders indicate not significant difference between treatments(P>0.05) 图 7 候选基因组织表达谱 Fig. 7 Tissue expression profiles of candidate genes |
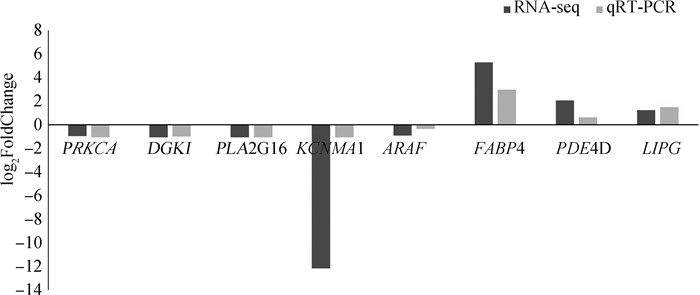
本研究随机选择了8个差异表达基因,在高、低组试验样品中(3个重复)运用qRT-PCR分析了它们的相对表达水平,通过log2FoldChange对差异倍数进行转换。结果如图 8所示,3个基因(FABP4、PDE4D、LIPG)表达上调,5个基因(PRKCA、DGKI、PLA2G16、KCNMA1、ARAF)表达下调。qRT-PCR试验结果与RNA-seq测序结果均表现一致,证实了转录组测序结果的可靠性。
|
图 8 qRT-PCR验证差异表达基因 Fig. 8 The differentially expressed genes confirmed by qRT-PCR |
乳脂是乳品质调控的主要目标性状,也是反映奶产品行业经济效益的主要指标之一。本研究以P < 0.05和|log2FoldChange|≥1.5为阈值,在荷斯坦奶牛高、低乳脂组之间共发现578个差异表达基因,其中上调基因332个,下调基因246个。功能富集分析发现,差异表达基因显著富集于长链脂肪酸的运输、磷脂酰肌醇介导的信号及乳腺肺泡的发育等GO条目。此外,与脂代谢密切相关的通路有15条,包括脂肪细胞脂解的调节、磷脂酶D信号通路、河马信号通路及Apelin信号通路等。在Apelin信号通路中,ADCY5基因通过对cAMP的调控间接对抑制脂质分解起到促进的作用[25]。PRKAA2基因通过AMPK信号通路间接促进脂肪的分解及褐色脂肪的形成[26]。FABP4参与甘油酯代谢,生成脂肪酸[27]。这说明ADCY5、PRKAA2及FABP4基因的表达与脂代谢密切相关。此外,利用String蛋白质互作数据库对候选基因蛋白进行蛋白互作分析,ID2、PRKAA2、FABP4基因与多个脂代谢相关基因存在较强的互作关系,它们在调控乳脂代谢方面发挥着相似的功能[28-29]。因此,本研究选取ID2、PRKAA2、FABP4、ADCY5基因作为乳脂代谢关键功能候选基因。
ID2目前在牛上未见报道,Liu等[30]研究发现ID2是鉴定DCIS(导管原位癌)非侵袭性乳腺癌起始的关键基因,在本研究中其在奶牛乳腺中的表达水平较高,显著高于小肠、肝、心和肾。PRKKA2基因对秦川牛部分体尺及肉质性状有显著影响,可作为秦川肉牛新品种早期选择的候选基因和分子辅助标记[31]。Khan等[32]研究发现,在奶牛泌乳早期,AMPK的完全激活需要PRKAA1和PRKAA2[33],PRKAA2表达的变化表明其参与了脂解的过程,脂肪生成的速率降低,从而刺激脂解的作用增加。此外,在牦牛泌乳周期中,PRKAA2在整个哺乳期内上调[34-35]。蛋白互作分析进一步发现,PRKAA2与ACACA、ACACB、PRKAA1、CAMKK2基因均有互作,这些都是与脂代谢密切相关的基因[36-37]。本研究发现,PRKAA2在乳腺中的表达水平处于中间水平,但其在乳脂合成中的作用是不可忽视的。Cai等[38]的研究表明,奶牛产奶量与生育力之间存在着遗传负相关性,使得在奶牛的育种中难以同时提高产奶量和生育力,奶牛产奶性状的快速改善伴随着奶牛生育能力的下降,这种相关性的遗传基础仍然知之甚少。ADCY5作为奶牛生育能力的候选基因,有助于剖析母牛生育能力的遗传学[39]。脂肪酸结合蛋白(fatty acid binding protein,FABP)可与长链脂肪酸结合,并将脂肪酸从质膜转运到脂肪酸氧化位点及其与甘油三酯和磷酸的合成位点,调节细胞内脂肪的合成与转运,并和其他脂质类调节因子调节细胞内信号通路[40-41]。在奶牛的乳腺组织中,FABP家族的FABP3、FABP4及FABP5表达量最高[42]。其中,脂肪酸结合蛋白4(FABP4)最早被Kacey等[43]在脂肪细胞中发现,是畜禽脂肪沉积与代谢的热门候选基因。本研究发现,FABP4在乳腺组织中的表达尤为突出,显著高于其它组织,但目前FABP4在奶牛乳脂合成中的作用机制还不明确,对其进行乳脂合成方面的深入探究是非常有必要的。
4 结论本研究共筛选到578个差异表达基因,其中上调基因有332个,下调基因有246个。通过功能富集分析和组织表达谱分析得到了4个乳脂代谢相关的关键候选基因,分别为ID2、PRKAA2、FABP4、ADCY5。本研究为阐明奶牛乳脂代谢的分子调控机制和调控网络提供了基础资料,也为荷斯坦奶牛分子选育工作提供了素材。
| [1] |
杨永新, 王加启, 卜登攀, 等. 牛乳重要营养品质特征的研究进展[J]. 食品科学, 2013, 34(1): 328-332. YANG Y X, WANG J Q, BU D P, et al. A review on research progress in key nutritional characteristics of milk[J]. Food Science, 2013, 34(1): 328-332. (in Chinese) |
| [2] |
CHEN Z, CHU S F, WANG X L, et al. MicroRNA-106b regulates milk fat metabolism via ATP binding cassette subfamily A member 1 (ABCA1) in bovine mammary epithelial cells[J]. J Agric Food Chem, 2019, 67(14): 3981-3990. DOI:10.1021/acs.jafc.9b00622 |
| [3] |
BAUMAN D E, GRⅡNARI J M. Nutritional regulation of milk fat synthesis[J]. Annu Rev Nutr, 2003, 23: 203-227. DOI:10.1146/annurev.nutr.23.011702.073408 |
| [4] |
MU T, HU H H, MA Y F, et al. Regulation of key genes for milk fat synthesis in ruminants[J]. Front Nutr, 2021, 8: 765147. DOI:10.3389/fnut.2021.765147 |
| [5] |
KLIEM K E, SHINGFIELD K J. Manipulation of milk fatty acid composition in lactating cows: opportunities and challenges[J]. Eur J Lipid Sci Technol, 2016, 118(11): 1661-1683. DOI:10.1002/ejlt.201400543 |
| [6] |
XIA L X, ZHAO Z H, YU X Z, et al. Integrative analysis of miRNAs and mRNAs revealed regulation of lipid metabolism in dairy cattle[J]. Funct Integr Genomics, 2021, 21(3-4): 393-404. DOI:10.1007/s10142-021-00786-9 |
| [7] |
杨仁辉, 余烨, 陶杨, 等. 奶牛乳脂合成分泌基因研究进展[J]. 中国畜牧杂志, 2021, 57(11): 11-15. YANG R H, YU Y, TAO Y, et al. Advances in the genes related to milk fat synthesis and secretion of dairy cows[J]. Chinese Journal of Animal Science, 2021, 57(11): 11-15. DOI:10.19556/j.0258-7033.20201119-01 (in Chinese) |
| [8] |
BIONAZ M, LOOR J J. Gene networks driving bovine milk fat synthesis during the lactation cycle[J]. BMC Genomics, 2008, 9: 366. DOI:10.1186/1471-2164-9-366 |
| [9] |
BERNARD L, LEROUX C, CHILLIARD Y. Expression and nutritional regulation of lipogenic genes in the ruminant lactating mammary gland[M]//BÖSZE Z. Bioactive Components of Milk. New York: Springer, 2008: 67-108.
|
| [10] |
王影, 文亮, 母童, 等. 荷斯坦牛高、低乳脂率牛乳代谢组分析[J]. 畜牧兽医学报, 2022, 53(5): 1396-1408. WANG Y, WEN L, MU T, et al. Metabolomic analysis of milk from Holstein cows with high and low milk fat percentage[J]. Acta Veterinaria et Zootechnica Sinica, 2022, 53(5): 1396-1408. (in Chinese) |
| [11] |
LI S S, WANG Q J, LIN X J, et al. The use of "Omics" in lactation research in dairy cows[J]. Int J Mol Sci, 2017, 18(5): 983. DOI:10.3390/ijms18050983 |
| [12] |
TAYLOR J F, WHITACRE L K, HOFF J L, et al. Lessons for livestock genomics from genome and transcriptome sequencing in cattle and other mammals[J]. Genet Sel Evol, 2016, 48(1): 59. DOI:10.1186/s12711-016-0237-6 |
| [13] |
CALVO L, BIRGAOANU M, PETTINI T, et al. The embryonic transcriptome of Parhyale hawaiensis reveals different dynamics of microRNAs and mRNAs during the maternal-zygotic transition[J]. Sci Rep, 2022, 12(1): 174. DOI:10.1038/s41598-021-03642-9 |
| [14] |
MANGIAVACCHI A, LIU P, VALLE F D, et al. New insights into the functional role of retrotransposon dynamics in mammalian somatic cells[J]. Cell Mol Life Sci, 2021, 78(13): 5245-5256. DOI:10.1007/s00018-021-03851-5 |
| [15] |
LI B J, LIU K Q, WENG Q N, et al. RNA-seq analysis reveals new candidate genes for drip loss in a Pietrain×Duroc×Landrace×Yorkshire population[J]. Anim Genet, 2016, 47(2): 192-199. DOI:10.1111/age.12401 |
| [16] |
SUN L M, BAI M, XIANG L J, et al. Comparative transcriptome profiling of longissimus muscle tissues from Qianhua Mutton Merino and Small Tail Han sheep[J]. Sci Rep, 2016, 6: 33586. DOI:10.1038/srep33586 |
| [17] |
DAI W T, ZOU Y X, WHITE R R, et al. Transcriptomic profiles of the bovine mammary gland during lactation and the dry period[J]. Funct Integr Genomics, 2018, 18(2): 125-140. DOI:10.1007/s10142-017-0580-x |
| [18] |
GAO Y Y, LIN X Y, SHI K R, et al. Bovine mammary gene expression profiling during the onset of lactation[J]. PLoS One, 2013, 8(8): e70393. DOI:10.1371/journal.pone.0070393 |
| [19] |
LIN Y, LV H, JIANG M H, et al. Functional analysis of the dairy cow mammary transcriptome between early lactation and mid-dry period[J]. J Dairy Res, 2019, 86(1): 63-67. DOI:10.1017/S0022029919000049 |
| [20] |
MU T, HU H H, FENG X F, et al. Screening and conjoint analysis of key lncRNAs for milk fat metabolism in dairy cows[J]. Front Genet, 2022, 13: 772115. DOI:10.3389/fgene.2022.772115 |
| [21] |
段安琴, 庞春英, 朱鹏, 等. 水牛乳汁中乳腺上皮细胞的分离培养及鉴定[J]. 中国畜牧兽医, 2017, 44(11): 3243-3249. DUAN A Q, PANG C Y, ZHU P, et al. Isolation, culture and identification of buffalo mammary epithelial cells from milk[J]. China Animal Husbandry & Veterinary Medicine, 2017, 44(11): 3243-3249. DOI:10.16431/j.cnki.1671-7236.2017.11.019 (in Chinese) |
| [22] |
SHEN B L, ZHANG L Y, LIAN C J, et al. Deep sequencing and screening of differentially expressed MicroRNAs related to milk fat metabolism in bovine primary mammary epithelial cells[J]. Int J Mol Sci, 2016, 17(2): 200. DOI:10.3390/ijms17020200 |
| [23] |
SZKLARCZYK D, GABLE A L, NASTOU K C, et al. The STRING database in 2021:customizable protein-protein networks, and functional characterization of user-uploaded gene/measurement sets[J]. Nucleic Acids Res, 2021, 49(D1): D605-D612. DOI:10.1093/nar/gkaa1074 |
| [24] |
RIO D C, ARES M JR, HANNON G J, et al. Purification of RNA using TRIzol (TRI reagent)[J]. Cold Spring Harb Protoc, 2010(6): pdb.prot5439. |
| [25] |
LV S H, QIU X C, LI J, et al. Glucagon-induced extracellular cAMP regulates hepatic lipid metabolism[J]. J Endocrinol, 2017, 234(2): 73-87. DOI:10.1530/JOE-16-0649 |
| [26] |
SIMINO L A P, PANZARIN C, FONTANA M F, et al. MicroRNA Let-7 targets AMPK and impairs hepatic lipid metabolism in offspring of maternal obese pregnancies[J]. Sci Rep, 2021, 11(1): 8980. DOI:10.1038/s41598-021-88518-8 |
| [27] |
LI Y H, ZHOU H T, CHENG L, et al. Effects of FABP4 variation on milk fatty-acid composition for dairy cattle grazed on pasture in late lactation[J]. J Dairy Res, 2020, 87(1): 32-36. DOI:10.1017/S0022029920000011 |
| [28] |
LIANG M Y, HOU X M, QU B, et al. Functional analysis of FABP3 in the milk fat synthesis signaling pathway of dairy cow mammary epithelial cells[J]. In Vitro Cell Dev Biol Anim, 2014, 50(9): 865-873. DOI:10.1007/s11626-014-9780-z |
| [29] |
HU X M, LUO P, PENG X W, et al. Molecular cloning, expression pattern analysis of porcine Rb1 gene and its regulatory roles during primary dedifferentiated fat cells adipogenic differentiation[J]. Gen Comp Endocrinol, 2015, 214: 77-86. DOI:10.1016/j.ygcen.2015.01.016 |
| [30] |
LIU Y, PANDEY P R, SHARMA S, et al. ID2 and GJB2 promote early-stage breast cancer progression by regulating cancer stemness[J]. Breast Cancer Res Treat, 2019, 175(1): 77-90. DOI:10.1007/s10549-018-05126-3 |
| [31] |
田万强, 梅楚刚, 付常振, 等. 牛PRKAA2基因SNP检测及与其生长和肉质性状的关联分析[J]. 西北农林科技大学学报: 自然科学版, 2016, 44(7): 1-9, 15. TIAN W Q, MEI C G, FU C Z, et al. SNPs of bovine PRKAA2 gene and its association with growth and meat quality traits[J]. Journal of Northwest A&F University (Natural Science Edition), 2016, 44(7): 1-9, 15. (in Chinese) |
| [32] |
KHAN M J, HOSSEINI A, BURRELL S, et al. Change in subcutaneous adipose tissue metabolism and gene network expression during the transition period in dairy cows, including differences due to sire genetic merit[J]. J Dairy Sci, 2013, 96(4): 2171-2182. DOI:10.3168/jds.2012-5794 |
| [33] |
CANTÓ C, AUWERX J. Calorie restriction: is AMPK a key sensor and effector?[J]. Physiology (Bethesda), 2011, 26(4): 214-224. |
| [34] |
XIA W, OSORIO J S, YANG Y X, et al. Short communication: characterization of gene expression profiles related to yak milk protein synthesis during the lactation cycle[J]. J Dairy Sci, 2018, 101(12): 11150-11158. DOI:10.3168/jds.2018-14715 |
| [35] |
BIONAZ M, LOOR J J. Gene networks driving bovine mammary protein synthesis during the lactation cycle[J]. Bioinform Biol Insights, 2011, 5: 83-98. |
| [36] |
孙成娟. 猪肉质性状相关基因FoxO4、PRKAA1的克隆表达及功能研究[D]. 贵阳: 贵州大学, 2017. SUN C J. Cloning expression and function analysis of FoxO4 and PRKAA1 genes related to pork quality traits[D]. Guiyang: Guizhou University, 2017. (in Chinese) |
| [37] |
LI D W, WANG H, LI Y M, et al. MicroRNA-378 regulates adipogenic differentiation in bovine intramuscular preadipocytes by targeting CaMKK2[J]. Adipocyte, 2021, 10(1): 483-492. DOI:10.1080/21623945.2021.1982526 |
| [38] |
CAI Z X, GULDBRANDTSEN B, LUND M S, et al. Prioritizing candidate genes for fertility in dairy cows using gene-based analysis, functional annotation and differential gene expression[J]. BMC Genomics, 2019, 20(1): 255. DOI:10.1186/s12864-019-5638-9 |
| [39] |
JIE L, SHEN C L, ZHANG K J, et al. Polymorphic variants of bovine ADCY5 gene identified in GWAS analysis were significantly associated with ovarian morphological related traits[J]. Gene, 2021, 766: 145158. DOI:10.1016/j.gene.2020.145158 |
| [40] |
OHBUCHI K, MORI Y, OGAWA K, et al. Detailed analysis of the binding mode of vanilloids to transient receptor potential vanilloid type I (TRPV1) by a mutational and computational study[J]. PLoS One, 2016, 11(9): e0162543. DOI:10.1371/journal.pone.0162543 |
| [41] |
LI B, HAO J Q, ZENG J, et al. SnapShot: FABP functions[J]. Cell, 2020, 182(4): 1066-1066.e1. DOI:10.1016/j.cell.2020.07.027 |
| [42] |
ZHOU H, CHENG L, AZIMU W, et al. Variation in the bovine FABP4 gene affects milk yield and milk protein content in dairy cows[J]. Sci Rep, 2015, 5: 10023. |
| [43] |
KACEY J P, JANI S, GÖKHAN S H. Adipokine FABP4 integrates energy stores and counterregulatory metabolic responses[J]. J Lipid Res, 2019, 60(4): 734-740. |
(编辑 郭云雁)



