2. 中国农业大学动物科学技术学院 畜禽育种国家工程实验室 农业农村部动物遗传育种与繁殖重点实验室, 北京 100193;
3. 河北省畜牧良种工作总站, 石家庄 050061;
4. 河南省畜牧总站, 郑州 450008
2. Key Laboratory of Animal Genetics And Breeding of Ministry of Agriculture and Rural Affairs, National Engineering Laboratory of Animal Breeding, College of Animal Science and Technology, China Agricultural University, Beijing 100193, China;
3. Hebei Province Animal Husbandry and Improved Breeds Work Station, Shijiazhuang 050061, China;
4. Henan Province Animal Husbandry Station, Zhengzhou 450008, China
单核苷酸多态性(SNP)是基因组上最常见的一种遗传变异,是由单个核苷酸突变引起,占所有已知多态性的90%以上,广泛存在于基因组中[1]。其具有密度高、分布范围广、遗传稳定、分型简单、可自动化检测等优点[2],被认为是目前最佳的分子标记。已广泛应用于全基因组关联分析[3](genome-wide association study)和基因组选择[4](genomic selection)等遗传分析和育种应用。尤其是基因组选择,高通量基因型检测是其实施的前提,正是由于2006年高通量SNP芯片的推出[5],基因组选择才正式应用于育种实践。SNP芯片标记密度对基因组选择准确性十分重要,然而高密度SNP芯片在大群体上的应用成本很高[6],为了降低成本,低密度SNP芯片应运而生,可通过基因型填充将其填充至高密度SNP芯片甚至测序数据,兼顾基因型检测成本和分子育种效率。在基因组选择育种实践中, 通过基因型填充技术,能有效地将低密度芯片填充到高密度水平,并保持相当高的填充准确率,不影响基因组选择准确性,从而大大提高了低密度芯片的使用价值和育种效果[7-8]。
基因型填充技术就是利用已有的分型信息对实际未分型的位点进行基因型预测[9]。目前,已经发展了很多基因型填充方法[10]。根据是否利用家系信息分为两种,一种是利用群体连锁不平衡信息构建单倍型,相应的软件有FAMHAP[11]、fastPHASE[12]、IMPUTE2[13]、Beagle[14]、PLINK[15]等;另一种是利用系谱信息和标记连锁信息构建单倍型,相应的软件有Find-hap[16]、Fimpute[17]、AlphaImpute[18]、PEDIMPUTE[19]等。其中Beagle主要利用隐性马尔科夫链技术,填充准确性高,应用广泛。本试验使用Beagle 4.1软件[20]进行填充。
液相芯片是继凝胶电泳、荧光检测、固相芯片之后的新型分子检测技术,该技术基于靶向测序基因型检测(GBTS)技术,因其能够使目标探针与靶向序列互补结合从而进行定点捕获,且能够在液相中同时快速完成成千上万个探针杂交反应的试剂盒,形象称之为液相芯片[21-22]。与传统的基因型检测技术和固相芯片相比,液相芯片具有平台广适性、标记灵活性、检测高效性、信息可加性、支撑便捷性和应用广谱性等优点[22-23],具有广阔的应用前景。中国农业大学先后开发了猪低密度SNP芯片(专利号:ZL201711190317.6)和猪50K液相芯片(专利申请号:202110359470.7),用于猪性状遗传解析和基因组选择。虽然从低密度SNP填充至高密度的研究很多[24],但主要以固相芯片为主,目前液相芯片基因型填充的研究并不多见。为此本研究利用中国农业大学开发的猪10K和50K液相芯片,研究液相芯片从10K到50K的基因型填充效果,为其他畜禽相关研究提供借鉴。
1 材料与方法 1.1 芯片数据本研究中3 761头猪只均来自河北大好河山养猪科技有限公司的健康大白群体,出生日期在2018—2021年间,日龄160 d左右,体重110 kg左右,采全血后,使用由中国农业大学开发的液相50K芯片(包含52 000个SNPs标记)进行基因型测定。
1.2 基因型质量控制采用PLINK软件对所有个体基因型数据进行质量控制,剔除性染色体和位置未知的位点及位点检出率(call rate) < 90%的SNP,剔除检出率 < 90%的个体。质量控制后,保留47 890个标记和全部个体。
1.3 基因型填充本试验使用Beagle4.1软件进行基因型填充。填充的流程如下:从质控后群体中随机抽取100头大白猪,从50K芯片中抽取标记生成10K芯片,作为填充群体。再从剩余群体中分别随机抽取800、2 000、3 600个个体作为参考群体,对100头填充群体进行基因型填充。相同流程重复10次。
基因型填充准确性是检验填充效率的一个重要指标。本研究用100头填充群体基因型填充后与原始50K基因型的一致性和相关系数衡量填充效果。基因型一致性是指正确填充的基因型占需要进行填充的基因型的比例[21]。基因型相关系数(Cor)是指推断的基因型和原始基因型之间的相关系数[25]。此外,Beagle 4.1软件计算出的理论填充准确性(DR2)也可作为参考[26]。
2 结果 2.1 10K和50K芯片的描述性统计表 1列出了质控后2款液相芯片18条常染色体的SNP标记数、平均间距和连锁不平衡水平统计。质控前液相10K和50K芯片18条常染色体共计9 823和49 886个位点,质控后分别有9 685和47 890个位点,位点的缺失率分别为0.40%和1.58%。如表 1所示,液相10K芯片的位点平均间距为226 394 bp,平均连锁不平衡(r2)为0.227;液相50K的平均位点间距为44 231 bp,平均连锁不平衡为0.258,说明10K与50K液相芯片相比,标记间距虽然从44 kb扩大到226 kb, 但是SNP标记间的连锁不平衡程度却几乎没有下降。虽然大部分染色体50K液相芯片的连锁不平衡程度高于10K芯片,但是8、12、18号染色体10K芯片的连锁不平衡程度高于50K芯片。
|
|
表 1 10K和50K液相芯片各染色体标记间距和连锁不平衡(r2)统计 Table 1 The markers distance and linkage disequilibrium (r2) of 10K and 50K SNP panels on each chromosome |
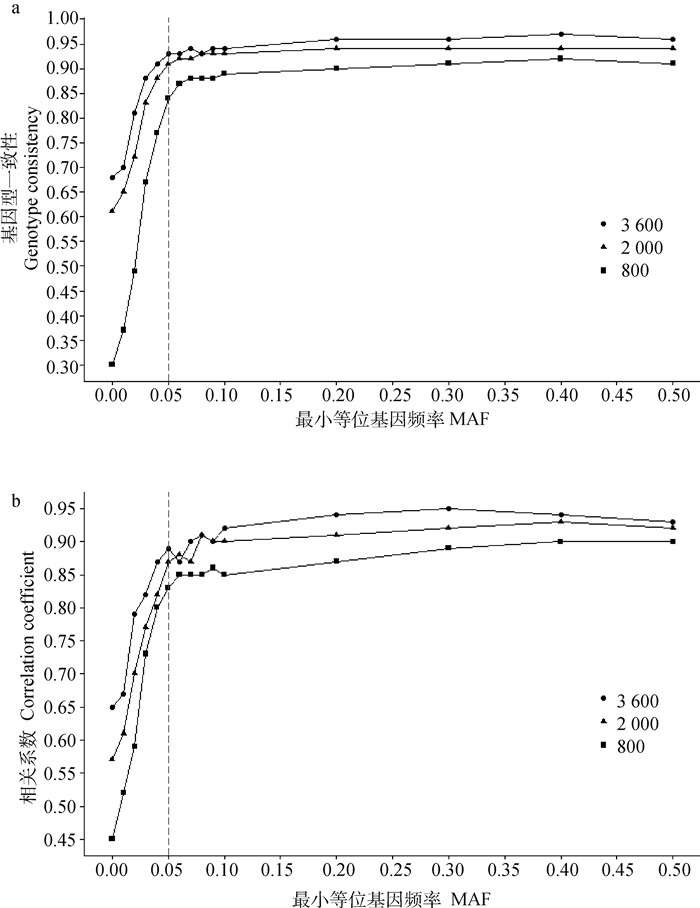
图 1显示了不同参考群体大小下最小等位基因频率(MAF)对基因型填充准确性的影响。从图 1a、1b可以看出,用来反映填充准确性的两个指标基因型一致性和相关系数展现了相同趋势。MAF为0.05时是填充准确性的拐点。当质控标准MAF小于0.05时,填充准确性都很低,随着MAF的增加,基因型填充的准确性急速上升;当MAF大于0.05时,虽然填充准确性依然随着MAF的上升有所增加,但增加幅度明显减缓。由于MAF < 0.05的标记填充准确性不高,因此本研究剔除MAF < 0.05的位点,用于后续分析。同时,从图 1中也可看出,随着参考群规模的增大,相同MAF下的填充准确性也随之升高,参考群体越大,填充准确性越高。
|
a.MAF 对基因型填充一致性的影响;b.MAF 对基因型填充相关系数的影响 a.Impact of MAF on genotype imputation consistency; b. Impact of MAF on genotype imputation correlation coefficients 图 1 MAF对基因型填充准确性的影响 Fig. 1 Impact of minor allele frequency (MAF) on genotype imputation accuracy |
表 2列出了删除10K和50K芯片中MAF < 0.05标记前后的基因型填充准确性。结果表明,删除MAF < 0.05的标记后,3种参考群体规模下,填充准确性都提高了。当参考群体大小为3 600时,DR2、基因型一致性和相关系数分别从0.976、0.941和0.922提高到0.978、0.965和0.948。同样,10次重复的每个填充准确性指标的标准差也变小了。参考群体大小为800和2 000时,也是如此。说明删除MAF < 0.05的标记可以进一步提高填充准确性。同图 1一样,表 2进一步表明了参考群体规模对填充准确性的影响。可以看出,当参考群体规模为800时,基因型一致性和相关系数分别为0.907和0.902;当参考群体规模增加至2 000时,基因型一致性和相关系数分别为0.939和0.924,相较于800群体,其基因型一致性和相关系数分别提升了3.53%和2.44%;继续增加参考群体规模至3 600时,基因型一致性和相关系数分别为0.965和0.948,较2 000规模群体提升了2.77%和2.60%,较800规模群体提升了6.39%和5.10%。填充准确性的上升幅度均比较明显。与基因型一致性和相关系数相比,DR2虽然也随着参考群体规模增大而提高,但是变化幅度不大,参考群体规模为2 000和3 600时,DR2几乎没有差异。
|
|
表 2 删除MAF < 0.05标记后填充准确性变化 Table 2 The comparison of the imputation accuracy before and after removal of SNPs with MAF < 0.05 |
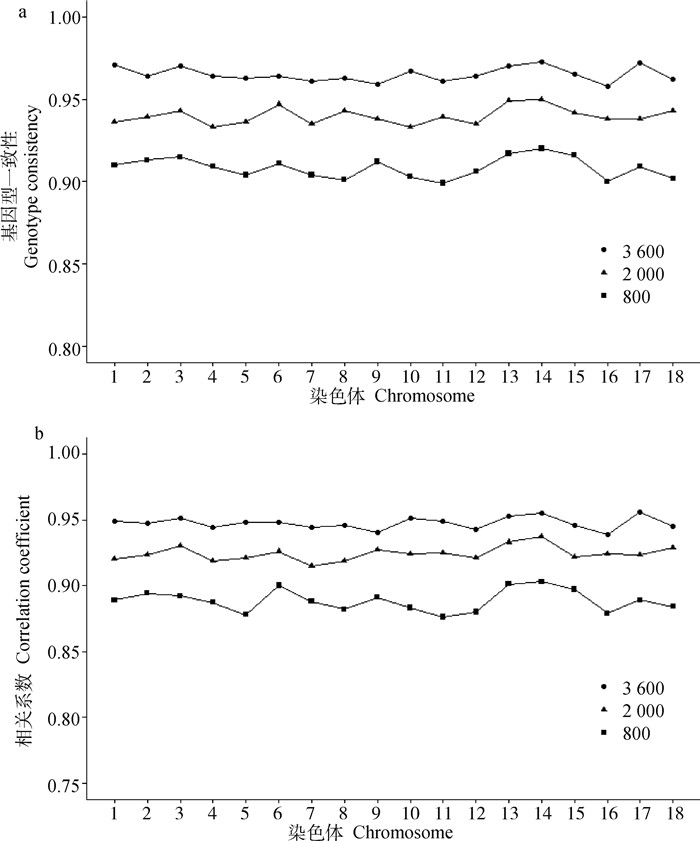
图 2a、2b展现了删除MAF < 0.05标记后每条常染色体上的基因型一致性(a)和相关系数(b), 可以看出,参考群体规模较小时,染色体填充准确性波动较大,随着参考群体规模增大,每条染色体填充准确性相差不大。
|
a.参考群大小对基因型填充一致性的影响;b.参考群大小对基因型填充相关系数的影响 a.Impact of reference population size on genotype imputation consistency; b.Impact of reference population size on genotype imputation correlation coefficients 图 2 参考群大小对基因型填充准确性的影响 Fig. 2 Impact of reference population size on genotype imputation accuracy |
已有很多研究表明,参考群大小会对基因型填充的准确性产生较大影响[27-29]。Lee等[30]使用了3 821头汉伍牛,以889头汉伍牛作为填充群体,以其余500、1 000、1 500、2 000、2 000余头汉伍牛作为参考群体,研究了3个低密度芯片(5K、10K、15K)到高密度(50K)芯片的基因型填充准确性,结果表明在参考群体规模为3 600时,基因型填充的准确性可达0.904~0.967。He等[24]在关于中国荷斯坦奶牛从6K到50K的填充效果研究中,比较了3种不同的填充软件对3种不同参考群下基因型填充准确性的影响,结果表明在fimpute软件中,所有情况下都表现最好,从公牛到女儿或半同胞之间的相关性由0.921提升至0.978。Weng等[31]也在2 108头中国荷斯坦奶牛3K到7K的填充效果研究中,发现使用3款填充软件无论以何种比例奶牛作参考群,Beagle的表现最稳定,平均填充准确性可达0.90。Badke等[32]用1 800头大白猪作参考群,从10K填充至60K,平均填充准确性可达到0.95。Xiang等[33]研究表明,使用4 263头大白猪作为参考群,从8K填充至60K,填充准确性可达0.94。这与本研究结果相似,当参考群体规模为2 000和3 600时,10K液相芯片填充到50K液相芯片的准确性为0.92、0.95(相关系数)和0.94、0.97(基因型一致性)。本研究用DR2、基因型一致性和相关系数作为衡量填充准确性指标,相同情况下,DR2的值最高,相关系数最低,通常使用相关系数评价填充准确性较多。
最小等位基因频率对基因型填充准确性的影响也有很多报道[34-36]。本研究结果表明,对于较低MAF的填充准确性会随着参考群体规模的增大提高,这是因为对于较低的MAF,更大的参考群体可以提供更多的稀有变异的单倍型种类,从而提高填充准确性。Lee等[30]在研究不同水平下低密度芯片到高密度芯片的填充效果时,发现在相同MAF下,提高参考群大小能显著提升基因型填充的准确性,且MAF等于0.015是该试验填充准确性的拐点。曾浩南等[26]研究了3款50K芯片填充至测序数据MAF对基因型填充准确性的影响时,发现MAF等于0.1是该试验填充准确性的拐点。马裴裴[37]在芬兰和瑞典红牛的混合群体中,比较了几种填充软件将标记从3K填充到54K时MAF对填充准确性的影响,发现无论使用哪种软件,MAF等于0.05是该试验基因型填充准确性的拐点。在本试验中,也发现MAF为0.05是填充准确性的拐点,MAF < 0.05时,基因型填充的准确性比较低,远小于MAF>0.05时的基因型填充准确性,当MAF>0.05后,基因型填充准确性会趋于平稳,这也与Heidaritabar等[38]和Zheng等[39]的研究结果相似。在大多数基于芯片的全基因组关联分析或基因组选择中,剔除MAF < 0.05的位点是常用的基因型质量控制标准,本研究也表明,剔除MAF < 0.05的位点后,液相芯片10K到50K的填充准确性得到提高。因此,液相芯片可同固相芯片一样,将剔除MAF < 0.05的标记作为基因型质量控制标准。
本研究表明,参考群体大小对基因型填充准确性的影响很大,当参考群体由800扩大到3 600时,填充准确性从0.90提高到0.95,10次重复的标准差也从0.006下降到0.002(表 2),说明不仅准确性提高了,而且填充效果稳定。进行基因型填充时,构建单倍型的准确性影响填充的效果,而参考群体的大小直接影响单倍型的构建。如果参考群较小,那么构建的单倍型种类可能不完全,填充时目标群体的单倍型就可能无法在参考群内找到[21]。Pausch等[40]在德系西门塔尔牛群体中分别利用50、100、200和400头牛作为参考群,将50K芯片填充至700K,结果也发现基因型填充准确性随着参考群的增加而增加。He等[24]在中国荷斯坦奶牛群体中,以不同参考群的10%、30%、60%、90%作为参考群,将6K芯片填充至50K,3款填充软件的基因型填充准确性都会随参考群的增大而增大。Weng等[31]也以2 108头中国荷斯坦奶牛的20%、40%、80%、95%作参考群,从3K填充至7K,基因型填充准确性逐渐增大。这与本研究的结果一致。此外,Ghoreishifar[27]等也发现,在填充群体一定时,参考群由小规模提升至中等规模,基因型填充的准确性提升很大,而当参考群继续增大至大规模时,填充准确性的提升缓慢。这与本研究的结果相似,在本试验中,随着参考群体的逐渐增大,基因型填充准确性的提升幅度会逐渐减小。
本研究探讨了猪液相芯片从10K到50K的基因型填充效果,结果表明,相同参考群体规模下,液相芯片填充准确性同研究较多的固相芯片一样。随着参考群体规模的扩大,基因型填充的准确性也会随之提升;但当参考群体扩大至一定程度时,继续扩大参考群体对基因型填充准确性的提升幅度会变小,但依旧可以提升基因型填充的准确性。因此,可以适当选取参考群规模,在降低检测成本的同时,保证基因型填充的准确性。另外,同固相芯片一样,MAF < 0.05是填充准确性的拐点,可以在基因型质量控制时剔除MAF < 0.05位点。
4 结论基因芯片的低密度化是有效降低测定成本的育种手段,也是国际动物育种的趋势。将低密度芯片与液相芯片结合可以实现基于检测成本的大幅降低,有助于推动基因组选择的实际应用。本研究结果表明了猪液相芯片从10K填充到50K是可行的,可以大规模用于基因组选择,进行早期选种,降低基因组选择育种成本。
| [1] |
叶绍潘. 基于全基因组测序数据的基因型填充准确性研究[D]. 广州: 华南农业大学, 2017. YE S P. Research on genotype imputation with whole-genome sequence data[D]. Guangzhou: South China Agricultural University, 2017. (in Chinese) |
| [2] |
KUMAR S, BANKS T W, CLOUTIER S. SNP discovery through next-generation sequencing and its applications[J]. Int J Plant Genomics, 2012, 2012: 831460. |
| [3] |
KLEIN R J, ZEISS C, CHEW E Y, et al. Complement factor H polymorphism in age-related macular degeneration[J]. Science, 2005, 308(5720): 385-389. DOI:10.1126/science.1109557 |
| [4] |
MEUWISSEN T H E, HAYES B J, GODDARD M E. Prediction of total genetic value using genome-wide dense marker maps[J]. Genetics, 2001, 157(4): 1819-1829. DOI:10.1093/genetics/157.4.1819 |
| [5] |
滕晓坤, 肖华胜. 基因芯片与高通量DNA测序技术前景分析[J]. 中国科学C辑: 生命科学, 2008, 38(10): 891-899. TENG X K, XIAO H S. Prospect analysis of gene chip and high-throughput DNA sequencing technology[J]. Science in China Series C: Life Sciences, 2008, 38(10): 891-899. DOI:10.3321/j.issn:1006-9259.2008.10.003 (in Chinese) |
| [6] |
王珏, 刘成琨, 刘德武, 等. 基于不同密度SNP芯片在杜洛克公猪中的全基因组选择效果分析[J]. 中国畜牧杂志, 2019, 55(12): 75-79. WANG J, LIU C K, LIU D W, et al. Analysis of genomic selection based on SNP data of various density chips in Duroc male pig population[J]. Chinese Journal of Animal Science, 2019, 55(12): 75-79. DOI:10.19556/j.0258-7033.20190927-02 (in Chinese) |
| [7] |
ZHANG Z, DRUET T. Marker imputation with low-density marker panels in Dutch Holstein cattle[J]. J Dairy Sci, 2010, 93(11): 5487-5494. DOI:10.3168/jds.2010-3501 |
| [8] |
WEIGEL K A, VAN TASSELL C P, O'CONNELL J R, et al. Prediction of unobserved single nucleotide polymorphism genotypes of Jersey cattle using reference panels and population-based imputation algorithms[J]. J Dairy Sci, 2010, 93(5): 2229-2238. DOI:10.3168/jds.2009-2849 |
| [9] |
MARCHINI J, HOWIE B. Genotype imputation for genome-wide association studies[J]. Nat Rev Genet, 2010, 11(7): 499-511. DOI:10.1038/nrg2796 |
| [10] |
何桑, 丁向东, 张勤. 基因型填充方法介绍及比较[J]. 中国畜牧杂志, 2013, 49(23): 95-100. HE S, DING X D, ZHANG Q. Comparison of different genotype imputation methods[J]. Chinese Journal of Animal Science, 2013, 49(23): 95-100. DOI:10.3969/j.issn.0258-7033.2013.23.022 (in Chinese) |
| [11] |
BECKER T, KNAPP M. Maximum-likelihood estimation of haplotype frequencies in nuclear families[J]. Genet Epidemiol, 2004, 27(1): 21-32. DOI:10.1002/gepi.10323 |
| [12] |
SCHEET P, STEPHENS M. A fast and flexible statistical model for large-scale population genotype data: Applications to inferring missing genotypes and haplotypic phase[J]. Am J Hum Genet, 2006, 78(4): 629-644. DOI:10.1086/502802 |
| [13] |
HOWIE B N, DONNELLY P, MARCHINI J. A flexible and accurate genotype imputation method for the next generation of genome-wide association studies[J]. PLoS Genet, 2009, 5(6): e1000529. DOI:10.1371/journal.pgen.1000529 |
| [14] |
BROWNING S R, BROWNING B L. Rapid and accurate haplotype phasing and missing-data inference for whole-genome association studies by use of localized haplotype clustering[J]. Am J Hum Genet, 2007, 81(5): 1084-1097. DOI:10.1086/521987 |
| [15] |
PURCELL S, NEALE B, TODD-BROWN K, et al. PLINK: a tool set for whole-genome association and population-based linkage analyses[J]. Am J Hum Genet, 2007, 81(3): 559-575. DOI:10.1086/519795 |
| [16] |
VANRADEN P M, O'CONNELL J R, WIGGANS G R, et al. Genomic evaluations with many more genotypes[J]. Genet Sel Evol, 2011, 43(1): 10. DOI:10.1186/1297-9686-43-10 |
| [17] |
SARGOLZAEI M, CHESNAIS J P, SCHENKEL F S. Accuracy of a family-based genotype imputation algorithm[C]//The 32nd Conference for the International Society for Animal Genetics. Edinburgh: ISAG, 2010.
|
| [18] |
HICKEY J M, KINGHORN B P, TIER B, et al. A combined long-range phasing and long haplotype imputation method to impute phase for SNP genotypes[J]. Genet Sel Evol, 2011, 43(1): 12. DOI:10.1186/1297-9686-43-12 |
| [19] |
NICOLAZZI E L, BIFFANI S, JANSEN G. Short communication: imputing genotypes using PedImpute fast algorithm combining pedigree and population information[J]. J Dairy Sci, 2013, 96(4): 2649-2653. DOI:10.3168/jds.2012-6062 |
| [20] |
阳文攀, 叶绍潘, 叶浩强, 等. 参考群筛选方法及规模对基因型填充准确性的影响[J]. 畜牧兽医学报, 2021, 52(12): 3357-3365. YANG W P, YE S P, YE H Q, et al. Effect of reference population selection method and size on genotype imputation accuracy[J]. Acta Veterinaria et Zootechnica Sinica, 2021, 52(12): 3357-3365. DOI:10.11843/j.issn.0366-6964.2021.012.004 (in Chinese) |
| [21] |
邱奥, 王雪, 孟庆利, 等. 3款猪50K SNP芯片基因型填充效果研究[J]. 中国畜牧杂志, 2021, 57(S1): 33-38. QIU A, WANG X, MENG Q L, et al. Impact of genotype imputation using the 50K SNP chip in three pigs[J]. Chinese Journal of Animal Science, 2021, 57(S1): 33-38. (in Chinese) |
| [22] |
徐云碧, 杨泉女, 郑洪建, 等. 靶向测序基因型检测(GBTS)技术及其应用[J]. 中国农业科学, 2020, 53(15): 2983-3004. XU Y B, YANG Q N, ZHENG H J, et al. Genotyping by target sequencing (GBTS) and its applications[J]. Scientia Agricultura Sinica, 2020, 53(15): 2983-3004. DOI:10.3864/j.issn.0578-1752.2020.15.001 (in Chinese) |
| [23] |
李欢, 张文洋, 田志强, 等. 高通量分子标记检测方法的研究进展[J]. 玉米科学, 2022, 30(3): 1-9. LI H, ZHANG W Y, TIAN Z Q, et al. Research progress of high-throughput molecular marker detection methods[J]. Journal of Maize Sciences, 2022, 30(3): 1-9. (in Chinese) |
| [24] |
HE S, WANG S, FU W, et al. Imputation of missing genotypes from low-to high-density SNP panel in different population designs[J]. Anim Genet, 2015, 46(1): 1-7. DOI:10.1111/age.12236 |
| [25] |
HICKEY J M, CROSSA J, BABU R, et al. Factors affecting the accuracy of genotype imputation in populations from several maize breeding programs[J]. Crop Sci, 2012, 52(2): 654-663. DOI:10.2135/cropsci2011.07.0358 |
| [26] |
曾浩南, 钟展明, 徐志婷, 等. 3款猪50KSNP芯片基因型填充至序列数据的效果研究[J]. 华南农业大学学报, 2022(4): 1-10. ZENG H N, ZHONG Z M, XU Z T, et al. Research on genotype imputation of three 50K SNP chips from chip data to sequencing data[J]. Journal of South China Agricultural University, 2022(4): 1-10. (in Chinese) |
| [27] |
GHOREISHIFAR S M, MORADI-SHAHRBABAK H, MORADI-SHAHRBABAK M, et al. Accuracy of imputation of single-nucleotide polymorphism marker genotypes for water buffaloes (Bubalus bubalis) using different reference population sizes and imputation tools[J]. Livest Sci, 2018, 216: 174-182. DOI:10.1016/j.livsci.2018.08.009 |
| [28] |
BUTTY A M, SARGOLZAEI M, MIGLIOR F, et al. Optimizing selection of the reference population for genotype imputation from array to sequence variants[J]. Front Genet, 2019, 10: 510. DOI:10.3389/fgene.2019.00510 |
| [29] |
SONG H L, YE S P, JIANG Y F, et al. Using imputation-based whole-genome sequencing data to improve the accuracy of genomic prediction for combined populations in pigs[J]. Genet Sel Evol, 2019, 51(1): 58. DOI:10.1186/s12711-019-0500-8 |
| [30] |
LEE D, KIM Y, CHUNG Y, et al. Accuracy of genotype imputation based on reference population size and marker density in Hanwoo cattle[J]. J Anim Sci Technol, 2021, 63(6): 1232-1246. DOI:10.5187/jast.2021.e117 |
| [31] |
WENG Z, ZHANG Z, ZHANG Q, et al. Comparison of different imputation methods from low- to high-density panels using Chinese Holstein cattle[J]. Animal, 2013, 7(5): 729-735. DOI:10.1017/S1751731112002224 |
| [32] |
BADKE Y M, BATES R O, ERNST C W, et al. Accuracy of estimation of genomic breeding values in pigs using low-density genotypes and imputation[J]. G3 (Bethesda), 2014, 4(4): 623-631. DOI:10.1534/g3.114.010504 |
| [33] |
XIANG T, MA P P, OSTERSEN T, et al. Imputation of genotypes in Danish purebred and two-way crossbred pigs using low-density panels[J]. Genet Sel Evol, 2015, 47(1): 54. DOI:10.1186/s12711-015-0134-4 |
| [34] |
RUBINACCI S, RIBEIRO D M, HOFMEISTER R J, et al. Efficient phasing and imputation of low-coverage sequencing data using large reference panels[J]. Nat Genet, 2021, 53(1): 120-126. DOI:10.1038/s41588-020-00756-0 |
| [35] |
DAVIES R W, KUCKA M, SU D W, et al. Rapid genotype imputation from sequence with reference panels[J]. Nat Genet, 2021, 53(7): 1104-1111. DOI:10.1038/s41588-021-00877-0 |
| [36] |
KREINER-MØLLER E, MEDINA-GOMEZ C, UITTERLINDEN A G, et al. Improving accuracy of rare variant imputation with a two-step imputation approach[J]. Eur J Hum Genet, 2015, 23(3): 395-400. DOI:10.1038/ejhg.2014.91 |
| [37] |
马裴裴. 通过基因型填充提高全基因组预测的方法和策略研究[D]. 北京: 中国农业大学, 2013. MA P P. Methods and strategies to impute missing genotypes for improving genomic prediction[D]. Beijing: China Agricultural University, 2013. (in Chinese) |
| [38] |
HEIDARITABAR M, CALUS M P L, VEREIJKEN A, et al. Accuracy of imputation using the most common sires as reference population in layer chickens[J]. BMC Genet, 2015, 16(1): 101. DOI:10.1186/s12863-015-0253-5 |
| [39] |
ZHENG H F, RONG J J, LIU M, et al. Performance of genotype imputation for low frequency and rare variants from the 1000 genomes[J]. PLoS One, 2015, 10(1): e0116487. DOI:10.1371/journal.pone.0116487 |
| [40] |
PAUSCH H, AIGNER B, EMMERLING R, et al. Imputation of high-density genotypes in the Fleckvieh cattle population[J]. Genet Sel Evol, 2013, 45(1): 3. DOI:10.1186/1297-9686-45-3 |
(编辑 郭云雁)



