2. 河南省农业科学院中英禽病国际研究中心,郑州 450002;
3. 河南牧业经济学院动物医药学院,郑州 450046
2. UK-China Centre of Excellence for Research on Avian Diseases, Henan Academy of Agricultural Sciences, Zhengzhou 450002, China;
3. College of Veterinary Medicine, Henan University of Animal Husbandry and Economy, Zhengzhou 450046, China
成簇的规律性间隔短回文重复序列(clustered regularly interspaced short palindromic repeats,CRISPR),是在细菌和古生菌中发现的一类重要外援基因,与CRISPR相关的(CRISPR associated,Cas)蛋白家族共同构成细菌自身适应性免疫系统的重要部分,用以抵御外来质粒或噬菌体的入侵。CRISPR/Cas9系统属于II型CRISPR/Cas系统,该系统只需一个与靶基因互补配对的单一向导RNA(single-guide RNA,sgRNA)的引导,Cas9蛋白即可特异性识别靶基因的前间隔序列邻近基序(protospacer adjacent motif,PAM)5′-NGG-3′(N指代任一碱基),从而激发其双链DNA切割活性,产生一个双链缺口(double-strand break,DSB),同时基因组DNA在复制时即可利用自我修复功能直接修复DSB,最终实现对靶基因的精准切割[1-2]。2013年,Cong等[3]首次将CRISPR/Cas9系统应用于真核细胞的基因编辑并取得巨大成功,从而大大推动了该技术在生命科学研究领域的应用,此后短短数年间,CRISPR/Cas9基因编辑技术就广泛应用于包括人类细胞、细菌、斑马鱼、酵母、小鼠、果蝇和寄生虫等多物种基因组的编辑[4-10]。最近,将CRISPR/Cas9基因编辑技术应用于病毒基因组的改造,尤其是一些致癌性病毒和疱疹病毒,已成为病毒学领域新的国际研究热点,如人类乙肝病毒(hepatitis B virus,HBV)、高危人乳头瘤病毒(high-risk human papillomavirus,HR-HPV)、单纯疱疹病毒I型(herpes simplex virus 1,HSV-1)、爱泼斯坦-巴尔病毒(Epstein-Barr virus,EBV)、人类巨细胞病毒(human cytomegalovirus,HCMV)、卡波西肉瘤相关疱疹病毒(Kaposi’s sarcoma-associated herpesvirus,KSHV)以及一些引起动物疫病的疱疹病毒,如鸡马立克病病毒(Marek's disease virus,MDV)、鸭肠炎病毒(duck enteritis virus,DEV)、猪伪狂犬病病毒(Pseudorabies virus,PRV)等[11-19]。
2 致瘤性家禽疱疹病毒的基因编辑 2.1 传统基因重组技术介导的基因编辑在CRISPR/Cas9系统广泛应用于大基因组DNA病毒的基因编辑之前,科学家们主要利用传统的基因同源重组技术(homologous recombination system,HRS)、黏粒(Cosmid)或F黏粒(Fosmid)基因组文库技术以及细菌人工染色体(bacterial artificial chromosome,BAC)同源重组技术来实现疱疹病毒的基因组改造[20-23]。MDV在病毒学分类上属于甲亚科疱疹病毒,是少数几种可在其自然感染宿主中诱导肿瘤发生并导致免疫抑制的一种致瘤性疱疹病毒。MDV早期感染雏鸡导致马立克病(Marek’s disease,MD),是目前生物界已知毒性最强的一种致瘤性病毒。根据抗原性的不同,MDV可以分为3种不同的血清型:MDV-1、MDV-2和MDV-3,其中只有MDV-1流行毒株具有致病性和致瘤性,MDV-2和MDV-3的毒株对鸡均无致病性,后者又被称为火鸡疱疹病毒(herpesvirus of turkey,HVT)。早在2002年,科学家们首先利用Cosmid基因组文库技术构建了MDV-1超强毒株Md5的感染性克隆,并进一步分别构建了pp38基因缺失株Md5Δpp38和meq基因缺失株Md5Δmeq,评估了该技术用于MDV病毒基因功能研究的可行性,并提出meq基因参与淋巴细胞转化但与病毒的复制无关[24-25]。由于用重组黏粒构建MDV感染性克隆重复性较差,且有可能形成不同的感染性病毒粒子,影响后续试验的稳定性。因此,黏粒系统很快被BAC克隆和基因同源重组技术所取代。Schumacher等[26]首次成功构建了MDV-1 584Ap80C株的感染性BAC克隆。此后,利用BAC和Rec E/T、En passant等一步或两步法同源重组技术[26-27],3种不同血清型的MDV疫苗株CVI988/Rispens[28]、HVT[20]、814[29]、SB-1[30]以及不同毒力MDV-1毒株的感染性BAC克隆陆续构建成功[31-35]。利用这些感染性BAC克隆,科学家们完成了7个MDV毒株的全基因组序列分析,为后续MDV病原学、流行病学、遗传进化及致病机制研究奠定了重要基础。
虽然HVT对宿主不具有致病性,但由于它与具有致病性的MDV-1毒株具有极强的抗原相关性[36],早在上世纪70年代就作为第一代商品疫苗应用于MD的免疫预防并取得了良好的防控效果,对MD的防控贡献巨大[37]。这是人类历史上第一个可以成功预防肿瘤发生的病毒性疫苗,至今仍在全球广泛使用。同时,基于传统的HRS或BAC重组技术,HVT也被广泛用于研发携带异源病毒抗原蛋白的基因工程载体疫苗,在其基因组中插入外源病毒基因,如鸡新城疫病毒(Newcastle disease virus,NDV)、禽流感病毒(avian influenza virus,AIV)、鸡传染性法氏囊病病毒(infectious bursal disease virus,IBDV)、鸡传染性喉气管炎病毒(infectious laryngotracheitis virus,ILTV)、禽白血病病毒(avian leucosis virus,ALV)和艾美耳球虫(Eimeria)等的主要抗原蛋白基因[38-42]。实验室研究数据显示,这些HVT重组疫苗对MDV-1和相关病毒引起的家禽疫病具有良好和持久的免疫保护。但是,由于MDV基因组比较庞大,在构建这些病毒基因组感染性BAC克隆的过程中,比较费时费力且成功率较低,而且该系统会将药物筛选标记基因和部分细菌质粒片段残留到重组病毒的基因组中,存在改变病毒原有生物学特性如病毒复制能力的可能性。在MDV基因工程疫苗毒株商品化之前,作为生物安全评价的重要前提,首先需要将相关抗性基因和外源片段从病毒基因组中剔除,这显然进一步增加了试验难度、工作量和研发成本。
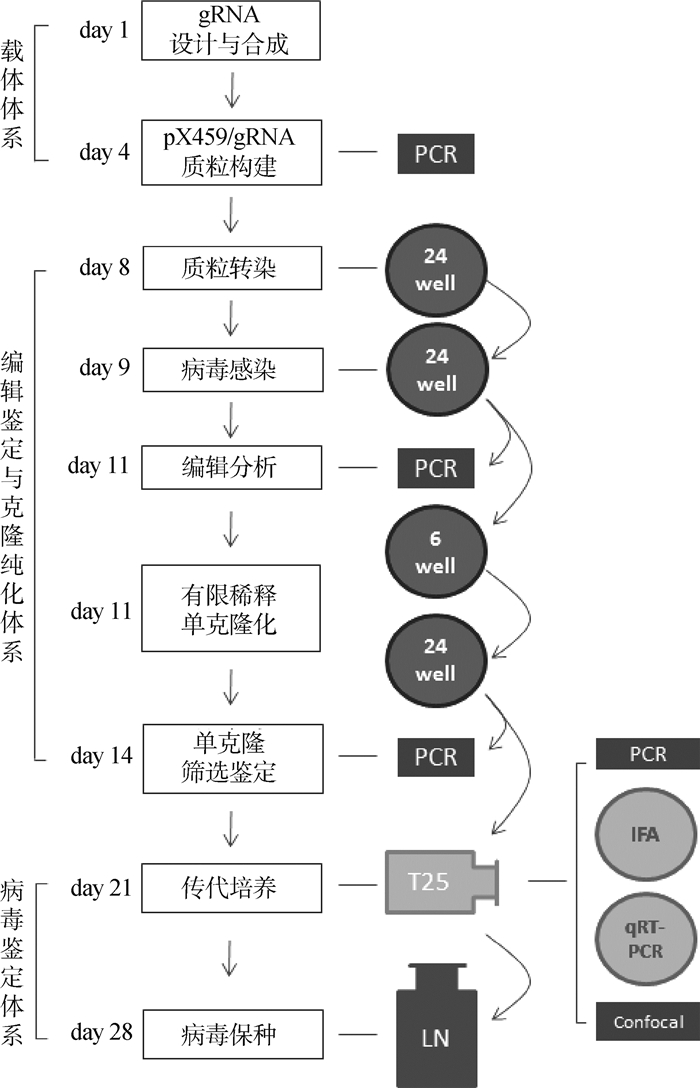
2.2 CRISPR/Cas9系统介导的基因编辑随着CRISPR/Cas9系统大量应用于大基因组DNA病毒,尤其是疱疹病毒的基因编辑以来,国内外的科学家们已经开始着手建立家禽疱疹病毒相关的CRISPR/Cas9基因编辑技术平台和体系(图 1),并在短短数年内就将其应用于绝大部分家禽疱疹病毒的基因编辑,而且已经取得了大量重要的研究进展(表 1)。
|
PCR.聚合酶链式反应;IFA.间接免疫荧光试验;qRT-PCR.实时荧光定量PCR;confocal.共聚焦分析;24 well.24孔细胞板;6 well.6孔细胞板;T25.T25细胞瓶;LN.液氮保存 PCR. Polymerase chain reaction; IFA. Indirect immunofluorescence assay; qRT-PCR. Quantitative real-time PCR; confocal. Confocal scanning; 24 well. 24-well plate; 6 well. 6-well plate; T25. T25 flask; LN. Liquid nitrogen storage 图 1 家禽疱疹病毒CRISPR/Cas9基因编辑技术体系与操作流程 Fig. 1 Schematic of the CRISPR/Cas9-based systems for avian herpesvirus gene editing |
|
|
表 1 CRISPR/Cas9系统编辑的家禽疱疹病毒 Table 1 List of the reconstituted avian herpesviruses using CRISPR/Cas9-based gene editing system |
2016年,Yao等[19]利用CRISPR/Cas9系统和单个病毒特异性的gRNA在预期的切割位点对HVT基因组的gB、gI和gE基因分别进行了靶向短缺失突变,证明CRISPR/Cas9系统可以替代Cosmid文库系统和BAC同源重组技术,高效精准地实现MDV基因组的编辑,这是CRISPR/Cas9基因编辑技术在家禽疱疹病毒基因组改造中的首次应用。紧接着,该团队又将CRISPR/Cas9系统应用于MDV-1疫苗株CVI988/Rispens的编辑,通过双gRNA表达质粒转染/病毒感染细胞的模式成功编辑敲除了meq基因和pp38基因,并证明meq基因或pp38基因的缺失均不影响CVI988/Rispens病毒的体外复制[43]。与此同时,国内学者也陆续有关于CRISPR/Cas9系统应用于MDV基因编辑的报道,先后构建了MDV-1超强毒株BS/15、Md5、GX0101和疫苗株CVI988/Rispens的meq基因部分或全部编辑缺失的毒株,相关研究同样证实meq基因的缺失不会影响MDV-1毒株的体外复制[44-46]。上述研究进一步证实了CRISPR/Cas9系统用于MDV基因组编辑的可行性、简便性和高效性。
除了病毒蛋白编码基因之外,Luo等[47]利用CRISPR/Cas9系统对MDV-1超强毒株RB-1B编码的一系列微小RNA(microRNA,miRNA)基因进行了编辑,成功构建了一系列Meq基因簇miRNAs和Mid基因簇单个miRNA编辑缺失的毒株。令人意外的是,在CEF上的体外复制动力学分析表明,Meq基因簇miRNAs及其单个miRNA的编辑缺失显著降低了MDV的体外复制能力,而Mid基因簇中miR-M11的编辑缺失却显著增强了病毒的体外复制能力,这与此前报道的利用BAC克隆和Rec E/T同源重组技术构建的相应miRNA基因缺失的GX0101毒株不影响其体外复制的结论完全不同,但却与其在攻毒感染宿主体内的复制动力学趋势一致[48-50]。随后,进一步对MDV-1超强毒株GX0101编码的第3个miRNA基因簇(即LAT基因簇miRNAs)的编辑发现,该基因簇miRNA的编辑缺失不影响MDV在CEF上的体外复制能力以及相邻病毒基因的表达[51],与此前研究报道LAT基因簇miRNAs具有调控潜伏感染的功能相一致。上述研究提示,与BAC克隆和Rec E/T同源重组技术可能改变MDV部分生物学特性(尤其是病毒复制能力)的缺陷相比[52],CRISPR/Cas9系统可能是一种更好地适用于MDV基因编辑的新技术,简便、低廉、高效,不仅可以用于较大的MDV蛋白编码基因的编辑,还可用于像miRNA这样仅仅21~24 nt的小片段非编码RNA基因的编辑。
近期,有学者将CRISPR/Cas9系统应用于编辑整合到MD淋巴瘤细胞染色体基因组的病毒基因。构建稳定表达Cas9蛋白的T淋巴肿瘤细胞系(lymphoblastoid cell line,LCL)MDCC-MSB-1-Cas9和MDCC-HP8-Cas9,然后用人工合成的特异性靶向gRNA转染细胞系,可以成功将宿主细胞染色体基因组中整合的pp38基因进行编辑,从而研究发现pp38基因的编辑缺失促进了肿瘤细胞系的增殖[53]。随后对MDCC-HP8-Cas9稳转细胞系基因组中整合的miR-M4(MDV编码的宿主癌基因miR-155的病毒同源基因)进行编辑敲除,进一步证实miR-M4不是MDV转化肿瘤细胞增殖的必需基因,但对淋巴瘤的诱导至关重要[54]。
在MDV基因工程疫苗研究方面,CRISPR/Cas9系统也已展现出极好的技术优势。2018年,利用CRISPR/Cas9系统和非同源末端连接(non-homologous end-joining,NHEJ),研究人员成功将IBDV的VP2基因和红色荧光蛋白(red fluorescent protein, RFP)串联表达盒编辑插入到HVT基因组的UL45/46位点,然后通过Cre-loxP重组酶系统去除外源筛选基因RFP,从而构建了HVT/IBDV-VP2二价重组疫苗候选毒株[55]。紧接着,利用同样的策略将ILTV-gD/gI和AIV-H9 N2-HA表达盒分别编辑插入HVT/IBDV-VP2重组病毒基因组的065/066和US2位点,成功构建了HVT-VP2-gDgI-HA三价重组疫苗候选毒株[56]。此外,有学者利用CRISPR/Cas9编辑系统和同源定向修复(homology-directed repair,HDR),首先构建了一个带有UL45/46两端同源臂的pHVT-UL45-46-eGFP表达盒,利用CRISPR/Cas9-HDR技术将其定向插入到HVT基因组中构建供体病毒HVT-GFP,然后利用同样的策略构建携带相同同源臂且可表达AIV H7 N9-HA基因的pHVT-UL45-46-H7 N9-HA表达盒,再次利用CRISPR/Cas9-HDR技术将该表达盒精准插入并替换重组病毒HVT-GFP中的绿色荧光蛋白(green fluorescent protein, GFP),成功构建了HVT-H7 N9-HA双价候选疫苗毒株[57]。与上述不同,也有学者利用传统的BAC和Rec E/T同源重组系统先将AIV BJ/15株的H9 N2-HA基因插入到HVT-BAC感染性克隆基因组的UL45/UL46之间,通过半乳糖激酶筛选系统在SW102大肠杆菌中构建HVT-BAC-H9 N2-HA供体DNA,然后再利用CRISPR/Cas9系统编辑剔除BAC残余序列,最终构建仅含AIV H9 N2-HA基因的rHVT-H9 N2-HA重组疫苗候选毒株[58]。
将CRISPR/Cas9系统应用于MDV的抗病毒治疗或抗病遗传育种,最近也已进行了一些探索。针对MDV编码的6个病毒复制必须基因(UL6、UL19、UL27、UL30、UL49和ICP4),研究人员设计了11个gRNAs分别对MDV超强毒株RB-1B进行编辑,研究显示,大多数单个的gRNA虽然可以显著抑制病毒的复制,但也可能导致逃逸突变体病毒的出现,两个或两个以上的gRNAs组合则可以完全消除病毒,即使经连续传代之后也没有观察到病毒逃逸突变体,这一发现说明CRISPR/Cas9系统可以有效地阻止MDV的复制,为未来MD的抗病毒治疗研究提供了可能性[59]。更有趣的是,最近还有科学家尝试培育了一种可在体内共表达Cas9蛋白和ICP4-gRNAs(gICP4)的转基因鸡[60],通过腹腔注射MDV进行动物攻毒试验,结果发现与仅转染Cas9基因的野生型对照鸡相比,表达ICP4-gRNAs/Cas9的转基因鸡体内MDV复制水平明显减少,提示CRISPR/Cas9或许可以作为抗病遗传育种的一种选择,用于控制鸡群MDV的流行和感染[60]。除了针对MDV自身,类似的抗病毒策略还被研究应用于同时抵抗其它的家禽免疫抑制病与肿瘤病病毒。比如,将特异性靶向ALV-J和禽网状内皮组织增生症病毒(reticuloendotheliosis virus,REV)长末端重复序列基因(long terminal repeat,LTR)的sgRNA,通过CRISPR/Cas9-HDR技术定向插入到814疫苗株的US2基因位点,分别构建重组病毒r814-Cas9-sgLTR-8和rMDV-Cas9-gLTR1/6,这两个重组病毒在宿主细胞中均能有效抑制ALV-J和REV的增殖,表明利用CRISPR/Cas9系统构建携带并能稳定表达针对异源病毒的sgRNA的MDV重组疫苗,免疫宿主鸡后可有效抵御相关病毒的攻击,为ALV-J和REV的共感染提供了一种全新的防控策略,具有潜在的市场应用前景[61-62]。
3 非致瘤性家禽疱疹病毒的CRISPR/Cas9基因编辑DEV感染鸭导致的病毒性肠炎流行广泛,传播迅速,发病率和死亡率都很高,是危害水禽养殖最严重的疫病之一。2017年,Zou等[18]利用CRISPR/Cas9系统和HDR介导的基因编辑策略,在DEV C-KCE毒株的UL27/UL26和US7/US8位点重组插入H5N1亚型高致病性AIV的HA基因和鸭坦布苏病毒(duck tembusu virus,DTMUV)的PrM和gE基因,构建的重组病毒C-KCE-HA/PrM-E可作为一种潜在的三价候选疫苗株,用于同时预防H5N1-AIV、DTMUV和DEV的感染。随后,有学者利用CRISPR/Cas9-NHEJ技术将AIV H5-HA抗原基因表达盒插入到DEV的UL27/UL26基因之间,构建了重组表达AIV H5-HA的DEV双价重组疫苗毒株[63]。此外,利用相同的技术策略,有研究人员在编辑敲除ILTV毒力因子TK和US4的同时,重组插入了NDV-F蛋白外源基因,构建的重组病毒既不影响ILTV的复制,NDV-F蛋白也能正常表达[64]。ILTV感染引起的鸡传染性喉气管炎(infectious laryngotracheitis,ILT)和NDV引起的鸡新城疫(Newcastle disease,ND),都是严重危害家禽养殖业健康发展的重要呼吸系统病,利用上述策略构建多价重组疫苗,为此类疫病的有效防控提供了新的思路。
4 前景与展望CRISPR/Cas系统是最新一代的基因组编辑技术,它能够特异、高效、几乎无所不能地改造(突变、敲除或插入)动植物乃至微生物的基因组。自2012年问世以来,该技术迅速成为生命科学领域的全球研究焦点,并于2020年获得诺贝尔化学奖。目前,发展最快、应用最广、效率最高的CRISPR/Cas9基因编辑技术已广泛应用于各种相关研究,如模式动物构建、人类疾病研究、基因治疗、动植物遗传育种等。CRISPR/Cas9基因编辑技术在基因组比较庞大的DNA病毒研究中的应用,也已成为新的国际研究热点,其中聚焦最多的是疱疹病毒[65-66]。家禽疱疹病毒主要包括MDV、HVT、DEV、ILTV等,CRISPR/Cas9基因编辑技术在这些家禽疱疹病毒的应用主要集中在3个方面:一是对部分重要的病毒基因进行突变或缺失,开展病毒基因功能和致病机制研究;二是利用NHEJ和/或HDR技术构建以家禽疱疹病毒基因缺失疫苗或基因重组多价疫苗;三是研究探索家禽抗病毒治疗及抗病遗传育种的新策略和新技术。
利用CRISPR/Cas9基因编辑技术构建一系列的MDV基因缺失疫苗、基因工程载体疫苗和多价疫苗,可能会成为今后相当一段时期内最受关注的研究方向和内容。以HVT作为载体构建的基因工程二价苗和三价苗,目前已在临床实践中得到应用。但是,近50年来在MD疫苗长期广泛的使用和高强度的疫苗免疫压力下,MDV的毒力正在不断增强甚至发生变异,今后若要取得良好的免疫防控效果,利用CRISPR/Cas9基因编辑技术对当前最新流行的MDV-1优势毒株进行基因组改造以构建能够有效防控vv+MDV或变异株流行的新型高效MD基因工程疫苗,是需要优先考虑的问题。同时,利用CRISPR/Cas9基因编辑系统对家禽疱疹病毒的全基因组进行gRNA设计、分析和扫描编辑,也有可能会为进一步发现和鉴定新的致病因子和毒力基因带来新的契机。显然,CRISPR/Cas9基因编辑技术在家禽疱疹病毒研究应用的大幕已经拉开,更多挑战和惊喜也正等待研究者们进一步努力揭晓。
| [1] |
DEVEAU H, GARNEAU J E, MOINEAU S. CRISPR/Cas system and its role in phage-bacteria interactions[J]. Annu Rev Microbiol, 2010, 64(1): 475-493. DOI:10.1146/annurev.micro.112408.134123 |
| [2] |
DELTCHEVA E, CHYLINSKI K, SHARMA C M, et al. CRISPR RNA maturation by trans-encoded small RNA and host factor RNase Ⅲ[J]. Nature, 2011, 471(7340): 602-607. DOI:10.1038/nature09886 |
| [3] |
CONG L, RAN F A, COX D, et al. Multiplex genome engineering using CRISPR/Cas systems[J]. Science, 2013, 339(6121): 819-823. DOI:10.1126/science.1231143 |
| [4] |
MALI P, ESVELT K M, CHURCH G M. Cas9 as a versatile tool for engineering biology[J]. Nat Methods, 2013, 10(10): 957-963. DOI:10.1038/nmeth.2649 |
| [5] |
JIANG W Y, BIKARD D, COX D, et al. RNA-guided editing of bacterial genomes using CRISPR-Cas systems[J]. Nat Biotechnol, 2013, 31(3): 233-239. DOI:10.1038/nbt.2508 |
| [6] |
CHANG N N, SUN C H, GAO L, et al. Genome editing with RNA-guided Cas9 nuclease in zebrafish embryos[J]. Cell Res, 2013, 23(4): 465-472. DOI:10.1038/cr.2013.45 |
| [7] |
RYAN O W, CATE J H D. Multiplex engineering of industrial yeast genomes using CRISPRm[J]. Methods Enzymol, 2014, 546: 473-489. |
| [8] |
WANG H Y, YANG H, SHIVALILA C S, et al. One-step generation of mice carrying mutations in multiple genes by CRISPR/Cas-mediated genome engineering[J]. Cell, 2013, 153(4): 910-918. DOI:10.1016/j.cell.2013.04.025 |
| [9] |
BASSETT A, LIU J L. CRISPR/Cas9 mediated genome engineering in Drosophila[J]. Methods, 2014, 69(2): 128-136. DOI:10.1016/j.ymeth.2014.02.019 |
| [10] |
WARD J D. Rendering the intractable more tractable: tools from Caenorhabditis elegans ripe for import into parasitic nematodes[J]. Genetics, 2015, 201(4): 1279-1294. DOI:10.1534/genetics.115.182717 |
| [11] |
KENNEDY E M, KORNEPATI A V R, CULLEN B R. Targeting hepatitis B virus cccDNA using CRISPR/Cas9[J]. Antiviral Res, 2015, 123: 188-192. DOI:10.1016/j.antiviral.2015.10.004 |
| [12] |
HU Z, YU L, ZHU D, et al. Disruption of HPV16-E7 by CRISPR/Cas system induces apoptosis and growth inhibition in HPV16 positive human cervical cancer cells[J]. Biomed Res Int, 2014, 2014: 612823. |
| [13] |
BI Y W, SUN L, GAO D D, et al. High-efficiency targeted editing of large viral genomes by RNA-guided nucleases[J]. PLoS Pathog, 2014, 10(5): e1004090. DOI:10.1371/journal.ppat.1004090 |
| [14] |
WANG J B, QUAKE S R. RNA-guided endonuclease provides a therapeutic strategy to cure latent herpesviridae infection[J]. Proc Natl Acad Sci U S A, 2014, 111(36): 13157-13162. DOI:10.1073/pnas.1410785111 |
| [15] |
GERGEN J, COULON F, CRENEGUY A, et al. Multiplex CRISPR/Cas9 system impairs HCMV replication by excising an essential viral gene[J]. PLoS One, 2018, 13(2): e0192602. DOI:10.1371/journal.pone.0192602 |
| [16] |
TSO F Y, WEST J T, WOOD C. Reduction of Kaposi's sarcoma-associated herpesvirus latency using CRISPR-Cas9 to edit the latency-associated nuclear antigen gene[J]. J Virol, 2019, 93(7): e02183-18. |
| [17] |
XU A T, QIN C, LANG Y, et al. A simple and rapid approach to manipulate pseudorabies virus genome by CRISPR/Cas9 system[J]. Biotechnol Lett, 2015, 37(6): 1265-1272. DOI:10.1007/s10529-015-1796-2 |
| [18] |
ZOU Z, HUANG K, WEI Y M, et al. Construction of a highly efficient CRISPR/Cas9-mediated duck enteritis virus-based vaccine against H5N1 avian influenza virus and duck Tembusu virus infection[J]. Sci Rep, 2017, 7(1): 1478. DOI:10.1038/s41598-017-01554-1 |
| [19] |
YAO Y, BASSETT A, NAIR V. Targeted editing of avian herpesvirus vaccine vector using CRISPR/Cas9 nucleases[J]. J Vaccine Technol, 2016, 1: 1-7. |
| [20] |
BAIGENT S J, PETHERBRIDGE L J, SMITH L P, et al. Herpesvirus of turkey reconstituted from bacterial artificial chromosome clones induces protection against Marek's disease[J]. J Gen Virol, 2006, 87(Pt 4): 769-776. |
| [21] |
LIU Y Z, LI K, GAO Y L, et al. Recombinant Marek's disease virus as a vector-based vaccine against avian leukosis virus subgroup J in chicken[J]. Viruses, 2016, 8(11): 301. DOI:10.3390/v8110301 |
| [22] |
BALZLI C L, BERTRAN K, LEE D H, et al. The efficacy of recombinant turkey herpesvirus vaccines targeting the H5 of highly pathogenic avian influenza virus from the 2014-2015 North American outbreak[J]. Vaccine, 2018, 36(1): 84-90. DOI:10.1016/j.vaccine.2017.11.026 |
| [23] |
LIU J X, CHEN P C, JIANG Y P, et al. A duck enteritis virus-vectored bivalent live vaccine provides fast and complete protection against H5N1 avian influenza virus infection in ducks[J]. J Virol, 2011, 85(21): 10989-10998. DOI:10.1128/JVI.05420-11 |
| [24] |
REDDY S M, LUPIANI B, GIMENO I M, et al. Rescue of a pathogenic Marek's disease virus with overlapping cosmid DNAs: use of a pp38 mutant to validate the technology for the study of gene function[J]. Proc Natl Acad Sci U S A, 2002, 99(10): 7054-7059. DOI:10.1073/pnas.092152699 |
| [25] |
LUPIANI B, LEE L F, CUI X P, et al. Marek's disease virus-encoded meq gene is involved in transformation of lymphocytes but is dispensable for replication[J]. Proc Natl Acad Sci U S A, 2004, 101(32): 11815-11820. DOI:10.1073/pnas.0404508101 |
| [26] |
SCHUMACHER D, TISCHER B K, FUCHS W, et al. Reconstitution of Marek's disease virus serotype 1 (MDV-1) from DNA cloned as a bacterial artificial chromosome and characterization of a glycoprotein B-negative MDV-1 mutant[J]. J Virol, 2000, 74(23): 11088-11098. DOI:10.1128/JVI.74.23.11088-11098.2000 |
| [27] |
TISCHER B K, SMITH G A, OSTERRIEDER N. En passant mutagenesis: a two step markerless red recombination system[M]//BRAMAN J. In Vitro Mutagenesis Protocols. 3rd ed. Humana Totowa: Springer, 2010: 421-430.
|
| [28] |
PETHERBRIDGE L, HOWES K, BAIGENT S J, et al. Replication-competent bacterial artificial chromosomes of Marek's disease virus: Novel tools for generation of molecularly defined herpesvirus vaccines[J]. J Virol, 2003, 77(16): 8712-8718. DOI:10.1128/JVI.77.16.8712-8718.2003 |
| [29] |
崔红玉. 鸡马立克氏病病毒细菌人工染色体的构建[D]. 哈尔滨: 中国农业科学院, 2007. CUI H Y. Cloning of Marek's disease virus genome as an infectious bacterial artificial chromosome[D]. Harbin: Chinese Academy of Agricultural Sciences, 2007. (in Chinese). |
| [30] |
PETHERBRIDGE L, XU H T, ZHAO Y G, et al. Cloning of Gallid herpesvirus 3 (Marek's disease virus serotype-2) genome as infectious bacterial artificial chromosomes for analysis of viral gene functions[J]. J Virol Methods, 2009, 158(1-2): 11-17. DOI:10.1016/j.jviromet.2009.01.009 |
| [31] |
PETHERBRIDGE L, BROWN A C, BAIGENT S J, et al. Oncogenicity of virulent Marek's disease virus cloned as bacterial artificial chromosomes[J]. J Virol, 2004, 78(23): 13376-13380. DOI:10.1128/JVI.78.23.13376-13380.2004 |
| [32] |
SUN A J, LAWRENCE P, ZHAO Y G, et al. A BAC clone of MDV strain GX0101 with REV-LTR integration retained its pathogenicity[J]. Chin Sci Bull, 2009, 54(15): 2641-2647. |
| [33] |
REDDY S M, SUN A J, KHAN O A, et al. Cloning of a very virulent plus, 686 strain of Marek's disease virus as a bacterial artificial chromosome[J]. Avian Dis, 2013, 57(2 Suppl): 469-473. |
| [34] |
JAROSINSKI K W, MARGULIS N G, KAMIL J P, et al. Horizontal transmission of Marek's disease virus requires US2, the UL13 protein kinase, and gC[J]. J Virol, 2007, 81(19): 10575-10587. DOI:10.1128/JVI.01065-07 |
| [35] |
SPATZ S J, SMITH L P, BAIGENT S J, et al. Genotypic characterization of two bacterial artificial chromosome clones derived from a single DNA source of the very virulent gallid herpesvirus-2 strain C12/130[J]. J Gen Virol, 2011, 92(Pt 7): 1500-1507. |
| [36] |
AFONSO C L, TULMAN E R, LU Z, et al. The genome of Turkey herpesvirus[J]. J Virol, 2001, 75(2): 971-978. DOI:10.1128/JVI.75.2.971-978.2001 |
| [37] |
BUBLOT M, SHARMA J. Vaccination against Marek's disease[M]//DAVISON F, NAIR V. Marek's Disease. Amsterdam: Elsevier, 2004: 168-185.
|
| [38] |
CRONENBERG A M, VAN GEFFEN C E, DORRESTEIN J, et al. Vaccination of broilers with HVT expressing an Eimeria acervulina antigen improves performance after challenge with Eimeria[J]. Acta Virol, 1999, 43(2-3): 192-197. |
| [39] |
LI Y Q, REDDY K, REID S M, et al. Recombinant herpesvirus of turkeys as a vector-based vaccine against highly pathogenic H7N1 avian influenza and Marek 's disease[J]. Vaccine, 2011, 29(46): 8257-8266. DOI:10.1016/j.vaccine.2011.08.115 |
| [40] |
REDDY S K, SHARMA J M, AHMAD J, et al. Protective efficacy of a recombinant herpesvirus of turkeys as an in ovo vaccine against Newcastle and Marek's diseases in specific-pathogen-free chickens[J]. Vaccine, 1996, 14(6): 469-477. DOI:10.1016/0264-410X(95)00242-S |
| [41] |
TSUKAMOTO K, SAITO S, SAEKI S, et al. Complete, long-lasting protection against lethal infectious bursal disease virus challenge by a single vaccination with an avian herpesvirus vector expressing VP2 antigens[J]. J Virol, 2002, 76(11): 5637-5645. DOI:10.1128/JVI.76.11.5637-5645.2002 |
| [42] |
HECKERT R A, RIVA J, COOK S, et al. Onset of protective immunity in chicks after vaccination with a recombinant herpesvirus of turkeys vaccine expressing Newcastle disease virus fusion and hemagglutinin-neuraminidase antigens[J]. Avian Dis, 1996, 40(4): 770-777. DOI:10.2307/1592296 |
| [43] |
ZHANG Y Y, TANG N, SADIGH Y, et al. Application of CRISPR/Cas9 gene editing system on MDV-1 genome for the study of gene function[J]. Viruses, 2018, 10(6): 279. DOI:10.3390/v10060279 |
| [44] |
张峰, 于正浩, 兰兴鸽, 等. 利用CRISPR/Cas9技术缺失MDV分离株meq基因的研究[J]. 中国家禽, 2019, 41(9): 26-32. ZHANG F, YU Z H, LAN X G, et al. Study on deletion of meq gene of MDV isolate by CRISPR/Cas9 system[J]. China Poultry, 2019, 41(9): 26-32. DOI:10.16372/j.issn.1004-6364.2019.09.005 (in Chinese) |
| [45] |
滕蔓, 郑鹿平, 刘金玲, 等. 利用CRISPR/Cas9基因编辑技术构建马立克病病毒超强毒株原癌基因meq缺失株及其鉴定[J]. 病毒学报, 2020, 36(4): 675-684. TENG M, ZHENG L P, LIU J L, et al. Editing of oncogenic meq of very-virulent Marek's disease virus by the CRISPR/Cas9 system[J]. Chinese Journal of Virology, 2020, 36(4): 675-684. DOI:10.13242/j.cnki.bingduxuebao.003763 (in Chinese) |
| [46] |
杨森, 滕蔓, 刘金玲, 等. 鸡马立克病疫苗株CVI988/Rispens meq基因编辑及缺失毒株的构建与鉴定[J]. 畜牧兽医学报, 2020, 51(8): 1970-1976. YANG S, TENG M, LIU J L, et al. Construction of meq deleted strain by gene editing of Marek's disease vaccine strain CVI988/Rispens via the CRISPR/Cas9 system and identification[J]. Acta Veterinaria et Zootechnica Sinica, 2020, 51(8): 1970-1976. (in Chinese) |
| [47] |
LUO J, TENG M, ZAI X S, et al. Efficient mutagenesis of Marek's disease virus-encoded microRNAs using a CRISPR/Cas9-based gene editing system[J]. Viruses, 2020, 12(4): 466. DOI:10.3390/v12040466 |
| [48] |
YU Z H, TENG M, SUN A J, et al. Virus-encoded miR-155 ortholog is an important potential regulator but not essential for the development of lymphomas induced by very virulent Marek's disease virus[J]. Virology, 2014, 448: 55-64. DOI:10.1016/j.virol.2013.09.017 |
| [49] |
TENG M, YU Z H, SUN A J, et al. The significance of the individual meq-clustered miRNAs of Marek's disease virus in oncogenesis[J]. J Gen Virol, 2015, 96(Pt 3): 637-649. |
| [50] |
TENG M, YU Z H, ZHAO P, et al. Putative roles as oncogene or tumour suppressor of the Mid-clustered microRNAs in Gallid alphaherpesvirus 2 (GaHV2) induced Marek's disease lymphomagenesis[J]. J Gen Virol, 2017, 98(5): 1097-1112. DOI:10.1099/jgv.0.000786 |
| [51] |
楚钰淑, 滕蔓, 周子誉, 等. LAT基因簇miRNA的CRISPR/Cas9基因编辑对马立克病病毒体外复制的影响分析[J]. 病毒学报, 2021, 37(6): 1428-1439. CHU Y S, TENG M, ZHOU Z Y, et al. CRISPR/Cas9 system-based gene editing of the LAT-clustered microRNAs and its influence on in vitro replication of Marek's disease virus[J]. Chinese Journal of Virology, 2021, 37(6): 1428-1439. DOI:10.13242/j.cnki.bingduxuebao.003947 (in Chinese) |
| [52] |
ZHAO Y G, PETHERBRIDGE L, SMITH L P, et al. Self-excision of the BAC sequences from the recombinant Marek's disease virus genome increases replication and pathogenicity[J]. Virol J, 2008, 5: 19. DOI:10.1186/1743-422X-5-19 |
| [53] |
ZHANG Y Y, LUO J, TANG N, et al. Targeted editing of the pp38 gene in Marek's disease virus-transformed cell lines using CRISPR/Cas9 system[J]. Viruses, 2019, 11(5): 391. DOI:10.3390/v11050391 |
| [54] |
ZHANG Y Y, TANG N, LUO J, et al. Marek's disease virus-encoded microRNA 155 ortholog critical for the induction of lymphomas is not essential for the proliferation of transformed cell lines[J]. J Virol, 2019, 93(17): e00713-19. |
| [55] |
TANG N, ZHANG Y Y, PEDRERA M, et al. A simple and rapid approach to develop recombinant avian herpesvirus vectored vaccines using CRISPR/Cas9 system[J]. Vaccine, 2018, 36(5): 716-722. DOI:10.1016/j.vaccine.2017.12.025 |
| [56] |
TANG N, ZHANG Y Y, SADIGH Y, et al. Generation of a triple insert live avian herpesvirus vectored vaccine using CRISPR/Cas9-based gene editing[J]. Vaccines (Basel), 2020, 8(1): 97. DOI:10.3390/vaccines8010097 |
| [57] |
CHANG P X, AMEEN F, SEALY J E, et al. Application of HDR-CRISPR/Cas9 and erythrocyte binding for rapid generation of recombinant turkey herpesvirus-vectored avian influenza virus vaccines[J]. Vaccines (Basel), 2019, 7(4): 192. DOI:10.3390/vaccines7040192 |
| [58] |
LIU L T, WANG T, WANG M Y, et al. Recombinant turkey herpesvirus expressing H9 hemagglutinin providing protection against H9N2 avian influenza[J]. Virology, 2019, 529: 7-15. DOI:10.1016/j.virol.2019.01.004 |
| [59] |
HAGAG I T, WIGHT D J, BARTSCH D, et al. Abrogation of Marek's disease virus replication using CRISPR/Cas9[J]. Sci Rep, 2020, 10(1): 10919. DOI:10.1038/s41598-020-67951-1 |
| [60] |
CHALLAGULLA A, JENKINS K A, O'NEIL T E, et al. In vivo inhibition of Marek's disease virus in transgenic chickens expressing Cas9 and gRNA against ICP4[J]. Microorganisms, 2021, 9(1): 164. DOI:10.3390/microorganisms9010164 |
| [61] |
LIU Y Z, XU Z K, ZHANG Y, et al. Marek's disease virus as a CRISPR/Cas9 delivery system to defend against avian leukosis virus infection in chickens[J]. Vet Microbiol, 2020, 242: 108589. DOI:10.1016/j.vetmic.2020.108589 |
| [62] |
LI K, LIU Y Z, XU Z K, et al. Prevention of avian retrovirus infection in chickens using CRISPR-Cas9 delivered by Marek's disease virus[J]. Mol Ther Nucleic Acids, 2020, 21: 343-353. DOI:10.1016/j.omtn.2020.06.009 |
| [63] |
CHANG P X, YAO Y X, TANG N, et al. The application of NHEJ-CRISPR/Cas9 and Cre-Lox system in the generation of bivalent duck enteritis virus vaccine against avian influenza virus[J]. Viruses, 2018, 10(2): 81. DOI:10.3390/v10020081 |
| [64] |
ATASOY M O, ROHAIM M A, MUNIR M. Simultaneous deletion of virulence factors and insertion of antigens into the infectious laryngotracheitis virus using NHEJ-CRISPR/Cas9 and Cre-Lox system for construction of a stable vaccine vector[J]. Vaccines (Basel), 2019, 7(4): 207. DOI:10.3390/vaccines7040207 |
| [65] |
滕蔓, 刘金玲, 郑鹿平, 等. CRISPR/Cas9基因编辑技术在病毒学研究中的应用及进展[J]. 病毒学报, 2020, 36(5): 946-954. TENG M, LIU J L, ZHENG L P, et al. Research progress in application of the CRISPR/Cas9 gene editing system in virology[J]. Chinese Journal of Virology, 2020, 36(5): 946-954. DOI:10.13242/j.cnki.bingduxuebao.003765 (in Chinese) |
| [66] |
TENG M, YAO Y X, NAIR V, et al. Latest advances of virology research using CRISPR/Cas9-based gene-editing technology and its application to vaccine development[J]. Viruses, 2021, 13(5): 779. DOI:10.3390/v13050779 |
(编辑 范子娟)



