一项关于碳青霉烯类抗生素敏感性值对医院病人死亡率影响的研究表明,MIC(minimum inhibitory concentration)值每增加2倍,病人死亡率也相应提高2倍[1],数据提示,随着抗菌药物的广泛使用,耐药细菌不断涌现并迅速扩散传播,严重弱化其疗效,威胁着人类健康及公共卫生安全。如何有效抗击抗生素的耐药性,成为全球科学家们当前最为关注的问题之一,新型抗菌策略的开发迫在眉睫。然而新药的研发速度远不及细菌耐药性进化的快速变迁,因此急需寻找其他途径来延缓耐药细菌所带来的后抗生素时代的危机[2]。通过联合用药于病原菌,并利用细菌的耐药性进化缺陷,如附属敏感性(collateral sensitivity)来优化耐药细菌及癌细胞的治疗方案,以更大程度地发挥药物疗效[3],延长现有药物的使用寿命是一种有效且被广泛认可的治疗思路。附属敏感性(collateral sensitivity)又称交互敏感性、协同敏感性、侧枝敏感性,由Szybalski和Bryso等于1952年首次在大肠杆菌中发现并提出,即细菌对某一药物敏感性下降时反而对另一药物敏感性提高的现象[3-5]。但可能由于以前细菌所展现的附属敏感性未能引起人们的注意、其产生机制较为复杂,以及多数附属敏感性的研究主要集中于肿瘤领域等原因,阐明细菌附属敏感性现象或机制的相关研究并不多见。直到2013年,Imamovic和Sommer[3]所绘制的大肠杆菌与各种药物间的附属敏感性关系图以及Lázár等[4]于分子层面上解释大肠杆菌对氨基糖苷类药物所产生的附属敏感性机制, 为附属敏感性的研究带来了重大的突破,细菌附属敏感性于近年又重新引起了科学家们的重视。后续多项关于附属敏感性广泛存在于细菌和癌细胞[5-6]以及利用附属敏感性来优化药物疗效方案的研究报道[3, 7-13]亦指示了其乐观的治疗前景。
由于细菌基因表达调控、代谢以及表型改变之间的互作较为复杂,细菌在耐药性进化过程中既可产生交叉耐药性(cross resistance),也可产生附属敏感性。随着对细菌附属敏感性的深入研究,人们发现其发生概率并不稳定且规律性不强[14]。因此,需要提升对细菌附属敏感性发生机制的认识,以更准确地预测细菌耐药性基因的出现、突变和传播的方法,从而更有针对性地利用附属敏感性制定新型治疗方案。本文对附属敏感性的产生规律及影响细菌附属敏感性的关键因素进行综述,并以此提出目前研究的不足以及未来发展的可能方向。
1 细菌耐药性进化与附属敏感性 1.1 附属敏感性体现了细菌耐药性进化路线选择的多样性细菌耐药性进化是极为复杂的过程,在特定抗生素存在的情况下,细菌可通过改变其结构、生理生化活动以适应抗生素给予的生存压力,进化出耐药性。从遗传学的角度而言,多种染色体的突变[4]、耐药质粒转导[15]、抗性基因的获得[16]等均是细菌常见的耐药性进化方式,而这些遗传物质改变后往往具有多效作用[17]。因此,在细菌耐药性进化过程中,其对单一药物产生耐药性的同时,也可能会对其他药物产生耐药性,即交叉耐药性;相反地,如果对其他药物的敏感性提高,则产生了附属敏感性。附属敏感性和交叉耐药性均是细菌耐药性进化过程中的存在形式,且交错存在于细菌耐药性进化过程中[8, 16], 是细菌耐药性进化路线选择多样性的一种体现。
1.2 附属敏感是细菌耐药性进化过程中的一种“进化代价”细菌耐药程度的变化及由此产生的适应性代价(fitness cost)是决定其耐药性进化是否成功的因素之一[18]。细菌对某种药物进化出耐药性利于其生存和传播的同时,也可能会因此导致其遗传负荷增加、细胞生理生化活动减弱等不利变化,即产生适应性代价。细菌在耐药性进化中的适应性代价普遍表现为发生耐药性进化的细菌在去除相应药物后,其生长速率依然下降[19]。适应性代价决定了细菌能否维持及传播其获得的耐药性[20]。
在生物进化过程中,一种性状上有益变化的积累常与其他性状的有害变化相关联[21-22]。在细菌对抗生素耐药性进化过程中,研究者也常能观察到由突变基因介导的一系列影响细胞渗透性、酸性、营养、膜和氧化应激等的防御反应[5, 23-24]。这些反应常导致许多细菌在无药情况下提高其适应性代价,或扰乱基因组表达,从而产生附属敏感性[25-28]。细菌附属敏感性机制可能涉及:1)细胞膜通透性的改变,如大肠杆菌减少对氨基糖苷类药物吸收以产生耐药性的同时也会相应减少对其他药物的排出而产生附属敏感现象[3]。2)耐药性进化提高胞内特殊活性蛋白(如酶)的表达,激活另一种前体药物提高有效药物量而产生附属敏感现象,如耐甲亚胺青霉素的大肠杆菌对呋喃妥因敏感性增强[29]。3)细胞耐药性进化致代谢通路改变,其他抗生素对细胞的毒性效应增强而产生附属敏感现象,如耐替加环素大肠杆菌的lon突变体对呋喃妥因敏感性大幅度增加[30-32]。4)耐药性进化令其他药物对作用靶点的亲和力增加而产生附属敏感现象,如耐甲氧西林金黄色葡萄球菌PBP2a蛋白上E246G突变使其在克拉维酸存在的情况下对青霉素的敏感性提高[33]。另外,不同菌株对某些药物的附属敏感性可能涉及一个或多个不同的作用机制[17]。
综上所述,细菌的附属敏感性以及适应性代价是耐药性进化过程中的一种取舍(trade-off),即“进化代价”。
1.3 细菌进化的适应性代价与附属敏感性之间的关系基于细菌附属敏感性设计药物治疗方案时,细菌的适应性代价能在一定程度上提高疗效,抑制细菌耐药性的发展[34];近期,也有研究人员利用数学模型,通过耐药细菌的适应性的变化来预测其附属敏感效应[35],但适应性代价与附属敏感性之间并无必然联系。换而言之,细菌耐药性进化过程中所产生的适应性代价不一定会引起细菌的附属敏感性,且细菌附属敏感性的产生不一定伴随着适应性代价[3, 34, 36-39]。主要原因是部分细菌可通过代偿突变来改善耐药性进化所产生的适应性代价[19, 40]。此外,一些利于提高耐药菌适应性的突变在相应野生株中反而是有害的[41],另外,一项对假单胞菌的研究显示,50%的常见rpoB突变所引起的适应性变化取决于其所在菌株的遗传背景[20]。不仅如此,特定的耐药突变在不同基因背景下,往往会因遗传上位效应而导致细菌不同的适应性变化结果[18, 20]。
2 附属敏感性发生的影响因素 2.1 细菌附属敏感性机制存在不稳定性Barbosa等[42]在试验中观察到,不同耐头孢磺啶铜绿假单胞杆菌PA14的平行样本对其他抗生素(如庆大霉素和链霉素)的敏感性呈现相反的变化。这是因为细菌发生耐药性进化的机制具有多样性,且细菌耐药性进化轨迹具有一定随机性,而细菌附属敏感性取决于细菌耐药性进化所选择的轨迹,因此细菌与药物间的附属敏感反应具有不确定性。这意味着同一株细菌,即使每次都用相同的处理条件,也可能会产生不一致的附属敏感性和交叉敏感性[14]。Wright的经典适应度景观图模型很好地解释了何为进化路线的多样性[43]。
假设将细菌的基因型当成一个二维平面(图 1),适应性表示为基因型平面上的高度。细菌的耐药性进化常有多个适应峰,我们可把细菌的耐药性进化看作是其随机朝着不同方向“上坡”的过程,而在不同的适应峰之间会有一个进化分支点(evolutionary branching point),当细菌在进化过程中的基因型在此进化分支点时,它未来的进化路线就有了多种可能[14, 44]。因此,对于仅由突变产生耐药性的细菌来说,其最终耐药性进化结果的显著差异与突变的随机性相关。也正由于基因突变的随机性,细菌在耐药性进化过程中衍生附属敏感性的概率并不稳定。同时,细菌产生附属敏感的可重复性还会受到药物直接作用的影响,某些种类的药物确实能诱导出较为稳定和固定的细菌附属敏感性[45]。
此外,Imamovic等[8]在一项纵向研究中发现,不同时间点从同一患者体内分离出来的同种菌株,其耐药特性也有较大差异。其中,最主要的原因是随着时间的推移,细菌在抗生素压力下产生并积累了多种突变。这些突变的逐步获得与积累,以及基因的上位效应等所带来的影响,造成细菌耐药性进化中交叉耐药性和附属敏感性的动态变化,增加了细菌附属敏感性的不稳定性[36]。
综上所述,细菌附属敏感性的不稳定性体现在其进化方向的不确定性,以及在时间维度上积累的不同突变而导致的动态变化。
2.2 细菌的遗传背景影响其附属敏感性发生研究者们常在细菌附属敏感性的研究中观察到,同一基因位点的突变可在不同种群造成一定的耐药性差异,即使两个种群对同种药物的耐药性进化方向相同,其耐药性变化程度及附属敏感性也常呈现一定的差异[36]。这些差异由两个细菌种群中目标突变位点所在的基因内部以及基因与基因间的遗传背景差异所影响。通常,遗传背景差异越大,细菌种群间的附属敏感性、交叉耐药性、适应性代价及耐药程度等的差异越明显[36]。
例如,大肠杆菌与沙门菌两者TrkH基因的相似度约为97.5%。TrkH基因主要编码钾离子转运体蛋白,而大肠杆菌摄取氨基糖苷类药物需通过呼吸作用[37]和质子驱动力(proton motive force,PMF)的介导,TrkH T350K突变基因可减少质子驱动力,导致大肠杆菌中依赖PMF介导的主要外排系统AcrAB-To1C的活性下降[3, 17],从而导致细菌摄取氨基糖苷类药物减少,产生相应耐药性,与此同时也会因此减弱对其他药物的外排作用,对其他类药物产生附属敏感性。将大肠杆菌和沙门菌的TrkH基因做相同突变后,大肠杆菌与沙门菌对氨苄西林和红霉素的耐药性变化方向并不一致[36]。并且,导入TrkH T350K突变基因片段后的大肠杆菌与沙门菌对不同种类药物所表现的附属敏感性也并不完全一致:导入突变基因片段的大肠杆菌对9种药物(氨苄青霉素、头孢西丁、强力霉素、四环素、氯霉素、红霉素、萘啶酮酸、呋喃妥英、环丙沙星)产生附属敏感性,然而导入突变基因的沙门菌仅对4种药物产生附属敏感性(头孢西丁、强力霉素、四环素、萘啶酮酸)。这种不同由两种细菌TrkH的基因内上位效应(intragenic epistasis)以及细菌基因间上位效应所引起,后者为主要的影响。这些上位效应的相互作用妨碍了人们预测附属敏感性出现的准确率。
2.3 环境因素影响细菌附属敏感性大量现有研究报道了影响细菌附属敏感性的重要遗传因素,除此以外,许多研究表明,环境因素也对细菌附属敏感性有重大影响。这些环境因素体现在细菌所生存环境中的抗生素压力强度、抗生素环境变化速率、不同的适应性进化方法、宿主内环境等方面。了解影响细菌附属敏感性的环境因素可令人们对其保守性有更深入的理解,从而在临床上更好地预测以及应用细菌附属敏感性。
2.3.1 抗生素压力强度影响细菌附属敏感性 Oz等[46]通过高/低强度抗生素持续诱导大肠杆菌的耐药性进化试验证明(图 2),抗生素的选择强度是导致抗生素耐药性问题复杂化的一个隐藏的重要参数,高强度的抗生素压力更利于细菌的直接耐药性、交叉耐药性以及附属敏感性的进化。在温和的抗生素压力下,部分细菌亚群仍能继续生长,此时,细菌基因突变的目的往往是增强其在这一条件下的生长适应性,如通过对多药耐药基因或者转录子的改变来适应环境的变化。而在高强度抗生素压力下,细菌的首要任务为生存,因此它们不惜采取具高适应性成本的基因突变,如以重要细胞器(核糖体)或者必须酶(RNA聚合酶)为目标的基因突变来提高生存概率[47-49]。不仅如此,细菌在较高强度抗生素压力下会比其在温和的抗生素压力下积累更多类型的基因突变。这些突变常影响不同细胞通路(相应药物作用抑制的或者非相应药物作用抑制的特殊通路)或提高耐药性进化过程的适应性,导致细菌出现更多交叉耐药或附属敏感反应。
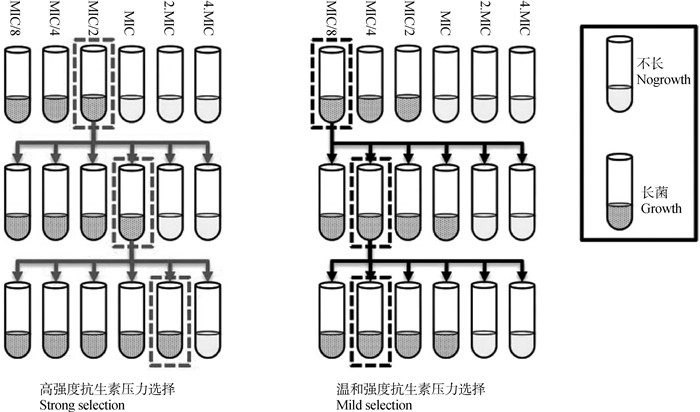
|
细菌在不同的药物浓度,且药物浓度梯度的跨度大于预期MIC的试管中生长。于高强度抗生素梯度以及温和强度的抗生素梯度压力下诱导培养细菌群体22 h,将能在最高药物浓度下存活的细菌群体转移到新的具有更高药物浓度的试管中培养,持续22 d Bacterial populations were grown in several tubes with increased drug concentrations that span the expected MIC of the population. Populations were grown for approximately 22 h and the populations surviving in the highest drug concentration were transferred to new culture vials with higher drug concentration, lasting for 22 days 图 2 强、弱选择条件下液体培养细菌进化试验示意图[46] Fig. 2 Schematic illustration of bacterial evolution experiments in liquid culture under strong and mild selection[46] |
2.3.2 抗生素环境变化速率影响细菌附属敏感性 除了抗生素强度,环境变化速率(rate of environmental change)也会影响细菌的耐药性进化。以抗生素浓度变化速率为例,Lindsey等[50]通过使用3种不同浓度增量速率(快速、适中、平缓)的利福平来诱导大肠杆菌的耐药性进化(后续将简称为抗生素增量法),并检测大肠杆菌对利福平耐药的相关基因(编码RNA聚合酶β亚基的rpoB基因)序列[51-53]。试验结果显示,诱导过程中大肠杆菌产生的所有类型的基因突变都是非同义的,平缓、适中的抗生素增量法诱导的细菌的核苷酸多样性显著高于快速抗生素增量法诱导的细菌,这可能是由于平缓和适中的药物浓度增量速率给细菌种群提供更多发生和传播耐药性突变的机会。除此以外,研究人员还发现由于受到基因符号上位效应(sign genetic epistasis)和环境上位效应(environmental epistasis),以及后一耐药突变的发生与否常取决于前一个关键的耐药突变是否发生等因素的影响[8],在快速抗生素增量法下诱导的细菌无法获得在平缓和适中药物浓度增量速率下所取得的耐药性进化路线,以至细菌最终无法获得可适应最高药物浓度的突变基因型,这表明循序渐进的低水平耐药突变更利于细菌的耐药性发展[54]。除抗生素浓度的增量速率以外,温度以及CO2等环境因素的变化速率不同,也可能会影响生物基因型的进化路线[50, 55-56],导致细菌附属敏感性差异。
2.3.3 不同适应性进化方法影响细菌附属敏感性 Jahn等[57]用不同的抗生素适应性进化方法(adaptive laboratory evolution,ALE),如梯度法和每日25%、50%、100%浓度增量法(后续将简称为增量法),来诱导大肠杆菌的耐药性(图 3)。结果表明,除了每日100%增量法,其他方法诱导后细菌耐药性均超过临床折点。在不同适应性进化方法诱导下,处于临床折点附近的大肠杆菌对其他药物的交叉耐药性及附属敏感性大部分相似。对比梯度法和25%增量法,梯度进化谱系生长速度较慢,25%增量法诱导的细菌增长速度最快;在25%增量法以及梯度法诱导后细菌基因型很相似,但是相对而言,25%增量法所诱导的细菌基因型更丰富且几乎携带了所有梯度法诱导的细菌所发生的突变,因此,推测在梯度法诱导后细菌所发生的突变可能是最重要的抗性表型。此项研究所展现的“较低浓度抗生素压力下细菌基因型更丰富”的结论与Oz等[46]所提出的“细菌在高浓度抗生素压力下会比其在低浓度抗生素压力下积累更多类型的基因突变”的结论不一致,原因尚不明确。可以确定的是细菌一旦有存活的机会,往往会朝着更利于生存的方向进化。因此适当的环境刺激及适应性代价反而常促进细菌的进化,让其得以不断自我完善。而附属敏感效应正是细菌不断进化以适应抗生素压力过程中的所产生的缺陷,人们利用此缺陷来延缓其耐药性进化速度以达到“减抗增效”的目的。
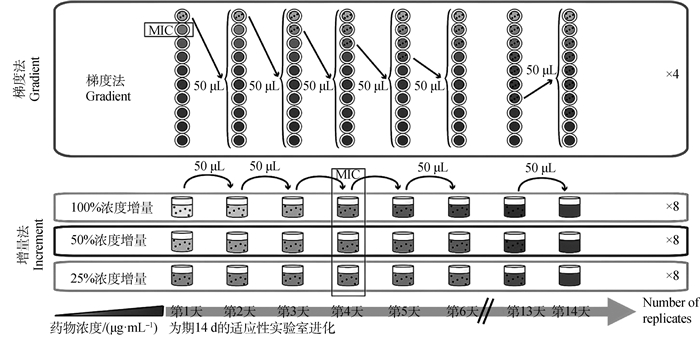
|
使用4种不同的适应性实验室进化法诱导大肠杆菌对不同药物的耐药性。这4种不同的方法分别为梯度法、100%浓度增量法、50%浓度增量法和25%浓度增量法。梯度法是将细菌接种到10个等药物浓度梯度,且每个梯度为2倍的96孔板中,第2天将生长在最高浓度的细胞接种到新的梯度中,持续14 d。在增量方法中,每转移一次,药物浓度提高100%、50%或25%。所有方法体系均在试验的第4天达到野生型菌株的最低抑菌浓度(MIC) Adaptation of E. coli K12 to three antibiotics following four adaptation protocols with different selection strengths. The four selection systems are named "gradient" "100% increment" "50% increment" and "25% increment". In the gradient approach, cells were inoculated into a 2-fold, 10-step drug gradient and cells growing in the highest concentration were used for inoculation of a fresh gradient. In the increment approaches the drug concentration was raised with every transfer either by 100%, 50%, or 25%. The wild type minimal inhibitory concentration (MIC) was reached on the fourth day of the experiment for all increment lineages 图 3 不同抗生素适应性实验室进化方法[57] Fig. 3 Different adaptive laboratory evolution experimental design of the study[57] |
此项研究表明,不同的环境因素对细菌的耐药性进化有决定性影响。其中,抗生素的增量速率常决定细菌的最大耐药水平[57],间接影响细菌的附属敏感性。而一旦细菌种群达到指定的耐药水平,无论其所采用的实验室进化方式如何,细菌种群间的基因型与表型都有一定的相似性。这是由于进化的本质是自然选择,虽然细菌耐药性进化具有一定的随机性从而丰富了其耐药性进化路线的多样性,但是细菌在适应一定选择压力的过程中可获得的变化数量有限且进化倾向于此类突变,细菌的进化具有一定的方向性,因此暴露在类似环境中的细菌所发生的变化具有一定的相似性[58]。而在25%浓度增量法以及梯度法诱导下,细菌种群间基因型的差异提示,不同的适应性进化方法也可能会造成其基因型有细微的不同,至于这些细微的不同是否会进一步地影响细菌后续的进化轨迹及附属敏感效应,仍需通过更深入的高通量试验研究解答。
2.3.4 细菌寄生环境的理化性质影响附属敏感性 除了上述常见于体外试验的环境因素,细菌寄生环境的理化性质对细菌的附属敏感性也有重大影响。例如:胆汁可上调细菌外排泵[59];锌可降低氨基糖苷类降解酶的活性[60];高温可改变无药条件下耐利福平细菌突变群体的生长状况[61];细菌可因不同的pH、温度、胆盐浓度等环境条件而更倾向于某种耐药性进化[62]。这意味着,宿主的某些生理条件也可能会影响细菌在有药以及无药环境下的进化路线,从而影响其附属敏感性。例如,耐庆大霉素大肠杆菌的cpxA基因突变群体在培养条件为37 ℃时对头孢呋辛表现出交叉耐药性,然而却在培养条件为42 ℃时表现出对头孢呋辛的附属敏感性;42 ℃,甲氧苄氨嘧啶存在条件下,大肠杆菌更倾向于进化成具有phoPQ突变的耐药群体,此群体在42 ℃培养条件下对呋喃妥因表现出交叉耐药性,但却在37 ℃培养条件下表现出相应的附属敏感性[62]。这提示细菌可因其所寄生环境的理化性质不同而表现出不同的附属敏感性。因此,应用附属敏感性设计临床治疗方案时,也需要充分考虑细菌寄生环境的理化性质对其附属敏感性的影响。
3 细菌附属敏感性的潜在应用前景 3.1 高度保守的附属敏感效应及其预测准确性是附属敏感性应用的前提慢性感染的特性决定其需要特定的治疗方案,因此附属敏感性在临床慢性感染的治疗上具有较大的发展前景[8]。然而,多项研究表明,细菌耐药性进化过程中附属敏感以及交叉耐药效应普遍是交替出现的[3, 63],这意味着若要将附属敏感性应用到临床治疗,需保证治疗过程中细菌总是或者大概率对某些药物对进化出稳定且固定的附属敏感性,即高度保守的附属敏感效应(conserved collateral sensitivity effects)。近期有多项研究报道了细菌与药物间高度保守的附属敏感反应,为细菌附属敏感性的应用前景带来曙光。例如,大肠杆菌对环丙沙星耐药的同时,对庆大霉素、磷霉素有较高保守程度的附属敏感性,而影响细菌外排泵系统的突变是引起其附属敏感性发生的重要因素[63]。无独有偶,Imamovic等[8]也在不同囊性纤维病患者体内分离出具有不同遗传背景的铜绿假单胞杆菌,并发现这些临床样本对环丙沙星耐药的同时,对庆大霉素、黏菌素具有较高保守程度的附属敏感性,这种附属敏感的产生与nfxB基因突变有关。nfxB基因将导致MexC转运体的过度表达,影响细菌的多重耐药外排系统,从而引起试验株的附属敏感效应。因此,nfxB基因突变或MexC蛋白的丰度可能是一种潜在的、可预测附属敏感效应的标志物。这些研究结果启示,在不同遗传背景的细菌种群中,附属敏感效应机制的相似性及可预测性是将细菌的附属敏感效应运用于临床的前提,而这种相似性与可预测性取决于由细菌种群的耐药机制所造成细菌生理变化的相似程度。或许,未来可通过检测细菌附属敏感效应的基因组或蛋白质组生物标志物来预测耐药细菌的附属反应,从而更科学地设计药物治疗方案,提高慢性感染治疗的成功率。
上文提到,细菌遗传背景的差异以及细菌耐药突变路线的多样性,是预测细菌种群形成高度保守的附属敏感效应的最大的障碍。针对此问题,Ardell和Kryazhimskiy[35]用联合适应度效应分布(joint distribution of fitness effects,JDFE)来量化细菌耐药突变的多效性所带来的多种影响。JDFE是指细菌在产生一个新突变后,将在初始使用的抗生素(用以诱导产生耐药性的抗生素)环境与之后使用的另一抗生素环境中表现出不同的适应性变化,将这两种变化的可能性组合起来,并计算其发生概率,这种概率称为JDFE[35]。利用JDFE和数学模型模拟短时间内细菌附属敏感效应的动态变化,并优化了先前用以预测附属敏感反应发生率及估算预测准确性的计算模型。优化后的计算模型减少了细菌遗传背景差异及耐药突变路线的多样性,但强调了细菌耐药突变种类对预测结果的影响。但由于缺乏高通量试验数据的支持以及体内试验的验证;计算模型仅局限于细菌多染色体的突变;且在设计模型时简化了许多参数对细菌耐药性进化路线的影响,该计算模型尚且不够完善,后续仍需对此类数学模型做更深入的研究。
3.2 基于细菌附属敏感性制定科学的用药策略基于细菌附属敏感性所设计的给药方案可在不降低疗效的前提下减少抗生素用量,从而降低抗生素相关的毒性风险,扩大抗生素的应用范围,并限制抗生素耐药性发展。附属敏感效应为提高药物疗效和延长现有药物使用寿命的问题提供了新思路,但是细菌的耐药性进化是复杂的,即使细菌对某些药物存在附属敏感性,不当的用药方案也可能会加速细菌耐药性的进化。因此,更需充分考虑以下问题:如何基于特定细菌的附属敏感效应制定科学而有效的药物治疗方案,在设计此方案的过程中需要重视哪些关键因素?
上文已强调各种环境因素对细菌附属敏感性有重大影响,需在制定用药方案时给予充分考虑。此外,另一项研究通过数学模型显示:在细菌和药物间存在附属敏感效应的情况下,同时用药和间隔1 d的轮换用药治疗法可显著抑制细菌的耐药性进化。对于间隔1 d的轮换用药疗法,开始使用时间依赖型的抗生素比最初使用浓度依赖型的抗生素更有效地抑制细菌耐药性进化;当细菌对药物的附属敏感性是单向的或细菌对所计划使用药物存有耐药性的情况下,需慎重考虑用药的先后次序。这提示,基于细菌附属敏感性设计药物治疗方案的效果,与用药间隔时间、药物类型、药物浓度、用药顺序、细菌现有耐药情况等多种因素密切相关[34]。因此若要形成成熟的基于细菌附属敏感性设计用药方案,我们仍需深入思考如病原菌和药物的特异性、药物间附属敏感效应的方向性、轮换用药的最佳切换时间、药物代谢动力学、药物效应动力学、药物间不同的相互作用[34, 45]等更多方面的因素对用药方案的影响。
4 小结近年来,借用细菌的附属敏感性来延长、优化现有药物的疗效是治疗耐药细菌的新思路之一,但目前对于细菌附属敏感性的研究尚处于初始阶段,由于进化轨迹的随机性所造成细菌附属敏感效应的异质性[9, 42]、较低重复性[14],以及细菌的表观遗传效应、基因缺失、基因水平转移等因素均增加了实际情况下细菌耐药性进化的复杂性及预测难度。为了打破体外试验的多种限制因素,更接近自然演化的耐药性进化情况,应着重收集临床治疗前后的菌株并对其进行药敏试验,分析其交叉耐药性和附属敏感性,并将分析结果与高通量实验室数据和数学模型相结合,以更好地评估细菌附属敏感性出现的保守程度,解释进化的内在随机性以及保证细菌附属敏感效应的高度保守性,从而更准确地解析且更好地应用细菌附属敏感效应。后续仍需要结合更多的前瞻性研究,提高对细菌耐药性进化以及附属敏感性的认识,以更加准确地预测及利用附属敏感效应突破由细菌耐药性进化引起的难点问题。
| [1] |
ESTERLY J S, WAGNER J, MCLAUGHLIN M M, et al. Evaluation of clinical outcomes in patients with bloodstream infections due to gram-negative bacteria according to carbapenem MIC stratification[J]. Antimicrob Agents Chemother, 2012, 56(9): 4885-4890. |
| [2] |
LAXMINARAYAN R. Antibiotic effectiveness: balancing conservation against innovation[J]. Science, 2014, 345(6202): 1299-1301. DOI:10.1126/science.1254163 |
| [3] |
IMAMOVIC L, SOMMER M O A. Use of collateral sensitivity networks to design drug cycling protocols that avoid resistance development[J]. Sci Transl Med, 2013, 5(204): 204ra132. |
| [4] |
LÁZÁR V, SINGH G P, SPOHN R, et al. Bacterial evolution of antibiotic hypersensitivity[J]. Mol Syst Biol, 2013, 9(1): 700. DOI:10.1038/msb.2013.57 |
| [5] |
PÁL C, PAPP B, LÁZÁR V. Collateral sensitivity of antibiotic-resistant microbes[J]. Trends Microbiol, 2015, 23(7): 401-407. DOI:10.1016/j.tim.2015.02.009 |
| [6] |
BAYM M, STONE L K, KISHONY R. Multidrug evolutionary strategies to reverse antibiotic resistance[J]. Science, 2016, 351(6268): eaad3292. DOI:10.1126/science.aad3292 |
| [7] |
ZHAO B Y, SEDLAK J C, SRINIVAS R, et al. Exploiting temporal collateral sensitivity in tumor clonal evolution[J]. Cell, 2016, 165(1): 234-246. DOI:10.1016/j.cell.2016.01.045 |
| [8] |
IMAMOVIC L, ELLABAAN M M H, MACHADO A M D, et al. Drug-driven phenotypic convergence supports rational treatment strategies of chronic infections[J]. Cell, 2018, 172(1-2): 121-134. DOI:10.1016/j.cell.2017.12.012 |
| [9] |
DHAWAN A, NICHOL D, KINOSE F, et al. Collateral sensitivity networks reveal evolutionary instability and novel treatment strategies in ALK mutated non-small cell lung cancer[J]. Sci Rep, 2017, 7(1): 1232. DOI:10.1038/s41598-017-00791-8 |
| [10] |
JENSEN P B, HOLM B, SORENSEN M, et al. In vitro cross-resistance and collateral sensitivity in seven resistant small-cell lung cancer cell lines: preclinical identification of suitable drug partners to taxotere, taxol, topotecan and gemcitabin[J]. Br J Cancer, 1997, 75(6): 869-877. DOI:10.1038/bjc.1997.154 |
| [11] |
PLUCHINO K M, HALL M D, GOLDSBOROUGH A S, et al. Collateral sensitivity as a strategy against cancer multidrug resistance[J]. Drug Resist Updat, 2012, 15(1-2): 98-105. |
| [12] |
WANG L Q, DE OLIVEIRA R L, HUIJBERTS S, et al. An acquired vulnerability of drug-resistant melanoma with therapeutic potential[J]. Cell, 2018, 173(6): 1413-1425. DOI:10.1016/j.cell.2018.04.012 |
| [13] |
KIM S, LIEBERMAN T D, KISHONY R. Alternating antibiotic treatments constrain evolutionary paths to multidrug resistance[J]. Proc Natl Acad Sci U S A, 2014, 111(40): 14494-14499. DOI:10.1073/pnas.1409800111 |
| [14] |
NICHOL D, RUTTER J, BRYANT C, et al. Antibiotic collateral sensitivity is contingent on the repeatability of evolution[J]. Nat Commun, 2019, 10(1): 334. |
| [15] |
HERENCIAS C, RODRÍGUEZ-BELTRÁN J, LEÓN-SAMPEDRO R, et al. Collateral sensitivity associated with antibiotic resistance plasmids[J]. eLife, 2021, 10: e65130. |
| [16] |
ROSENKILDE C E H, MUNCK C, PORSE A, et al. Collateral sensitivity constrains resistance evolution of the CTX-M-15 β-lactamase[J]. Nat Commun, 2019, 10(1): 618. DOI:10.1038/s41467-019-08529-y |
| [17] |
ROEMHILD R, ANDERSSON D I. Mechanisms and therapeutic potential of collateral sensitivity to antibiotics[J]. PLoS Pathog, 2021, 17(1): e1009172. DOI:10.1371/journal.ppat.1009172 |
| [18] |
KNOPP M, ANDERSSON D I. Predictable phenotypes of antibiotic resistance mutations[J]. mBio, 2018, 9(3): e00770-18. |
| [19] |
ANDERSSON D I, HUGHES D. Antibiotic resistance and its cost: is it possible to reverse resistance?[J]. Nat Rev Microbiol, 2010, 8(4): 260-271. |
| [20] |
VOGWILL T, KOJADINOVIC M, MACLEAN R C. Epistasis between antibiotic resistance mutations and genetic background shape the fitness effect of resistance across species of Pseudomonas[J]. Proc Biol Sci, 2016, 283(1830): 20160151. |
| [21] |
BENNETT A F, LENSKI R E. An experimental test of evolutionary trade-offs during temperature adaptation[J]. Proc Natl Acad Sci U S A, 2007, 104(S1): 8649-8654. |
| [22] |
VELICER G J, SCHMIDT T M, LENSKI R E. Application of traditional and phylogenetically based comparative methods to test for a trade-off in bacterial growth rate at low versus high substrate concentration[J]. Microb Ecol, 1999, 38(3): 191-200. |
| [23] |
PALMER A C, KISHONY R. Opposing effects of target overexpression reveal drug mechanisms[J]. Nat Commun, 2014, 5(1): 4296. |
| [24] |
TOPRAK E, VERES A, MICHEL J B, et al. Evolutionary paths to antibiotic resistance under dynamically sustained drug selection[J]. Nat Genet, 2012, 44(1): 101-105. |
| [25] |
LÁZÁR V, NAGY I, SPOHN R, et al. Genome-wide analysis captures the determinants of the antibiotic cross-resistance interaction network[J]. Nat Commun, 2014, 5(1): 4352. |
| [26] |
DRAGOSITS M, MOZHAYSKIY V, QUINONES-SOTO S, et al. Evolutionary potential, cross-stress behavior and the genetic basis of acquired stress resistance in Escherichia coli[J]. Mol Syst Biol, 2013, 9: 643. |
| [27] |
OSHIMA T, AIBA H, MASUDA Y, et al. Transcriptome analysis of all two-component regulatory system mutants of Escherichia coli K-12[J]. Mol Microbiol, 2002, 46(1): 281-291. |
| [28] |
VENKATESH B, BABUJEE L, LIU H, et al. The Erwinia chrysanthemi 3937 PhoQ sensor kinase regulates several virulence determinants[J]. J Bacteriol, 2006, 188(8): 3088-3098. |
| [29] |
ROEMHILD R, LINKEVICIUS M, ANDERSSON D I. Molecular mechanisms of collateral sensitivity to the antibiotic nitrofurantoin[J]. PLoS Biol, 2020, 18(1): e3000612. |
| [30] |
BRYANT D W, MCCALLA D R. Nitrofuran induced mutagenesis and error prone repair in Escherichia coli[J]. Chem-Biol Interact, 1980, 31(2): 151-166. |
| [31] |
HUISMAN O, D'ARI R. An inducible DNA replication-cell division coupling mechanism in E. coli[J]. Nature, 1981, 290(5809): 797-799.
|
| [32] |
BI E, LUTKENHAUS J. Cell division inhibitors SulA and MinCD prevent formation of the FtsZ ring[J]. J Bacteriol, 1993, 175(4): 1118-1125. |
| [33] |
MIZUSAWA S, GOTTESMAN S. Protein degradation in Escherichia coli: The lon gene controls the stability of sulA protein[J]. Proc Natl Acad Sci U S A, 1983, 80(2): 358-362. |
| [34] |
AULIN L B S, LIAKOPOULOS A, VAN DER GRAAF P H, et al. Design principles of collateral sensitivity-based dosing strategies[J]. Nat Commun, 2021, 12(1): 5691. |
| [35] |
ARDELL S M, KRYAZHIMSKIY S. The population genetics of collateral resistance and sensitivity[J]. eLife, 2021, 10: e73250. |
| [36] |
APJOK G, BOROSS G, NYERGES Á, et al. Limited evolutionary conservation of the phenotypic effects of antibiotic resistance mutations[J]. Mol Biol Evol, 2019, 36(8): 1601-1611. |
| [37] |
CHEN H L, JIANG Y, LI M M, et al. Acquisition of tigecycline resistance by carbapenem-resistant Klebsiella pneumoniae confers collateral hypersensitivity to aminoglycosides[J]. Front Microbiol, 2021, 12: 674502. |
| [38] |
GAGNEUX S, LONG C D, SMALL P M, et al. The competitive cost of antibiotic resistance in Mycobacterium tuberculosis[J]. Science, 2006, 312(5782): 1944-1946. |
| [39] |
RODRíGUEZ-ROJAS A, MACIÁ M D, COUCE A, et al. Assessing the emergence of resistance: the absence of biological cost in vivo may compromise fosfomycin treatments for P. Aeruginosa infections[J]. PLoS One, 2010, 5(4): e10193. |
| [40] |
BJOÖRKMAN J, NAGAEV I, BERG O G, et al. Effects of environment on compensatory mutations to ameliorate costs of antibiotic resistance[J]. Science, 2000, 287(5457): 1479-1482. |
| [41] |
MAISNIER-PATIN S, PAULANDER W, PENNHAG A, et al. Compensatory evolution reveals functional interactions between ribosomal proteins S12, L14 and L19[J]. J Mol Biol, 2007, 366(1): 207-215. |
| [42] |
BARBOSA C, TREBOSC V, KEMMER C, et al. Alternative evolutionary paths to bacterial antibiotic resistance cause distinct collateral effects[J]. Mol Biol Evol, 2017, 34(9): 2229-2244. |
| [43] |
JONES D F. Proceedings of the sixth International congress of genetics[M]. New York: Brooklyn Botanic Garden, 1932.
|
| [44] |
MIRA P M, CRONA K, GREENE D, et al. Rational design of antibiotic treatment plans: a treatment strategy for managing evolution and reversing resistance[J]. PLoS One, 2015, 10(9): e0139387. |
| [45] |
MALTAS J, WOOD K B. Pervasive and diverse collateral sensitivity profiles inform optimal strategies to limit antibiotic resistance[J]. PLoS Biol, 2019, 17(10): e3000515. |
| [46] |
OZ T, GUVENEK A, YILDIZ S, et al. Strength of selection pressure is an important parameter contributing to the complexity of antibiotic resistance evolution[J]. Mol Biol Evol, 2014, 31(9): 2387-2401. |
| [47] |
BILGIN N, RICHTER A A, EHRENBERG M, et al. Ribosomal RNA and protein mutants resistant to spectinomycin[J]. EMBO J, 1990, 9(3): 735-739. |
| [48] |
BABA T, ARA T, HASEGAWA M, et al. Construction of Escherichia coli K-12 in-frame, single-gene knockout mutants: the Keio collection[J]. Mol Syst Biol, 2006, 2(1): 2006.0008. |
| [49] |
LOZOVSKY E R, CHOOKAJORN T, BROWN K M, et al. Stepwise acquisition of pyrimethamine resistance in the malaria parasite[J]. Proc Natl Acad Sci U S A, 2009, 106(29): 12025-12030. |
| [50] |
LINDSEY H A, GALLIE J, TAYLOR S, et al. Evolutionary rescue from extinction is contingent on a lower rate of environmental change[J]. Nature, 2013, 494(7438): 463-467. |
| [51] |
REYNOLDS M G. Compensatory evolution in rifampin-resistant Escherichia coli[J]. Genetics, 2000, 156(4): 1471-1481. |
| [52] |
MACLEAN R C, PERRON G G, GARDNER A. Diminishing returns from beneficial mutations and pervasive epistasis shape the fitness landscape for rifampicin resistance in Pseudomonas aeruginosa[J]. Genetics, 2010, 186(4): 1345-1354. |
| [53] |
HALL A R, MACLEAN R C. Epistasis buffers the fitness effects of rifampicin- resistance mutations in Pseudomonas aeruginosa[J]. Evolution, 2011, 65(8): 2370-2379. |
| [54] |
SOLÉ M, FÀBREGA A, COBOS-TRIGUEROS N, et al. In vivo evolution of resistance of Pseudomonas aeruginosa strains isolated from patients admitted to an intensive care unit: mechanisms of resistance and antimicrobial exposure[J]. J Antimicrob Chemother, 2015, 70(11): 3004-3013. |
| [55] |
DALLINGER W H. The president's address[J]. J Roy Microsc Soc, 1887, 7(2): 185-199. |
| [56] |
KLIRONOMOS J N, ALLEN M F, RILLIG M C, et al. Abrupt rise in atmospheric CO2 overestimates community response in a model plant-soil system[J]. Nature, 2005, 433(7026): 621-624. |
| [57] |
JAHN L J, MUNCK C, ELLABAAN M M H, et al. Adaptive laboratory evolution of antibiotic resistance using different selection regimes lead to similar phenotypes and genotypes[J]. Front Microbiol, 2017, 8: 816. |
| [58] |
LOSOS J B. Convergence, adaptation, and constraint[J]. Evolution, 2011, 65(7): 1827-1840. |
| [59] |
ROSENBERG E Y, BERTENTHAL D, NILLES M L, et al. Bile salts and fatty acids induce the expression of Escherichia coli AcrAB multidrug efflux pump through their interaction with Rob regulatory protein[J]. Mol Microbiol, 2003, 48(6): 1609-1619. |
| [60] |
LIN D L, TRAN T, ALAM J Y, et al. Inhibition of aminoglycoside 6'-N-acetyltransferase type Ib by zinc: reversal of amikacin resistance in Acinetobacter baumannii and Escherichia coli by a zinc ionophore[J]. Antimicrob Agents Chemother, 2014, 58(7): 4238-4241. |
| [61] |
RODRÍGUEZ-VERDUGO A, GAUT B S, TENAILLON O. Evolution of Escherichia coli rifampicin resistance in an antibiotic-free environment during thermal stress[J]. BMC Evol Biol, 2013, 13(1): 50. |
| [62] |
ALLEN R C, PFRUNDER-CARDOZO K R, HALL A R. Collateral sensitivity interactions between antibiotics depend on local abiotic conditions[J]. mSystems, 2021, 6(6): e01055-21. |
| [63] |
PODNECKY N L, FREDHEIM E G A, KLOOS J, et al. Conserved collateral antibiotic susceptibility networks in diverse clinical strains of Escherichia coli[J]. Nat Commun, 2018, 9(1): 3673. |
(编辑 白永平)




