肠球菌(Enterococcus)为革兰阳性菌,广泛分布于自然环境(如土壤、水和植物)中,也是人和动物胃肠道内常见的共生菌[1]。由于其参与宿主多项生理功能,因此被认为是对人类无害的共栖菌。但当机体的免疫环境遭到抑制或破坏时,肠球菌则会引起感染,主要包括伤口、尿道、腹腔、血流和外科手术感染[2-3]。而肠球菌中的粪肠球菌和屎肠球菌是引发外科术后和尿道感染的主要因素,是导致菌血症的第3大因素。目前肠球菌是院内感染的重要病原菌。由于肠球菌具有固有耐药特征,并且具有获得耐药性和毒力因子及形成生物被膜的能力和倾向[4-5],因此给临床治疗带来困难和挑战。在2019年的关于我国临床细菌耐药检测的调查中,屎肠球菌的分离率为53.7%(10 413/19 403),粪肠球菌的分离率为39.6%(7 676/19 403)。在动物源性肠球菌中,粪肠球菌和屎肠球菌同样是分离率最高的两种肠球菌。随着抗生素在养殖业的大量、长期使用,尤其动物养殖过程中抗生素作为促生长剂的应用,导致动物源细菌耐药性快速增长。而抗生素耐药性可随动物肠道细菌传播给人类的潜在风险已经成为全球关注的食品安全和公共卫生问题。肠球菌具有天然的将耐药性及毒力因子以水平转移方式传播给其他细菌的能力,因此养殖场长期使用抗生素选择出的耐药肠球菌,可成为人类耐药条件致病菌的储库。通常认为肠球菌的致病能力比较复杂。有研究表明,肠球菌自身分泌的毒力因子与其致病力有关,毒力因子主要包括胶原蛋白黏附素(ace)、溶血素(cly)、心内膜炎抗原(efaA)、表面蛋白(esp)、明胶酶E(gelE)、聚集物质(asal)、透明质酸(hyl)等[6-7]。
加强养殖场动物肠道共生菌抗生素耐药监测对遏制耐药菌/耐药基因传播具有重要意义。泛欧盟一项长期(2004—2014年)的食品动物中抗生素敏感性监测项目(EASSA),监测从屠宰的健康食品动物(肉鸡、屠宰猪和肉牛)中分离的人畜共患病原菌(沙门菌和弯曲杆菌)和共生菌(大肠杆菌和肠球菌)的抗生素敏感性表明,健康食品动物中肠球菌耐药性风险较低,对医学临床上至关重要的抗生素仍然敏感或者低水平耐药[8]。食品动物与人类有密不可分的联系,耐药细菌可通过人与动物直接接触或食物链完成耐药基因从动物到人类转移,从而对公共卫生产生威胁[9]。目前我国食品动物源肠球菌抗生素耐药性相关研究报道较少,尤其对医学临床重要抗生素的耐药水平、传播扩散风险尚不清楚。广东地区是养殖大省,随着该地养殖产业的发展,滥用抗生素的现象不断出现,有关肠球菌耐药菌株的报道也屡见不鲜,给养殖行业和公共卫生带来潜在风险。本研究采集了广东省养殖场鸡和鸭的肠道样品,以分离率较高且具有重要公共卫生意义的肠球菌(尤其屎肠球菌和粪肠球菌)为对象,分析其耐药现状、耐药基因和毒力基因的流行分布,研究结果为动物源细菌耐药性监控提供数据,也为评估由此带来的食品安全和公共卫生风险提供理论依据。
1 材料与方法 1.1 试验菌株2018年8—10月, 从广东省4个不同养禽场鸡和鸭的肠道样品中分离的125株肠球菌,质控菌ATCC29212保存于华南农业大学药理教研室。
1.2 主要试剂和仪器MH固体培养基、MH液体培养基、BHI固体培养基、BHI液体培养基、叠氮钠-结晶紫-七叶苷固体培养基均购自广东环凯微生物科技有限公司;2×Hieff-PCR Master Mix购自翌圣生物科技(上海)有限公司;微生物质谱鉴定仪MALDI-TOF-MS(基质辅助激光解析电离飞行时间质谱仪)购自岛津公司。
1.3 菌株的分离鉴定2018年8—10月间,采集了广东省4个不同养禽场鸡和鸭的肠道粪便样品493份,样品置于冰盒,于24 h内送至实验室进行肠球菌的分离和鉴定:肠道样品在6.5% NaCl高盐BHI肉汤中,37 ℃过夜增菌,划线接种于叠氮钠-结晶紫-七叶苷琼脂平板。37 ℃培养18~24 h后,观察其菌落形态及培养基颜色,在培养基上大小中等、表面光滑,且能使周围培养基变黑的典型菌落,可初步判断为肠球菌。采用PCR方法对菌株进行分子鉴定:扩增粪肠球菌、屎肠球菌特异性持家基因[10-11]。引物由北京擎科生物科技有限公司合成。采用微生物质谱鉴定仪MALDI-TOF-MS(基质辅助激光解析电离飞行时间质谱仪)对PCR扩增阳性的粪肠球菌和屎肠球菌进行鉴定。
1.4 药物敏感性试验按照2018年美国临床实验室标准委员会(CLSI)推荐的抗菌药物敏感性实验执行标准[12],对分离出的粪肠球菌和屎肠球菌进行氨苄西林、左氧氟沙星、环丙沙星、四环素、多西环素、替加环素、万古霉素、利奈唑胺、利福平、红霉素、氯霉素、氟苯尼考等8大类12种药物的MIC测定。同时由于肠球菌对氨基糖苷类药物低水平固有耐药,但氨基糖苷类药物与青霉素、糖肽类合用对肠球菌有协同治疗作用,在临床治疗上有应用价值[13]。本研究也对分离株进行了高水平氨基糖苷类药物耐药(high-level aminoglycoside resistant,HLAR)、高水平链霉素耐药(high-level streptomycin resistant,HLSR)检测。抗生素敏感性结果依据CLSI的耐药折点来判断。
1.5 耐药基因的检测采用PCR的方法检测噁唑烷酮类(poxtA、optrA和cfr)、四环素类[tet(M)和tet(L)]氯霉素类(fexA、fexB和floR)、大环内酯类(ermB)、喹诺酮类(qnrA、qnrB、qnrS)、糖肽类(vanA和vanB)等抗生素耐药基因,引物设计与退火温度参照文献[14]。扩增后的产物通过凝胶电泳进行检测,阳性产物送北京擎科生物科技有限公司测序,测序结果通过NCBI网站(https://www.ncbi.nlm.nih.gov)进行Blast分析。
1.6 毒力基因的检测本研究对携带重要抗生素耐药基因poxtA、optrA、cfr的肠球菌株,依照相关参考文献[15-16],采用PCR的方法,对ace、cylA、cylB、cylM、efaA、esp、gelE、asal、hyl和agg等10种常见的毒力基因进行筛查。
1.7 统计学分析应用SPSS 22.0软件进行数据分析,组间比较采用χ2检验,P < 0.05为差异有统计学意义,其中0.05≤P≤0.01为差异显著,P≤0.01表示差异极显著。
2 结果 2.1 细菌的分离鉴定从采集的493份肠道样品中,共分离鉴定出125株肠球菌,其中粪肠球菌84株,66株来自鸡肠道样本,18株来自鸭肠道样本;屎肠球菌41株,均来自鸡肠道样本。
2.2 药敏试验结果分析125株粪肠球菌、屎肠球菌的药敏试验结果显示,所有菌株对替加环素均敏感,对万古霉素、氨苄西林也很敏感,而对四环素、多西环素和红霉素几乎全部耐药;对氟苯尼考和氯霉素的耐药率高达89.60%和74.40%。此外,对利奈唑胺、利福平、左氧氟沙星、环丙沙星的耐药率为25%~33%(表 1)。
|
|
表 1 125株肠球菌对抗菌药物的耐药率 Table 1 The antimicrobial resistance rate of 125 Enterococci strains |
2.2.1 不同动物来源耐药性比较 如表 1所示,分离自鸭的肠球菌的耐药情况比鸡肠球菌严重,鸭源分离株对氯霉素和氟苯尼考全耐药。鸭源肠球菌对利奈唑胺的耐药率也高达94%,显著高于鸡源分离株(P < 0.01);对左氧氟沙星、环丙沙星、利福平的耐药率也显著高于(P < 0.05)鸡源分离株。从鸡分离的1株粪肠球菌对万古霉素耐药(GA22)。两种动物来源的肠球菌几乎均对四环素、多西环素、红霉素耐药。
2.2.2 不同亚型肠球菌耐药性比较 屎肠球菌的耐药率普遍高于粪肠球菌。屎肠球菌对AMP、LVX、CIP、HLGR、HLSR的耐药率显著高于粪肠球菌(P < 0.05)。但粪肠球菌对利奈唑胺和利福平的耐药率却极显著的高于(P < 0.01)屎肠球菌。
2.2.3 不同来源、不同亚型肠球菌多重耐药性比较 以对3种或3种以上的抗菌药物种类出现耐药的情况判断为多重耐药,绝大多数菌株(98.40%;123/125)呈现多重耐药表型。来源于鸭的肠球菌的多重耐药情况比鸡的严重,鸭源肠球菌大多都表现为同时耐4种以上抗菌药的多重耐药表型,而且耐7~9种药物的菌株明显多于鸡源肠球菌相比粪肠球菌,屎肠球菌多重耐药现象较为严重,尤其对4种以上药物耐药的菌株比例大于粪肠球菌(图 1)。

|
图 1 不同来源、不同亚型肠球菌多重耐药统计 Fig. 1 Detection of resistant genes in enterococcus strains from different animal sources |
耐药基因检测结果显示,tetL、fexA、ermB最流行,检出率均高于90%。有92株肠球菌检测出optrA耐药基因,检出率为73.60%。tetL的检出率为96.00%,且其中46.67%的菌株同时携带有tetM基因。poxtA和fexB的检出率均低于20%。floR、qnrA、qnrB、qnrS、vanA、vanB等基因均未检出。大多数菌株同时携带多种耐药基因。值得注意的是,有3株鸭源粪肠球菌(JDS2、JDS15、JDS18)中检测出cfr基因。
比较不同动物来源肠球菌中耐药基因的流行分布发现(图 2),来自鸭源肠球菌的耐药基因更复杂,检测到的8种耐药基因的检出率均高于鸡源肠球菌,甚至有3株检测出cfr(16.67%)。
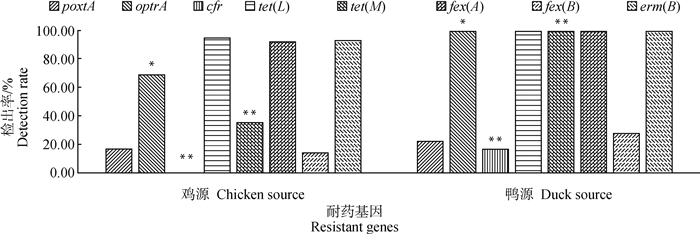
|
*.0.01≤P≤0.05;**.P≤0.01,下同 * Indicates significant difference among detection rates(0.01≤P≤0.05);** Indicates significant extreme difference among detection rates(P≤0.01). The same as below 图 2 不同动物来源肠球菌菌株中耐药基因检出情况 Fig. 2 Detection of resistant genes in enterococcus strains from different animal sources |
比较各耐药基因在不同亚型肠球菌之间的差异特点(图 3),耐药基因optrA的检出率在两种菌种之间的差异不明显(P>0.05);tetL、ermB、fexA在两种菌种中的流行率均为最高,且都呈现出屎肠球菌中的检出率高于粪肠球菌的特点;屎肠球菌中poxtA的检出率明显高于粪肠球菌;多重耐药基因cfr仅在粪肠球菌中被检测到。
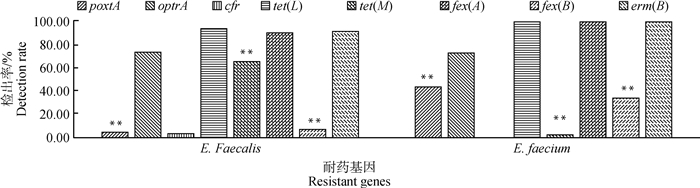
|
图 3 不同亚型肠球菌菌株中耐药基因检出情况 Fig. 3 Detection of resistant genes in subtypes of enterococcus strains |
对92株optrA阳性菌株进行毒力基因筛查,被检的10种毒力基因中检测出7种,其中efaA的检出率最高,达到63.04%(58/92);其他依次为gelE(54.35%,50/92)、aggA(50.00%, 56/92)、ace(47.83%, 44/92)、asa1(44.57%, 41/92)、cylM(4.35%, 4/92)。毒力基esp仅在1株鸭源粪肠球菌中检出。检测的肠球菌往往携带多种毒力基因,42.39%(39/92)菌株携带1~2种毒力基因,23.91%(22/92)的菌株携带3~4种毒力基因,有1株鸭源粪肠球菌(JDS1)同时携带6种毒力基因(cylM、efaA、gelE、esp、ace、asal)。菌株携带的多个毒力基因以efaA-gelE-ace组合最为常见。未检测到cylA、cylB、hyl 3种毒力基因。
不同来源的肠球菌携带的毒力基因也有差异。由图 4可以看出,毒力基因ace在鸭源分离株中的检出率(88.89%)明显高于鸡源分离株(37.84%)(P < 0.01)。鸡源分离株的gelE的检出率(59.46%)显著高于鸭源(33.33%)(P < 0.05)。鸭源分离株中检测到的毒力基因的种类也多于鸡源。鸭源中以5种的毒力基因组合最多,鸡源中携带4种毒力基因的较多。
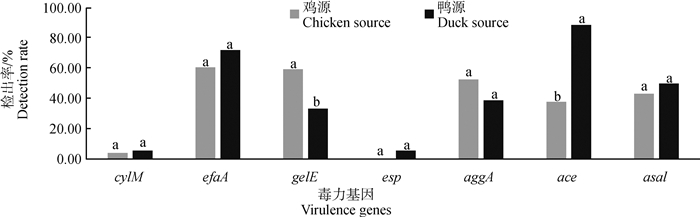
|
同种基因,如两组之间标记字母相同,说明两组之间不存在差异;两组之间标记字母不同,则说明两组之间存在差异 Different letters in the same gene means significant difference between the animal sources, the same letter in the same gene means not significant difference between animal sources. The same as below 图 4 不同动物来源肠球菌菌株中毒力基因检出情况 Fig. 4 Detection of virulence genes in enterococcus strains from different animal sources |
同种基因,不同亚型肠球菌,携带的毒力基因也存在差异。粪肠球菌的毒力基因检出率除cylM外,其余均高于屎肠球菌,且efaA、gelE、ace、asal的检出率显著高于屎肠球菌(图 5)。粪肠球菌携带毒力基因的种类多于屎肠球菌,粪肠球菌携带3~4种毒力基因的最常见,而屎肠球菌中只携带1种毒力基因的最普遍,粪肠球菌中携带至少1种毒力基因的菌株数明显高于屎肠球菌。
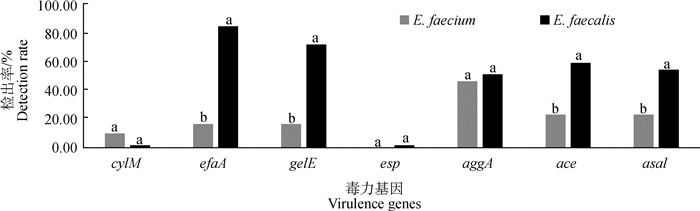
|
图 5 不同亚型肠球菌菌株中毒力基因检出情况 Fig. 5 Detection of virulence genes in subtypes of enterococcus strains |
比较抗菌药物敏感性和毒力基因之间的关系,携带毒力基因数≤3种的肠球菌对CIP及HLAR的耐药率高于携带毒力基因数≥4种的肠球菌见表 2。对CIP及HLAR耐药的菌株大多数携带aggA、ace或asal。对万古霉素耐药的菌株(GA22)仅检出1种毒力基因(gelE),3株携带cfr基因的分离株(JDS2、JDS15、JDS18),有1株不携带毒力基因,其他两株分别携带ace-asal、aggA-ace-asal毒力基因。
|
|
表 2 携带不同毒力基因数目的肠球菌对抗菌药物耐药率 Table 2 Antimicrobial resistance rate of enterococci with different number of virulence genes |
由于临床和畜牧养殖过程中大量使用抗菌药物,使耐药菌和耐药基因普遍存在于人、动物及整个生态环境微生物中。基于“one health”理念下,人、动物和环境形成紧密关联的整体,耐药细菌及耐药基因能在这个整体环境中不断转移、进化和传播。
本研究结果显示,从养禽场分离的肠球菌的耐药水平较高。几乎所有菌株对四环素、红霉素耐药,均高于侯书宝等[17]、Barlow等[18]报道的耐药率。本研究中广东养禽场粪肠球菌的HLAR和HLSR为38.1%和61.9%,而屎肠球菌高达85.3%和82.9%,高于医院临床株[19],也比其他国家食品源肠球菌耐药率高[20-21]。出现这些差异的原因可能是因为不同地区的用药水平不同,在广东养殖场中经常使用该类药物,导致细菌选择压力增大,因此耐药率较高。而本研究鸭源分离株耐药水平高于鸡源,这可能是因为不同动物的养殖环境、用药习惯不同,鸭的饲养环境更为复杂、用药相对较多导致的。与医院临床分离的肠球菌结果相似[19],本研究从养禽场分离的屎肠球菌的耐药率和耐药水平均高于粪肠球菌,而且还从鸡粪便中分离到1株耐万古霉素的粪肠球菌。虽然目前粪肠球菌的分离率高于屎肠球菌,但由于屎肠球菌天然更容易获得耐药基因、表现出高水平耐药等特点,其分离率逐渐增高[22-23],这也与本试验结果一致。动物养殖过程中不合理使用抗菌药物,导致动物源和环境细菌耐药性日趋严重。肠球菌“作为抗生素耐药基因储库”可以将耐药基因转移至其他肠球菌和其他革兰阳性细菌。而食源性耐药肠球菌可能在消费者的肠道中定殖并将耐药基因转移到肠道微生物群中。因此加强鸡场肠球菌耐药性监测对控制耐药性传播、保障公共卫生和人类健康具有重要意义。
万古霉素用于治疗对青霉素和高水平氨基糖苷类耐药的肠球菌所引起的感染,曾被称为“抗感染的最后一道防线”[24]。目前利奈唑胺是临床多重耐药革兰阳性菌,尤其是耐甲氧西林金黄色葡萄球菌(MASA)及耐万古霉素肠球菌(VRE)感染的有效治疗药物[25]。耐利奈唑胺肠球菌(LRE)和VRE是目前最受人们关注的两类耐药肠球菌。利奈唑胺的获得性耐药机制主要由cfr和optrtA、poxtA这几种基因介导。本研究中,73.60%(92/125)的菌株分离到optrA基因,远远高于先前报道中的检出率[26-27],而且鸭源粪肠球菌对利奈唑胺耐药率远高于鸡源粪肠球菌。与之前研究报道相似[27-28],肠球菌菌株携带optrA基因,但对利奈唑胺仍然敏感。目前poxtA基因主要分离自动物源细菌。本研究中17.6%(22/125)菌株检测到poxtA,而且屎肠球菌的分离率(42.5%)明显高于粪肠球菌(4.6%),也高于Lei等[29]2017年分离自食品动物源的检出率。LEADER(The Linezolid Experience and Accurate Determination of Resistance)项目表明,对从2004—2011年医院病人中分离的革兰阳性菌,cfr阳性检出率不足0.03%[30]。但是,我国从养殖场猪分离的葡萄球菌和肠球菌的检出率达到5%[31-32]。本研究从鸭分离的肠球菌中也检出3株cfr阳性菌株(2.4%;3/125)。畜禽专用药物氟苯尼考被证明与在动物中发现耐利奈唑胺肠球菌,有着密切的关系[27, 33],本研究中与利奈唑胺耐药相关的耐药基因检出率较高的原因,可能与饲养过程中氟苯尼考的使用情况有关。因此,在本地区饲养过程选择用药时,应谨慎选择。
肠球菌致病是由多种毒力基因和多重耐药性协同作用的结果[16]。2019年,朱宏等[34]对尿路感染肠球菌进行毒力基因的筛查后发现,粪肠球菌中的毒力基因携带量要显著高于屎肠球菌,这与本研究结果一致。毒力基因esp在肠球菌尿道感染中它能使肠球菌定植在宿主细胞上,并且延长肠球菌细菌在膀胱内停留的时间。有研究认为esp基因更能在临床分离屎肠球菌中检测到,在非临床株以及动物分离株中并未检测到esp基因[35],这也与本试验结果相符。由gelE基因编码的明胶酶是一种含锌的蛋白酶,可以引起细菌感染并在组织和器官间扩散,参与感染和炎症的进程[7]。efaA基因编码一种脂蛋白,在一定条件下帮助肠球菌黏附在宿主非寄居器官,从而导致宿主的感染;Low等[36]研究证明,efaA是粪肠球菌感染性心内膜炎的优势抗原之一。ace(胶原蛋白黏附素)是肠球菌表面的黏附素,在宿主细胞受损伤时,黏附蛋白暴露出来后,会促进细菌黏附到宿主细胞上,有研究证明携带ace基因的肠球菌黏附能力更强,有更强形成生物膜的能力[37]。本研究肠球菌中毒力基因普遍存在,不同动物携带的毒力基因种类存在差异。禽源肠球菌携带的毒力基因主要是efaA、gelE、ace,与猪源肠球菌毒力基因检测结果[6-7]相似,而且鸭源分离株携带的毒力基因的检出率大多高于鸡源菌株。此外,本研究发现在对环丙沙星、高浓度氨基糖苷类耐药的菌株及携带cfr基因的菌株中,大多携带了aggA、asal、ace或gelE基因,推测这几个基因与耐药菌的致病力有关。而在此前祝进[38]、黄雯奕等[7]的研究中,肠球菌对环丙沙星、红霉素、四环素、多西环素、青霉素和氟苯尼考等的耐药率与毒力基因数无相关联。因此,肠球菌的耐药性与携带的毒力基因之间的关系仍需进一步研究确定。
4 结论广东地区禽类养殖场中屎肠球菌耐药率较粪肠球菌严重,而粪肠球菌对环丙沙星和利奈唑胺的耐药率高于屎肠球菌。鸭源粪肠球菌对利奈唑胺耐药率显著高于鸡源。肠球菌中检测出对人医临床重要抗生素耐药基因optrA、poxtA、cfr。对环丙沙星及高浓度氨基糖苷类耐药的菌株,及携带cfr基因的菌株,大多携带agg、asal、gelE或ace。养殖场用药可能筛选出利奈唑胺耐药肠球菌,这些肠球菌可能在人、动物甚至是环境之间进行相互传播,导致optrA、poxtA、cfr等进一步扩散,对公共卫生带来潜在危险,因此需要加强养禽场,尤其养鸭场肠球菌耐药性监测。
| [1] |
BYAPPANAHALLI M N, NEVERS M B, KORAJKIC A, et al. Enterococci in the environment[J]. Microbiol Mol Biol Rev, 2012, 76(4): 685-706. DOI:10.1128/MMBR.00023-12 |
| [2] |
MOHAMED J A, HUANG D B. Biofilm formation by enterococci[J]. J Med Microbiol, 2007, 56(12): 1581-1588. DOI:10.1099/jmm.0.47331-0 |
| [3] |
POH C H, OH H M L, TAN A L. Epidemiology and clinical outcome of enterococcal bacteraemia in an acute care hospital[J]. J Infect, 2006, 52(5): 383-386. DOI:10.1016/j.jinf.2005.07.011 |
| [4] |
MOHAMED J A, TENG F, NALLAPAREDDY S R, et al. Pleiotrophic effects of 2 Enterococcus faecalis sagA: like genes, salA and salB, which encode proteins that are antigenic during human infection, on biofilm formation and binding to collagen type I and fibronectin[J]. J Infect Dis, 2006, 193(2): 231-240. DOI:10.1086/498871 |
| [5] |
LOW D E, KELLER N, BARTH A, et al. Clinical prevalence, antimicrobial susceptibility, and geographic resistance patterns of enterococci: results from the SENTRY Antimicrobial Surveillance Program, 1997-1999[J]. Clin Infect Dis, 2001, 32(S2): S133-S145. DOI:10.1086/320185 |
| [6] |
朱娜, 夏宇飞, 张磊, 等. 湖南健康猪源与流行性腹泻病猪肠球菌毒力基因检测分析[J]. 中国预防兽医学报, 2017, 39(3): 196-200. ZHU N, XIA Y F, ZHANG L, et al. Detection and analysis of Enterococcus virulence gene from health and porcine epidemic diarrhea virus infected of swine in Hunan[J]. Chinese Journal of Preventive Veterinary Medicine, 2017, 39(3): 196-200. (in Chinese) |
| [7] |
黄奕雯, 幸文定, 陶政, 等. 江西省猪源粪肠球菌毒力基因及耐药性分析[J]. 中国兽医学报, 2018, 38(3): 476-480, 514. HUANG Y W, XING W D, TAO Z, et al. Detection and analysis of virulence genes and antibiotic resistence of porcine E. faecalis in Jiangxi[J]. Chinese Journal of Veterinary Science, 2018, 38(3): 476-480, 514. (in Chinese) |
| [8] |
DE JONG A, SIMJEE S, ROSE M, et al. Antimicrobial resistance monitoring in commensal enterococci from healthy cattle, pigs and chickens across Europe during 2004-14 (EASSA Study)[J]. J Antimicrob Chemother, 2019, 74(4): 921-930. DOI:10.1093/jac/dky537 |
| [9] |
YANG B W, QU D, ZHANG X L, et al. Prevalence and characterization of Salmonella serovars in retail meats of marketplace in Shaanxi, China[J]. Int J Food Microbiol, 2010, 141(1-2): 63-72. DOI:10.1016/j.ijfoodmicro.2010.04.015 |
| [10] |
KASIMOGLU-DOGRU A, GENCAY Y E, AYAZ N D. Prevalence and antibiotic resistance profiles of Enterococcus species in chicken at slaughter level; absence of vanA and vanB genes in E. faecalis and E. faecium[J]. Res Vet Sci, 2010, 89(2): 153-158. DOI:10.1016/j.rvsc.2010.02.005 |
| [11] |
CHENG S, MCCLESKEY F K, GRESS M J, et al. A PCR assay for identification of Enterococcus faecium[J]. J Clin Microbiol, 1997, 35(5): 1248-1250. DOI:10.1128/jcm.35.5.1248-1250.1997 |
| [12] |
WEINSTEIN M P, PATEL J B, CAMPEAU S, et al. M 100 Performance standards for antimicrobial susceptibility testing; 28th informational supplement[S]. Wayne, PA, USA: Clinical and Laboratory Standards Institute, 2018.
|
| [13] |
黄华山. 肠球菌对氨基糖苷类的耐药机制研究[D]. 合肥: 安徽医科大学, 2007. HUANG H S. Study on the mechanism of resistance to aminoglycosides in enterococcus[D]. Hefei: Anhui Medical University, 2007. (in Chinese) |
| [14] |
黄奕雯. 粪肠球菌江西分离株对氟喹诺酮类药物的耐药性检测及分析[D]. 南昌: 江西农业大学, 2018. HUANG Y W. Detection and analysis of antimicrobial resistance of fluoroquinolone of porcine E. faecalis in Jiangxi[D]. Nanchang: Jiangxi Agricultural University, 2018. (in Chinese) |
| [15] |
廉鲁昕, 牛沁雅, 曹晨阳, 等. 陕西扶风某生猪养殖场肠球菌分型与毒力基因检测[J]. 西北农林科技大学学报(自然科学版), 2021, 49(4): 10-18. LIAN L X, NIU Q Y, CAO C Y, et al. Subtyping and virulence gene detection of Enterococci isolates derived from a pig farm in Fufeng, Shaanxi[J]. Journal of Northwest A&F University (Natural Science Edition), 2021, 49(4): 10-18. (in Chinese) |
| [16] |
王佳慧, 贾丹, 李贺海, 等. 屎肠球菌携带毒力基因及其耐药性检测[J]. 中国兽医科学, 2020, 50(11): 1428-1432. WANG J H, JIA D, LI H H, et al. Detection of virulence genes and drug resistance of Enterococcus faecium[J]. Chinese Veterinary Science, 2020, 50(11): 1428-1432. (in Chinese) |
| [17] |
候书宝, 武小虎, 张勇, 等. 牛源肠球菌耐药基因与表型关系及16S rDNA同源性分析[J]. 中国兽医学报, 2015, 35(8): 1269-1274. HOU S B, WU X H, ZHANG Y, et al. Phenotype and genotype of bovine Enterococci antimicrobial properties and its 16S rDNA homology[J]. Chinese Journal of Veterinary Science, 2015, 35(8): 1269-1274. (in Chinese) |
| [18] |
BARLOW R S, MCMILLAN K E, DUFFY L L, et al. Antimicrobial resistance status of Enterococcus from Australian cattle populations at slaughter[J]. PLoS One, 2017, 12(5): e0177728. DOI:10.1371/journal.pone.0177728 |
| [19] |
胡付品, 郭燕, 朱德妹, 等. 2019年CHINET三级医院细菌耐药监测[J]. 中国感染与化疗杂志, 2020, 20(3): 233-243. HU F P, GUO Y, ZHU D M, et al. CHINET surveillance of bacterial resistance across tertiary hospitals in 2019[J]. Chinese Journal of Infection and Chemotherapy, 2020, 20(3): 233-243. (in Chinese) |
| [20] |
UNNO T, HAN D, JANG J, et al. Genotypic and phenotypic trends in antibiotic resistant pathogenic Escherichia coli isolated from humans and farm animals in South Korea[J]. Microbes Environ, 2011, 26(3): 198-204. DOI:10.1264/jsme2.ME10194 |
| [21] |
TREMBLAY C L, LETELLIER A, QUESSY S, et al. Multiple-antibiotic resistance of Enterococcus faecalis and Enterococcus faecium from cecal contents in broiler chicken and Turkey flocks slaughtered in Canada and plasmid colocalization of tetO and ermB genes[J]. J Food Protect, 2011, 74(10): 1639-1648. DOI:10.4315/0362-028X.JFP-10-451 |
| [22] |
罗燕萍, 白立彦, 李俊玲, 等. 211株肠球菌属细菌对18种抗生素的耐药性特征[J]. 中华医院感染学杂志, 2004, 14(1): 80-82. LUO Y P, BAI L Y, LI J L, et al. Resistance of 211 isolates of Enterococci to 18 kinds of antibiotics[J]. Chinese Journal of Nosocomiology, 2004, 14(1): 80-82. DOI:10.3321/j.issn:1005-4529.2004.01.026 (in Chinese) |
| [23] |
JOHNSTON L M, JAYKUS L A. Antimicrobial resistance of Enterococcus species isolated from produce[J]. Appl Environ Microbiol, 2004, 70(5): 3133-3137. DOI:10.1128/AEM.70.5.3133-3137.2004 |
| [24] |
董朕, 陈晨, 李冰, 等. 糖肽类抗生素的研究进展[J]. 畜牧兽医学报, 2020, 51(7): 1488-1498. DONG Z, CHEN C, LI B, et al. Research progress of glycopeptide antibiotics[J]. Acta Veterinaria et Zootechnica Sinica, 2020, 51(7): 1488-1498. (in Chinese) |
| [25] |
李培, 林东昉. 肠球菌对利奈唑胺耐药机制研究进展[J]. 中国感染与化疗杂志, 2018, 18(3): 341-344. LI P, LIN D F. Research updates on the mechanisms of linezolid resistance in Enterococcus[J]. Chinese Journal of Infection and Chemotherapy, 2018, 18(3): 341-344. (in Chinese) |
| [26] |
CAI J, WANG Y, SCHWARZ S, et al. Enterococcal isolates carrying the novel oxazolidinone resistance gene optrA from hospitals in Zhejiang, Guangdong, and Henan, China, 2010-2014[J]. Clin Microbiol Infect, 2015, 21(12): 1095. |
| [27] |
WANG Y, LV Y, CAI J C, et al. A novel gene, optrA, that confers transferable resistance to oxazolidinones and phenicols and its presence in Enterococcus faecalis and Enterococcus faecium of human and animal origin[J]. J Antimicrob Chemother, 2015, 70(8): 2182-2190. DOI:10.1093/jac/dkv116 |
| [28] |
LI S M, ZHOU Y F, LI L, et al. Characterization of the multi-drug resistance gene cfr in Methicillin-Resistant Staphylococcus aureus (MRSA) strains isolated from animals and humans in China[J]. Front Microbiol, 2018, 9: 2925. DOI:10.3389/fmicb.2018.02925 |
| [29] |
LEI C W, KANG Z Z, WU S K, et al. Detection of the phenicol-oxazolidinone-tetracycline resistance gene poxtA in Enterococcus faecium and Enterococcus faecalis of food-producing animal origin in China[J]. J Antimicrob Chemother, 2019, 74(8): 2459-2461. DOI:10.1093/jac/dkz198 |
| [30] |
FARRELL D J, MENDES R E, ROSS J E, et al. LEADER program results for 2009:an activity and spectrum analysis of Linezolid using 6, 414 clinical isolates from 56 medical centers in the United States[J]. Antimicrob Agents Chemother, 2011, 55(8): 3684-3690. DOI:10.1128/AAC.01729-10 |
| [31] |
WANG J, LIN D C, GUO X M, et al. Distribution of the multidrug resistance gene cfr in Staphylococcus isolates from pigs, workers, and the environment of a hog market and a slaughterhouse in Guangzhou, China[J]. Foodborne Pathog Dis, 2015, 12(7): 598-605. DOI:10.1089/fpd.2014.1891 |
| [32] |
幸文定. 江西部分地区猪源粪肠球菌耐药性及毒力基因检测与分析[D]. 南昌: 江西农业大学, 2016. XING W D. Detection and analysis of antimicrobial resistance and virulence gene of porcine E. faecalis in Jiangxi[D]. Nanchang: Jiangxi Agricultural University, 2016. (in Chinese) |
| [33] |
魏海涛, 方秋华, 曾振灵, 等. 氟苯尼考药剂学研究进展[J]. 中国兽药杂志, 2010, 44(7): 44-46. WEI H T, FANG Q H, ZENG Z L, et al. Research progress on pharmaceutics of florfenicol[J]. Chinese Journal of Veterinary Drug, 2010, 44(7): 44-46. DOI:10.3969/j.issn.1002-1280.2010.07.014 (in Chinese) |
| [34] |
朱宏, 高硕, 周辉, 等. 尿路感染中肠球菌耐药性及毒力基因分析[J]. 东南国防医药, 2019, 21(5): 466-469. ZHU H, GAO S, ZHOU H, et al. Analysis of drug resistance and virulence genes of enterococci isolated from urinary tract[J]. Military Medical Journal of Southeast China, 2019, 21(5): 466-469. DOI:10.3969/j.issn.1672-271X.2019.05.004 (in Chinese) |
| [35] |
RATHNAYAKE I U, HARGREAVES M, HUYGENS F. Antibiotic resistance and virulence traits in clinical and environmental Enterococcus faecalis and Enterococcus faecium isolates[J]. Syst Appl Microbiol, 2012, 35(5): 326-333. DOI:10.1016/j.syapm.2012.05.004 |
| [36] |
LOW Y L, JAKUBOVICS N S, FLATMAN J C, et al. Manganese-dependent regulation of the endocarditis-associated virulence factor EfaA of Enterococcus faecalis[J]. J Med Microbiol, 2003, 52(2): 113-119. DOI:10.1099/jmm.0.05039-0 |
| [37] |
SOARES R O, FEDI A C, REITER K C, et al. Correlation between biofilm formation and gelE, esp and agg genes in Enterococcus spp. clinical isolates[J]. Virulence, 2014, 5(5): 634-637. DOI:10.4161/viru.28998 |
| [38] |
祝进, 白永凤, 陆军, 等. 粪肠球菌毒力基因及耐药性分析[J]. 放射免疫学杂志, 2012, 25(3): 276-279. ZHU J, BAI Y F, LU J, et al. The incidence of virulence genes and antibiotic resistence in Enterococcus faecalis strains[J]. Journal of Radioimmunology, 2012, 25(3): 276-279. DOI:10.3969/j.issn.1008-9810.2012.03.022 (in Chinese) |
编辑 白永平



