林麝(Moschus berezovskii)又名香獐,其雄麝香腺囊分泌的麝香是中医药的名贵药材,亦是一种高级香料,具有极高的药用价值和经济价值。由于过度猎捕和栖息地的破坏,野生林麝种群数量急剧减少,因而我国将其列为国家一级重点保护动物,《国际濒危动植物贸易公约》(CITES)亦将其列为极危级保护动物[1-2]。为有效保护林麝资源,我国从1958年即开始林麝的人工驯养,但随着生活方式的改变和饲养种群的扩大,麝群中传染性疾病亦频繁发生,给林麝驯养业造成了重大损失,也严重制约了相关产业的发展[3-4]。因此,开展林麝传染性疾病相关的研究,将为我国林麝驯养业的健康发展提供有效保障。
绿脓杆菌(Pseudomonas aeruginosa)又称铜绿假单胞菌,是自然界中广泛存在的一种条件致病菌[5]。当人或动物机体免疫力下降或伤口暴露时,该菌极易感染并导致相应的疾病[6-10]。2019年7月,甘肃省天水市某林麝养殖场出现疫情,数头林麝突发死亡。剖检病死林麝可见肝、肺等脏器肿大、出血,其中肺出血尤为严重,且有大小不等、散在分布的化脓灶。本课题组采集病死林麝肺组织,从中分离到1株绿脓杆菌,并在分离菌株致病性和药物敏感性分析的基础上,完成了分离菌全基因组测序、序列的组装与注释及毒力基因toxA和exoT的遗传进化分析,为林麝绿脓杆菌感染相关疾病的防治提供理论依据,也为绿脓杆菌致病机制和耐药机制的深入研究奠定了基础。
1 材料与方法 1.1 病料来源病料为采自甘肃省天水市某林麝养殖场病死林麝的肺组织。
1.2 主要试剂5%兔鲜血琼脂培养基、普通琼脂培养基由本实验室配制;细菌微量生化反应管、药敏纸片购自杭州滨和微生物试剂有限公司;细菌全基因组提取试剂盒、DNA回收试剂盒购自北京天根生化科技有限公司;pMD19-T载体、T4 DNA连接酶为TaKaRa公司产品;大肠杆菌DH5α感受态细胞购自北京全式金生物有限公司;DNA Marker购自北京康为世纪生物科技有限公司。
1.3 实验动物BALB/c小鼠,5周龄,雌雄各20只,购自中国农业科学院兰州兽医研究所。
1.4 细菌的分离及镜检无菌取相应组织病料划线接种于5%兔鲜血琼脂培养基,37 ℃培养24 h,观察菌落形态特征,挑选可疑菌落涂片染色镜检,并转接种于肉汤培养基中增殖。
1.5 生化试验应用生化微量反应管进行分离菌的葡萄糖、麦芽糖等糖发酵试验,枸橼酸盐利用试验,VP试验,MR试验,硝酸盐还原试验,H2S试验和吲哚试验,即将分离菌株分别接种于上述生化微量反应管中,置37 ℃培养24~48 h后,参照产品说明书判定结果。
1.6 16SrRNA序列测定及分析以提取的分离菌株全基因组为模板,应用16S rRNA基因通用引物对(16S-F:5′-AGAGTTTGATCMTGGCTCAG-3′;16S-R:5′-TACGGYTAC CTTGTTACGACTT-3′),利用PCR扩增分离菌株16S rRNA基因。PCR程序:98 ℃预变性5 min;98 ℃ 1 min,55 ℃ 45 s,72 ℃ 1 min,共30个循环;72 ℃ 10 min。PCR扩增产物与pMD19-T载体连接后转化大肠杆菌DH5α感受态细胞,经PCR鉴定的阳性克隆送北京擎科生物有限公司测序,并将测序结果登陆NCBI,通过BLAST与GenBank中的已知序列进行比对。
1.7 小鼠致病性试验将分离菌划线接种于普通琼脂培养基,置37 ℃恒温培养12 h,挑取单菌落接种于营养肉汤培养基中培养至细菌对数期,并进行菌落计数。将36只BALB/c小鼠随机分为6组,每组雌雄各3只,第1组小鼠腹腔的细菌接种量为2.64×108CFU、第2组小鼠接种量为2.64×107CFU、第3组小鼠接种量为2.64×106CFU、第4组小鼠接种量为2.64×105CFU、第5组小鼠接种量为2.64×104CFU,对照组每只小鼠则分别腹腔注射0.2 mL营养肉汤培养基。每天观察小鼠健康状况,并记录小鼠发病情况及死亡数量,连续7 d,用改良Karber法计算分离菌的半数致死量(LD50)。同时,无菌剖检死亡小鼠并分离鉴定病原菌。
1.8 药敏试验参照葛爱民等[11]所述方法,将分离菌纯培养物涂布于普通琼脂培养基,5 min后将药敏纸片贴于培养基表面,37 ℃恒温培养16~18 h后测量各药敏片的抑菌圈直径并判定结果。
1.9 病原菌全基因组测序及序列分析提取病原菌基因组后送至西安奥纳斯特生物科技有限公司进行全基因组测序。应用Unicycler(0.4.8)软件过滤测序结果并将所得reads进行组装、连接,进而获得分离菌的基因组完成图。利用Prokka、CRISPRfinder、IslandViewer 4等软件对组装后的基因组进行功能元件预测分析,并将预测的基因序列与GO、COG、KEGG、CARD、VFDB等功能数据库通过BLAST比对,从而得到相应基因功能的注释。
1.10 毒力基因的遗传进化分析在全基因组测序并分析的基础上,将TS2019的毒力基因toxA和exoT序列分别登录NCBI,通过BLAST与GenBank中的已知序列比对,选取AE004091.2、LN831024.1、CP065412.1等数十株相似性高的绿脓杆菌相应基因序列,利用DNAstar软件构建系统进化树。
2 结果 2.1 细菌的分离及镜检用接种针蘸取肺组织病料,划线接种于5%兔鲜血琼脂培养基,37 ℃培养18~24 h,可见表面光滑、湿润、边缘整齐的淡黄绿色菌落生长,且呈β溶血,并伴有特殊芳香气味。挑取单菌落接种于营养肉汤培养基中,37 ℃ 200 r·min-1增菌培养12 h后可见培养基呈均匀浑浊。菌液涂片,革兰染色、镜检见单个或成对排列的革兰阴性短杆菌。
2.2 生化试验利用微量生化反应管进行分离菌的生化试验。结果显示(结果未展示,备索):分离菌葡萄糖发酵试验、枸橼酸盐利用试验和硝酸盐还原试验为阳性,而麦芽糖和乳糖发酵试验、VP试验、MR试验、H2S试验及吲哚试验结果均为阴性。试验结果均与绿脓杆菌一致,因此,初步判定分离菌为绿脓杆菌。
2.3 16SrRNA基因序列分析利用16S rRNA通用引物扩增分离菌16S rRNA基因,结果显示PCR产物约为1 500 bp。测序结果表明,分离菌株16S rRNA基因序列与绿脓杆菌PA01(登录号:AE004091.2)的16S rRNA基因序列相似性为99.5%,且与GenBank中其他绿脓杆菌分离株16S rRNA基因均具有极高的相似性。因此,确定该分离菌株为绿脓杆菌,并将其命名为TS2019。
2.4 小鼠致病性试验利用小鼠进行分离菌致病性分析,试验结果表明,在攻毒12 h后,2.64×108CFU和2.64×107CFU接种组小鼠全部死亡,2.64×106CFU接种组的小鼠死亡1只;其余接种组小鼠均出现不同程度症状,但至7 d未见死亡;对照组小鼠无任何异常表现。所有死亡小鼠剖检见肺轻微肿大并明显出血,并从肺中检出TS2019菌(见图 1)。根据公式LD50=lg-1[Xm-i (∑P-0.5)],计算得到TS2019对小鼠的半数致死量LD50为2.82×107CFU·mL-1。

|
A. 正常小鼠肺形态及组织抹片镜检(1 000×); B. 感染组小鼠肺形态及组织抹片镜检(1 000×) A. Lung of uninfected mice and corresponding microscopical characteristic(1 000×); B. Lung of infected mice and corresponding microscopical characteristic(1 000×) 图 1 攻毒小鼠肺剖检病变及组织抹片革兰染色镜检结果Fig. 1 Autopsy symptoms of infected mice lung and microscopical characteristic of isolated strain |
利用K-B法进行分离菌的药敏试验,结果显示TS2019对环丙沙星、洛美沙星等4种药物高度敏感,对头孢曲松、大观霉素等5种药物敏感性次之,而对新霉素、强力霉素等7种药物存在耐药性(表 1)。
|
|
表 1 药敏试验结果 Table 1 Antibiotic sensitivity test |
全基因组测序获得TS2019菌株全基因组大小为6 308 327 bp,其中G+C含量占66.51%,共编码5 929个基因,含tRNA 73个、rRNA 12个、tmRNA 1个。全基因组中共预测到5条CRISPRs结构,6个基因岛。
2.7 TS2019基因功能分析2.7.1 GO注释分析 TS2019全基因组经GO数据库注释分析,共有3 647个基因得到注释,占总基因的61.5%。其中生物学过程注释条目约占总功能注释的33.1%,主要与代谢过程(metabotic process)、转录调控(regulation of transcription)、跨膜转运(transmembrane transport)等有关;细胞组分注释条目约占总功能条目的23.2%,其中主要为细胞质(cytoplasm)和质膜(plasma membrane)等;而分子功能注释约占总注释条目的43.7%,主要被注释到转运活性(transporter activity)和结合(binding)单元。具体结果见图 2A(每个分类选取前20个注释最多的条目)。

|
A. GO功能分类; B. COG功能分类; C. KEGG功能分类 A. Functional classification of GO annotation; B. Functional classification of COG annotation; C. Functional classification of KEGG annotation 图 2 TS2019菌株的GO、COG、KEGG功能分类 Fig. 2 GO, COG, KEGG functional classification of TS2019 strain |
2.7.2 COG功能统计分析 通过与COG数据库比对,发现TS2019中共有2 352个基因得到功能注释,约占总基因数的39.7%。其中预测蛋白功能注释主要集中在氨基酸转运和代谢过程(291个蛋白质)、能量生产与转化(211个蛋白质)、核苷酸转运和代谢(61个蛋白质)等分类单元。具体COG功能注释分类见图 2B。
2.7.3 KEGG功能统计分析 经KEGG数据库注释,TS2019中共有2 021个基因参与相应的代谢通路,约占总基因数的34.1%。其中参与新陈代谢途径的蛋白质最多,共有1 035个,约占注释蛋白总数的51.2%。另外,有500个蛋白质参与环境信息处理,约占注释蛋白总数的24.7%。具体注释分类可见图 2C。
2.7.4 耐药基因注释分析 经CARD数据库比对注释,发现TS2019共有5 288个ORF分别为四环素类、氨基糖苷类、氟喹诺酮类等药物的耐药相关基因。其中多数耐药相关基因为抗菌药物的特有外排泵和抗生素靶位点修饰酶或水解酶。具体结果见表 2。
|
|
表 2 TS2019菌株主要耐药基因类型 Table 2 The main types of resistance genes in TS2019 strain |
2.7.5 毒力因子注释分析 经VFDB数据库比对注释,得到TS2019全基因组中共有875个序列可编码与TS2019致病性相关的基因。且这些基因产物多具有黏附、调控或铁摄取等功能,其中部分基因产物存在功能交叉。根据功能不同可将毒力因子进行分类,具体结果见表 3。
|
|
表 3 TS2019菌株主要毒力因子类型 Table 3 The main types of virulence factors in TS2019 strain |
TS2019毒力基因toxA和exoT的序列分析结果表明,toxA基因和exoT基因序列与GenBank中大多数绿脓杆菌分离株的相应基因序列相似性均高于99%。toxA基因进化树表明,TS2019与绿脓杆菌NCTC10332株(登录号:LN831024.1)位于同一分支上,其亲缘关系最近(图 3A)。而exoT基因进化树表明,TS2019与中国杭州人源分离株P33(登录号:CP065412.1)亲缘关系较近,并与中国苏州人源分离株SE5416(登录号:CP046404.1)、美国人源分离株PABL077(登录号:MK509304.1)位于同一大分支(图 3B)。推测TS2019分离株可能来源于人,并通过人与林麝的接触而导致林麝的感染。
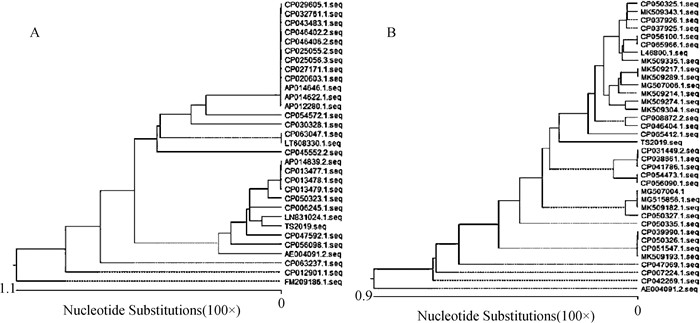
|
A. toxA基因; B. exoT基因 A. toxA gene; B. exoT gene 图 3 toxA和exoT基因的系统发育树 Fig. 3 Phylogenetic tree of toxA and exoT genes |
麝香是我国传统名贵中药材,其具有抗炎、抑菌、抗肿瘤等诸多功效,药用和经济价值极高[12-13]。但随着野生麝类资源的锐减,我国将所有麝属动物列为一级重点保护野生动物,并全面禁止野生麝类动物猎捕和取香。因此,麝属动物的人工繁育及活体取香便成为获取天然麝香的唯一途径。林麝是我国人工驯养种群数量最多的麝属动物,但随着生境的变化和种群密度的增加,传染性疾病逐渐成为影响林麝种群稳定增长的主要因素[14-15]。因此,开展林麝传染病及其防控相关的研究,将为林麝资源的保护和合理利用提供理论支持和技术支撑。
绿脓杆菌的致病性是由众多胞内及胞外毒力因子协同作用所形成,鞭毛、菌毛可通过发挥运动、黏附功能来参与细菌定植过程;铁摄取系统可通过各种策略获取铁元素以满足细菌生长需求[16];Ⅲ型分泌系统可直接使效应蛋白exoY、exoS、exoU、exoT进入宿主细胞内,从而破坏宿主细胞防御系统和信号转导系统[17];而弹性蛋白酶和碱性蛋白酶协同作用可使绿脓杆菌逃逸宿主的免疫识别过程[18]。本研究毒力基因注释结果显示,TS2019菌株Ⅲ型分泌系统所携带的效应蛋白为exoY、exoS、exoT,而exoU缺失。其可能是因exoU基因和exoS基因在基因组中的位点存在一定的拮抗作用而导致通常只能携带二者之一[19]。将TS2019与20株水貂源绿脓杆菌分离株对比,发现所有菌株携带的毒力基因(如exoS、exoT、aprA、phzM、plcH等)均有一定差异[20]。由于本研究分离菌株样本量不足,因此未能发现不同动物源性绿脓杆菌的毒力因子携带类型与宿主的直接关联性。
外毒素A为绿脓杆菌最主要的毒力因子之一,由绿脓杆菌II型分泌系统所分泌,可通过阻断宿主细胞蛋白质的合成导致细胞坏死或凋亡[21-22]。exoT作为一种基础的T3SS效应蛋白,几乎在所有的绿脓杆菌临床分离株中都表达,并作为一种通用且有效的细胞毒素可诱导多种形式的宿主细胞凋亡[23-24]。基于此,本研究对toxA和exoT基因分别进行了遗传进化分析。结果表明,toxA和exoT分别与GenBank中绿脓杆菌相应基因序列具有高度相似性,但TS2019的exoT基因处于独立分支,这可能与该基因碱基突变、缺失等所造成的遗传距离变化有关。另外,发现TS2019的toxA基因在第426、553位氨基酸位点均发生突变。有研究表明,外毒素A的关键氨基酸位点改变将导致其毒力下降[25-26],这与TS2019相比国内部分绿脓杆菌分离株毒力较弱的结果相符合[7, 27]。但两者之间是否存在必然联系,需进一步研究。
药敏试验结果表明TS2019对强力霉素、四环素等药物存在显著耐药性。而葛爱民等[11]的研究结果显示,大多数绿脓杆菌对强力霉素敏感。其可能是由于不同地域使用不同抗菌药物而导致细菌耐药谱产生了变化。耐药基因注释结果显示,TS2019可编码氨基糖苷类、氟喹诺酮类等药物的耐药基因,但试验结果显示分离菌对庆大霉素、左氧氟沙星等药物敏感。此类差异可能与细菌生物被膜的生理异质性有关,即氧气和各种营养物质浓度发生变化使生物被膜内不同区域菌细胞的基因表达、代谢活性出现差异,最终导致基因型和表型之间出现不一致[28]。
本研究中,预测基因的COG功能注释结果表明,TS2019中有大量基因的编码产物可参与氨基酸转运和代谢、能量生产与转化等生物学过程,这将提供充足的能源物质来保证细菌的增殖、生长。另外,KEGG功能注释结果显示,TS2019中有超过50%的预测蛋白参与新陈代谢途径。这将极有助于细菌建立强大的代谢调控系统来适应不同的生存环境,以便在感染过程中形成绝对优势[29]。
4 结论从病死林麝肺组织中分离到1株绿脓杆菌TS2019,该菌有较强的致病性,且对多种抗生素耐药;全基因组序列显示TS2019携带耐药相关基因5 288个、毒力基因875个,其中毒力基因toxA、exoT与GenBank中所有绿脓杆菌toxA、exoT基因序列具有高度相似性。本研究结果为林麝绿脓杆菌感染相关疾病的防治提供了理论支持,也为绿脓杆菌致病机制和耐药机制的深入研究奠定了基础。
| [1] |
LI Y M, SHI M H, ZHANG T X, et al. Dynamic changes in intestinal microbiota in young forest musk deer during weaning[J]. PeerJ, 2020, 8: e8923. DOI:10.7717/peerj.8923 |
| [2] |
QI W H, LU T, ZHENG C L, et al. Distribution patterns of microsatellites and development of its marker in different genomic regions of forest musk deer genome based on high throughput sequencing[J]. Aging (Albany NY), 2020, 12(5): 4445-4462. |
| [3] |
ZHAO K L, MA J N, WANG X R, et al. Population divergence of Pseudomonas aeruginosa can lead to the coexistence with Escherichia coli in animal suppurative lesions[J]. Vet Microbiol, 2019, 231: 169-176. DOI:10.1016/j.vetmic.2019.03.014 |
| [4] |
袁阳, 李静, 张爱雪, 等. 林麝化脓病病原菌化脓隐秘杆菌与铜绿假单胞菌互作机制研究[J]. 四川动物, 2020, 39(3): 241-248. YUAN Y, LI J, ZHANG A X, et al. Interspecific interaction between Pseudomonas aeruginosa and Trueperella pyogenes from the abscesses disease of Moschus berezovskii[J]. Sichuan Journal of Zoology, 2020, 39(3): 241-248. (in Chinese) |
| [5] |
张传美, 王颖, 秦晓冰, 等. 貂源绿脓杆菌流行株toxA基因克隆序列分析及原核表达[J]. 中国兽医杂志, 2015, 51(10): 38-41. ZHANG C M, WANG Y, QIN X B, et al. Cloning, sequence analysis and Prokaryotic expression of the toxA gene of Pseudomonas aeruginosa from minks[J]. Chinese Journal of Veterinary Medicine, 2015, 51(10): 38-41. DOI:10.3969/j.issn.0529-6005.2015.10.011 (in Chinese) |
| [6] |
KHALIFA M M, ELKHAWAGA A A, HASSAN M A, et al. Highly specific electrochemical sensing of Pseudomonas aeruginosa in patients suffering from corneal ulcers: a comparative study[J]. Sci Rep, 2019, 9(1): 18320. DOI:10.1038/s41598-019-54667-0 |
| [7] |
QIAN Z, HUI P, HAN L, et al. Serotypes and virulence genes of Pseudomonas aeruginosa isolated from mink and its pathogenicity in mink[J]. Microb Pathog, 2020, 139: 103904. DOI:10.1016/j.micpath.2019.103904 |
| [8] |
李珮瑶, 高晶萍, 田勇, 等. 鸽源绿脓杆菌的分离鉴定及致病性试验[J]. 江苏农业科学, 2020, 48(8): 189-191. LI P Y, GAO J P, TIAN Y, et al. Isolation, identification and pathogenicity test of Pseudomonas aeruginosa from pigeon[J]. Jiangsu Agricultural Sciences, 2020, 48(8): 189-191. (in Chinese) |
| [9] |
SLIGL W I, DRAGAN T, SMITH S W. Nosocomial Gram-negative bacteremia in intensive care: epidemiology, antimicrobial susceptibilities, and outcomes[J]. Int J Infect Dis, 2015, 37: 129-134. DOI:10.1016/j.ijid.2015.06.024 |
| [10] |
TUMBARELLO M, DE PASCALE G, TRECARICHI E M, et al. Clinical outcomes of Pseudomonas aeruginosa pneumonia in intensive care unit patients[J]. Intens Care Med, 2013, 39(4): 682-692. DOI:10.1007/s00134-013-2828-9 |
| [11] |
葛爱民, 姜八一, 崔晓娜, 等. 威海地区水貂绿脓杆菌的血清型鉴定及药敏试验[J]. 江苏农业科学, 2015, 43(6): 202-203. GE A M, JIANG B Y, CUI X N, et al. Serotype identification and drug sensitivity test of Pseudomonas aeruginosa from minks in Weihai area[J]. Jiangsu Agricultural Sciences, 2015, 43(6): 202-203. (in Chinese) |
| [12] |
吴家炎, 王伟. 中国麝类[M]. 北京: 中国林业出版社, 2006: 179-185. WU J Y, WANG W. The Musk Deer of China[M]. Beijing: China Forestry Publishing House, 2006: 179-185. (in Chinese) |
| [13] |
王玉玲, 哈成勇. 林麝的人工繁殖新技术及麝香研究进展[J]. 中国中药杂志, 2018, 43(19): 3806-3810. WANG Y L, HA C Y. Research progress on musk and artificial propagation technique of forest musk deer[J]. China J Chin Mater Med, 2018, 43(19): 3806-3810. (in Chinese) |
| [14] |
赵位. 2株林麝源肺炎克雷伯氏菌的全基因组序列分析和新基因kp05372的原核表达[D]. 雅安: 四川农业大学, 2018. ZHAO W. Whole genome analysis of two Klebsiella pneumoniae strains isolated from Moschus berezovskii and prokaryotic expression of novel gene kp05372[D]. Ya'an: Sichuan Agricultural University, 2018. (in Chinese) |
| [15] |
曾红梅, 赵位, 王翔, 等. 一株林麝肺源创口博德特氏杆菌全基因组及细胞致死性肿胀毒素分析[J/OL]. 微生物学报, 2020: 1-14. (2020-10-22)[2021-03-31]. https://doi.org/10.13343/j.cnki.wsxb.20200482. ZENG H M, ZHAO W, WANG X, et al. Whole genome sequence and Cytolethal distending toxin analysis of a Bordetella trematum from the lung of a forest musk deer[J/OL]. Acta Microbiologica Sinica, 2020: 1-14. (2020-10-22)[2021-03-31]. https://doi.org/10.13343/j.cnki.wsxb.20200482. (in Chinese) |
| [16] |
于珊, 马旅雁. 铜绿假单胞菌铁摄取与生物被膜形成研究进展[J]. 生物工程学报, 2017, 33(9): 1489-1512. YU S, MA L Y. Iron uptake and biofilm formation in Pseudomonas aeruginosa[J]. Chin J Biotechnol, 2017, 33(9): 1489-1512. (in Chinese) |
| [17] |
AZIMI S, KAFIL H S, BAGHI H B, et al. Presence of exoY, exoS, exoU and exoT genes, antibiotic resistance and biofilm production among Pseudomonas aeruginosa isolates in Northwest Iran[J]. GMS Hyg Infect Control, 2016, 11: Doc04. |
| [18] |
CASILAG F, LORENZ A, KRUEGER J, et al. The LasB Elastase of Pseudomonas aeruginosa acts in concert with alkaline protease AprA to prevent flagellin-mediated immune recognition[J]. Infect Immun, 2016, 84(1): 162-171. DOI:10.1128/IAI.00939-15 |
| [19] |
LIEW S M, RAJASEKARAM G, PUTHUCHEARY S A, et al. Antimicrobial susceptibility and virulence genes of clinical and environmental isolates of Pseudomonas aeruginosa[J]. PeerJ, 2019, 7: e6217. DOI:10.7717/peerj.6217 |
| [20] |
彭辉. 貂源绿脓杆菌血清型和毒力基因的检测及其致病性研究[D]. 泰安: 山东农业大学, 2018. PENG H. Identification of serotypes and distribution of virulence genes in Pseudomonas aeruginosa isolated from mink and its pathogenesis in mink[D]. Taian: Shandong Agricultural University, 2018. (in Chinese) |
| [21] |
MICHALSKA M, WOLF P. Pseudomonas Exotoxin A: optimized by evolution for effective killing[J]. Front Microbiol, 2015, 6: 963. DOI:10.3389/fmicb.2015.00963/pdf |
| [22] |
TEHRANIZADEH Z A, SANKIAN M, BAZZAZ B S F, et al. The immunotoxin activity of exotoxin A is sensitive to domain modifications[J]. Int J Biol Macromol, 2019, 134: 1120-1131. DOI:10.1016/j.ijbiomac.2019.05.137 |
| [23] |
ZOU M X, WANG H C, SHUI J, et al. Characterization of clinical extensively drug resistant Pseudomonas aeruginosa from a Chinese teaching hospital[J]. J Infect Dev Ctries, 2018, 12(10): 835-841. DOI:10.3855/jidc.10743 |
| [24] |
WOOD S J, GOLDUFSKY J W, BELLO D, et al. Pseudomonas aeruginosa ExoT induces mitochondrial apoptosis in target host cells in a manner that depends on its GTPase-activating Protein (GAP) domain activity[J]. J Biol Chem, 2015, 290(48): 29063-29073. DOI:10.1074/jbc.M115.689950 |
| [25] |
WICK M J, COOK J M, IGLEWSKI B H. Structure-function analysis of exotoxin A proteins with mutations at histidine 426[J]. Infect Immun, 1992, 60(3): 1128-1139. DOI:10.1128/iai.60.3.1128-1139.1992 |
| [26] |
DOUGLAS C M, COLLIER R J. Pseudomonas aeruginosa exotoxin A: alterations of biological and biochemical properties resulting from mutation of glutamic acid 553 to aspartic acid[J]. Biochemistry, 1990, 29(21): 5043-5049. DOI:10.1021/bi00473a007 |
| [27] |
伍茜, 杨威, 赵位, 等. 林麝肺源ST1249型铜绿假单胞菌的分离鉴定及其全基因组序列分析[J]. 西北农林科技大学学报: 自然科学版, 2020, 48(10): 9-17. WU Q, YANG W, ZHAO W, et al. Isolation, identification and whole genome sequence analysis of Pseudomonas aeruginosa (ST1249) from lung of forest musk deer[J]. Journal of Northwest A&F University: Natural Science Edition, 2020, 48(10): 9-17. (in Chinese) |
| [28] |
邱潇. 生物膜群感效应调控对细菌耐药性转移和毒力因子表达影响机制研究[D]. 济南: 山东大学, 2020. QIU X. The mechanism of biofilm quorum sensing regulation on the antibiotic resistance genes transfer and virulence factors expression[D]. Ji'nan: Shandong University, 2020. (in Chinese) |
| [29] |
BOOPATHI S, VASHISTH R, MANOHARAN P, et al. Stigmatellin Y-an anti-biofilm compound from Bacillus subtilis BR4 possibly interferes in PQS-PqsR mediated quorum sensing system in Pseudomonas aeruginosa[J]. Bioorg Med Chem Lett, 2017, 27(10): 2113-2118. DOI:10.1016/j.bmcl.2017.03.074 |
(编辑 白永平)



