2. 广西大学动物科技学院 亚热带农业生物资源保护与利用国家重点实验室, 南宁 530004;
3. 成都中科奥格生物科技有限公司, 成都 610000
2. State Key Laboratory for Conservation and Utilization of Subtropical Agro-Bioresources, College of Animal Science and Technology, Guangxi University, Nanning 530004, China;
3. Chengdu Clonorgan Biotechnology Co. Ltd., Chengdu 610000, China
猪肉是人类膳食中蛋白质的主要来源,在我国国民经济中占有重要地位。随着人们膳食水平的提高,消费者越来越追求食用瘦肉。为提高中国地方猪种的瘦肉含量,利用转基因技术制备瘦肉型猪是遗传育种领域的研究热点。巴马香猪是我国主要的地方猪品种,被广泛用作动物模型以研究哺乳动物的基因功能。锌指蛋白6(ZBED6)是一种转录因子,能够通过与IGF2基因相互作用,从而调节肌肉的生长发育。虽然目前已有少量的基因修饰动物模型研究ZBED6-IGF2轴对肌肉发育的影响,但ZBED6在猪模型中的功能作用尚未被完全探索。本文以ZBED6基因敲除巴马香猪为试验模型,在体内水平研究ZBED6对脾组织的作用功能,进一步探究转录因子调控生长发育等相关的生物学功能的分子机制,从而为研究ZBED6基因的更多功能提供理论基础,同时,为我国瘦肉型家猪的育种改良提供思路。ZBED6是一个转录因子,位于ZC3H11A的第一内含子中,是所有胎盘哺乳动物所特有的。研究发现,猪胰岛素样生长因子2(IGF2)内含子3的G突变为A,使得ZBED6无法与之结合,从而增加肌肉重量和心的大小,同时减少脂肪沉积[1-3]。随后的研究发现,ZBED6为IGF2的转录抑制因子,可影响胎盘哺乳动物的细胞增殖、发育和生长[4]。最近的一项研究表明,ZBED6基因敲除和IGF2敲入导致IGF2的表达上调,促进小鼠骨骼肌和内脏的生长[5]。在中国巴马香猪中,IGF2敲入使得其产肉量有所提高[6]。目前,ZBED6在中国本土家猪中的研究比较匮乏,其对巴马香猪脾发育的影响和分子机制尚未明确。本研究以ZBED6基因敲除巴马香猪为模型载体,对ZBED6基因敲除巴马香猪和野生型巴马香猪的脾组织进行表型鉴定以及转录组学分析,以揭示ZBED6在脾发育中的影响和调控作用,为进一步探究ZBED6的分子机制提供更多的理论依据。
1 材料与方法 1.1 试验动物ZBED6基因敲除巴马香猪(ZBED6-KO)和同日龄野生型巴马香猪(WT)来自于中国农业科学院北京畜牧兽医研究所大兴猪场实验基地,ZBED6基因的敲除由成都中科奥格生物科技有限公司利用CRISPR/Cas9技术完成。
1.2 样品采集及表型鉴定对3头ZBED6基因敲除巴马香猪(ZBED6-KO)(spleen1、spleen3、spleen6)和3头同日龄野生型巴马香猪(WT)(spleen2、spleen4、spleen5)进行屠宰,称取组织重量,同时采集脾组织样品,置于RNA-later中,用于RNA的提取。
1.3 RT-qPCR分析IGF2在脾组织中的表达量1.3.1 脾组织总RNA提取与反转录 使用Qiagen公司试剂盒进行脾组织RNA的提取,具体操作步骤参照试剂盒使用说明书。每个样本取2~3 mg的组织进行裂解匀浆,60 μL的洗脱液洗脱RNA。使用NanoDrop1000检测RNA浓度及质量,OD260 nm/OD280 nm在1.9~2.1之间,RNA浓度>100 ng·μL-1,可用于后续分析。
1.3.2 定量引物的设计合成 NCBI在线网站下载猪IGF2基因的转录本序列,以β-actin为内参基因,利用Primer3.0在线网站设计引物(表 1)。引物由北京天一辉远生物科技有限公司合成。
|
|
表 1 本试验中使用的RT-qPCR引物序列 Table 1 RT-qPCR primer sequence used in this experiment |
1.3.3 RT-qPCR检测 用TaKaRa公司的SYBR Green Master Mix试剂,以cDNA为模板。在ABI Q7实时荧光定量仪(Applied Biosystems, Forest City, CA, SA)上进行基因定量分析,每个样品设置3个生物学重复。使用2-ΔΔct方法计算相对表达量,使用t检验进行分析,若P < 0.05,则认为差异显著。
1.4 RNA-Seq1.4.1 总RNA提取 RNA提取及反转录方法同“1.3.1”,使用Agilent 2100 bioanalyzer检测RNA的总量、浓度和完整度(RIN值),RNA浓度> 100 ng·μL-1,总量≥2.0 μg,且RIN值>7.0,则可以用于后续转录组分析。
1.4.2 RNA-seq文库构建及测序 使用Illumina TruSeq RNA样品制备试剂盒,按照说明书要求对RNA-seq测序所需的cDNA文库进行构建。用TruSeq PE Cluster Kit v3-cBot-HS试剂盒,严格按照试剂盒说明书要求,用cBot Cluster Generation系统对含有索引接头的样品进行聚类,不同文库按照目标下机数量进行pooling,用Illumina Hiseq 2500测序平台进行测序,测序读长为pair-end (PE) 125 bp。
1.5 测序数据分析1.5.1 测序数据及原始数据的质量控制 利用NGSQC Toolkit v2.3.3软件对下机后的原始读数(reads)进行过滤,去除含有索引接头的reads,去除低质量的reads(包括去除N的比例大于10%的reads;去除质量值Q≤10的碱基数占整条read的50%以上的reads),以及过滤后长度小于25 bp的reads。经过上述质量控制后得到高质量的Clean Data。
1.5.2 测序数据的比对与拼接 从Ensemble数据库下载猪11.1版本参考基因组以及基因组注释文件进行数据分析,利用Hisat2[7]的默认参数将clean reads与参考基因组进行序列比对,获取在参考基因组或基因上的位置消息,以及测序样品特有的序列特征信息。用StringTie[8]软件进行转录本的组装,通过Mapped Reads在基因上的位置信息,对转录本和基因的表达水平进行定量,滤掉样本少于一个转录本表达量的数据。继续使用StringTie软件将所有样本的转录本及参考基因组组装成新的注释文件,用于后续分析。
1.5.3 基因差异表达分析及层次聚类分析 用R语言的Ballgown包(v2.14.1)对两组转录本数据进行差异基因表达分析。用FDR法进行多重校正,同时满足FDR < 0.05并且|Fold change|>1.5的基因作为两组之间的显著差异表达基因。
用R语言中pheatmap包的聚类分析函数对ZBED6基因敲除猪(ZBED6-KO)及野生(WT)猪脾组织的显著差异表达基因进行层次聚类分析[9]。
1.5.4 差异表达基因的GO和KEGG富集分析 使用DAVID(v6.8)在线分析软件对脾组织的差异表达基因进行GO和KEGG富集分析[10],按照富集倍数(Fold change)从小到大的原则,使用P值小于0.05作为显著富集基因的阈值。
1.6 RT-qPCR验证差异表达基因随机选取7个差异表达基因,采用RT-qPCR方法验证基因表达水平及测序结果的准确性,验证方法同“1.3”。
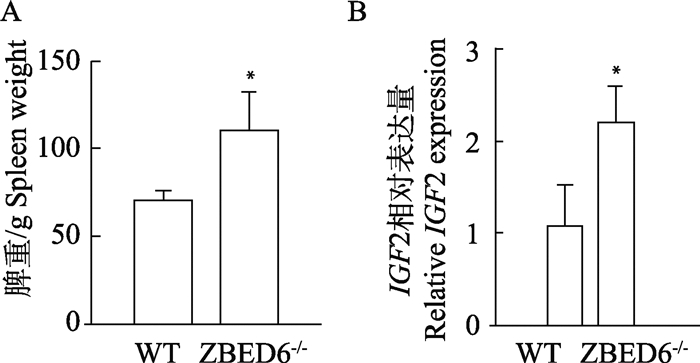
2 结果 2.1 表型分析结果ZBED6-KO猪的脾组织重量显著高于WT猪(P < 0.05,图 1A),推测ZBED6的敲除对巴马香猪脾组织的生长发育有一定的促进作用。
|
*P < 0.05 图 1 脾组织重量鉴定结果(A)和IGF2基因在脾中的相对表达量(B) Fig. 1 Results of identification of spleen tissue weight (A) and the relative expression of IGF2 in spleen (B) |
IGF2基因在ZBED6-KO猪和WT猪脾组织中的基因相对表达量如图 1B所示,结果显示,IGF2在ZBED6-KO猪中的表达量显著高于WT猪(P < 0.05)。
2.3 测序数据质量评估每个样本测序获得至少4G的数据量,Clean data在去除比对上核糖体数据库的数据后,共得到298 929 423个clean reads,单个文库的总reads在30 134 829~62 565 114个之间。各样本clean ratio及Q30 data均在90%以上,测序数据符合后续生物信息分析要求(表 2)。
|
|
表 2 转录组测序数据质量检测分析 Table 2 The quality analysis of sequencing data of transcriptome |
通过Hisat比对,对所得reads在猪参考基因组及基因上的匹配情况进行了统计(表 3)。测序得到的clean data中,比对率均在83.94%以上,可以进行下一步分析。
|
|
表 3 转录组测序基因组比对分析 Table 3 The genome mapping analysis of transcriptome sequencing |
以FDR < 0.05,Fold Change>2的标准进行筛选,ZBED6-KO猪与WT猪之间共得到161个差异表达基因,其中上调的基因90个,下调的基因71个。用R语言的ggplot2软件包对差异基因进行可视化,结果如图 2所示。

|
图 2 差异表达基因数统计 Fig. 2 The number statistics of differentially expressed genes |
ZBED6-KO猪(spleen1、spleen3、spleen6)和WT猪(spleen2、spleen4、spleen5)脾组织之间的差异基因进行聚类分析,结果显示(图 3),spleen1和spleen6聚类在一起且基因的上下调趋势一致,spleen4先与spleen2聚类在一起,再与spleen5聚类在一起,且spleen4、spleen2和spleen5各个基因的上下调趋势相一致。spleen3与WT猪的3个样本聚类在一起,有可能是由于spleen3样品的个体差异,总体上,ZBED6-KO猪(spleen1、spleen3、spleen6)的3个个体表达模式相似,ZBED6-WT猪(spleen2、spleen4、spleen5)的3个个体表达模更相似,证明实验样品和RNA-seq数据可靠。

|
图 3 161个差异表达基因的聚类结果 Fig. 3 The clustering of 161 differentially expressed genes |
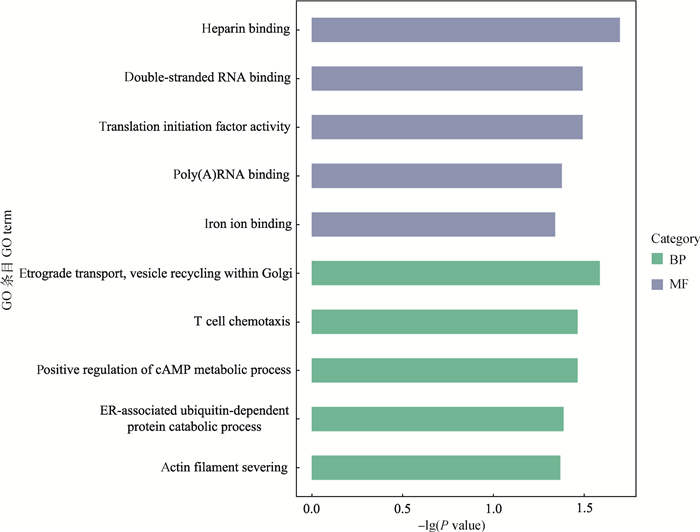
利用在线富集分析软件DAVID对获得的差异表达基因进行GO功能注释,共富集到10条显著的条目,其中包括5个生物学过程条目和5个分子功能条目(表 4)。用R语言的ggplot2软件包对GO功能注释结果进行可视化(图 4),筛选到一条与肌肉发育相关的条目(actin filament severing)。同时,对差异基因进行KEGG富集分析,富集到5条显著相关的信号通路(表 5),同样用R语言程序包进行可视化(图 5),筛选到一条与肌肉发育相关的信号通路(regulation of actin cytoskeleton)。
|
|
表 4 差异基因的GO分析 Table 4 GO analysis of differentially expressed genes |
|
图 4 差异基因的GO分析 Fig. 4 GO analysis of differentially expressed genes |
|
|
表 5 差异基因的KEGG通路富集分析 Table 5 KEGG pathway enrichment analysis of differentially expressed genes |

|
图 5 差异基因的KEGG通路富集分析 Fig. 5 KEGG pathway enrichment analysis of differentially expressed genes |
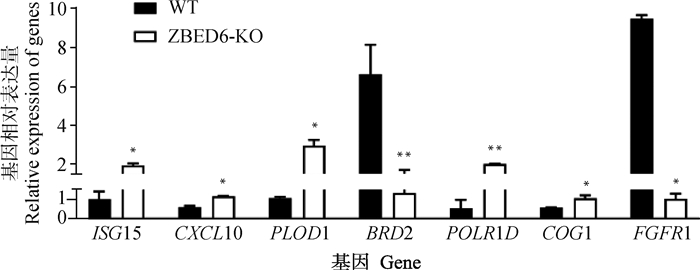
随机选取ISG15、CXCL10、PLOD1、BROD2、POLRLD、COG1、FGFR1共7个基因进行RT-qPCR验证,以β-actin为内参基因,7个差异基因的RT-qPCR结果(图 6)的表达模式与测序数据的结果一致,证实RNA-seq测序的准确性。
|
*.P < 0.05; **.P < 0.01 图 6 7个差异表达基因的qRT-PCR验证 Fig. 6 qRT-PCR verification of 7 differentially expressed genes |
Jeon等[3, 11-12]在猪中发现,IGF2基因内含子3的一个单碱基突变(G突变为A)可增加猪3%~4%的瘦肉量,并且内含子3突变的区域内还存在一个未知抑制因子。后来发现,这个未知因子为转录因子ZBED6[4]。在哺乳动物中,ZBED6基因的序列具有高度的保守性和广泛的组织分布,并可调节肌肉的生长发育。在小鼠、牛、山羊等不同种哺乳动物中对ZBED6的影响与调控进行了一定程度的研究,表明ZBED6能够调控肌肉的生长发育[5, 13-14]。近年来的一项研究发现,将IGF2敲入中国地方巴马香猪后,发现其产肉量有所提高[6]。但ZBED6基因对猪生长发育的影响尚不清楚,对脾生长发育的作用机制也不明晰。
本试验结果显示,ZBED6-KO猪脾组织的IGF2表达量显著高于野生型,这与之前在小鼠和肉牛中的结果相符[4, 15-21]。ZBED6-KO猪的脾组织重量较WT猪增加,且差异显著。这些结果暗示,ZBED6基因的敲除能够促进脾组织的生长发育。ZBED6-KO猪和WT猪脾组织的转录组中共得到161个差异基因,这些差异基因主要参与肌动蛋白丝切断、肌动蛋白细胞骨架的调节、T细胞的趋化作用等于肌肉发育相关及免疫反应的生物学过程和通路。
在筛选到的与肌肉发育相关的通路中,涉及DSTN、FGFR1、LIMK1、MAPK3、CFL1、NCKAP1L、ITGB3、MSN、PPP1CC基因,推测ZBED6可能通过影响这些基因的表达及其所在通路进而调控脾组织的生长发育。
在涉及的9个基因中,FGFR1(成纤维细胞因子Ⅰ型受体)为成纤维细胞生长因子受体(FGFR)之一,其作为受体酪氨酸激酶超家族的成员,能与成纤维细胞生长因子(FGF)结合,激活下游信号转导通路,调控细胞的增殖、分化和存活,对胚胎发育、器官形成、血管生成、组织修复及代谢调控等生理功能发挥重要作用[22-23]。研究发现,FGFR1功能丧失性突变可引起人类Kallmann综合征,导致生长缓慢,个子矮小[24]。最近一项研究表明,FGFR1是肢芽发生、发育和趾骨图式发育所必需的。在胚胎发育过程中,FGFR1基因在肢芽的间质和中胚层侧板、体节和其它退化器官中表达,表明在骨骼发育中可能有一定的作用[25-27]。本研究中,ZBED6-KO猪FGFR1基因的表达量是野生型的1.98倍,推测ZBED6基因的敲除通过上调FGFR1基因的表达,促进巴马香猪脾组织的生长发育。
肌动蛋白丝(也称微丝)为肌动蛋白的组成元件之一,是真核生物细胞骨架的必需成分。它不仅参与了细胞内物质的运输和肌肉的收缩,还与细胞的移动以及细胞分裂等活动有关,其形成的大规模网络结构,对细胞器的运动、形状提供机械支持以及传导信号等诸多功能[28],对细胞的增殖也有重要影响[29]。哺乳动物的减数分裂高度依赖于完整的微丝网络[28]。DSTN又称丝切蛋白(ADF),属于肌动蛋白结合蛋白ADF家族,广泛存在于真核生物中,对细胞生长发育至关重要,是组织肌丝中必需成分[30]。在细胞运动、胞质分裂和其他细胞运动过程中起着加速微丝翻转的作用[28, 31-33],能提高肌动蛋白的周转速度。ADF蛋白能够与肌动蛋白丝(F-肌动蛋白)以1∶1的比例结合,诱导微丝解聚[34-38],切断肌动蛋白丝(F-肌动蛋白)并与肌动蛋白单体(G-肌动蛋白)结合[29, 38-40]。DSTN在ZBED6-KO猪脾组织中的表达量较WT显著升高,推测在巴马香猪脾组织中ZBED6基因通过调控DSTN基因的表达,促进脾组织中细胞的增殖分裂活动及生长发育。
ZBED6基因敲除后,除了影响脾和肌肉发育,同样影响了巴马香猪脾组织免疫相关的生物学进程,这与脾组织作为淋巴器官之一也有一定的关系。在与免疫反应相关的通路中,富集到的主要基因为CXCL10、CXCL11、PLD2、LIMK1、MAPK3、CFL1。
LIMK1为一种位于Rho家族下游的丝氨酸、苏氨酸蛋白激酶,是丝切蛋白的上游调控因子,通过影响丝切蛋白与肌动蛋白结合蛋白的活性,从而防止肌动蛋白细胞骨架的降解,还可抑制肿瘤的迁移[41]。LIMK1在多种肿瘤中,如恶性黑色素瘤、乳腺癌和前列腺癌等过表达[42]。在上游信号分子介导的信号通路中,LIMK1处于中心地位,可能为肿瘤细胞迁移与侵袭的重要靶点[43-44]。而沉默LIMK1基因可减少胰腺癌细胞及肿瘤诱导的血管形成[45]。LIMK1基因在ZBED6-KO猪脾组织中的表达量较野生型有显著降低,因此推测,ZBED6基因的敲除有可能降低巴马香猪脾组织中肿瘤发生的可能性,进而提高其抗病能力。
CXCL10基因(CXC趋化因子配体10)又称干扰素诱导蛋白(IP-10),是CXC趋化因子超家族中的一员,具有调节免疫应答、血管生成、细胞凋亡、细胞周期以及细胞增殖等作用。多项研究表明,CXCL10与多种肿瘤的发生、发展、治疗疗效以及预后相关[46-47]。CXCL10与CXCR3受体结合,抑制黑色素瘤血管生成作用,降低肿瘤内血管密度,增加癌细胞凋亡,使癌组织坏死[48]。Giuliani等[46]研究发现,骨髓瘤细胞可通过自分泌方式分泌CXCL10,随后与骨髓瘤细胞上的CXCR3B受体结合,缩短肿瘤细胞的生存时间发挥抗增殖效应。本研究中CXCL10在ZBED6-KO猪脾组织中表达量是WT猪的1.87倍,CXCL10表达量显著升高增强了家猪抵御肿瘤的抗病能力。
4 结论与野生型巴马香猪相比,敲除ZBED6基因之后,显著提高了猪的脾组织重量。IGF2作为ZBED6转录因子的调控基因,与组织的生长发育密切相关。在ZBED6-KO猪中,脾组织的IGF2基因表达量显著升高。ZBED6-KO猪与WT猪相比较共筛选到161个差异表达基因,其中上调基因90个,下调基因71个;差异表达基因的GO和KEGG富集分析筛选到2条与肌肉生长发育显著相关的通路,即肌动蛋白丝切断和肌动蛋白细胞骨架的调节。由此推测,ZBED6基因的敲除可能通过上述生物学过程和通路影响了脾组织的生长发育。
| [1] |
VAN LAERE A S, COPPIETERS W, GEORGES M. Characterization of the bovine pseudoautosomal boundary: documenting the evolutionary history of mammalian sex chromosomes[J]. Genome Res, 2008, 18(12): 1884-1895. DOI:10.1101/gr.082487.108 |
| [2] |
WANG X, JIANG L, WALLERMAN O, et al. Transcription factor ZBED6 affects gene expression, proliferation, and cell death in pancreatic beta cells[J]. Proc Natl Acad Sci U S A, 2013, 110(40): 15997-16002. DOI:10.1073/pnas.1303625110 |
| [3] |
JEON J T, CARLBORG O, TÖRNSTEN A, et al. A paternally expressed QTL affecting skeletal and cardiac muscle mass in pigs maps to the IGF2 locus[J]. Nat Genet, 1999, 21(2): 157-158. DOI:10.1038/5938 |
| [4] |
MARKLJUNG E, JIANG L, JAFFE J D, et al. ZBED6, a novel transcription factor derived from a domesticated DNA transposon regulates IGF2 expression and muscle growth[J]. PLoS Biol, 2009, 7(12): e1000256. DOI:10.1371/journal.pbio.1000256 |
| [5] |
YOUNIS S, SCHÖNKE M, MASSART J, et al. The ZBED6-IGF2 axis has a major effect on growth of skeletal muscle and internal organs in placental mammals[J]. Proc Natl Acad Sci U S A, 2018, 115(9): E2048-E2057. DOI:10.1073/pnas.1719278115 |
| [6] |
XIANG G H, REN J L, HAI T, et al. Editing porcine IGF2 regulatory element improved meat production in Chinese Bama pigs[J]. Cell Mol Life Sci, 2018, 75(24): 4619-4628. DOI:10.1007/s00018-018-2917-6 |
| [7] |
KIM D, PAGGI J M, PARK C, et al. Graph-based genome alignment and genotyping with HISAT2 and HISAT-genotype[J]. Nat Biotechnol, 2019, 37(8): 907-915. DOI:10.1038/s41587-019-0201-4 |
| [8] |
PERTEA M, KIM D, PERTEA G M, et al. Transcript-level expression analysis of RNA-seq experiments with HISAT, StringTie and Ballgown[J]. Nat Protoc, 2016, 11(9): 1650-1667. DOI:10.1038/nprot.2016.095 |
| [9] |
TRAPNELL C, ROBERTS A, GOFF L, et al. Differential gene and transcript expression analysis of RNA-seq experiments with TopHat and Cufflinks[J]. Nat Protoc, 2012, 7(3): 562-578. DOI:10.1038/nprot.2012.016 |
| [10] |
DENNIS JR G, SHERMAN B T, HOSACK D A, et al. DAVID: database for annotation, visualization, and integrated discovery[J]. Genome Biol, 2003, 4(5): P3. DOI:10.1186/gb-2003-4-5-p3 |
| [11] |
NEZER C, MOREAU L, BROUWERS B, et al. An imprinted QTL with major effect on muscle mass and fat deposition maps to the IGF2 locus in pigs[J]. Nat Genet, 1999, 21(2): 155-156. DOI:10.1038/5935 |
| [12] |
VAN LAERE A S, NGUYEN M, BRAUNSCHWEIG M, et al. A regulatory mutation in IGF2 causes a major QTL effect on muscle growth in the pig[J]. Nature, 2003, 425(6960): 832-836. DOI:10.1038/nature02064 |
| [13] |
SAMUELS Y, WANG Z H, BARDELLI A, et al. High frequency of mutations of the PIK3CA gene in human cancers[J]. Science, 2004, 304(5670): 554. DOI:10.1126/science.1096502 |
| [14] |
党云龙. 辽宁绒山羊IGF2和ZBED6基因分子特征、表达及甲基化研究[D]. 沈阳: 沈阳农业大学, 2017. DANG Y L. Molecular Characteristics, expression and methylation analysis of IGF2 and ZBED6 genes in Liaoning Cashmere goats[D]. Shenyang: Shenyang Agricultural University, 2017. (in Chinese) |
| [15] |
BUTTER F, KAPPEI D, BUCHHOLZ F, et al. A domesticated transposon mediates the effects of a single-nucleotide polymorphism responsible for enhanced muscle growth[J]. EMBO Rep, 2010, 11(4): 305-311. DOI:10.1038/embor.2010.6 |
| [16] |
HUANG Y Z, SUN Y J, LI M X, et al. Evaluation of the causality of the zinc finger BED-type containing 6 gene (ZBED6) for six important growth traits in Nanyang beef cattle[J]. Anim Genet, 2015, 46(2): 225-226. DOI:10.1111/age.12265 |
| [17] |
HUANG Y Z, SUN Y J, ZHAN Z Y, et al. Expression, SNP identification, linkage disequilibrium, and haplotype association analysis of the growth suppressor gene ZBED6 in Qinchuan beef cattle[J]. Anim Biotechnol, 2014, 25(1): 35-54. DOI:10.1080/10495398.2013.814572 |
| [18] |
HUANG Y Z, ZHAN Z Y, SUN Y J, et al. Comparative analysis of the IGF2 and ZBED6 gene variants and haplotypes reveals significant effect of growth traits in cattle[J]. Genome, 2013, 56(6): 327-334. DOI:10.1139/gen-2013-0062 |
| [19] |
HUANG Y Z, ZHANG L Z, LAI X S, et al. Transcription factor ZBED6 mediates IGF2 gene expression by regulating promoter activity and DNA methylation in myoblasts[J]. Sci Rep, 2014, 4: 4570. |
| [20] |
HUANG Y Z, LI M X, WANG J, et al. A 5'-regulatory region and two coding region polymorphisms modulate promoter activity and gene expression of the growth suppressor gene ZBED6 in cattle[J]. PLoS One, 2013, 8(11): e79744. DOI:10.1371/journal.pone.0079744 |
| [21] |
JIANG L, WALLERMAN O, YOUNIS S, et al. ZBED6 modulates the transcription of myogenic genes in mouse myoblast cells[J]. PLoS One, 2014, 9(4): e94187. DOI:10.1371/journal.pone.0094187 |
| [22] |
SARGAR K M, SINGH A K, KAO S C. Imaging of skeletal disorders caused by fibroblast growth factor receptor gene mutations[J]. RadioGraphics, 2017, 37(6): 1813-1830. DOI:10.1148/rg.2017170017 |
| [23] |
GOETZ R, MOHAMMADI M. Exploring mechanisms of FGF signalling through the lens of structural biology[J]. Nat Rev Mol Cell Biol, 2013, 14(3): 166-180. DOI:10.1038/nrm3528 |
| [24] |
JOHNSON D E, LU J, CHEN H, et al. The human fibroblast growth factor receptor genes: a common structural arrangement underlies the mechanisms for generating receptor forms that differ in their third immunoglobulin domain[J]. Mol Cell Biol, 1991, 11(9): 4627-4634. |
| [25] |
PETERS K G, WERNER S, CHEN G, et al. Two FGF receptor genes are differentially expressed in epithelial and mesenchymal tissues during limb formation and organogenesis in the mouse[J]. Development, 1992, 114(1): 233-243. DOI:10.1242/dev.114.1.233 |
| [26] |
VERHEYDEN J M, LEWANDOSKI M, DENG C X, et al. Conditional inactivation of Fgfr1 in mouse defines its role in limb bud establishment, outgrowth and digit patterning[J]. Development, 2005, 132(19): 4235-4245. DOI:10.1242/dev.02001 |
| [27] |
KINDBLOM J M, GEVERS E F, SKRTIC S M, et al. Increased adipogenesis in bone marrow but decreased bone mineral density in mice devoid of thyroid hormone receptors[J]. Bone, 2005, 36(4): 607-616. DOI:10.1016/j.bone.2005.01.017 |
| [28] |
BAMBURG J R. Proteins of the ADF/cofilin family: essential regulators of actin dynamics[J]. Annu Rev Cell Dev Biol, 1999, 15: 185-230. DOI:10.1146/annurev.cellbio.15.1.185 |
| [29] |
YONEZAWA N, HOMMA Y, YAHARA I, et al. A short sequence responsible for both phosphoinositide binding and actin binding activities of cofilin[J]. J Biol Chem, 1991, 266(26): 17218-17221. DOI:10.1016/S0021-9258(19)47361-4 |
| [30] |
ONO S, BAILLIE D L, BENIAN G M. UNC-60B, an ADF/cofilin family protein, is required for proper assembly of actin into myofibrils in Caenorhabditis elegans body wall muscle[J]. J Cell Biol, 1999, 145(3): 491-502. DOI:10.1083/jcb.145.3.491 |
| [31] |
MACIVER S K. How ADF/cofilin depolymerizes actin filament[J]. Curr Opin Cell Biol, 1998, 10(1): 140-144. DOI:10.1016/S0955-0674(98)80097-5 |
| [32] |
CARLIER M F, RESSAD F, PANTALONI D. Control of actin dynamics in cell motility[J]. J Biol Chem, 1999, 274(48): 33827-33830. DOI:10.1074/jbc.274.48.33827 |
| [33] |
MCGOUGH A. Pope B, Weeds A. The ADF/cofilin family: accelerators of actin reorganization[M]//DOS REMEDIOS C G, THOMAS D D. Molecular Interactions of Actin: Actin Structure and Actin-Binding Proteins. Berlin, Heidelberg: Springer, 2001: 135-154.
|
| [34] |
MACIVER S K, POPE B J, WHYTOCK S, et al. The effect of two actin depolymerizing factors (ADF/cofilins) on actin filament turnover: pH sensitivity of F-actin binding by human ADF, but not of Acanthamoeba actophorin[J]. Eur J Biochem, 1998, 256(2): 388-397. DOI:10.1046/j.1432-1327.1998.2560388.x |
| [35] |
DU J Y, FRIEDEN C. Kinetic studies on the effect of yeast cofilin on yeast actin polymerization[J]. Biochemistry, 1998, 37(38): 13276-13284. DOI:10.1021/bi981117r |
| [36] |
BLANCHOIN L, POLLARD T D. Mechanism of interaction of Acanthamoeba actophorin (ADF/cofilin) with actin filaments[J]. J Biol Chem, 1999, 274(22): 15538-15546. DOI:10.1074/jbc.274.22.15538 |
| [37] |
ICHETOVKIN I, HAN J H, PANG K M, et al. Actin filaments are severed by both native and recombinant Dictyostelium cofilin but to different extents[J]. Cell Motil Cytoskeleton, 2000, 45(4): 293-306. DOI:10.1002/(SICI)1097-0169(200004)45:4<293::AID-CM5>3.0.CO;2-1 |
| [38] |
POPE B J, GONSIOR S M, YEOH S, et al. Uncoupling actin filament fragmentation by cofilin from increased subunit turnover[J]. J Mol Biol, 2000, 298(4): 649-661. DOI:10.1006/jmbi.2000.3688 |
| [39] |
LAPPALAINEN P, FEDOROV E V, FEDOROV A A, et al. Essential functions and actin-binding surfaces of yeast cofilin revealed by systematic mutagenesis[J]. EMBO J, 1997, 16(18): 5520-5530. DOI:10.1093/emboj/16.18.5520 |
| [40] |
ONO S, MCGOUGH A, POPE B J, et al. The C-terminal tail of UNC-60B (actin depolymerizing factor/cofilin) is critical for maintaining its stable association with F-actin and is implicated in the second actin-binding site[J]. J Biol Chem, 2001, 276(8): 5952-5958. DOI:10.1074/jbc.M007563200 |
| [41] |
TAKAHASHI H, FUNAKOSHI H, Nakamura T. LIM-kinase as a regulator of actin dynamics in spermatogenesis[J]. Cytogenet Genome Res, 2003, 103(3-4): 290-298. DOI:10.1159/000076815 |
| [42] |
HANAHAN D, WEINBERG R A. Hallmarks of cancer: the next generation[J]. Cell, 2011, 144(5): 646-674. DOI:10.1016/j.cell.2011.02.013 |
| [43] |
SCOTT R W, HOOPER S, CRIGHTON D, et al. LIM kinases are required for invasive path generation by tumor and tumor-associated stromal cells[J]. J Cell Biol, 2010, 191(1): 169-185. DOI:10.1083/jcb.201002041 |
| [44] |
HORITA Y, OHASHI K, MUKAI M, et al. Suppression of the invasive capacity of rat ascites hepatoma cells by knockdown of Slingshot or LIM kinase[J]. J Biol Chem, 2008, 283(10): 6013-6021. DOI:10.1074/jbc.M706538200 |
| [45] |
BRYAN B A, DENNSTEDT E, MITCHELL D C, et al. RhoA/ROCK signaling is essential for multiple aspects of VEGF-mediated angiogenesis[J]. FASEB J, 2010, 24(9): 3186-3195. DOI:10.1096/fj.09-145102 |
| [46] |
GIULIANI N, BONOMINI S, ROMAGNANI P, et al. CXCR3 and its binding chemokines in myeloma cells: expression of isoforms and potential relationships with myeloma cell proliferation and survival[J]. Haematologica, 2006, 91(11): 1489-1497. |
| [47] |
LI G, TIAN L, HOU J M, et al. Improved therapeutic effectiveness by combining recombinant CXC chemokine ligand 10 with Cisplatin in solid tumors[J]. Clin Cancer Res, 2005, 11(11): 4217-4224. DOI:10.1158/1078-0432.CCR-04-2117 |
| [48] |
YANG J M, RICHMOND A. The angiostatic activity of interferon-inducible protein-10/CXCL10 in human melanoma depends on binding to CXCR3 but not to glycosaminoglycan[J]. Mol Ther, 2004, 9(6): 846-855. DOI:10.1016/j.ymthe.2004.01.010 |
(编辑 范子娟)



