当来自双亲中任何一方的两个等位基因传递给后代的机会不均等时,就会观察到偏分离现象,导致统计上偏离孟德尔遗传率0.5[1-3]。偏分离是自然界中广泛存在的现象,是遗传和进化的基础。随着分子标记和测序技术的发展,许多偏分离研究被报道。在植物中的偏分离研究对象包括玉米[4]、棉花[5]、小麦[6]、水稻[7]等,在动物中的偏分离研究对象主要是模式动物,如果蝇[8]、老鼠[9-10]等。大型家养动物中偏分离研究相对较少,目前仅在牛中有些报道[11]。大型家养动物较少研究偏分离机制,其主要原因是研究偏分离需要完整的两代以上大规模的家系数据及基因型数据,而对大规模个体的基因分型耗费较大。随着基因芯片技术的发展,快速检测个体高密度基因型的价格不断下降,对大规模群体高密度基因分析也已实现,使得偏分离研究在大型家养动物中成为可能。猪作为伴随人类进化历程的驯养动物,其生理特性和器官大小与人非常相似,对猪进行偏分离研究,不仅让我们对猪的偏分离有基本的认识,也为大型家养动物的偏分离研究提供一定的参考。
由于偏分离悖离了孟德尔分离定律(父母遗传给后代的等位基因的比例为1∶1),因此我们能在基于家系的研究中观察到偏分离现象。通常情况下,传统的传递不平衡检验(transmission disequilibrium test,TDT)可以用在父亲-母亲-后代的三元家系中检测偏分离效应。但是,当父母双方都是杂合子时,TDT的检验效力会降低[12]。所以,研究人员提出了新的检验父母特异性来源的偏分离方法,包括传递不对称检验(transmission asymmetry test,TAT)、亲本来源的似然比检验[13](parent-of-origin likelihood ratio test,PO-LRT)、基于贝叶斯模型的方法[14]等。本研究使用基于贝叶斯模型的偏分离分析软件TRDscan v.1.0[15]以及用自编R语言脚本实现的TDT和TDTMVsF方法分别分析了总的偏分离位点和父母特异性偏分离位点。对所鉴定的显著性偏分离位点周围100 kb内利用BioMart挖掘工具筛选功能相关基因,并结合生物信息学工具DAVID[16]与GeneCard(https://www.genecards.org/)注释相应基因的功能和通路。另外,提出一种基于单倍型连锁相分离不平衡的检测方法,通过PHASEBOOK[17]单倍型分型软件包中的LinkPHASE3[18]和HiddenPHASE构建单倍型,得到后代个体继承父母的连锁相信息,分别对后代父源、母源染色体的继承模式计数并进行皮尔逊卡方检验,从而得到父母源连锁相的偏分离效应估计。
本研究以2头白色杜洛克公猪和17头中国二花脸母猪构建的三代资源家系1 020个个体为研究对象,利用60K芯片分型数据,结合基于贝叶斯的TRDscan软件和TDT方法,并通过生物信息学分析,探究在两方法中都出现显著信号的位点100 kb区域的基因。此外,基于单倍型继承模式,提出一种能够鉴别父母源同系物的偏分离效应的启发式方法。
1 材料与方法 1.1 数据来源本研究以2头白色杜洛克公猪和17头中国二花脸母猪构建的三代资源家系群体[19]共1 020个个体为研究对象。用常规的酚氯仿试剂提取法,从耳组织或血液中提取DNA,并将DNA浓度稀释至50 ng·μL-1。经质控后符合要求的基因组DNA利用猪Illumina 60K SNP芯片进行基因型分型,本研究共检测了1 020个个体,每个个体获得了62 163个SNPs位点的基因型。
1.2 数据处理利用Plink V1.9[20]对原始基因型数据进行质控,删除基因型缺失率大于5%、次等位基因型频率小于0.5%的位点以及删除基因型缺失大于5%的个体。由于性染色体分离与性别相关,在偏分离估算时对结果影响较大,质控时过滤了性染色体标记。质控后,1 020个个体共45 966个SNPs位点用于后续分析。质控后的数据使用TRDScan软件进行偏分离分析,TDT和分析父母特异性的TDTMVsF方法使用自编的R语言脚本实现偏分离分析。在基于单倍型连锁相偏分离算法中,除上述质控条件外,将孟德尔错误率大于0.06的位点和孟德尔错误率大于0.1的家系过滤,利用PHASEBOOK软件包中的LinkPhase和HiddenPhase构建单倍型并追溯后代单倍型的继承信息。
1.3 统计模型利用TRDscan v.1.0软件对总的偏分离效应、父系偏分离效应、母系偏分离效应进行分析,统计模型为[15]:
| $ \begin{aligned} &p(\alpha \mid y) \propto p(y \mid \alpha) p(\alpha) \text { 以及 } \\ &p\left(\alpha_{s}, \alpha_{d} \mid y\right) \propto p\left(y \mid \alpha_{s}, \alpha_{d}\right) p\left(\alpha_{s}\right) p\left(\alpha_{d}\right) \end{aligned} $ |
式中,α表示服从均匀分布的总的偏分离参数;αs表示服从均匀分布的父系偏分离参数;αd表示服从均匀分布的母系偏分离参数;y表示后代基因型的列向量。
TDT和TDTMVsF方法利用皮尔逊卡方检验分别对总偏分离效应、父系偏分离效应、母系偏分离效应进行显著性检验。TDT和分析父母特异性的TDTMVsF方法的卡方值类似[13]:
| $ \chi_{i}^{2}=\left(T S_{i}-N T_{i}\right)^{2} /\left(T S_{i}+N T_{i}\right) $ | (1-1) |
式中,χi2为第i个标记的卡方值;TSi为第i个标记父母传递给后代等位基因的数量;NTi为第i个标记父母没有传递给后代等位基因的数量。
式中父母传递给后代等位基因指的是在一个包含父母和后代的三元单元中,杂合子父母传递给后代等位基因的数量。总的TDT和考虑父母特异性的TDTMVsF差异是TDT中的数量为杂合子父亲和母亲传递给后代的和,而TDTMVsF中分别是父亲或母亲传递给后代等位基因的数量[21-22]。
基于单倍型继承模式的偏分离方法的卡方值:
| $ \chi_{k i}^{2}=\left(N_{k i}^{1}-N_{k i}^{2}\right)^{2} / N_{k i}^{1}+N_{k i}^{2} $ | (1-2) |
式中,χki2: 第i个标记的k单倍型的卡方值, (k= 1、2);Nki1为第i个标记处单倍型k中继承亲本(左/右)侧染色体的群体数;Nki2为第i个标记处单倍型k中继承模亲本(右/左)侧染色体的群体数。
k=1时,分析的是父源染色体中继承自父亲左侧单倍型和右侧单倍型的偏分离效应,k=2时对应母源染色体中继承自母亲左侧单倍型1和右侧单倍型的偏分离效应。
2 结果 2.1 总的偏分离效应分析通过使用两种不同的方法,可以定位猪中全基因组范围的偏分离位点。在本研究中,质控后共有45 966个SNPs,分别使用贝叶斯模型的TRDscan软件统计推断通过贝叶斯因子(Bayes factor, BF)和R语言的TDT脚本用于偏分离分析。两种检测方法的显著性阈值分别为BF>100和P < 0.001[23]。结果表明,在所有的染色体上都存在显著位点(图 1A、1B),特别是在2和4号染色体上的显著位点较集中,而其他染色体的显著位点无明显聚集现象。对两种偏分离结果取交集,共得到在两种方法中都表现为显著偏分离的SNPs位点44个(表 1)。

|
A. 以TDT方法的偏分离分析曼哈顿图;B.TRDScan软件的偏分离分析曼哈顿图 A. The TRD Manhattan plot was analyzed by TDT method; B. The TRD Manhattan plot was analyzed by TRDScan software 图 1 总的偏分离分析曼哈顿图 Fig. 1 Total TRD Manhattan diagram |
|
|
表 1 与总TRD密切相关的SNPs(BF>100,P < 0.01) Table 1 SNPs closely related to all TRD(BF>100, P < 0.01) |
偏分离效应常常与性别有关[24-25],因此,分析时区分父母的特异性偏分离有助于得到更准确的结果。本研究利用基于贝叶斯算法的TRDscan和基于传递不平衡方法的R脚本分别对父源和母源的偏分离位点进行分析,结果如图 2所示。两种方法的偏分离效应结果相似,父系与母系偏分离在所有染色体中都存在显著位点,特别是在父母特异性的结果中2号染色体都存在明显的成簇显著信号(图 2)。另外,还发现不考虑父母特异性偏分离时,相比区分父母特异性偏分离效应时更显著,这与区分父母特异性结果更准确的理论一致。通过结合TRDscan v.1.0和TDTMVsF方法得到的父母特异性偏分离结果,在父系特异性偏分离分析中共得到在两方法中都显示显著性偏分离的27个SNPs位点, 母系特异性偏分离分析中共得到35个显著偏分离的SNPs位点。
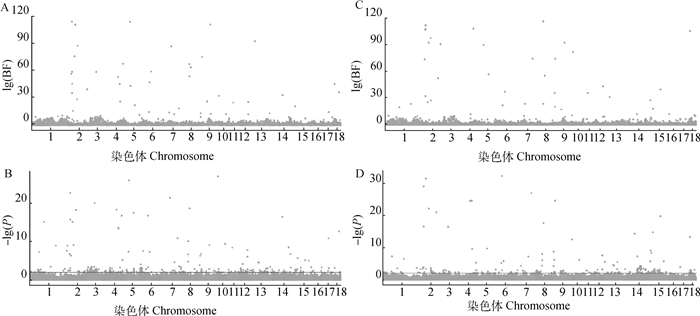
|
A. TRDScan母系偏分离分析曼哈顿图;B. TDTMVsF母系偏分离分析曼哈顿图;C. TRDScan父系偏分离分析曼哈顿图;D. TDTMVsF父系偏分离分析曼哈顿图 A. The maternal TRD Manhattan plot was analyzed by TRDScan software; B. The maternal TRD Manhattan plot was analyzed by using TDTMVsF; C. The paternal TRD Manhattan plot was analyzed by TRDScan software; D. The paternal TRD Manhattan plot was analyzed by using TDTMVsF 图 2 父母特异性偏分离分析曼哈顿图 Fig. 2 Manhattan graph of parental specific TRD analysis |
为了探究引起猪发生偏分离的潜在基因,本研究在Ensembl猪基因组数据库中查找了显著位点100 kb左右区域的基因(http://uswest.ensembl.org/Sus_scrofa/Info/Index)。
不考虑父母特异性偏分离效应时,共筛选到23个基因;考虑父母特异性偏分离效应时,其中父系特异性偏分离位点中筛选出11个基因,母系特异性偏分离位点中筛选出25个基因,比较分析3种偏分离效应所鉴定到的基因,发现5个基因(CRACDL、ISCA2、KIT、MOGAT2、NANOG)在总的偏分离、父系特异性偏分离、母系特异性偏分离分析中都被筛选到(表 2)。2个基因(LMNB1、MCM6)在总的和父系特异性偏分离分析中都被检索到。9个基因(PBXIP1、PMVK、RP9、SHC1、SLC25A27、TDRD6、C20orf194、DCST2、GNAT2)被发现在总的和母系偏分离效应分析结果中存在,相比2个在总的和父系偏分离效应分析中的基因,表明母系偏分离效应在本试验群体中占主要部分。3个(OR51F1、OR51C1P、OR51E2)在父系和母系偏分离效应分析中都出现的基因与完整精子组装,精子细胞运动等有关。1个只在总的偏分离分析中出现的基因(SPAG6)和2个只在母系特异性偏分离分析中出现的基因(ALX4、ADAM22)。
|
|
表 2 引起猪偏分离的候选基因 Table 2 Candidate genes causing porcine TRD |
基于单倍型继承父母染色体信息,可以得到染色体水平的偏分离效应估计。对质控后的44 864个SNPs进行单倍型偏分离分析,以0.01作为显著水平。结果显示,在父本偏分离分析中, 5和13号染色体出现少数显著偏分离的位点,而母本偏分离分析中, 4、6、12号染色体有较多位点表现出显著偏分离的现象(图 3)。为了搜寻这些区域出现显著偏分离的潜在候选基因,在猪QTL数据库animalQTLdb(https://www.animalgenome.org/)中查询显著性偏分离区域内相关的QTLs。结果显示,父源染色体的显著偏分离区域与3个繁殖性状QTLs区域重叠,分别为4号染色体的QTL: 178849、5号染色体的QTL: 18128和13号染色体QTL: 493。而母源染色体的显著偏分离区域与5个繁殖性状QTLs区域重叠,包括3号染色体的QTL: 515、4号染色体的QTL: 450和QTL: 18337、6号染色体的QTL: 160544和12号染色体的QTL: 120292 (表 3)。此外,为了检验得到的繁殖性状相关QTL的随机性,随机抽取与鉴定到的显著区域相当的区域1 000次,并检索猪QTL数据库,结果显示,只出现了一次得到8个繁殖性状QTL,表明偏分离位点倾向于与繁殖性状QTL重叠。
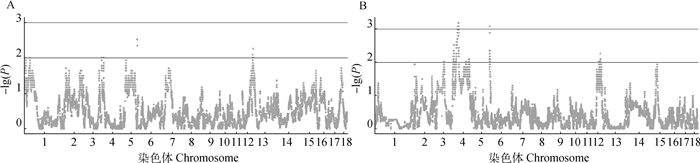
|
A. 父传染色体偏分离分析曼哈顿图;B. 母传染色体偏分离分析曼哈顿图 A. Manhattan plot of paternal chromosomal TRD analysis; B. Manhattan plot of maternal chromosomal TRD analysis 图 3 基于单倍型继承模式的偏分离曼哈顿图 Fig. 3 Manhattan graph of TRD based on haplotype inheritance pattern |
|
|
表 3 基于单倍型继承模式显著偏分离区域相关QTL分析 Table 3 The significant TRD of region-related QTLs based on haplotype inheritance pattern |
引起后代产生偏分离的机制比较复杂,在很多生物中偏分离的具体机制仍不清楚[26]。总的来说,从精子或卵子发生的减数分裂开始到配子形成合子,到胚胎发育再到形成后代,偏分离的机制包括:1)不对称的减数分裂,在大多数动物和植物中,雌性的减数分裂是不对称的,减数分裂的4个单倍体只有一个继续成为卵母细胞,这种细胞命运的不对称性是一种潜在的偏分离来源,任何可以优先分离到卵母细胞的变异都会获得传播优势[27];2)配子的偏分离主要是精子竞争引起的,雄性与雌性不同的是,雄性会产生大量的较小的配子(精子或花粉),因此,雄性配子之间的竞争尤其激烈,这既是自然选择的主要场所,也是偏分离的可能来源[28];3)精子/卵子致死,是单倍体基因产物自私的杀死或禁止配子成功受精;4)单倍体不兼容,是两个单倍体配子结合后由于等位基因间之间负的相互作用导致的差异;5)合子的偏分离机制包括近交衰退和杂种优势带来的差异;6)母胎相容性,不同二倍体基因型的胚胎存活率差异[29];7)由于印记基因错误导致的胚胎存活差异; 8)诸多环境因素的影响,其中,温度是影响配子体选择并导致生物遗传分化的重要因子[30]。
以上机制表明,偏分离偏向于影响繁殖性状,即偏分离现象的产生与繁殖性状紧密相关,而繁殖性状在猪中一直是影响经济效应最重要的因素之一,与养殖场的经济效益密切相关。基因组偏分离的存在会减少特定基因型存活率,导致母猪繁殖性状的指标如总产仔数、产活仔数、受胎率等降低。对猪偏分离的研究让我们可能鉴定到基因组上引起偏分离现象的位点,这些位点可作为后续育种工作的选择位点,结合基因组选择等技术为提高猪的繁殖性能提供参考和应用价值。
基于单倍型继承模式,本研究同时分析了父传和母传同系物的偏分离现象,通过查询猪QTL数据库,在结果中分别找到了3个和5个繁殖性状相关的QTLs。与其他性状如胴体性状相关的QTLs没有在表中展示,原因是,尽管引起偏分离的机制比较复杂,但最后都会直接或间接影响生物的繁殖性状。亲本的左右系同系物中存在偏分离现象,且这些繁殖性状相关的QTLs可能存在潜在的候选基因。
3.2 引起猪偏分离候选基因的分析本研究鉴定出一些引起猪偏分离的候选基因。CRACDL是蛋白编码基因,与睾丸白血病有关,影响睾丸的正常生理功能。KIT是编码受体酪氨酸激酶蛋白的基因,通过KIT发出的信号在细胞存活、增殖和分化中起作用。例如,KIT信号传导是黑色素细胞存活所必需的,此外它还涉及机体造血和配子发生[31]。NANOG是胚胎干细胞的转录因子,被认为是维持多能性的关键基因。NANOG基因与中胚层细胞命运,胚胎模式规范,干细胞分裂等分子过程相关[32]。ISCA2基因编码的蛋白质是线粒体中的一种A型铁硫簇(ISC)蛋白质,该蛋白似乎与线粒体铁硫蛋白质的成熟有关。LMNB1基因编码核纤层蛋白B1,核纤层蛋白B1被认为与核稳定性,染色质核基因表达有关[33]。MCM6基因编码DNA复制许可因子MCM6,是高度保守的微型染色体维持蛋白(MCM)之一,在真核基因组启动复制中起着至关重要的作用[34]。PBXIP1编码的蛋白质主要在细胞质中,但可以穿梭至细胞核,还可以与雌激素受体α和β相互作用,并促进乳腺癌、脑瘤和肺癌的增殖[35]。TDRD6是一种包含Tudor域蛋白质编码基因,含有Tudor域的蛋白质与生殖细胞发育,包括精子形成过程中类染色体的形成,卵子形成过程中的巴尔比尼亚体的形成,受精后生殖细胞的细胞质形成,以及适当的miRNA表达和剪接体成熟,可见TDTR6基因在生殖细胞的形成过程中起着重要的作用[36]。C20orf194基因编码具有C末端卷曲螺旋区的未鉴定蛋白,DCST2基因与机体发育有关,GNAT2基因编码鸟嘌呤核苷酸结合蛋白G的α亚基,在视觉冲动中刺激视紫红质和cGMP磷酸二酯酶的偶联[37]。3个(OR51F1、OR51C1P、OR51E2)在父系和母系偏分离效应分析中都出现的基因,这3个基因为气味受体51家族基因,编码嗅觉受体蛋白,负责识别和G蛋白介导的气味信号转导[38]。从机理上来看,气味受体51家族基因与偏分离并不存在直接的关系,实际上,本研究得到的许多基因从功能上看与偏分离的潜在机制并没有很明显的直接联系,但是,这些基因可能间接的引起基因组偏分离现象,或者与引起偏分离的基因存在一定程度的连锁不平衡。另外,只利用一种方法所鉴定的特异性基因与偏分离的潜在机制存在一定的关联,比如在总的偏分离分析中,SPAG16基因与完整精子组装、精子细胞运动等有关,推测可能影响精子与卵子结合的能力[39];ADAM22基因编码整合素和金属钛酶结构域家族成员,参与调节细胞黏附和扩散以及抑制细胞增殖[40]。
前人的研究表明,编码胚胎发生的转录因子的基因与偏分离有关,如PAX5,它对中脑和小脑的发生至关重要[41],HOXD基因(HOXD1、HOXD3、HOXD4、HOXD8、HOXD9、HOXD12、HOXD13)对后肢神经支配来说是重要的[42],以及参与体细胞发生的DMRT2基因,DMRT1基因的功能缺失与否与人类的精子发生存在重要关联[43]。虽然本研究中没有显著的SNPs处在上述基因内,但是找到了一些可能引起基因组偏分离的标记。
3.3 单倍型偏分离分析与传统偏分离分析方法的比较传统的偏分离分析方法主要从基因型着手,通过对群体中父亲-母亲-后代三元单元基因传递的分析得到基因组偏分离景观。不论是基于贝叶斯模型的方法,还是基于传递不平衡方法或者是其他方法,偏分离的检出效力明显都会受到样本量大小的影响,小样本量所提供信息的位点更少,在检验中很容易产生假阳性的结果。本研究基于单倍型继承模式的偏分离分析方法从单倍型着手,利用的是后代标记中单倍型的继承信息。无论纯合子与否都可以获得单倍型分型结果,但是纯合的位点在传统的偏分离方法中不提供信息,另外与传统的方法相比,单倍型偏分离分析在使用基因型构建单倍型时利用了连锁不平衡的信息。所以,对比传统方法的分析结果(图 1、图 2)和单倍型方法的分析结果(图 3),前者的结果为分散的位点,且几乎所有的染色体上都存在显著的偏分离位点,可能存在假阳性位点。而后者的结果显示,位点之间相对更为连续,且只有个别染色体存在偏分离区域,结果稳定性更高。
4 结论本研究结合两种偏分离分析方法分析了杜洛克×二花脸三代杂交群体的60K基因型数据的非特异性和父母特异性的全基因组偏分离位点,并利用生物信息学工具分析了引起偏分离的候选基因。此外,还提出一种新的基于单倍型继承模式的偏分离分析方法,研究了父母传染色体的偏分离现象,并利用animalQTLdb分析了可能的原因。本研究为进一步解析猪群中的偏分离现象和探究其生物学机制以及其它家养动物的偏分离研究提供了基础资料和参考。
致谢: 感谢博士生导师黄路生院士在F2资源群体构建、基因型及表型性状测定及论文修订方面提供的帮助。
| [1] |
SILVER L M. The peculiar journey of a selfish chromosome: mouse t haplotypes and meiotic drive[J]. Trends Genet, 1993, 9(7): 250-254. DOI:10.1016/0168-9525(93)90090-5 |
| [2] |
CORBETT-DETIG R, MEDINA P, FRÉROT H, et al. Bulk pollen sequencing reveals rapid evolution of segregation distortion in the male germline of Arabidopsis hybrids[J]. Evol Lett, 2019, 3(1): 93-103. DOI:10.1002/evl3.96 |
| [3] |
CASELLAS J, GULARTE R J, FARBER C R, et al. Genome scans for transmission ratio distortion regions in mice[J]. Genetics, 2012, 191(1): 247-259. DOI:10.1534/genetics.111.135988 |
| [4] |
付忠军, 官玲, 张丕辉, 等. 玉米RIL群体SNP偏分离分析[J]. 农业生物技术学报, 2019, 27(9): 1542-1549. FU Z J, GUAN L, ZHANG P H, et al. Segregation distortion analysis on maize (Zea mays) RIL population by SNP[J]. Journal of Agricultural Biotechnology, 2019, 27(9): 1542-1549. (in Chinese) |
| [5] |
鲁宁宁, 张凯, 赵云雷, 等. 棉花F2群体及RIL群体SSR标记偏分离的比较剖析[J]. 分子植物育种, 2018, 16(19): 6372-6379. LU N N, ZHANG K, ZHAO Y L, et al. Comparative analysis of segregation distortion of SSR markers in F2 and RIL populations of cotton[J]. Molecular Plant Breeding, 2018, 16(19): 6372-6379. (in Chinese) |
| [6] |
郭继元, 王凤涛, 蔺瑞明, 等. 小麦SSR和SNP标记在Taichung29×白芒麦重组自交系群体中的偏分离分析[J]. 植物保护学报, 2018, 45(4): 795-803. GUO J Y, WANG F T, LIN R M, et al. Genetic analysis of segregation distortion of SNP and SSR markers in wheat RIL population from the Taichung29×Baimangmai[J]. Journal of Plant Protection, 2018, 45(4): 795-803. (in Chinese) |
| [7] |
LI G W, JIN J Y, ZHOU Y, et al. Genome-wide dissection of segregation distortion using multiple inter-subspecific crosses in rice[J]. Sci China Life Sci, 2019, 62(4): 507-516. DOI:10.1007/s11427-018-9452-8 |
| [8] |
ZIMMERING S. A genetic study of segregation in a translocation heterozygote in drosophila[J]. Genetics, 1955, 40(6): 809-825. DOI:10.1093/genetics/40.6.809 |
| [9] |
UNDERKOFFLER L A, MITCHELL L E, ABDULALI Z S, et al. Transmission ratio distortion in offspring of mouse heterozygous carriers of a (7.18) robertsonian translocation[J]. Genetics, 2005, 169(2): 843-848. DOI:10.1534/genetics.104.032755 |
| [10] |
CORBETT-DETIG R, JACOBS-PALMER E, HARTL D, et al. Direct gamete sequencing reveals no evidence for segregation distortion in house mouse hybrids[J]. PLoS One, 2015, 10(6): e0131933. DOI:10.1371/journal.pone.0131933 |
| [11] |
HOFF J L, DECKER J E, SCHNABEL R D, et al. Candidate lethal haplotypes and causal mutations in Angus cattle[J]. BMC Genomics, 2017, 18: 799. DOI:10.1186/s12864-017-4196-2 |
| [12] |
SPIELMAN R S, MCGINNIS R E, EWENS W J. Transmission test for linkage disequilibrium: the insulin gene region and insulin-dependent diabetes mellitus (IDDM)[J]. Am J Hum Genet, 1993, 52(3): 506-516. |
| [13] |
WEINBERG C R. Methods for detection of parent-of-origin effects in genetic studies of case-parents triads[J]. Am J Hum Genet, 1999, 65(1): 229-235. DOI:10.1086/302466 |
| [14] |
COULTON A, PRZEWIESLIK-ALLEN A M, BURRIDGE A J, et al. Segregation distortion: utilizing simulated genotyping data to evaluate statistical methods[J]. PLoS One, 2020, 15(2): e0228951. DOI:10.1371/journal.pone.0228951 |
| [15] |
ID-LAHOUCINE S, CÁNOVAS A, JATON C, et al. Implementation of Bayesian methods to identify SNP and haplotype regions with transmission ratio distortion across the whole genome: TRDscan v.1.0[J]. J Dairy Sci, 2019, 102(4): 3175-3188. DOI:10.3168/jds.2018-15296 |
| [16] |
HUANG D W, SHERMAN B T, LEMPICKI R A. Systematic and integrative analysis of large gene lists using DAVID bioinformatics resources[J]. Nat Protoc, 2009, 4(1): 44-57. DOI:10.1038/nprot.2008.211 |
| [17] |
DRUET T, GEORGES M. A hidden Markov model combining linkage and linkage disequilibrium information for haplotype reconstruction and quantitative trait locus fine mapping[J]. Genetics, 2010, 184(3): 789-798. DOI:10.1534/genetics.109.108431 |
| [18] |
DRUET T, GEORGES M. LINKPHASE3:an improved pedigree-based phasing algorithm robust to genotyping and map errors[J]. Bioinformatics, 2015, 31(10): 1677-1679. DOI:10.1093/bioinformatics/btu859 |
| [19] |
GUO Y, MAO H, REN J, et al. A linkage map of the porcine genome from a large-scale White Duroc×Erhualian resource population and evaluation of factors affecting recombination rates[J]. Anim Genet, 2009, 40(1): 47-52. DOI:10.1111/j.1365-2052.2008.01802.x |
| [20] |
PURCELL S, NEALE B, TODD-BROWN K, et al. PLINK: a tool set for whole-genome association and population-based linkage analyses[J]. Am J Hum Genet, 2007, 81(3): 559-575. DOI:10.1086/519795 |
| [21] |
WEINBERG C R, WILCOX A J, LIE R T, et al. A log-linear approach to case-parent-triad data: assessing effects of disease genes that act either directly or through maternal effects and that may be subject to parental imprinting[J]. Am J Hum Genet, 1998, 62(4): 969-978. DOI:10.1086/301802 |
| [22] |
HUANG L O, LABBE A, INFANTE-RIVARD C. Transmission ratio distortion: review of concept and implications for genetic association studies[J]. Hum Genet, 2013, 132(3): 245-263. DOI:10.1007/s00439-012-1257-0 |
| [23] |
BHATNAGAR S R, GREENWOOD C M T, LABBE A. Assessing transmission ratio distortion in extended families: a comparison of analysis methods[J]. BMC Proc, 2016, 10(Suppl 7): 197-202. |
| [24] |
CASELLAS J, FARBER C R, VERDUGO R A, et al. Segregation analysis of a sex ratio distortion locus in congenic mice[J]. J Hered, 2010, 101(3): 351-359. DOI:10.1093/jhered/esp118 |
| [25] |
HOANG K P, TEO T M, HO T X, et al. Mechanisms of sex determination and transmission ratio distortion in Aedes aegypti[J]. Parasit Vectors, 2016, 9: 49. DOI:10.1186/s13071-016-1331-x |
| [26] |
FISHMAN L, MCINTOSH M. Standard deviations: the biological bases of transmission ratio distortion[J]. Annu Rev Genet, 2019, 53: 347-372. DOI:10.1146/annurev-genet-112618-043905 |
| [27] |
DE VILLENA F P M, SAPIENZA C. Nonrandom segregation during meiosis: the unfairness of females[J]. Mamm Genome, 2001, 12(5): 331-339. DOI:10.1007/s003350040003 |
| [28] |
CARBALLO L, BATTISTOTTI A, TELTSCHER K, et al. Sperm morphology and evidence for sperm competition among parrots[J]. J Evol Biol, 2019, 32(8): 856-867. DOI:10.1111/jeb.13487 |
| [29] |
MAISONNEUVE E, SAUVÉE C, MAILLOUX A, et al. Intrauterine transfusions for maternofetal red-blood-cell incompatibility in monochorionic diamniotic twin pregnancy[J]. Ultrasound Obstet Gynecol, 2020, 55(6): 838-839. DOI:10.1002/uog.20418 |
| [30] |
李振, 雷伟, 普世皇, 等. 温度对水稻雌配子体基因型选择及其后代表型遗传分离的影响[J]. 云南农业大学学报, 2015, 30(4): 511-521. LI Z, LEI W, PU S H, et al. Female gametophyte genotype selection and its progenies phenotypic genetic segregation in rice under extreme temperature[J]. Journal of Yunnan Agricultural University, 2015, 30(4): 511-521. (in Chinese) |
| [31] |
BROOKS S A. Studies of genetic variation at the kit locus and white spotting patterns in the horse[D]. Lexington, Kentucky: University of Kentucky, 102.
|
| [32] |
ZHANG W, SUI Y, NI J, et al. Insights into the Nanog gene: a propeller for stemness in primitive stem cells[J]. Int J Biol Sci, 2016, 12(11): 1372-1381. DOI:10.7150/ijbs.16349 |
| [33] |
FURUKAWA K, KONDO T. Identification of the lamina-associated-polypeptide-2-binding domain of B-type lamin[J]. Eur J Biochem, 1998, 251(3): 729-733. DOI:10.1046/j.1432-1327.1998.2510729.x |
| [34] |
YOU Z Y, ISHIMI Y, MASAI H, et al. Roles of Mcm7 and Mcm4 subunits in the DNA helicase activity of the mouse Mcm4/6/7 complex[J]. J Biol Chem, 2002, 277(45): 42471-42479. DOI:10.1074/jbc.M205769200 |
| [35] |
赵一鸣, 杨刚, 由磊, 等. 敲低造血前B细胞白血病转录因子相互作用蛋白对胰腺癌细胞增殖、周期和凋亡的影响[J]. 中国医学科学院学报, 2020, 42(1): 7-15. ZHAO Y M, YANG G, YOU L, et al. Effect of Hematopoietic Pre-B-cell leukemia transcription factor interacting protein knockdown on proliferation, cell cycle and apoptosis in pancreatic cancer cells[J]. Acta Academiae Medicinae Sinica, 2020, 42(1): 7-15. (in Chinese) |
| [36] |
AKPINAR M, LESCHE M, FANOURGAKIS G, et al. TDRD6 mediates early steps of spliceosome maturation in primary spermatocytes[J]. PLoS Genet, 2017, 13(3): e1006660. DOI:10.1371/journal.pgen.1006660 |
| [37] |
GEORGIOU M, SINGH N, KANE T, et al. Photoreceptor structure in GNAT2-associated achromatopsia[J]. Invest Ophthalmol Vis Sci, 2020, 61(3): 40. DOI:10.1167/iovs.61.3.40 |
| [38] |
GILAD Y, LANCET D. Population differences in the human functional olfactory repertoire[J]. Mol Biol Evol, 2003, 20(3): 307-314. DOI:10.1093/molbev/msg013 |
| [39] |
NAGARKATTI-GUDE D R, JAIMEZ R, HENDERSON S C, et al. Spag16, an axonemal central apparatus gene, encodes a male germ cell nuclear speckle protein that regulates SPAG16 mRNA expression[J]. PLoS One, 2011, 6(5): e20625. DOI:10.1371/journal.pone.0020625 |
| [40] |
HIVERT B, MARIEN L, AGBAM K N, et al. ADAM22 and ADAM23 modulate the targeting of the Kv1 channel-associated protein LGI1 to the axon initial segment[J]. J Cell Sci, 2019, 132(2): jcs219774. |
| [41] |
PFEFFER P L, BOUCHARD M, BUSSLINGER M. Pax2 and homeodomain proteins cooperatively regulate a 435 bp enhancer of the mouse Pax5 gene at the midbrain-hindbrain boundary[J]. Development, 2000, 127(5): 1017-1028. DOI:10.1242/dev.127.5.1017 |
| [42] |
LI L L, WANG Y, ZHANG X Y, et al. Long non-coding RNA HOXD-AS1 in cancer[J]. Clin Chim Acta, 2018, 487: 197-201. DOI:10.1016/j.cca.2018.10.002 |
| [43] |
KIKKAWA T, SAKAYORI N, YUUKI H, et al. Dmrt genes participate in the development of Cajal-Retzius cells derived from the cortical hem in the telencephalon[J]. Dev Dyn, 2020, 249(6): 698-710. DOI:10.1002/dvdy.156 |
(编辑 郭云雁)



