氨基糖苷类药物(aminoglycosides,AGs)是一类作用于细菌核糖体,并抑制其蛋白合成的药物,如阿米卡星和庆大霉素等。在临床上,AG通常与β-内酰胺类药物联用来治疗革兰阴性菌感染。在畜牧业生产中,AG在抗菌和促生长等方面均具有重要作用。按照化学结构,氨基糖苷类药物可以分为4, 6-二取代-2-脱氧链胺(deoxystreptamine,DOS)、4, 5-二取代-2- DOS、单取代DOS和无DOS环。随着这类药物在临床上的广泛应用,其耐药性问题日益突出,耐药机制也日益复杂。AG主要的耐药机制:1)酶修饰/失活;2)结合位点的突变或修饰;3)细胞膜通透性下降;4)细胞质外排增加。而与临床相关最常见的耐药机制是修饰酶使药物失活,根据其分子机制,修饰酶又进一步分为乙酰基转移酶、核苷酸转移酶和磷酸转移酶,它们可通过细菌特异性DNA重组系统及可转移质粒在病原菌之间传播。近几年又发现了一类新的耐药决定因子,即16S rRNA甲基化酶(16S rRNA methyltransferases,16S-RMTases),其耐药机制是在16S rRNA A-位点内的特定核苷酸残基上添加来自SAM的CH3基团,甲基化的16S rRNA与某些氨基糖苷类药物的结合亲和力明显降低,从而导致对氨基糖苷类药物的耐药性显著提高[1]。
放线菌是自然界产生氨基糖苷类化合物的细菌,如链霉菌属和小单孢菌。这些细菌含有内源性16S-RMTases基因,天生对氨基糖苷类药物具有耐药性。虽然产生氨基糖苷类化合物的细菌可以通过16S-RMTases来保护自己,但之前在引起人或动物感染性疾病的病原菌中尚未发现这一特性[2]。然而在2003年,日本报道称铜绿假单胞菌能够产获得性16S-RMTases,使菌株能够对氨基糖苷类药物产生广泛且高水平耐药性[3]。需要引起高度重视的是,NDM-1金属β-内酰胺酶(metallo-β-lactamase,MBL)和16S-RMTases在肠杆菌中常常偶联存在,近年来携带这两种酶的病原菌在全球范围内迅速传播,对人类健康造成了严重威胁。
本文对引起氨基糖苷类药物耐药的16S-RMTases进行综述,并着重讨论其作用机制、基因环境、流行病学及与其偶联的抗性决定簇,同时介绍了如何检测产16S-RMTases阳性菌的方法。
1 内源性16S-RMTases20世纪80年代,研究者在部分产氨基糖苷类化合物的放线菌中发现了16S-RMTases基因。这些放线菌对氨基糖苷类化合物所表现出的高水平的耐药性,可以保护它们不受自身产生的氨基糖苷类化合物的伤害。近年来,随着遗传分析技术的发展,放线菌中与氨基糖苷类化合物合成有关的基因簇得到了很大的挖掘,遗传分析结果表明,16S-RMTases基因大部分位于放线菌氨基糖苷的生物合成基因簇中。例如,在棘孢小单孢菌ATCC 15835的庆大霉素生物合成基因簇中发现了16S-RMTasess基因——grmA和grmO(GenBank登录号:AY524043.1);同时,在链霉菌菌株DSM 44523T的安普霉素生物合成基因簇中又发现了另一个16S-RMTases基因——kamB(GenBank登录号:AJ875019.1)。在各种微生物竞争激烈的生存环境中,合成基因和耐药基因偶联给放线菌带来了一定的生存优势。
根据在16S rRNA的A-位点上修饰的核苷酸位置不同,内源性16S-RMTases主要分为两类:N7-G1405 16S-RMTases和N1-A1408 16S-RMTases。KgmB、GrmO和Sgm等N7-G1405 16S-RMTases修饰了16S rRNA内G1405核苷酸的N7位,并使菌株对4, 6-二取代-2-DOS产生抗性,但对4, 5-二取代-2-DOS、安普霉素和链霉素不产生抗性。Kmr和KamB等N1-A1408 16S-RMTases修饰了16S rRNA内A1408核苷酸的N1位,并使菌株具有对多种氨基糖苷类药物的广泛耐药性。内源性N1-A1408 16S-RMTases目前仅在链霉菌属中发现,而内源性N7-G1405 16S-RMTases在链霉菌属和小单孢菌属中均有发现。
2 获得性16S-RMTases 2.1 获得性16S-RMTases的发现日本学者于1997年从唾液中分离到1株铜绿假单胞菌,该菌株对多种氨基糖苷类药物表现出极强的耐药性。进一步研究发现,介导该菌株对氨基糖苷类药物高度耐药的是1种16S-RMTases,其后被命名为RmtA[3]。Galimand等[4]在2003年报道了1株临床分离的肺炎克雷伯菌,该菌株对临床上的氨基糖苷类药物表现出高水平耐药,研究发现,是armA造成了对氨基糖苷类药物的耐药。随后进一步研究发现,armA可能与之前检测到的pCTX-M3质粒中的RMTase基因相同。armA和rmtA基因揭示了外源获得性16S-RMTases的出现,这种酶使致病性革兰阴性菌对氨基糖苷类药物产生广泛且高水平的耐药性。Liou等[5]详细描述了ArmA的酶功能,并发现ArmA属于N7-G1405 16S-RMTases的一员。基于氨基酸序列的相似性,RmtA也被认为是N7-G1405 16S-RMTases的一员。
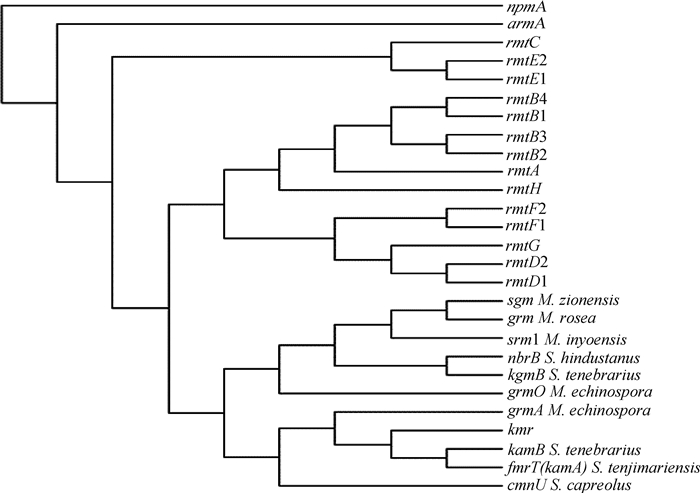
RmtB首次发现于日本的黏质沙雷菌中,随后在墨西哥的阴沟杆菌中鉴定出RmtB2(参考序列号:WP_063854854.1),其与RmtB在41位和229位氨基酸存在差异;而美国的大肠杆菌临床分离株鉴定到的RmtB3(参考序列号:WP_048266647.1),与RmtB相比存在7个氨基酸差异;尼泊尔的奇异变形菌临床分离株鉴定到的RmtB4(参考序列号:WP_032634099.1),与RmtB相比存在6个氨基酸差异[6]。2006年,在日本的奇异变形杆菌的临床分离株中首次发现了RmtC[7]。2005年,RmtD(现在被称为RmtD1)首次发现于巴西的1株铜绿假单胞菌临床菌株[8]。之后阿根廷报告了1株产气肠杆菌临床分离株能够产RmtD2,其与RmtD相比存在9个氨基酸差异[9]。2012年,在美国的牛源共生大肠杆菌菌株中首次发现了RmtE[10],随后从中国食品动物源大肠杆菌中鉴定出RmtE2[11],其与RmtE相比存在1个氨基酸的差异。2011年,从法国分离的肺炎克雷伯菌鉴定出RmtF[12];2017年,尼泊尔报道的铜绿假单胞菌产生的RmtF2与RmtF相比有1个氨基酸差异[13]。2013年,巴西圣保罗州医院从骨中分离的肺炎克雷伯菌首次鉴定出RmtG[14]。同年在美国,研究者从1名男性士兵分离到的肺炎克雷伯菌中首次鉴定出RmtH[15]。迄今为止,在病原菌中发现的16S-RMTases基因大多数位于可转移质粒上,或者是与转座子等细菌特异性DNA重组系统相关(表 1)。16S-RMTases的进化树见图 1。
|
|
表 1 首次分离获得性16S-RMTases的信息 Table 1 The information of first isolation of acquired 16S-RMTases |
|
图 1 N7-G1405 16S-RMTases和N1-A1408 16S-RMTases的进化树 Fig. 1 Evolutionary tree of N7-G1405 16S-RMTases and N1-A1408 16S-RMTases |
截至目前,报道了9种N7-G1405 16S-RMTases:ArmA、RmtA、RmtB、RmtC、RmtD、RmtE、RmtF、RmtG和RmtH。这些酶能使菌株对4, 6-二取代-2DOS具有专一耐药性(表 2)。虽然armA、rmtB和rmtC编码的氨基酸序列之间的相似性最高仅30%,但RmtB、RmtC和ArmA的三维结构相似性却很高[16]。在体外,用纯化的蛋白质探究RmtB、RmtC和ArmA的酶功能,结果证明,这些16S-RMTases可以把SAM的甲基作为辅助因子,添加到成熟的30S核糖体亚单位16S rRNA内的G1405的N7位上,该位点的甲基化对4, 6-二取代-2-DOS的III环3′端造成空间位阻[17],但该酶无法将甲基转移到裸露的16S rRNA分子或成熟的70S核糖体上,表明酶识别或修饰靶位需要16S rRNA和核糖体蛋白组成的三级结构才能进行[18]。
|
|
表 2 N7-G1405和N1-A1408 16S-RMTases氨基糖苷类药物耐药谱 Table 2 Aminoglycoside resistance profile provided by N7-G1405 and N1-A1408 16S-RMTases |
N7-G1405 16S-RMTases使菌株对4, 6-二取代-2-DOS耐药,但对4, 5-二取代-2-DOS或其他氨基糖苷类药物(如链霉素)不能产生耐药性,这一特性可以从氨基糖苷类药物和16S rRNA之间的结合模式来解释。G1405残基的N7位最接近4, 6-二取代-2-DOS III环中3′端,在N7位引入CH3残基可能会导致与III环侧链的空间碰撞或静电排斥,从而降低氨基糖苷类药物结合的亲和力,导致对4, 6-二取代-2-DOS耐药。与此相反,4, 5-二取代-2-DOS的III环和IV环通常距离G1405的N7位置较远,16S-RMTases引入CH3不会干扰它们之间的结合,因此不会对4, 5-二取代-2-DOS耐药。其他与16S rRNA结合但未与G1405位点发生相互作用的氨基糖苷类化合物,仍可通过N7-G1405位点与16S rRNA结合并表现出正常活性[19]。
与N7-G1405 16S-RMTases相比,获得性N1-A1408 16S-RMTases在临床分离的病原菌中很少被发现。2007年,首次从大肠杆菌临床菌株ARS3中发现了NpmA,该酶是目前唯一的获得性N1-A1408 16S-RMTases[20]。在30S核糖体亚基中,NpmA修饰16S rRNA的N1-A1408和N1-G1408位点,其中N1-A1408位点靠近4, 5-二取代-2-DOS、4, 6-二取代-2-DOS和安普霉素的I环[20]。虽然氨基糖苷类药物的结构差异性很大,但现有氨基糖苷类药物的I环的空间位置是一样的。因此相对N7-G1405 16S-RMTases而言,N1-A1408 16S-RMTases NpmA可赋予细菌更广泛的氨基糖苷类药物耐药性(表 2)。Kanazawa等[21]指出,在N1-A1408位点引入CH3基团可以阻止A1408与氨基糖苷类药物I环氢键的形成,特别是N1正电原子可以阻止带有NH3+的I环与A1408结合。试验还模拟了I环中带有6′-OH基团的氨基糖苷类药物(如巴龙霉素)和N1-A1408之间的结合,结果表明这种氨基糖苷类药物对NpmA阳性菌仍然有药物效应。所以I环中带有6′-OH基团的4, 5-二取代-2-DOS可以作为设计下一代氨基糖苷类药物的良好靶点,用于治疗16S-RMTases阳性菌引发的感染[21]。
3 16S-RMTases的基因环境 3.1 armA从多种来源中分离得到的armA,其周围基因都较为保守。armA位于ISCR1的下游,ISCR1之后是由dhfrA12、aadA2和sul1组成的Ι类整合子,armA的下游经常发现trpA、mel和mph等耐药基因。Galimand等[22]报道,armA两侧是IS6的两个拷贝,定位于复合转座子Tn1548中,比较容易嵌入另一个DNA靶位点,这对于armA的传播意义重大,可能是armA成为最常见的16S-RMTases基因的原因。
3.2 rmtArmtA位于铜绿假单胞菌中1个6.2 kb的遗传区域内,该区域的两侧各分布着1个长为262 bp的κ-γ可移动元件,该元件是也曾被发现存在于假单胞菌属携带汞耐药基因的复合转座子Tn5041中。据此推断,包含rmtA和两个κ-γ元件的6.2 kb遗传区域可能可以嵌入Tn5041中。
3.3 rmtB几乎所有rmtB的上游都有携带blaTEM-1的Tn3转座子,而rmtB的下游区域的变异性较大,但通常与喹诺酮外排转运蛋白基因qepA和ISCR3插入序列相关。近期研究表明,在rmtB的上游还存在IS26插入序列,该插入序列位于Tn3转座子之前。与此同时,还在rmtB的下游区域发现了编码Na+/K+转运蛋白和401-氨基酸转座酶的基因,该转座酶基因属于ISL3超家族,这可能与rmtB的水平传播有关[23]。
3.4 rmtC在奇异变形杆菌中发现的rmtC位于ISEcp1元件附近,该元件含有转座酶基因tnpA,主要参与邻近rmtC的转移,并为其表达提供启动子。ISEcp1元件通常与blaCTX-M和blaCMY等β-内酰胺酶基因偶联,有研究发现ISEcp1元件与肠杆菌中blaCTX-M基因的传播有关。在几个携带NDM-1的分离株中也发现了RmtC,所以rmtC很可能通过ISEcp1元件的转座活性在肠杆菌中广泛传播[24]。
3.5 rmtD发现于巴西铜绿假单胞菌[25]的rmtD下游是ISCR14元件,以及由qacEΔ1和sul1组成的3′保守片段。其上游还有orfA、ΔgroEL和另一个ISCR14元件,故rmtD与两个同向重复的ISCR14组合。在肺炎克雷伯菌中,rmtD附近的区域与铜绿假单胞菌相同,但两端的ISCR14都被IS26截短。发现于阿根廷的rmtD2与巴西的rmtD的遗传背景相似,但rmtD2上游的ΔgroEL的5′尾大部分被切除。Tijet等[9]推测,rmtD和rmtD2的两端是由ISCR14组成的不同基因组装的,而不是由同一基因共同组装。
3.6 rmtE和rmtFrmtE位于Ι类整合子中,定位于携带blaCMY-2基因的质粒上,质粒序列显示,rmtE可与ISCR20-like元件结合,也可与IS1294-like插入序列结合。这些转座元件通常与16S-RMTases等耐药基因相关,如rmtF的两侧有ISCR5-like和ISCR14元件[9, 12, 26],这一现象表明,这些元件在rmtE的启动过程中可能起到一定的作用。
现有研究表明,rmtF位于blaNDM下游的Ι类整合子中。
3.7 rmtG和rmtHrmtG位于ISVsa3下游,但该序列可能是携带rmtG的组件的插入位点,而不是rmtG本身所携带的。有研究证明,rmtG由不同的Inc质粒(N,A/C,B/O)携带,通常位于具有其他耐药基因的转座子中。
rmtH的侧翼是两个拷贝的ISCR2,该元件可能与rmtH的启动有关。
3.8 npmAnpmA位于1个9.1 kb大小的遗传区域中,其两侧是两个拷贝的IS26,位于可转移的IncF质粒上,该元件可能与npmA的启动有关。遗传区域的两侧可能是转座元件,该元件与数据库中各种多重耐药质粒的部分序列有着很大的序列同源性。
4 16S-RMTases的流行病学携带16S-RMTases基因的可移动遗传元件已经嵌入到各种可转移质粒中,并且拥有广泛的质粒宿主,例如IncN和IncA/C等。这些质粒除了携带16S-RMTases基因之外,还含有多种其他耐药基因。研究表明,肠杆菌通常能共同携带NDM-1 MBL和16S-RMTases (RmtB、RmtC和ArmA)[27]。目前碳青霉烯类药物是治疗革兰阴性菌所致感染性疾病的重要药物,而细菌通过碳青霉烯酶水解β-内酰胺类药物,给临床治疗带来很大的困难。需要引起重视的是,这两种抗性决定簇通常位于肠杆菌的同一种接合质粒上[28],而这些质粒的快速转移大大加快了多重耐药基因的传播。
4.1 armAarmA是最常见的16S-RMTases基因之一,广泛分布于肠杆菌和鲍曼不动杆菌中。在肠杆菌中,它主要分布在与公共卫生相关的病原菌中,如肺炎克雷伯菌。此外,在食源性和腹泻疾病的相关致病菌中也有armA的相关报道,如肠沙门菌和福氏志贺菌[22]。研究人员推测,食用动物中可能存在armA的储存库。例如,在中国的鸡源大肠杆菌和留尼旺岛的鸡源肠沙门菌分离株中都鉴定出armA[29]。不过,armA在食用动物中的传播程度目前还不清楚。
在携带碳青霉烯酶基因blaOXA-23的鲍曼不动杆菌耐药菌株中,经常能鉴定出armA,但两个基因位于不同的质粒上[30]。2007年,美国在多重耐药鲍曼不动杆菌(multidrug-resistant A. baumannii,MDRA)中首次发现了blaOXA-23和armA的偶联[31]。此后,中国和印度有MDRA联合生产OXA-23和ArmA的少数报道,有些MDRA还可以生产NDM-1[32-33]。最近,希腊的一项研究收集了耐碳青霉烯类鲍曼不动杆菌的相关数据,其中 armA的流行率高达93.7%,且其阳性分离株绝大多数(98.5%)都携带blaOXA-23[34]。另一个armA流行率较高的是产NDM型碳青霉烯酶肠杆菌,许多携带NDM-1或其突变体的肠杆菌对4, 6-二取代-2-DOS类药物也具有耐药性[35]。在肺炎克雷伯菌中研究者还发现blaNDM和blaOXA-48-like两个基因同时与两个16S-RMTases基因(其中包含armA)偶联,并且这些基因且位于同一质粒上[36]。在英国进行的一项全国性调查显示,16S-RMTases阳性菌大多数(93.4%)也携带碳青霉烯酶基因,其中以blaNDM最为常见(83.1%)[37]。对携带NDM的质粒进行分析表明,blaNDM常常与armA或其他16S-RMTases基因(特别是rmtB、rmtC和 rmtF)位于同一质粒上,而且有些质粒还携带氟喹诺酮耐药基因,例如qnrB1[38]。携带这些多重耐药质粒会同时对大多数药物产生耐药性,包括碳青霉烯类、氨基糖苷类和氟喹诺酮类抗菌药物,而这三类药物对临床治疗革兰阴性菌感染具有重要意义。
其他耐药基因也有与armA偶联的报道。在韩国报道了blaIMP-1和armA偶联的铜绿假单胞菌;中国报道了blaKPC-2和armA偶联的肺炎克雷伯菌和其他肠杆菌[39];在质粒pCTX-M3和肺炎克雷伯菌质粒pIP1204上鉴定出了armA和blaCTX-M-3[22];土耳其还报道了黏质葡萄球菌中blaOXA-48与armA的偶联[40];2015年,我国首次在armA阳性的分离株中同时检出blaTEM、blaSHV和blaCTX-M-3[41]。
4.2 rmtArmtA在东亚(如日本和韩国)的铜绿假单胞菌中报道较多[42-43],但最近在其他地区也有关于rmtA的相关报道。伊朗西北部在大肠杆菌中鉴定出rmtA,但流行率较低,仅为0.91%[44];印度北部在超广谱β-内酰胺酶(extended-spectrum β-lactamases,ESBLs)阳性的大肠杆菌和肺炎克雷伯菌中分离出rmtA[45];最近由我国湘雅二院收集的多重耐药鲍曼不动菌的rmtA分离率竟达到了94.12%,而且还携带了其他多种耐药基因,包括Ι类整合子和编码碳青霉烯酶、ESBLs和其他16S-RMTases的基因[46]。Ι类整合子在耐药基因的水平传播中具有重要意义,多种耐药基因与整合子偶联使细菌能够对抗多种临床治疗方案。如果这种“超级细菌”进一步传播,其后果将不堪设想。
4.3 rmtBrmtB是继armA之后编码16S-RMTases最常见的基因,主要分布在东亚、欧洲、南北美洲和大洋洲的肠杆菌中[43, 47-53]。携带rmtB的细菌不仅能从人的临床环境中分离出来,还从可以家畜和宠物中分离出来,这表明该耐药基因可能会在人和动物之间相互传播[54]。在畜牧业中,rmtB似乎比armA更常见。rmtB在猪、农场工人及其环境[54]和宠物[55]的大肠杆菌中检出率较高。近年来国内有学者发现,rmtB能够在食品性动物中持续传播[56]。
肠杆菌中CTX-M型ESBLs基因,特别是blaCTX-M-15,是一种全球流行的ESBLs基因,经常与armA/rmtB同时检出[26]。有研究表明,rmtB和blaCTX-M-15位于同一质粒上[23]。研究者还发现rmtB经常与另一CTX-M型(CTX-M-9和CTX-M-14)基因偶联[57]。rmtB在碳青霉烯酶阳性的肠杆菌的分布尤为普遍,如中国报道了blaKPC-2和rmtB偶联的肺炎克雷伯菌分离株[58];安哥拉报道了rmtB和blaNDM偶联的大肠杆菌和肺炎克雷伯菌[59]。2015年,我国研究者首次从牛乳房炎大肠杆菌中分离出rmtB,且所有阳性菌株均携带blaTEM-1和blaCTX-M-15[60];同年,我国还报道了一株同时携带rmtB、喹诺酮类耐药基因(gyrA和parC)和多种β-内酰胺酶基因(blaCTX-M-14、blaTEM-1和blaOXA-1)的黏质链球菌[61];最近,国内又报道了一株耐碳青霉烯类超强毒力肺炎克雷伯菌(carbapenem-resistant hypervirulent Klebsiella pneumoniae,CR-hvKP),该菌株同时携带编码碳青霉烯酶、ESBLs和16S-RMTases的基因以及喹诺酮类耐药基因[62]。拥有这些多重耐药质粒的细菌会同时对大多数抗菌药物产生耐药性,给临床治疗带来极大的困难。尽管我国研究者们在越来越多的物种中检测出16S-RMTases,但目前还缺乏全国范围内的流行病学调查,很多潜在的携带16S-RMTases的物种及其流行地区尚待进一步明确。
4.4 rmtC最初rmtC仅在日本和澳大利亚的奇异变形杆菌中检出[7, 63]。随后在英国发现了13株显著表达rmtC的肠炎沙门菌[64],这13例肠炎沙门菌感染的患者中,4例患者存在印度旅游史。此后不久,开始出现rmtC与blaNDM-1偶联的报道。在英国、印度[65]和新西兰[66]的大肠杆菌分离株中,研究者发现blaNDM-1和 rmtC有显著相关性。其他致病性革兰阴性菌中也有rmtC的相关报道,如安哥拉[59]、伊朗[44]的肺炎克雷伯菌临床分离株和巴西的鲍曼不动杆菌[66]。
大量数据表明,rmtC已经在多药耐药(multidrug-resistant,MDR)和广泛耐药(extensively drug-resistant,XDR)肠杆菌,尤其是在携带NDM型碳青霉烯酶的肠杆菌之间传播。一项来自英国和爱尔兰的全国性调查结果显示,在大肠杆菌、弗氏柠檬酸杆菌、阴沟肠杆菌、肺炎克雷伯菌等多种革兰阴性菌中检测出rmtC,其分离率为19.2%,其中一些菌株除blaNDM之外还携带blaOXA-48-like[37]。
4.5 rmtD和rmtD2rmtD和rmtD2分布于南美国家分离的铜绿假单胞菌和肠杆菌中,巴西、智利和阿根廷[9, 48, 68]都有rmtD和rmtD2的相关报道。而最近缅甸[69]和波兰[70]分别报道的携带rmtD3的铜绿假单胞菌临床分离株,表明rmtD开始向南美洲以外地区传播。blaSPM-1是一种与rmtD偶联的MBL基因[8],这一现象在巴西的铜绿假单胞菌临床分离株中得到了证实[71]。而且在来自河流的铜绿假单胞菌中也检出了blaSPM-1和rmtD,这表明它可能可以通过环境进行传播[72]。与此同时,在肺炎克雷伯菌和其他肠杆菌中也发现了rmtD,表明该基因具有跨物种迁移的能力[9, 48]。从巴西分离出的肺炎克雷伯菌中,发现了rmtD与blaKPC-2的偶联[14],与产NDM-1的肠杆菌不同,rmtD和blaKPC位于不同的质粒上。研究人员对携带rmtD的质粒序列进行的分析表明,质粒中还同时存在ESBL类(blaTEM-1、blaCTX-M-8)、修饰酶类[aac(6′)-Ⅰb、ant(3′′)-Ⅰa)]和磺胺类(sul1)耐药基因[73]。在印度北部的大肠杆菌[74]中发现了rmtD与ESBL类(blaCTX-M-15)和碳青霉烯类(blaNDM、blaOXA-48)的共携带。最近我国研究者在禽源沙门菌中还发现了rmtD与ESBL类(blaCTX-M)、修饰酶类[aac(6′)-Ib-cr]和PMQR(qepA、qnrB、qnrD、oqxAB)的偶联[75]。
4.6 rmtE和rmtF关于rmtE (rmtE1~rmtE3)的报道非常有限,其中3例来自美国(均为大肠杆菌)[10, 76-77],1例来自中国的大肠杆菌[11],还有最近1例来自缅甸的铜绿假单胞菌[78]。
rmtF是继armA、rmtB、rmtC之后最流行的16S-RMTases基因。2011年,首次发现于法国的肺炎克雷伯菌分离株中[12],随后陆续在印度[65]、英国[37, 65]、澳大利亚[79]、埃及[80]和爱尔兰[37]的肠杆菌和尼泊尔[13]的铜绿假单胞菌鉴定出rmtF。rmtF与blaNDM的关系非常密切,比迄今为止已经发现的任何其他的16S-RMTases基因都有更强的相关性。第一例rmtF是从blaNDM阳性的肺炎克雷伯菌分离株中发现的[80],并且来自印度[65]和埃及[80]的数据显示,blaNDM阳性菌株中rmtF的检出率更高。此外也有rmtF与其他基因偶联的情况,在土耳其曾报道了阴沟杆菌中rmtF和blaOXA-48的偶联[40]。
4.7 rmtG和rmtH最初在巴西的blaKPC-2和blaCTX-M偶联的肺炎克雷伯菌临床分离株中发现了rmtG[14],随后在智利[81]、美国[14]和瑞士[82]出现rmtG的相关报道,均为肺炎克雷伯菌。但之后在铜绿假单胞菌和产气克雷伯菌临床分离株中也报道了rmtG[83]。最近,从印度东北部[74]的大肠杆菌中鉴定出rmtG,并发现其与blaCTX-M-15、blaNDM、blaOXA-48偶联。
rmtH的来源非常有限,首次分离自美国的肺炎克雷伯菌[7]。另一例也是肺炎克雷伯菌,分离自黎巴嫩的一名新生儿[84]。最近在印度东北部发现了rmtH与blaCTX-M-15、blaNDM、blaOXA-48偶联的大肠杆菌[74]。
4.8 npmA自2007年首次报道npmA以来,与armA、rmtB和rmtC等N7-G1405 MTases基因相比,很少有关于npmA的报道[44]。npmA首次分离自日本的大肠杆菌[20],随后在沙特阿拉伯、伊朗、印度东北部的肠杆菌中鉴定到npmA[44, 74]。值得注意的是,最近在艰难梭菌中发现了NpmA2,与NpmA相比有1个氨基酸的差异[85]。
4.9 两类16S-RMTases流行率比较armA、rmtB、rmtC等N7-G1405 16S-RMTases基因广泛存在,而有关npmA N1-A1408 16S-RMTases基因的报道却非常有限。为了探索这一差异,一些研究人员研究了携带16S-RMTases的细菌的适应性代价与流行率之间的关系。
在大肠杆菌中,16S-RMTases能够修饰G1405或A1408位点,这两个位点接近内源性甲基化残基C1402位(由内源性16S-RMTases RsmI修饰)和C1407位(由内源性16S-RMTases RsmF修饰)。G1405和A1408的甲基化可能会影响C1402和C1407位点的正常内源性甲基化过程,从而降低最佳的核糖体功能。ArmA引起的G1405甲基化阻碍了C1402位点的甲基化,而RmtC和NpmA则阻碍C1407的甲基化[86]。不同内源性甲基化缺陷对细胞的影响不同:C1407甲基化的缺陷不会影响细胞生长,而C1402甲基化缺陷会导致不同程度的生长障碍[87]。
armA已见诸于世界范围内的许多细菌种属,而npmA则很少被报道,这与内源性甲基化缺陷对生长的影响结果大相径庭。可能的原因是armA是Tn1548功能转座子的一部分[88],由pIP1204接合质粒携带[4],与那些β-内酰胺类和氟喹诺酮类耐药基因偶联[89],而这两类药物被广泛应用,这种基因环境有利于armA在抗生素的选择压力下传播。而npmA位于没有其他耐药基因的质粒上[90],由于基因环境缺乏共抗性基因,导致携带该基因的细菌在抗生素的压力下被淘汰,从而限制了其传播。不过,npmA的表达不会影响宿主的生长,因此该基因在没有抗生素选择压力的情况下得以保留。
5 16S-RMTases阳性菌的检测 5.1 N7-G1405 16S-RMTases的检测开发实用的16S-RMTases病原菌检测技术,有助于临床微生物学实验室对其进行流行病学研究和快速鉴定。N7-G1405 16S-RMTases阳性菌的特点是对4, 6-二取代-2-DOS组具有高度耐药性(表 2)。阿米卡星和庆大霉素的高MIC值(≥128 μg·mL-1)是检测N7-G1405 16S-RMTases阳性菌的良好指标[91]。所以检测N7-G1405 16S-RMTases阳性菌时,首先通过常规药敏试验,确定菌株同时对阿米卡星和庆大霉素耐药,然后进行高MIC范围的药敏试验。可以采用纸片扩散法筛选,N7-G1405 16S-RMTases阳性菌在阿米卡星和庆大霉素中几乎没有抑菌圈。到目前为止,这种检测方法应用于肠杆菌和部分非发酵革兰阴性杆菌(如铜绿假单胞菌和鲍曼不动杆菌)的试验已被证明有效。对4, 5-二取代-2-DOS和链霉素的敏感性不能作为检测N7-G1405 16S-RMTases阳性菌的指标,因为N7-G1405 16S-RMTases对这类氨基糖苷类药物没有明显的抗性,而氨基糖苷磷酸化酶和核苷酸转移酶可以使菌株对这些药物产生抗性。所以先通过药敏试验对N7-G1405 16S-RMTases阳性菌进行初期筛选,然后进行PCR扩增相应耐药基因,可能是目前唯一检测N7-G1405 16S-RMTases基因的方法。
5.2 N1-A1408 16S-RMTases的检测目前关于该酶阳性菌的报道较少,尚未建立实用的N1-A1408 16S-RMTases阳性菌检测方法。单凭常规药敏试验结果,很难找到N1A1408 16S-RMTases阳性菌,因为携带N1-A1408 16S-RMTases的菌株几乎没有任何显著特征,对临床使用的氨基糖苷类抗生素的耐药谱与多种氨基糖苷类修饰酶联合产生的耐药谱相似。N1-A1408 16S-RMTases阳性菌可能对单取代DOS和安普霉素具有很高的耐药性,实际上,在对安普霉素耐药菌的检测中,已经鉴定出一株产N1-A1408 16S-RMTases(即NpmA)的大肠杆菌临床分离株(ARS3)。对安普霉素耐药还存在另一种耐药机制,即aac(3)-IV修饰酶,这是大肠杆菌中对安普霉素耐药最普遍的基因[92],因而对安普霉素的敏感性不能作为检测N1-A1408 16S-RMTases基因的指标。因此,有必要进一步收集N1-A1408 16S-RMTases非克隆阳性菌株并进行大量检测,以建立一种实用可行的N1-A1408 16S-RMTases阳性菌检测方法。
6 展望 6.1 16S-RMTases在革兰阳性菌中的潜在转移革兰阴性菌的多重耐药已成为严重威胁人类健康的问题之一。引起氨基糖苷类药物耐药的遗传因素中,16S-RMTases阳性菌对氨基糖苷类药物的耐药性高于目前所知的其他耐药决定因素,且范围更广,是导致肠杆菌、铜绿假单胞菌和鲍曼不动杆菌对氨基糖苷类抗生素耐药的主要因素。到目前为止,仅在革兰阴性致病菌中鉴定出了16S-RMTases,而对革兰阳性致病菌如葡萄球菌属、链球菌和肠球菌等尚未见报道。然而已有研究表明,在天然启动子的作用下,革兰阴性病原菌的16S-RMTases在异源革兰阳性菌、枯草芽胞杆菌和金黄色葡萄球菌中具有高水平的氨基糖苷类药物耐药性[17]。因此16S-RMTases阳性菌的出现和传播应该引起人们的密切关注。
6.2 新型抗16S-RMTases阳性菌强效剂的研制针对16S-RMTases所带来的问题,开发能特异性阻断16S-RMTases活性的有效抑制剂是一种可行的方法。因此,有必要通过X射线晶体学分析16S-RMTases三维结构,了解该酶与其底物之间的相互作用方式。到目前为止,从放线菌和病原菌中分离出的N7-G1405 16S-RMTases (Sgm、ArmA、RmtB)和N1-A1408 16S-RMTases (KamB、NpmA)的结构已被解析并保存在蛋白质数据库中。尽管放线菌和病原菌之间的氨基酸残基差异很大(相似性不到30%),但它们的三维结构非常相似,活性中心的关键氨基酸残基具有很高的保守性。9个获得性N7-G1405 16S-RMTases在氨基酸序列水平上有相当大的相似性,一般至少有6个保守性氨基酸残基,初步推测这些保守性残基形成了酶的活性中心,在酶反应中发挥重要作用。其中一些氨基酸残基后来被发现位于甲基转移所涉及的酶的催化基团中[93-94]。包括NpmA在内的N1-A1408 16S-RMTases之间也存在保守的氨基酸残基,但N7-G1405和N1-A1408 16S-RMTases之间的氨基酸序列没有太大的相似性。进一步明确16S-RMTases的精细三维结构将为开发有效的特异性抑制剂奠定基础,还可用于建立临床和兽医微生物学实验室检测16S-RMTases阳性菌的检测方法。
6.3 16S-RMTases阳性菌的监测如上所述,产16S-RMTases的革兰阴性病原菌已经在世界范围内传播,它们通过获得blaNDM-1和blaCTX-M-15等各种耐药基因表现出多重耐药表型。此外,沙门菌和志贺菌属等高致病性微生物已经携带16S-RMTases基因,并且已经从家畜和宠物的养殖环境甚至食物分离得到。因此,在全国范围的人和动物监测计划下,继续监测16S-RMTases阳性菌的传播趋势是非常重要的。
7 小结自21世纪初首次发现介导高水平氨基糖苷类药物耐药性的产16S-RMTases革兰阴性菌以来,获得性16S-RMTases已经在全球范围内广泛传播,且出现了新的16S-RMTases变体。16S-RMTases阳性菌经常携带其他临床相关的耐药基因,包括碳青霉烯酶基因(如blaNDM和blaKPC)、ESBLs基因和大肠杆菌素耐药基因(mcr),使得其治疗变得更为复杂。因此,获得性16S-RMTases在革兰阴性病原菌中的出现和传播必须引起高度重视。
| [1] |
PFISTER P, HOBBIE S, VICENS Q, et al. The molecular basis for A-site mutations conferring aminoglycoside resistance: relationship between ribosomal susceptibility and X-ray crystal structures[J]. ChemBioChem, 2003, 4(10): 1078-1088. DOI:10.1002/cbic.200300657 |
| [2] |
WACHINO J I, ARAKAWA Y. Exogenously acquired 16S rRNA methyltransferases found in aminoglycoside-resistant pathogenic Gram-negative bacteria: an update[J]. Drug Resist Updat, 2012, 15(3): 133-148. DOI:10.1016/j.drup.2012.05.001 |
| [3] |
YOKOYAMA K, DOI Y, YAMANE K, et al. Acquisition of 16S rRNA methylase gene in Pseudomonas aeruginosa[J]. Lancet, 2003, 362(9399): 1888-1893. DOI:10.1016/S0140-6736(03)14959-8 |
| [4] |
GALIMAND M, COURVALIN P, LAMBERT T. Plasmid-mediated high-level resistance to aminoglycosides in Enterobacteriaceae due to 16S rRNA methylation[J]. Antimicrob Agents Chemother, 2003, 47(8): 2565-2571. DOI:10.1128/AAC.47.8.2565-2571.2003 |
| [5] |
LIOU G F, YOSHIZAWA S, COURVALIN P, et al. Aminoglycoside resistance by ArmA-mediated ribosomal 16S methylation in human bacterial pathogens[J]. J Mol Biol, 2006, 359(2): 358-364. DOI:10.1016/j.jmb.2006.03.038 |
| [6] |
DOI Y, YOKOYAMA K, YAMANE K, et al. Plasmid-mediated 16S rRNA methylase in Serratia marcescens conferring high-level resistance to aminoglycosides[J]. Antimicrob Agents Chemother, 2004, 48(2): 491-496. DOI:10.1128/AAC.48.2.491-496.2004 |
| [7] |
WACHINO J I, YAMANE K, SHIBAYAMA K, et al. Novel plasmid-mediated 16S rRNA methylase, RmtC, found in a Proteus mirabilis isolate demonstrating extraordinary high-level resistance against various aminoglycosides[J]. Antimicrob Agents Chemother, 2006, 50(1): 178-184. DOI:10.1128/AAC.50.1.178-184.2006 |
| [8] |
DOI Y, DE OLIVEIRA GARCIA D, ADAMS J, et al. Coproduction of novel 16S rRNA methylase RmtD and metallo-β-lactamase SPM-1 in a panresistant Pseudomonas aeruginosa isolate from Brazil[J]. Antimicrob Agents Chemother, 2007, 51(3): 852-856. DOI:10.1128/AAC.01345-06 |
| [9] |
TIJET N, ANDRES P, CHUNG C, et al. rmtD2, a new allele of a 16S rRNA methylase gene, has been present in Enterobacteriaceae isolates from Argentina for more than a decade[J]. Antimicrob Agents Chemother, 2011, 55(2): 904-909. DOI:10.1128/AAC.00962-10 |
| [10] |
DAVIS M A, BAKER K N K, ORFE L H, et al. Discovery of a gene conferring multiple-aminoglycoside resistance in Escherichia coli[J]. Antimicrob Agents Chemother, 2010, 54(6): 2666-2669. DOI:10.1128/AAC.01743-09 |
| [11] |
XIA J, SUN J, LI L, et al. First report of the IncI1/ST898 conjugative plasmid carrying rmtE2 16S rRNA methyltransferase gene in Escherichia coli[J]. Antimicrob Agents Chemother, 2015, 59(12): 7921-7922. DOI:10.1128/AAC.01235-15 |
| [12] |
GALIMAND M, COURVALIN P, LAMBERT T. RmtF, a New member of the aminoglycoside resistance 16S rRNA N7 G1405 methyltransferase family[J]. Antimicrob Agents Chemother, 2012, 56(7): 3960-3962. DOI:10.1128/AAC.00660-12 |
| [13] |
TADA T, SHIMADA K, SATOU K, et al. Pseudomonas aeruginosa clinical isolates in nepal coproducing metallo-β-lactamases and 16S rRNA methyltransferases[J]. Antimicrob Agents Chemother, 2017, 61(9): e00694-17. |
| [14] |
BUENO M F C, FRANCISCO G R, O'HARA J A, et al. Coproduction of 16S rRNA methyltransferase RmtD or RmtG with KPC-2 and CTX-M group extended-spectrum β-lactamases in Klebsiella pneumoniae[J]. Antimicrob Agents Chemother, 2013, 57(5): 2397-2400. DOI:10.1128/AAC.02108-12 |
| [15] |
O'HARA J A, MCGANN P, SNESRUD E C, et al. Novel 16S rRNA methyltransferase RmtH produced by Klebsiella pneumoniae associated with war-related trauma[J]. Antimicrob Agents Chemother, 2013, 57(5): 2413-2416. DOI:10.1128/AAC.00266-13 |
| [16] |
NOSRATI M, DEY D, MEHRANI A, et al. Functionally critical residues in the aminoglycoside resistance-associated methyltransferase RmtC play distinct roles in 30S substrate recognition[J]. J Biol Chem, 2019, 294(46): 17642-17653. DOI:10.1074/jbc.RA119.011181 |
| [17] |
WACHINO J I, SHIBAYAMA K, KIMURA K, et al. RmtC introduces G1405 methylation in 16S rRNA and confers high-level aminoglycoside resistance on Gram-positive microorganisms[J]. FEMS Microbiol Lett, 2010, 311(1): 56-60. DOI:10.1111/j.1574-6968.2010.02068.x |
| [18] |
WACHINO J I, YAMANE K, KIMURA K, et al. Mode of transposition and expression of 16S rRNA methyltransferase gene rmtC accompanied by ISEcp1[J]. Antimicrob Agents Chemother, 2006, 50(9): 3212-3215. DOI:10.1128/AAC.00550-06 |
| [19] |
WACHINO J I, DOI Y, ARAKAWA Y. Aminoglycoside resistance: updates with a focus on acquired 16S ribosomal RNA methyltransferases[J]. Infect Dis Clin North Am, 2020, 34(4): 887-902. DOI:10.1016/j.idc.2020.06.002 |
| [20] |
WACHINO J I, SHIBAYAMA K, KUROKAWA H, et al. Novel plasmid-mediated 16S rRNA m1A1408 methyltransferase, NpmA, found in a clinically isolated Escherichia coli strain resistant to structurally diverse aminoglycosides[J]. Antimicrob Agents Chemother, 2007, 51(12): 4401-4409. DOI:10.1128/AAC.00926-07 |
| [21] |
KANAZAWA H, BABA F, KOGANEI M, et al. A structural basis for the antibiotic resistance conferred by an N1-methylation of A1408 in 16S rRNA[J]. Nucleic Acids Res, 2017, 45(21): 12529-12535. DOI:10.1093/nar/gkx882 |
| [22] |
GALIMAND M, SABTCHEVA S, COURVALIN P, et al. Worldwide disseminated armA aminoglycoside resistance methylase gene is borne by composite transposon Tn1548[J]. Antimicrob Agents Chemother, 2005, 49(7): 2949-2953. DOI:10.1128/AAC.49.7.2949-2953.2005 |
| [23] |
MOREL F, DECOUSSER J W, KUMANSKI S, et al. Association of the 16S rRNA methylase gene rmtB with a novel insertion sequence element belonging to the ISL3 family[J]. Int J Antimicrob Agents, 2017, 49(1): 117-118. DOI:10.1016/j.ijantimicag.2016.10.002 |
| [24] |
HUANG J W, DENG S S, REN J M, et al. Characterization of a blaNDM-1-harboring plasmid from a Salmonella enterica clinical isolate in China[J]. Mol Med Rep, 2017, 16(2): 1087-1092. DOI:10.3892/mmr.2017.6733 |
| [25] |
DOI Y, ADAMS-HADUCH J M, PATERSON D L. Genetic environment of 16S rRNA methylase gene rmtD[J]. Antimicrob Agents Chemother, 2008, 52(6): 2270-2272. DOI:10.1128/AAC.00037-08 |
| [26] |
TOLEMAN M A, BENNETT P M, WALSH T R. ISCR elements: novel gene-capturing systems of the 21st century?[J]. Microbiol Mol Biol Rev, 2006, 70(2): 296-316. DOI:10.1128/MMBR.00048-05 |
| [27] |
POIREL L, BONNIN R A, NORDMANN P. Analysis of the resistome of a multidrug-resistant NDM-1-producing Escherichia coli strain by high-throughput genome sequencing[J]. Antimicrob Agents Chemother, 2011, 55(9): 4224-4229. DOI:10.1128/AAC.00165-11 |
| [28] |
HO P L, LO W U, YEUNG M K, et al. Complete sequencing of pNDM-HK encoding NDM-1 carbapenemase from a multidrug-resistant Escherichia coli strain isolated in Hong Kong[J]. PLoS One, 2011, 6(3): e17989. DOI:10.1371/journal.pone.0017989 |
| [29] |
GRANIER S A, HIDALGO L, SAN MILLAN A, et al. ArmA methyltransferase in a monophasic Salmonella enterica isolate from food[J]. Antimicrob Agents Chemother, 2011, 55(11): 5262-5266. DOI:10.1128/AAC.00308-11 |
| [30] |
WRIGHT M S, HAFT D H, HARKINS D M, et al. New insights into dissemination and variation of the health care-associated pathogen Acinetobacter baumannii from genomic analysis[J]. mBio, 2014, 5(1): e00963-13. |
| [31] |
DOI Y, ADAMS J M, YAMANE K, et al. Identification of 16S rRNA methylase-producing Acinetobacter baumannii clinical strains in North America[J]. Antimicrob Agents Chemother, 2007, 51(11): 4209-4210. DOI:10.1128/AAC.00560-07 |
| [32] |
ZHAO W S, LIU G Y, MI Z H, et al. Coexistence of blaOXA-23 with armA and novel gyrA mutation in a pandrug-resistant Acinetobacter baumannii isolate from the blood of a patient with haematological disease in China[J]. J Hosp Infect, 2011, 77(3): 278-279. DOI:10.1016/j.jhin.2010.11.006 |
| [33] |
KARTHIKEYAN K, THIRUNARAYAN M A, KRISHNAN P. Coexistence of blaOXA-23 with blaNDM-1 and armA in clinical isolates of Acinetobacter baumannii from India[J]. J Antimicrob Chemother, 2010, 65(10): 2253-2254. DOI:10.1093/jac/dkq273 |
| [34] |
NAFPLIOTI K, GALANI I, ANGELIDIS E, et al. Dissemination of international clone Ⅱ Acinetobacter baumannii strains coproducing OXA-23 carbapenemase and 16S rRNA methylase ArmA in Athens, Greece[J]. Microb Drug Resist, 2020, 26(1): 9-13. DOI:10.1089/mdr.2019.0075 |
| [35] |
KUMARASAMY K K, TOLEMAN M A, WALSH T R, et al. Emergence of a new antibiotic resistance mechanism in India, Pakistan, and the UK: a molecular, biological, and epidemiological study[J]. Lancet Infect Dis, 2010, 10(9): 597-602. DOI:10.1016/S1473-3099(10)70143-2 |
| [36] |
AISHWARYA K V L, GEETHA P V, SHANTHI M, et al. Co occurrence of two 16S rRNA methyltrasferases along with NDM and OXA 48 like carbapenamases on a single plasmid in Klebsiella pneumoniae[J]. J Lab Physicians, 2019, 11(4): 305-311. DOI:10.4103/JLP.JLP_59_19 |
| [37] |
TAYLOR E, SRISKANDAN S, WOODFORD N, et al. High prevalence of 16S rRNA methyltransferases among carbapenemase-producing Enterobacteriaceae in the UK and Ireland[J]. Int J Antimicrob Agents, 2018, 52(2): 278-282. DOI:10.1016/j.ijantimicag.2018.03.016 |
| [38] |
DOI Y, HAZEN T H, BOITANO M, et al. Whole-genome assembly of Klebsiella pneumoniae coproducing NDM-1 and OXA-232 carbapenemases using single-molecule, real-time sequencing[J]. Antimicrob Agents Chemother, 2014, 58(10): 5947-5953. DOI:10.1128/AAC.03180-14 |
| [39] |
LUO Y P, YANG J Y, YE L Y, et al. Characterization of KPC-2-producing Escherichia coli, Citrobacter freundii, Enterobacter cloacae, Enterobacter aerogenes, and Klebsiella oxytoca isolates from a Chinese Hospital[J]. Microb Drug Resist, 2014, 20(4): 264-269. DOI:10.1089/mdr.2013.0150 |
| [40] |
GÜR D, HASDEMIR U, ÇAKAR A, et al. Comparative in vitro activity of plazomicin and older aminoglyosides against Enterobacterales isolates; prevalence of aminoglycoside modifying enzymes and 16S rRNA methyltransferases[J]. Diagn Microbiol Infect Dis, 2020, 97(4): 115092. DOI:10.1016/j.diagmicrobio.2020.115092 |
| [41] |
LIU Z R, LING B D, ZHOU L M. Prevalence of 16S rRNA methylase, modifying enzyme, and extended-spectrum beta-lactamase genes among Acinetobacter baumannii isolates[J]. J Chemother, 2015, 27(4): 207-212. DOI:10.1179/1973947814Y.0000000190 |
| [42] |
JIN J S, KWON K T, MOON D C, et al. Emergence of 16S rRNA methylase rmtA in colistin-only-sensitive Pseudomonas aeruginosa in South Korea[J]. Int J Antimicrob Agents, 2009, 33(5): 490-491. DOI:10.1016/j.ijantimicag.2008.10.024 |
| [43] |
YAMANE K, WACHINO J I, SUZUKI S, et al. 16S rRNA methylase-producing, gram-negative pathogens, Japan[J]. Emerg Infect Dis, 2007, 13(4): 642-646. DOI:10.3201/eid1304.060501 |
| [44] |
YEGANEH SEFIDAN F, MOHAMMADZADEH-ASL Y, GHOTASLOU R. High-level resistance to aminoglycosides due to 16S rRNA methylation in Enterobacteriaceae Isolates[J]. Microb Drug Resist, 2019, 25(9): 1261-1265. DOI:10.1089/mdr.2018.0171 |
| [45] |
GAJAMER V R, BHATTACHARJEE A, PAUL D, et al. High prevalence of carbapenemase, AmpC β-lactamase and aminoglycoside resistance genes in extended-spectrum β-lactamase-positive uropathogens from Northern India[J]. J Glob Antimicrob Resist, 2020, 20: 197-203. DOI:10.1016/j.jgar.2019.07.029 |
| [46] |
CHEN F, WANG L, WANG M, et al. Genetic characterization and in vitro activity of antimicrobial combinations of multidrug-resistant Acinetobacter baumannii from a general hospital in China[J]. Oncol Lett, 2018, 15(2): 2305-2315. |
| [47] |
BOGAERTS P, GALIMAND M, BAURAING C, et al. Emergence of ArmA and RmtB aminoglycoside resistance 16S rRNA methylases in Belgium[J]. J Antimicrob Chemother, 2007, 59(3): 459-464. DOI:10.1093/jac/dkl527 |
| [48] |
FRITSCHE T R, CASTANHEIRA M, MILLER G H, et al. Detection of methyltransferases conferring high-level resistance to aminoglycosides in Enterobacteriaceae from Europe, North America, and Latin America[J]. Antimicrob Agents Chemother, 2008, 52(5): 1843-1845. DOI:10.1128/AAC.01477-07 |
| [49] |
KANG H Y, KIM K Y, KIM J, et al. Distribution of conjugative-plasmid-mediated 16S rRNA methylase genes among amikacin-resistant Enterobacteriaceae isolates collected in 1995 to 1998 and 2001 to 2006 at a university hospital in South Korea and identification of conjugative plasmids mediating dissemination of 16S rRNA methylase[J]. J Clin Microbiol, 2008, 46(2): 700-706. DOI:10.1128/JCM.01677-07 |
| [50] |
POIREL L, LAGRUTTA E, TAYLOR P, et al. Emergence of metallo-β-lactamase NDM-1-producing multidrug-resistant Escherichia coli in Australia[J]. Antimicrob Agents Chemother, 2010, 54(11): 4914-4916. DOI:10.1128/AAC.00878-10 |
| [51] |
TIAN G B, RIVERA J I, PARK Y S, et al. Sequence type ST405 Escherichia coli isolate producing QepA1, CTX-M-15, and RmtB from Detroit, Michigan[J]. Antimicrob Agents Chemother, 2011, 55(8): 3966-3967. DOI:10.1128/AAC.00652-11 |
| [52] |
WU Q, ZHANG Y B, HAN L Z, et al. Plasmid-mediated 16S rRNA methylases in aminoglycoside-resistant Enterobacteriaceae isolates in Shanghai, China[J]. Antimicrob Agents Chemother, 2009, 53(1): 271-272. DOI:10.1128/AAC.00748-08 |
| [53] |
YAMANE K, WACHINO J I, SUZUKI S, et al. Plasmid-mediated qepA gene among Escherichia coli clinical isolates from Japan[J]. Antimicrob Agents Chemother, 2008, 52(4): 1564-1566. DOI:10.1128/AAC.01137-07 |
| [54] |
DENG Y, ZENG Z, CHEN S, et al. Dissemination of IncFⅡ plasmids carrying rmtB and qepA in Escherichia coli from pigs, farm workers and the environment[J]. Clin Microbiol Infect, 2011, 17(11): 1740-1745. DOI:10.1111/j.1469-0691.2011.03472.x |
| [55] |
XIA J, FANG L X, CHENG K, et al. Clonal spread of 16S rRNA methyltransferase-producing Klebsiella pneumoniae ST37 with high prevalence of ESBLs from companion animals in China[J]. Front Microbiol, 2017, 8: 529. |
| [56] |
XIA J, SUN J, CHENG K, et al. Persistent spread of the rmtB 16S rRNA methyltransferase gene among Escherichia coli isolates from diseased food-producing animals in China[J]. Vet Microbiol, 2016, 188: 41-46. DOI:10.1016/j.vetmic.2016.03.018 |
| [57] |
DENG Y T, HE L Y, CHEN S, et al. F33:A-: B- and F2:A-: B- plasmids mediate dissemination of rmtB-blaCTX-M-9 group genes and rmtB-qepA in Enterobacteriaceae isolates from pets in China[J]. Antimicrob Agents Chemother, 2011, 55(10): 4926-4929. DOI:10.1128/AAC.00133-11 |
| [58] |
SHENG J F, LI J J, TU S, et al. blaKPC and rmtB on a single plasmid in Enterobacter amnigenus and Klebsiella pneumoniae isolates from the same patient[J]. Eur J Clin Microbiol Infect Dis, 2012, 31(7): 1585-1591. DOI:10.1007/s10096-011-1481-x |
| [59] |
POIREL L, GOUTINES J, AIRES-DE-SOUSA M, et al. High rate of association of 16S rRNA methylases and carbapenemases in Enterobacteriaceae recovered from hospitalized children in Angola[J]. Antimicrob Agents Chemother, 2018, 62(4): e00021-18. |
| [60] |
YU T, HE T, YAO H, et al. Prevalence of 16S rRNA methylase gene rmtB among Escherichia coli isolated from bovine mastitis in Ningxia, China[J]. Foodborne Pathog Dis, 2015, 12(9): 770-777. DOI:10.1089/fpd.2015.1983 |
| [61] |
MA X J, YANG H F, LIU Y Y, et al. The emergence of the 16S rRNA methyltransferase RmtB in a multidrug-resistant Serratia marcescens isolate in China[J]. Ann Lab Med, 2015, 35(1): 172-174. DOI:10.3343/alm.2015.35.1.172 |
| [62] |
LIAO W J, DE WANG L, LI D, et al. High prevalence of 16S rRNA methylase genes among carbapenem-resistant hypervirulent Klebsiella pneumoniae isolates in a Chinese tertiary hospital[J]. Microb Drug Resist, 2020, 27(1): 44-52. |
| [63] |
ZONG Z Y, PARTRIDGE S R, IREDELL J R. RmtC 16S rRNA methyltransferase in Australia[J]. Antimicrob Agents Chemother, 2008, 52(2): 794-795. DOI:10.1128/AAC.01399-07 |
| [64] |
HOPKINS K L, ESCUDERO J A, HIDALGO L, et al. 16S rRNA methyltransferase RmtC in Salmonella enterica serovar Virchow[J]. Emerg Infect Dis, 2010, 16(4): 712-715. DOI:10.3201/eid1604.090736 |
| [65] |
HIDALGO L, HOPKINS K L, GUTIERREZ B, et al. Association of the novel aminoglycoside resistance determinant RmtF with NDM carbapenemase in Enterobacteriaceae isolated in India and the UK[J]. J Antimicrob Chemother, 2013, 68(7): 1543-1550. DOI:10.1093/jac/dkt078 |
| [66] |
WILLIAMSON D A, SIDJABAT H E, FREEMAN J T, et al. Identification and molecular characterisation of New Delhi metallo-β-lactamase-1 (NDM-1)- and NDM-6-producing Enterobacteriaceae from New Zealand hospitals[J]. Int J Antimicrob Agents, 2012, 39(6): 529-533. DOI:10.1016/j.ijantimicag.2012.02.017 |
| [67] |
DA PAZ PEREIRA J N, DE ANDRADE C A D N, DA COSTA LIMA J L, et al. Clonal dissemination of clinical isolates of Acinetobacter baumannii carriers of 16S rRNA methylase genes in an Oncological Hospital in Recife, Brazil[J]. Curr Microbiol, 2020, 77(1): 32-39. DOI:10.1007/s00284-019-01786-6 |
| [68] |
YAMANE K, ROSSI F, BARBERINO M G M A, et al. 16S ribosomal RNA methylase RmtD produced by Klebsiella pneumoniae in Brazil[J]. J Antimicrob Chemother, 2008, 61(3): 746-747. DOI:10.1093/jac/dkm526 |
| [69] |
TADA T, SHIMADA K, MYA S, et al. A new variant of 16S rRNA methylase, RmtD3, in a Clinical Isolate of Pseudomonas aeruginosa in Myanmar[J]. Antimicrob Agents Chemother, 2018, 62(1): e01806-17. |
| [70] |
URBANOWICZ P, IZDEBSKI R, BARANIAK A, et al. Pseudomonas aeruginosa with NDM-1, DIM-1 and PME-1 β-lactamases, and RmtD3 16S rRNA methylase, encoded by new genomic islands[J]. J Antimicrob Chemother, 2019, 74(10): 3117-3119. DOI:10.1093/jac/dkz262 |
| [71] |
DOI Y, GHILARDI A C R, ADAMS J, et al. High prevalence of metallo-beta-lactamase and 16S rRNA methylase coproduction among imipenem-resistant Pseudomonas aeruginosa isolates in Brazil[J]. Antimicrob Agents Chemother, 2007, 51(9): 3388-3390. DOI:10.1128/AAC.00443-07 |
| [72] |
FONTES L C, NEVES P R, OLIVEIRA S, et al. Isolation of Pseudomonas aeruginosa coproducing metallo-β-lactamase SPM-1 and 16S rRNA methylase RmtD1 in an urban river[J]. Antimicrob Agents Chemother, 2011, 55(6): 3063-3064. DOI:10.1128/AAC.00138-11 |
| [73] |
BUENO M F C, FRANCISCO G R, DE OLIVEIRA GARCIA D, et al. Complete sequences of multidrug resistance plasmids bearing rmtD1 and rmtD2 16S rRNA methyltransferase genes[J]. Antimicrob Agents Chemother, 2016, 60(3): 1928-1931. DOI:10.1128/AAC.02562-15 |
| [74] |
WANGKHEIMAYUM J, BHATTACHARJEE M, DAS B J, et al. Correction to: expansion of acquired 16S rRNA methytransferases along with CTX-M-15, NDM and OXA-48 within three sequence types of Escherichia coli from northeast India[J]. BMC Infect Dis, 2020, 20(1): 620. DOI:10.1186/s12879-020-05326-7 |
| [75] |
WANG Y X, ZHANG A Y, YANG Y Q, et al. Emergence of Salmonella enterica serovar Indiana and California isolates with concurrent resistance to cefotaxime, amikacin and ciprofloxacin from chickens in China[J]. Int J Food Microbiol, 2017, 262: 23-30. DOI:10.1016/j.ijfoodmicro.2017.09.012 |
| [76] |
LI B, PACEY M P, DOI Y. Chromosomal 16S ribosomal RNA methyltransferase RmtE1 in Escherichia coli sequence Type 448[J]. Emerg Infect Dis, 2017, 23(5): 876-878. |
| [77] |
LEE C S, LI J J, DOI Y. Complete sequence of conjugative IncA/C plasmid encoding CMY-2 β-lactamase and RmtE 16S rRNA methyltransferase[J]. Antimicrob Agents Chemother, 2015, 59(7): 4360-4361. DOI:10.1128/AAC.00852-15 |
| [78] |
TADA T, HISHINUMA T, WATANABE S, et al. Molecular characterization of multidrug-resistant Pseudomonas aeruginosa isolates in Hospitals in Myanmar[J]. Antimicrob Agents Chemother, 2019, 63(5): e02397-18. |
| [79] |
SIDJABAT H E, TOWNELL N, NIMMO G R, et al. Dominance of IMP-4-producing Enterobacter cloacae among carbapenemase-producing Enterobacteriaceae in Australia[J]. Antimicrob Agents Chemother, 2015, 59(7): 4059-4066. DOI:10.1128/AAC.04378-14 |
| [80] |
GAMAL D, FERNÁNDEZ-MARTÍNEZ M, SALEM D, et al. Carbapenem-resistant Klebsiella pneumoniae isolates from Egypt containing blaNDM-1 on IncR plasmids and its association with rmtF[J]. Int J Infect Dis, 2016, 43: 17-20. DOI:10.1016/j.ijid.2015.12.003 |
| [81] |
POIREL L, LABARCA J, BELLO H, et al. Emergence of the 16S rRNA methylase RmtG in an extended-spectrum-β-lactamase-producing and colistin-resistant Klebsiella pneumoniae isolate in Chile[J]. Antimicrob Agents Chemother, 2014, 58(1): 618-619. DOI:10.1128/AAC.02059-13 |
| [82] |
MANCINI S, POIREL L, CORTHESY M, et al. Klebsiella pneumoniae co-producing KPC and RmtG, finally targeting Switzerland[J]. Diagn Microbiol Infect Dis, 2018, 90(2): 151-152. DOI:10.1016/j.diagmicrobio.2017.10.019 |
| [83] |
PASSARELLI-ARAUJO H, PALMEIRO J K, MOHARANA K C, et al. Molecular epidemiology of 16S rRNA methyltransferase in Brazil: RmtG in Klebsiella aerogenes ST93 (CC4)[J]. An Acad Bras Cienc, 2019, 91(S1): e20180762. |
| [84] |
BEYROUTHY R, ROBIN F, HAMZE M, et al. IncFⅡk plasmid harbouring an amplification of 16S rRNA methyltransferase-encoding gene rmtH associated with mobile element ISCR2[J]. J Antimicrob Chemother, 2017, 72(2): 402-406. DOI:10.1093/jac/dkw435 |
| [85] |
MARSH J W, PACEY M P, EZEONWUKA C, et al. Clostridioides difficile: a potential source of NpmA in the clinical environment[J]. J Antimicrob Chemother, 2019, 74(2): 521-523. DOI:10.1093/jac/dky420 |
| [86] |
LIOY V S, GOUSSARD S, GUERINEAU V, et al. Aminoglycoside resistance 16S rRNA methyltransferases block endogenous methylation, affect translation efficiency and fitness of the host[J]. RNA, 2014, 20(3): 382-391. DOI:10.1261/rna.042572.113 |
| [87] |
KIMURA S, SUZUKI T. Fine-tuning of the ribosomal decoding center by conserved methyl-modifications in the Escherichia coli 16S rRNA[J]. Nucleic Acids Res, 2010, 38(4): 1341-1352. DOI:10.1093/nar/gkp1073 |
| [88] |
YUAN M, CHEN H, ZHU X, et al. pSY153-MDR, a p12969-DIM-related mega plasmid carrying blaIMP-45 and armA, from clinical Pseudomonas putida[J]. Oncotarget, 2017, 8(40): 68439-68447. DOI:10.18632/oncotarget.19496 |
| [89] |
SHANKAR C, SHANKAR B A, MANESH A, et al. KPC-2 producing ST101 Klebsiella pneumoniae from bloodstream infection in India[J]. J Med Microbiol, 2018, 67(7): 927-930. DOI:10.1099/jmm.0.000767 |
| [90] |
HUSAIN N, TULSIAN N K, CHIEN W L, et al. Ligand-mediated changes in conformational dynamics of NpmA: implications for ribosomal interactions[J]. Sci Rep, 2016, 6(1): 37061. DOI:10.1038/srep37061 |
| [91] |
DOI Y, ARAKAWA Y. 16S ribosomal RNA methylation: emerging resistance mechanism against aminoglycosides[J]. Clin Infect Dis, 2007, 45(1): 88-94. DOI:10.1086/518605 |
| [92] |
PLATTNER M, GYSIN M, HALDIMANN K, et al. Epidemiologic, phenotypic, and structural characterization of aminoglycoside-resistance gene aac(3)-IV[J]. Int J Mol Sci, 2020, 21(17): 6133. DOI:10.3390/ijms21176133 |
| [93] |
MACMASTER R, ZELINSKAYA N, SAVIC M, et al. Structural insights into the function of aminoglycoside-resistance A1408 16S rRNA methyltransferases from antibiotic-producing and human pathogenic bacteria[J]. Nucleic Acids Res, 2010, 38(4): 7791-7799. |
| [94] |
SCHMITT E, GALIMAND M, PANVERT M, et al. Structural bases for 16S rRNA methylation catalyzed by ArmA and RmtB methyltransferases[J]. J Mol Biol, 2009, 388(3): 570-582. DOI:10.1016/j.jmb.2009.03.034 |
(编辑 白永平)



