皮肤是人体最大的器官,由表皮、真皮和皮下组织构成,并含有毛囊、汗腺、皮脂腺、指甲等附属物,具有保护身体、维持水分、感觉冷暖等功能[1]。由于羊毛囊的形态发生类似于人的头部毛囊,而绵羊的躯干皮肤所含有的毛囊和汗腺与人类腋窝皮肤的解剖结构相似[2],所以,绵羊可成为研究人类毛发生长的优良动物模型。
毛囊是由上皮细胞和间充质之间通过信号分子介导的一系列相互作用形成的[3]。成熟的毛囊包括真皮乳头、外根鞘、内根鞘、毛球、毛干等结构[4],且在发育形成的过程中通常伴随着汗腺、皮脂腺和立毛肌的出现。立毛肌是一类平滑肌,由皮肤结缔组织的成纤维细胞分化形成[5],位于表皮下方,始于毛囊底部隆起处,终止于真皮细胞外基质[6]。在发育过程中,立毛肌与毛囊和交感神经的关系十分密切。立毛肌附着于毛囊干细胞所在的毛囊隆起区域,交感神经使立毛肌平滑肌细胞束化,交感神经与立毛肌和毛囊一起形成三系单元(tri-lineage unit)[7-8]。Shwartz等[9]对小鼠皮肤的研究进一步阐述了三系单元的作用机制:在发育过程中,毛囊干细胞会分泌音猬因子(Sonic hedgehog)指导立毛肌交感神经小生境的形成,进而控制成体毛囊再生。Torkamani等[10]研究发现,脱发患者的毛囊附着的立毛肌结构残缺,表明立毛肌可以影响成熟毛囊的完整性。也有研究表明,皮肤组织中立毛肌肌动蛋白的存在有利于毛囊调控毛干的运动[11]。除此之外,Sato等[12]研究表明,立毛肌在毛发移植中与毛囊的再生有关。可见立毛肌对皮肤其他附属器官的发育过程起重要作用。
藏羊是我国特有的地方绵羊品种,近些年来,已经有研究人员报道了关于胚胎期藏羊皮肤中毛囊、汗腺等皮肤附属物发育的分子调控机制[13-15],但目前还没有关于调控藏羊胚胎期立毛肌发育分子机制的报道。本研究初步探索藏羊立毛肌发生及发育的过程,通过形态学变化明确立毛肌发生的关键时期,并通过转录组数据分析该时期的整体基因表达变化,探索影响立毛肌发生过程的动态基因调控网络和潜在的候选基因。该结果将丰富有关立毛肌形态发生过程中组织学和分子变化的研究,为了解人类毛发发育的分子调控机制提供参考,对了解皮肤生物学复杂性具有重要的参考意义。
1 材料与方法 1.1 试验材料藏羊(西藏羊)取自青海当地屠宰场,采集30只75~110日龄健康情况良好的藏羊胚胎并解剖背部皮肤,将样品平均分为两部分,一部分立即放在4 ℃用4%的多聚甲醛固定,另一部分冻存在液氮中[13-14]。
1.2 绵羊背部皮肤石蜡组织切片及HE染色为了确定藏羊皮肤中立毛肌的发生阶段,将藏羊不同胚龄背部皮肤样品用梯度酒精脱水,二甲苯透明,石蜡包埋并切成5 μm组织切片。然后将背部皮肤切片进行脱蜡和HE(苏木精和伊红)染色,并用中性树脂固定。对染色的皮肤切片进行显微镜拍照记录,根据形态学初步推断样品胚胎日龄。
1.3 绵羊背部皮肤组织转录组测序挑选藏羊胚龄大约在75和85 d(E75和E85)各3个背部皮肤组织样品,使用TRIzol试剂(美国Invitrogen公司)分别从6个样品中提取总RNA,并使用带有Bioanalyzer 2100系统(美国加利福尼亚州安捷伦科技公司)的RNA Nano 6000分析试剂盒评估RNA完整性。按照制造商的程序,使用用于Illumina的NEBNext9 UltraTM RNA文库制备试剂盒(美国NEB),在Novogene(中国北京)构建测序文库,利用Agilent Bioanalyzer 2100系统评估文库质量,在Illumina Hiseq平台(Hiseq X ten)上进行150个碱基对配对末端序列的测序。
1.4 绵羊背部皮肤组织转录组测序数据分析原始数据的上游处理均在服务器中进行,利用fastqc和trimglore软件进行数据的质控和过滤;通过Hisat2[16](版本: 2.1.0)软件将clean reads映射到藏羊参考基因组(版本:Oarv3.1);用Samtools软件对比对结果进行格式转换、排序;使用Htseq[17]软件对clean reads进行计数;用Stringtie[18](版本:1.3.5)软件计算每个基因的FPKM值。使用R语言(版本:3.6.3)进行转录组下游数据分析;用DESeq2软件包进行基因表达量差异分析[19]。对于生物学重复,将p < 0.05和|log2(fold change)|≥1设置为差异表达基因的阈值,获得差异表达的基因。使用bioDBnet网站进行基因ID的转换。因为藏羊是非模式生物,先用AnnotationHub R包获取绵羊注释文件,再使用clusterProfiler[20]R包对DEGs进行GO富集分析,差异表达基因显著富集于校正后p < 0.05的GO条目,使用KOBAS[21]网站进行KEGG通路中差异表达基因的富集分析。使用Cytoscape StringApp软件[22]实现蛋白质相互作用网络的可视化。
1.5 差异表达基因实时荧光定量PCR验证1.5.1 引物的设计与合成 随机选出6个差异表达基因,以羊的GAPDH为内参基因进行实时荧光定量PCR(RT-qPCR)验证。利用NCBI网站中的primer-BLAST在线工具设计引物(表 1),引物由生工生物工程(上海)股份有限公司合成。
|
|
表 1 RT-qPCR引物序列信息 Table 1 Primers information for RT-qPCR |
1.5.2 荧光定量PCR 每个时期的样本设置3个技术性重复(n=3)。按照Vazyme公司的产品ChamQ Universal SYBR qPCR Master Mix说明书对目的基因进行RT-qPCR检测。反应体系为:cDNA 6.6 μL,酶Mix 7.5 μL,上、下游引物各0.45 μL;反应程序:95 ℃预变性30 s;95 ℃变性10 s,62 ℃退火30 s,72 ℃延申30 s,40个循环;65~95 ℃,每5 s增加0.5 ℃,5 min制作熔解曲线。用Bio-Rad CFX Manager 3.1软件收集处理数据,采用2-ΔΔCT方法对mRNA进行定量数据分析,并用t检验进行统计分析。
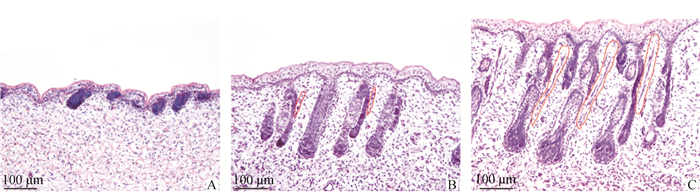
2 结果 2.1 藏羊背部皮肤中立毛肌的形态发生及发育本试验选择西藏地毯羊(一种典型的粗羊毛绵羊)为研究对象,详细研究皮肤中立毛肌的发生过程。通过组织学HE染色观察到立毛肌从无到有直至结构完整的3个时期(图 1A~C)。根据Rogers[2]的描述确定TF2a(约E75,图 1A,T代表藏羊)和TF3a(约E85,图 1B)为立毛肌开始形成的关键时期。
|
A.TF2a(E75)皮肤组织切片图:没有观察到明显的立毛肌;B.TF3a(E85)皮肤组织切片图:显示形成早期立毛肌;C.TF4(E100)皮肤组织切片图:显示立毛肌的延伸。B、C图红色虚线内标识部位为立毛肌 A. TF2a (E75) skin tissue section image: no obvious arrector pili muscle; B. TF3a (E85) skin tissue section image: the early formation of arrector pili muscle; C. TF4 (E100) skin tissue section image: the down-growth of arrector pili muscle. In B, C figures, the parts in the red dashed line are the arrector pili muscle 图 1 藏羊胚胎期皮肤组织切片显示立毛肌的早期形态发生过程图 Fig. 1 The histological identification of arrector pili muscle development in Tibetan sheep skin during embryo period |
取两个时期(TF2a、TF3a),每个时期3个样品,共6个藏羊背部皮肤组织样品进行RNA测序。将数据上传于服务器中进行质控、比对、计数。每个样品产生超过12G的原始数据,测序结果如表 2所示,其中Q30数据基本都在90%以上,表明测序数据满足后续分析的条件。
|
|
表 2 转录组测序数据质量检测分析 Table 2 Quality analysis of transcriptome sequencing data |
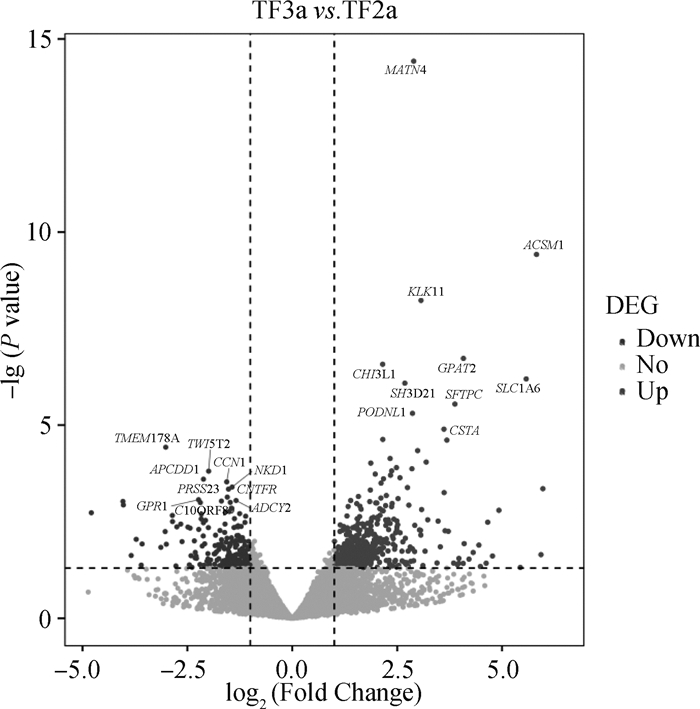
利用R软件包DESeq2进行差异分析得到初步差异分析的结果。设置筛选差异基因的标准为|log2(fold change)|≥1和p < 0.05,得到的差异基因结果如图 2所示,共获得1 159个差异表达基因,其中900个基因上调表达,259个基因下调表达。
|
虚线左上侧代表显著下调基因,虚线右上侧代表显著上调基因。TF2a代表E75;TF3a代表E85 The upper left side of the dotted line represents significantly down-regulated genes, and the upper right side of the dotted line represents significantly up-regulated genes. TF2a stands for E75; TF3a stands for E85 图 2 立毛肌早期形态发生期藏羊皮肤中差异表达基因火山图(立毛肌雏形vs. 无立毛肌) Fig. 2 Volcano map of differentially expressed genes in Tibetan sheep skin during early morphogenesis of arrector pili muscle (the germ of arrector pili muscle vs. no arrector pili muscle) |
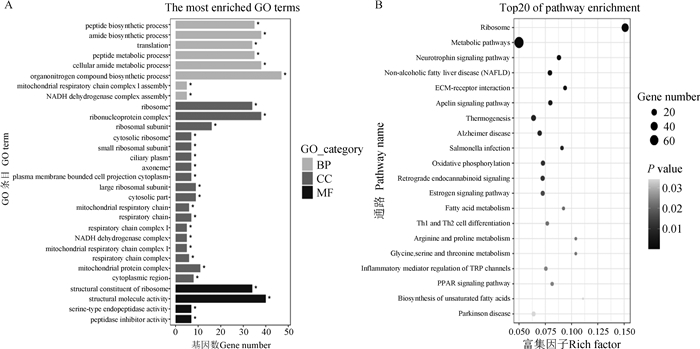
对1 159个差异基因进行GO和KEGG功能富集分析,一共显著富集到71个GO条目,包括29个生物学过程(BP),27个细胞组分(CC),15个生物学功能(MF)。前30条显著富集到的GO条目如图 3A所示,在生物学过程中,差异基因显著富集到肽生物合成过程(peptide biosynthetic process,35个基因)、酰胺生物合成过程(amide biosynthetic process,38个基因)、翻译(translation,34个基因)。在生物学功能归类中,差异基因显著富集到结构分子活性(structural molecule activity,40个基因)。所有富集条目大部分都与转录和翻译过程紧密相关,表明细胞在此时期的活动,如细胞增殖和分化等非常活跃,此时期可能为器官发育形成的重要时期。
|
BP、MF、CC分别代表生物学过程、生物学功能、细胞组分 BP, MF, and CC represent biological process, molecular function, and cellular component, respectively 图 3 立毛肌早期形态发生期差异表达基因富集分析中前30个GO条目(A)与前20条KEGG通路图(B)(TF3a vs. TF2a) Fig. 3 Representation of top 30 GO terms(A) and top 20 KEGG pathways(B) in the enrichment analysis of differentially expressed genes in early morphogenesis of arrector pili muscle(TF3a vs. TF2a) |
在KEGG富集分析中,共显著富集到28条信号通路,其中图 3B列出了前20个显著富集到的通路,显著富集到的通路为代谢途径(metabolic pathways,77个基因)、神经营养蛋白信号通路(neurotrophin signaling pathway,11个基因)、ECM-受体相互作用(ECM-receptor interaction,9个基因)等。
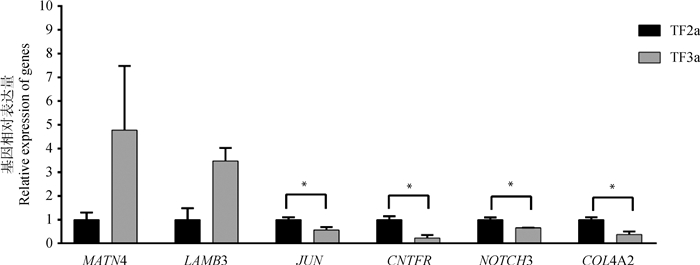
2.5 RT-qPCR验证对随机选取的6个差异基因进行RT-qPCR验证,结果如图 4所示,2个基因(MATN4、LAMB3)上调表达,4个基因(JUN、CNTFR、NOTCH3、COL4A2)下调表达,6个基因在立毛肌发育的2个时期(TF3a vs. TF2a)的藏羊皮肤组织中表达趋势与转录组测序结果一致。进一步表明测序结果具有可靠性。
|
*. p < 0.05 图 4 RT-qPCR检测6个差异表达基因在藏羊皮肤组织中的表达模式(TF3a vs. TF2a) Fig. 4 RT-qPCR verification of expression tendency of 6 differentially expressed genes in Tibetan sheep skin(TF3a vs. TF2a) |
立毛肌发生的初期伴随着毛囊的成熟和汗腺的出现,这些器官的协同变化表明,一些参与调控毛囊或腺体发育的转录因子也可能会对立毛肌的发育有影响。本研究富集到了与管发育、腺体发育、肌肉发育、干细胞发育以及皮肤发育相关的GO条目中共23个非冗余差异基因,见表 3。这些条目均属于生物学过程类别。
|
|
表 3 参与立毛肌发育的候选GO条目及基因 Table 3 GO terms and genes related to arrector pili muscle development |
KEGG富集分析得到了可能与立毛肌发育有关的通路基因(表 4)。这些通路包括粘着斑(focal adhesion)信号通路、Wnt信号通路(Wnt signaling pathway)、Notch信号通路(Notch signaling pathway)、TGF-β信号通路(TGF-beta signaling pathway)和钙离子信号通路(calcium signaling pathway)。
|
|
表 4 参与立毛肌发育的候选信号通路及基因 Table 4 Signaling pathways and genes related to arrector pili muscle development |
通过筛选GO和KGEE富集到的条目以及信号通路,共得到47个非冗余的差异基因(图5A),并对其进行了蛋白网络互作分析。共有29个基因之间有互作关系,其中有16个上调基因,13个下调基因。这些基因构成复杂的调控网络,例如,Wnt信号通路中的LEF1基因参与到肌肉结构发育和腺体发育等生物学过程;粘着斑信号通路中的JUN基因参与到腺体发育以及管发育等生物学过程(图 5B)。
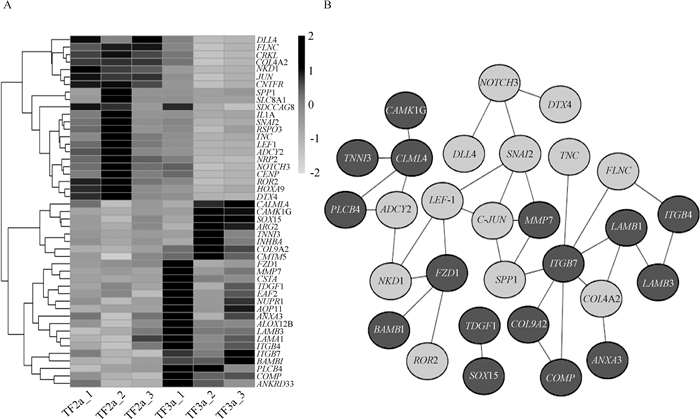
|
黑色代表表达量上调的基因;浅灰色代表表达量下调的基因 Black signal represents genes with up-regulated expression; Light gray signal represents genes with down-regulated expression 图 5 差异表达基因聚类热图(A)及网络互作图(B) Fig. 5 Heat map of clustering (A) and protein-protein network interaction (B)of differentially expressed genes |
组织切片结果展示出立毛肌发育过程中3个比较重要的发育时期的动态变化。根据已有文献的描述[2]以及试验观察[13-14],推测藏羊TF2a和TF3a两个时期分别对应E75和E85左右,TF4则对应E100左右的胚龄。其中,E75和E85左右时期立毛肌分别处于未发育和发育初期阶段,在E100左右时立毛肌已经发育较为成熟。因此,E75和E85藏羊立毛肌组织形态学的明显差异为后续的转录组数据分析提供了基础。
KEGG富集分析得到了候选信号通路。粘着斑通路(focal adhesion)可以与肌动蛋白细胞骨架途径协同调节平滑肌的迁移[23],粘着斑是连接细胞骨架与细胞外基质的一种细胞结构,是信号传导过程中许多信号分子组装的平台[24],差异基因在该信号通路富集表明,在立毛肌细胞形成后向真皮外基质方向扩展的过程中,该信号通路中某些基因的表达可能促进了立毛肌细胞的分化与形态的变化。Wnt分泌型糖脂蛋白家族介导的信号传导通路在胚胎发育和组织稳态过程中起着调节细胞增殖、分化、迁移和极性等重要作用[25],它被证明是调节癌症的重要信号通路之一[26-27],Wnt信号通路还广泛参与到皮肤组织与毛囊等器官的发育过程并调节该程中的细胞稳态[28-29]。Notch信号通路是一种进化上保守的细胞内途径,参与细胞的增殖、分化、迁移和细胞命运等过程,该途径不仅参与皮肤组织器官的发育,而且与人类皮肤疾病的发生密切相关[30],另外它也参与了肠道平滑肌的发育[31]。转化生长因子β(TGF-β) 信号通路在动物中高度保守,被认为是多细胞动物进化初期最早出现的信号通路之一,其在胚胎期可以介导组织特异性的分化和增殖等活动[32],TGF-β信号通路在调节肌肉的生长和重塑方面也起着重要作用[33]。Ca2+信号通路则是促进肌肉形成、肌肉动态平衡和再生的信号传导网络的重要组成部分[34]。上述信号通路被富集表明,TF2a到TF3a时期这一发育过程中与肌肉发育相关的基因活跃表达,各种信号通路之间的相互通讯参与了肌肉细胞发育的调节,立毛肌展现出的显著形态变化也证实了这一点。
在47个候选差异表达基因中,有研究表明一些基因与肌肉发育相关。其中,SPP1在立毛肌发生期间高表达,有文献报道SPP1与人类肌肉炎症以及肌肉再生有关[35],它也可能参与调控鸡成肌细胞的增殖发育过程[36],表明SPP1可能会在立毛肌发生早期诱导肌肉发育。BAMBI基因在立毛肌发生期间表达量显著上调,Yao等[37]的研究也证明,BAMBI蛋白在肌肉生长和再生期间富集在细胞膜上,这表明BAMBI介导的重要信号通路可能是肌肉生长和再生的重要组成部分。同样,作为表达量显著上调的TNNI3基因,在动物心肌形成过程中发挥着重要的作用,是影响肌纤维类型的一种肌小节基因,它的突变常常会引发肥厚型心肌病的发生[38],该基因的高表达也暗示了其可能与立毛肌的发育密切相关。HOX基因家族是胚胎发生过程中调控干细胞和组织模式的关键调节因子,本研究结果表明,HOXA9基因在E85天时显著下调,这与Schwörer等[39]的试验结果一致,敲低HOXA9基因的小鼠呈现肌纤维显著增加,通过抑制HOXA9基因的表达可以使肌肉再生,表明HOXA9基因的下调促进了肌肉干细胞的分化。SOX15基因在E85天时显著上调,Lee等[40]的研究表明,SOX15基因对肌肉的再生有着重要的作用,Savage等[41]的研究显示,SOX15基因可能是早期肌肉前体细胞的调节因子,它的表达对肌肉发生是正向调控作用,表明SOX15在E85天时的上调可能促进立毛肌的发生过程。因此,推测SPP1、BAMBI、TNNI3、HOXA9、SOX15为影响藏羊立毛肌发生的候选基因。
4 结论本研究利用组织切片和HE染色技术推测出藏羊胚胎期立毛肌发生的关键时期为E75~E85,通过皮肤转录组数据分析,得到1 159个差异表达基因,其中有47个可能参与到肌肉发育过程,进一步筛选得到5个与立毛肌发育显著相关的候选基因,分别为SPP1、BAMBI、TNNI3、HOXA9、SOX15。该结果为进一步阐述藏羊立毛肌发生的分子作用机制以及皮肤调控分子网络奠定了基础。
| [1] | BELKAID Y, TAMOUTOUNOUR S. The influence of skin microorganisms on cutaneous immunity[J]. Nat Rev Immunol, 2016, 16(6): 353–366. DOI: 10.1038/nri.2016.48 |
| [2] | ROGERS G E. Biology of the wool follicle: an excursion into a unique tissue interaction system waiting to be re-discovered[J]. Exp Dermatol, 2006, 15(12): 931–949. DOI: 10.1111/j.1600-0625.2006.00512.x |
| [3] | SAXENA N, MOK K W, RENDL M. An updated classification of hair follicle morphogenesis[J]. Exp Dermatol, 2019, 28(4): 332–344. DOI: 10.1111/exd.13913 |
| [4] | HOUSCHYAR K S, BORRELLI M R, TAPKING C, et al. Molecular mechanisms of hair growth and regeneration: current understanding and novel paradigms[J]. Dermatology, 2020, 236(4): 271–280. DOI: 10.1159/000506155 |
| [5] | DRISKELL R R, LICHTENBERGER B M, HOSTE E, et al. Distinct fibroblast lineages determine dermal architecture in skin development and repair[J]. Nature, 2013, 504(7479): 277–281. DOI: 10.1038/nature12783 |
| [6] | POBLET E, ORTEGA F, JIMÉNEZ F. The arrector pili muscle and the follicular unit of the scalp: a microscopic anatomy study[J]. Dermatol Surg, 2002, 28(9): 800–803. |
| [7] | FURLAN A, LA MANNO G, LVBKE M, et al. Visceral motor neuron diversity delineates a cellular basis for nipple-and pilo-erection muscle control[J]. Nat Neurosci, 2016, 19(10): 1331–1340. DOI: 10.1038/nn.4376 |
| [8] | FUJIWARA H, FERREIRA1 M, DONATI G, et al. The basement membrane of hair follicle stem cells is a muscle cell niche[J]. Cell, 2011, 144(4): 577–589. DOI: 10.1016/j.cell.2011.01.014 |
| [9] | SHWARTZ Y, GONZALEZ-CELEIRO M, CHEN C L, et al. Cell Types promoting goosebumps form a niche to regulate hair follicle stem cells[J]. Cell, 2020, 182(3): 578–593. DOI: 10.1016/j.cell.2020.06.031 |
| [10] | TORKAMANI N, RUFAUT N, JONES L, et al. The arrector pili muscle, the bridge between the follicular stem cell niche and the interfollicular epidermis[J]. Anat Sci Int, 2017, 92(1): 151–158. DOI: 10.1007/s12565-016-0359-5 |
| [11] | MORIOKA K, ARAI M, IHARA S. Steady and temporary expressions of smooth muscle actin in hair, vibrissa, arrector pili muscle, and other hair appendages of developing rats[J]. Acta Histochem Cytochem, 2011, 44(3): 141–153. DOI: 10.1267/ahc.11013 |
| [12] | SATO A, TOYOSHIMA K E, TOKI H, et al. Single follicular unit transplantation reconstructs arrector pili muscle and nerve connections and restores functional hair follicle piloerection[J]. J Dermatol, 2012, 39(8): 682–687. DOI: 10.1111/j.1346-8138.2012.01505.x |
| [13] | LI S M, ZHENG X T, NIE Y F, et al. Defining key genes regulating morphogenesis of apocrine sweat gland in sheepskin[J]. Front Genet, 2018, 9: 739. |
| [14] | NIE Y F, LI S M, ZHENG X T, et al. Transcriptome reveals long non-coding RNAs and mRNAs involved in primary wool follicle induction in carpet sheep fetal skin[J]. Front Physiol, 2018, 9: 446. DOI: 10.3389/fphys.2018.00446 |
| [15] | YUE Y, GUO T, YUAN C, et al. Integrated analysis of the roles of long noncoding RNA and coding RNA expression in sheep (Ovis aries) skin during initiation of secondary hair follicle[J]. PLoS One, 2016, 11(6): e156890. |
| [16] | KIM D, LANGMEAD B, SALZBERG SL. HISAT: a fast spliced aligner with low memory requirements[J]. Nat Methods, 2015, 12(4): 357–360. DOI: 10.1038/nmeth.3317 |
| [17] | ANDERS S, PYL P T, HUBER W. HTSeq—a Python framework to work with high-throughput sequencing data[J]. Bioinformatics, 2015, 31(2): 166–169. DOI: 10.1093/bioinformatics/btu638 |
| [18] | PERTEA M, PERTEA G M, ANTONESCU C M, et al. StringTie enables improved reconstruction of a transcriptome from RNA-seq reads[J]. Nat Biotechnol, 2015, 33(3): 290–295. DOI: 10.1038/nbt.3122 |
| [19] | COSTA-SILVA J, DOMINGUES D, LOPES F M. RNA-Seq differential expression analysis: an extended review and a software tool[J]. PLoS One, 2017, 12(12): e0190152. DOI: 10.1371/journal.pone.0190152 |
| [20] | YU G C, WANG LG G, HAN Y Y, et al. clusterProfiler: an R package for comparing biological themes among gene clusters[J]. OMICS, 2012, 16(5): 284–287. DOI: 10.1089/omi.2011.0118 |
| [21] | MAO X Z, CAI T, OLYARCHUK J G, et al. Automated genome annotation and pathway identification using the KEGG Orthology (KO) as a controlled vocabulary[J]. Bioinformatics, 2005, 21(19): 3787–3793. DOI: 10.1093/bioinformatics/bti430 |
| [22] | DONCHEVA N T, MORRIS J H, GORODKIN J, et al. Cytoscape stringapp: network analysis and visualization of proteomics data[J]. J Proteome Res, 2019, 18(2): 623–632. DOI: 10.1021/acs.jproteome.8b00702 |
| [23] | TAND D D, GERLACH B D. The roles and regulation of the actin cytoskeleton, intermediate filaments and microtubules in smooth muscle cell migration[J]. Respir Res, 2017, 18(1): 54. DOI: 10.1186/s12931-017-0544-7 |
| [24] | BURRIDGE K. Focal adhesions: a personal perspective on a half century of progress[J]. FEBS J, 2017, 284(20): 3355–3361. DOI: 10.1111/febs.14195 |
| [25] | MACDONALD B T, TAMAI K, HE X. Wnt/β-catenin signaling: components, mechanisms, and diseases[J]. Dev Cell, 2009, 17(1): 9–26. DOI: 10.1016/j.devcel.2009.06.016 |
| [26] | FRENQUELLI M, TONON G. WNT signaling in hematological malignancies[J]. Front Oncol, 2020, 10: 615190. DOI: 10.3389/fonc.2020.615190 |
| [27] | KRISHNAMURTHY N, KURZROCK R. Targeting the Wnt/beta-catenin pathway in cancer: Update on effectors and inhibitors[J]. Cancer Treat Rev, 2018, 62: 50–60. DOI: 10.1016/j.ctrv.2017.11.002 |
| [28] | RISHIKAYSH P, DEV K, DIAZ D, et al. Signaling involved in hair follicle morphogenesis and development[J]. Int J Mol Sci, 2014, 15(1): 1647–1670. DOI: 10.3390/ijms15011647 |
| [29] | VELTRI A, LANG C, LIEN W H. Concise review: wnt signaling pathways in skin development and epidermal stem cells[J]. Stem Cells, 2018, 36(1): 22–35. DOI: 10.1002/stem.2723 |
| [30] | GRATTON R, TRICARICO P M, MOLTRASIO C, et al. Pleiotropic role of notch signaling in human skin diseases[J]. Int J Mol Sci, 2020, 21(12): 4214. DOI: 10.3390/ijms21124214 |
| [31] | HUYCKE T R, MILLER B M, GILL H K, et al. Genetic and mechanical regulation of intestinal smooth muscle development[J]. Cell, 2019, 179(1): 90–105. DOI: 10.1016/j.cell.2019.08.041 |
| [32] | TZAVLAKI K, MOUSTAKAS A. TGF-β signaling[J]. Biomolecules, 2020, 10(3): 487. DOI: 10.3390/biom10030487 |
| [33] | CHEN J L, COLGAN T D, WALTON K L, et al. The TGF-β signalling network in muscle development, adaptation and disease[J]. Adv Exp Med Biol, 2016, 900: 97–131. |
| [34] | TU M K, LEVIN J B, HAMILTON A M, et al. Calcium signaling in skeletal muscle development, maintenance and regeneration[J]. Cell Calcium, 2016, 59(2-3): 91–97. DOI: 10.1016/j.ceca.2016.02.005 |
| [35] | RYALL J G, CHURCH J E, LYNCH G S. Novel role for β-adrenergic signalling in skeletal muscle growth, development and regeneration[J]. Clin Exp Pharmacol Physiol, 2010, 37(3): 397–401. DOI: 10.1111/j.1440-1681.2009.05312.x |
| [36] |
刘龙洲, 梁忠, 黄华云, 等. 焦点黏连信号通路相关基因在鸡肌肉发育中的表达研究[J]. 中国家禽, 2018, 40(17): 8–12.
LIU L Z, LIANG Z, HUANG H Y, et al. Study on the related genes expression of focal adhesion signal pathway in development of chicken muscle[J]. China Poultry, 2018, 40(17): 8–12. (in Chinese) |
| [37] | YAO X P, YU T Y, XI F X, et al. BAMBI shuttling between cytosol and membrane is required for skeletal muscle development and regeneration[J]. Biochem Biophys Res Commun, 2019, 509(1): 125–132. DOI: 10.1016/j.bbrc.2018.12.082 |
| [38] | BIAGINI E, OLIVOTTO I, IASCONE M, et al. Significance of sarcomere gene mutations analysis in the end-stage phase of hypertrophic cardiomyopathy[J]. Am J Cardiol, 2014, 114(5): 769–776. DOI: 10.1016/j.amjcard.2014.05.065 |
| [39] | SCHWÖRER S, BECKER F, FELLER C, et al. Epigenetic stress responses induce muscle stem-cell ageing by Hoxa9 developmental signals[J]. Nature, 2016, 540(7633): 428–432. DOI: 10.1038/nature20603 |
| [40] | LEE H J, GÖRING W, OCHS M, et al. Sox15 is required for skeletal muscle regeneration[J]. Mol Cell Biol, 2004, 24(19): 8428–8436. DOI: 10.1128/MCB.24.19.8428-8436.2004 |
| [41] | SAVAGE J, CONLEY A J, BLAIS A, et al. SOX15 and SOX7 differentially regulate the myogenic program in P19 cells[J]. Stem Cells, 2009, 27(6): 1231–1243. DOI: 10.1002/stem.57 |



