养猪业在我国畜牧业,甚至整个国民经济中都占着举足轻重的地位。经过长期发展,我国养猪业规模化程度越来越高,但是随着饲养密度增大,猪群的健康开始恶化,严重限制了生产效率,降低了猪场收益[1]。研究发现,中外猪种对疾病的抗性以及敏感性不同[2-3]。因此,通过对我国地方猪抗病机理的研究,可以提高猪的免疫能力、生活力及生产力[1]。
动物的免疫识别、对疾病的抵抗能力以及免疫系统的调节一定程度上依赖于主要组织相容性复合体(major histocompatibility complex, MHC,在猪中称为猪白细胞抗原(swine leukocyte antigen, SLA))[4-5]。SLA对感染疾病后的免疫反应以及对病毒的识别和清除具有很重要的作用[6-7]。SLA基因位于猪7号染色体着丝粒两端,分为Ⅰ、Ⅱ、Ⅲ类[8-9],具有高度多态性、单倍型遗传、连锁不平衡等特征[10]。SLAⅠ类基因主要分为3类:Ⅰa、Ⅰb和Ⅰc。Ⅰa包括SLA-1、SLA-2、SLA-3、SLA-4、SLA-5、SLA-9、SLA-11;Ⅰb包括SLA-6、SLA-7、SLA-8;Ⅰc包括MIC1和MIC2[11]。Ⅰa类基因中只有SLA-1、SLA-2、SLA-3具有完整的Ⅰ类基因结构,可编码SLAⅠ类分子。经典SLAⅠ类基因包含8个外显子,其中前导多肽链是由外显子1编码,α1-3功能区域分别由外显子2~4编码,跨膜区由外显子5编码,胞质区由外显子6~8编码。SLAⅠ类分子由重链和轻链(β2 m)组成,重链分为3个区,为胞浆区、跨膜区、胞外区。胞外区包含α1、α2和α3三个结构域,多态性主要集中在α1、α2[12]。α1、α2形成抗原肽结合槽,α1和α2功能区的α螺旋结构为该槽的两侧壁,底部为与α螺旋呈直角排列的8条反平行β片层链。肽结合槽底部有6个不同的口袋,决定了T淋巴细胞表位结合的特异性,不同的等位基因与不同类型的T细胞表位结合是由锚定残基和氨基酸共同决定的[13]。SLAⅠ类分子多态性位点与HLAⅠ类分子多态性位点几乎完全重叠,这些氨基酸多态性位点在HLAⅠ类分子中参与抗原肽递呈或与T细胞受体结合[14]。在MHC分子中,与抗原肽结合起递呈作用的主要是抗原识别位点(ARSs)。在人类MHC(HLA)Ⅰ类分子中,ARSs由57个氨基酸组成,分为3部分,即抗原结合槽、T细胞受体结合区、外侧的氨基酸残基,其侧链远离抗原结合槽及TCR。MHCⅠ类分子在疾病发生中的作用研究除了人和其他哺乳动物以及禽类,同样对鱼类、两栖类、鸟类、哺乳类的MHC的研究也有很多[15-23]。
我国地方猪资源丰富,高抗病力是地方猪种的重要特性。研究发现,在感染不同的病原体之后,中国地方猪种对疾病的敏感性比其他引入品种要强,具有较为明显的抗病力,其症状和病变也相对较轻[24-26]。一些中国地方猪种,如中国合作猪的SLAⅠ类分子的多态性要高于商品化白猪,这与合作猪对周围环境高度的适应性、耐粗饲、抗病性强等特点一致[27]。民猪是分布在中国东北地区的优良品种,具有免疫力较高、抗病性强等特征。为找出该猪种与大白猪经典SLAⅠ类基因及其编码蛋白的差异,为地方猪抗病力的研究提供分子基础,本研究选取了具有高抗病能力的地方猪种——民猪,与大白猪进行基因组重测序,拟分析不同品种经典SLAⅠ类基因的遗传多态性及对抗病潜能的影响,旨在为阐明民猪的抗病机理提供参考。
1 材料与方法 1.1 试验材料本研究所使用的样本均来自于中国农业科学院北京畜牧兽医研究所昌平种猪场,饲养条件、饲喂方式和饲料等均始终保持一致。试验样本包括8头民猪和4头大白猪的耳组织。
1.2 组织DNA提取及测序对大白猪和民猪的耳组织采用常规的酚仿法提取DNA,利用NanoDrop 1000 (Thermo Scientific, USA)测定DNA的浓度与质量。将质检合格的样品利用Illumina Hi-seq2500进行双末端测序,测序深度为5×~7×。
1.3 测序数据的质量控制与多态位点的检测和注释获得原始测序序列后,利用GATK软件中的Base Recalibration进行质控,对reads的碱基质量值进行校正。对接头序列及poly N,poly A等序列进行删除,将过滤后的reads数通过BWA比对到猪参考基因组(Sus scrofa 11.1),并进行SNP calling,进一步质控得到可用于后续分析的SNP,在ENSEMBL的VEP工具中导入SNP,使用Gene symbol、Transcript version、Transcript biotype、Transcript support level、APPRIS、MANE参数进行注释,并统计突变类型和数量。
1.4 SLAⅠ类基因单倍型块分析使用VCFtools中的-vcf、-out、-chr、-from-bp、-to-bp参数提取SNP后利用GEVALT[28]软件Gerbil模块中的-pedfile、-blockputput参数对SNP进行单倍型块分析[29]。利用R(R version 3.6.1)包的circlize进行单倍型图谱构建,进一步统计单倍型长度和数目作为单倍型结构的指标。
1.5 经典SLAⅠ类基因核苷酸序列比对和核苷酸多态性分析在Mega 7[30]软件中利用Clustal W进行分析,得到核苷酸以及氨基酸序列的比对结果。利用DnaSP 6.12[31]统计序列的核苷酸多态性。
1.6 经典SLAⅠ类基因编码蛋白质一般特性分析运用Expasy服务器上的ProtParam工具(https://web.expasy.org/protparam)对经典SLAⅠ类基因所编码蛋白质的理化性质进行预测和分析,同时利用Expasy服务器上的Protscale程序(https://web.expasy.org/protscale)进行疏水性、亲水性预测,分析不同突变对经典SLAⅠ类基因所编码蛋白质性质的影响。
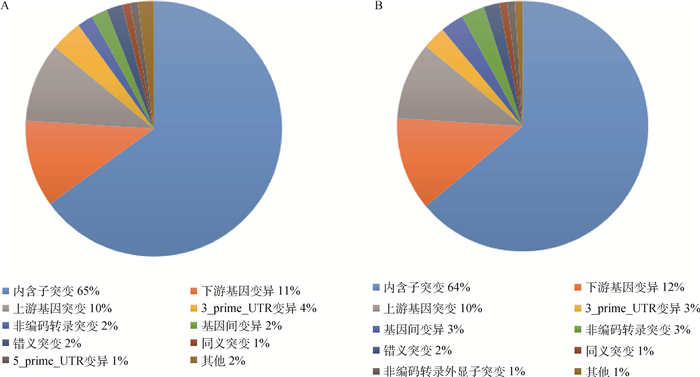
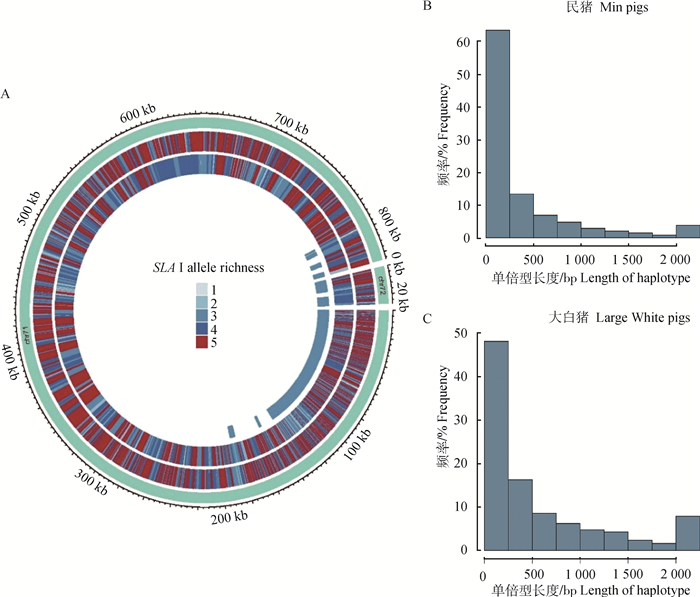
2 结果 2.1 民猪和大白猪SLAⅠ类基因的SNP和单倍型比较分析本研究使用单倍型块长度作为单倍型结构多样性的指标。本试验对民猪和大白猪的SLAⅠ类基因进行SNP和单倍型块的分析,其中单倍型块的长度越短、数量越多代表其多样性越高。表 1显示,民猪SLAⅠ类基因的SNP数量要多于大白猪。通过对两猪种SNP突变类型所占比例的分析发现,民猪中错义突变共248个占比为2%,内含子变异共8 069个占比65%(图 1A);大白猪共有196个SNP为错义突变占比2%,内含子变异共6 266个占比64%(图 1B)。通过对不同品种的单倍型长度及每一个单倍型块中单倍型个数的分析发现,民猪的SLAⅠ类基因平均单倍型个数(4.107)要多于大白猪的SLAⅠ类基因平均单倍型个数(3.547)(图 2A)。民猪中长度较短的单倍型所占比例(>60%,图 2B)要大于大白猪的占比(>50%,图 2C)。
|
|
表 1 东北民猪和大白猪SLAⅠ类基因单核苷酸多态性分布表 Table 1 Single nucleotide polymorphism distribution of SLA class Ⅰ genes of the Min pigs and Large White pigs |
|
A.民猪碱基突变类型所占比例;B.大白猪碱基突变类型所占比例 A. Proportion of base mutation types in Min pigs; B. Proportion of base mutation types in Large White pigs 图 1 民猪和大白猪SLAⅠ类基因突变类型统计图 Fig. 1 Statistics of mutation types of SLAⅠ gene in Min pigs and Large White pigs |
|
A.民猪和大白猪单倍型分布;B.民猪不同单倍型块长度所占比例;C.大白猪不同单倍型块长度所占比例 A.Haplotype distribution of Min pigs and Large White pigs; B. Proportion of different haplotype blocks length of Min pigs; C. Proportion of different haplotype blocks length of Large White pigs 图 2 民猪和大白猪SLAⅠ类基因的单倍型及单倍型块长度百分比 Fig. 2 The percentage of haplotypes and haplotype blocks length of the SLA classⅠ genes of Min pigs and Large White pigs |
本研究对基因SLA-1、SLA-2和SLA-3等位基因序列进行分析,并计算核苷酸多样性(Pi),其结果如表 2所示。对于3个经典SLAⅠ类基因,民猪的核苷酸多样性都要高于大白猪,说明民猪的遗传多样性高于大白猪。
|
|
表 2 SLA-1、SLA-2、SLA-3基因的核苷酸多态性 Table 2 Nucleotide polymorphisms of SLA-1, SLA-2 and SLA-3 genes |
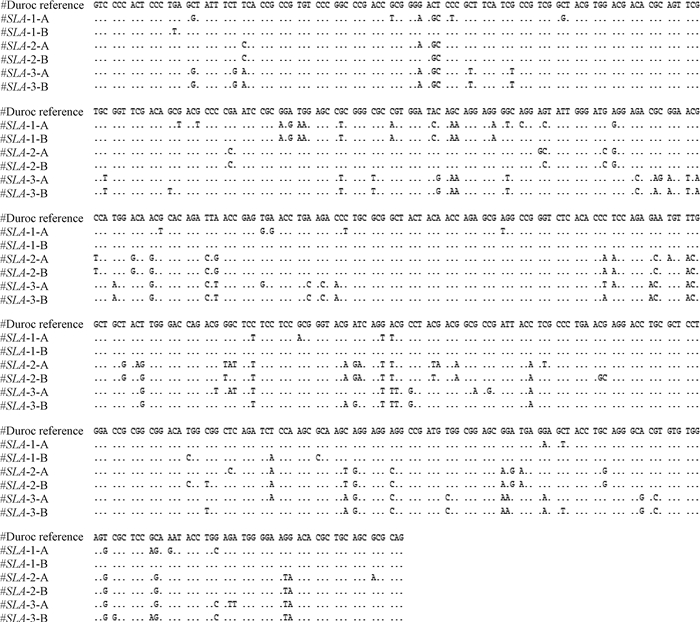
对民猪和大白猪12个样本所测得的SLA-1、SLA-2、SLA-3的第2和3外显子的核苷酸序列与杜洛克猪基因序列进行多态性比较分析。结果表明,在3个基因中民猪的碱基突变及位于抗原结合位点的氨基酸突变数量都要多于大白猪的数量。
2.3.1 两猪种SLA-1基因及氨基酸多态性比较 在SLA-1基因中共发现了2种等位基因。民猪中共有39个碱基发生突变,共导致10个氨基酸发生改变,其中有4个位于抗原结合位点上;大白猪中共有14个碱基发生突变,共有3个氨基酸发生改变,并无氨基酸位于抗原结合位点上(图 3和图 4)。SLA-1基因所编码的20种氨基酸中,精氨酸(Arg)数目最多,在民猪中占整个氨基酸组成的18.9%,在大白猪中占18.4%。民猪负电荷残基总数(Asp+ Glu)为20,正电荷残基总数(Arg+Lys)为34,大白猪分别为20和33,民猪和大白猪中编码产物的不稳定指数分别为76.24和80.37,不稳定指数大于40,则两品种猪SLA-1基因编码产物都是不稳定的。氨基酸序列亲水性/疏水性预测结果如图 5A和5B所示,民猪第73位的苏氨酸和第74位的丙氨酸分值最低(-2.989),亲水性最强,第161位的丝氨酸疏水性最强(1.211);大白猪中第26位的半胱氨酸亲水性最强(-2.833),第160位的丝氨酸疏水性最强(1.211)。在整条氨基酸肽链中,民猪的总平均亲水性为-0.931,大白猪的总平均亲水性为-0.901,二者都表现为亲水性。
|
A.民猪; B. 大白猪, 图 4同。圆点代表碱基相同,字母代表碱基发生突变 A. Min pigs; B. Large White pigs, the same as figure 4. Dots indicate the same base as reference, and letters show substitutions of base 图 3 SLA-1、SLA-2、SLA-3等位基因外显子2和3序列比对 Fig. 3 Alignment of the SLA-1, SLA-2, SLA-3 allele sequences in exon 2 and 3 |

|
圆点代表氨基酸相同,字母代表氨基酸发生改变, 星号表示抗原结合位点 Dots indicate the same amino acid as reference, letters show substitutions of amino acid, and the putative 图 4 SLA-1、SLA-2、SLA-3基因第2、3外显子编码氨基酸序列比对 Fig. 4 Alignment of the SLA-1, SLA-2, SLA-3 amino acid sequences in exon 2 and 3 |

|
A. 民猪SLA-1; B. 大白猪SLA-1; C. 民猪SLA-2; D.大白猪SLA-2; E.民猪SLA-3; F.大白猪SLA-3 A. SLA-1 of Min pigs; B. SLA-1 of Large White pigs; C. SLA-2 of Min pigs; D. SLA-2 of Large White pigs; E. SLA-3 of Min pigs; F. SLA-3 of Large White pigs 图 5 SLA-1、SLA-2、SLA-3蛋白的疏水性预测结果 Fig. 5 The hydrophobicity profile of SLA-1, SLA-2, SLA-3 analyzed by ProtScale program |
2.3.2 两猪种SLA-2基因及氨基酸多态性比较 在SLA-2基因中共发现了2种等位基因。民猪中共有25个碱基发生突变,共导致18个氨基酸发生改变,其中有3个位于抗原结合位点上;大白猪中共有17个碱基发生突变,共有13个氨基酸发生改变,并无氨基酸位于抗原结合位点上(图 3和图 4)。SLA-2基因所编码的20种氨基酸中,丙氨酸在民猪中占比最多为9.4%,精氨酸在大白猪中占比最多为9.4%,半胱氨酸占比最低,在两种猪中均为1.1%,民猪负电荷残基总数(Asp+Glu)为24,正电荷残基总数(Arg+Lys)为22,大白猪分别为26和22,民猪和大白猪的氨基酸理论等电点(PI)分别为5.97和5.27,编码产物的不稳定指数分别为24.88和32.02,不稳定指数小于40,则两品种猪SLA-2基因编码产物都是稳定的。氨基酸序列亲水性/疏水性预测结果如图 5C和图 5D所示,民猪第16位的丙氨酸分值最低(-2.811),亲水性最强,第23位的甘氨酸疏水性最强(1.078)。大白猪第60位的亮氨酸亲水性最强(-2.878),同时第105位的亮氨酸疏水性最强(1.089)。在整条氨基酸肽链中,民猪的总平均亲水性为-0.835,大白猪的总平均亲水性为-0.823,二者都表现为亲水性。
2.3.3 两猪种SLA-3基因及氨基酸多态性比较 在SLA-3基因中共发现了2种等位基因。对于民猪,共有39个碱基发生突变,共导致9个氨基酸发生改变,其中有7个位于抗原结合位点上;对于大白猪,共有30个碱基发生突变,共有8个氨基酸发生改变,有2个氨基酸位于抗原结合位点上(图 3和图 4)。SLA-3基因所编码的20种氨基酸中,精氨酸(Arg)数目最多,在民猪中占整个氨基酸组成的19.6%,在大白猪中占19.00%。民猪负电荷残基总数(Asp+Glu)为18,正电荷残基总数(Arg+Lys)为36,大白猪分别为18和35,民猪和大白猪的氨基酸理论等电点(PI)分别为11.82和11.79,编码产物的不稳定指数分别为76.44和79.12,不稳定指数大于40,则两品种猪SLA-3基因编码产物都是不稳定的。氨基酸序列亲水性/疏水性预测结果如图 5E和图 5F所示,民猪和大白猪第121位的半胱氨酸和第122位的丝氨酸分值最低(-3.000),亲水性最强。民猪和大白猪第166位的甘氨酸疏水性最强(1.122)。在整条氨基酸肽链中,民猪的总平均亲水性为-1.000,大白猪的总平均亲水性为-0.965,二者都表现为亲水性。
3 讨论在2006年全国蓝耳病爆发期间,某项调查数据显示,莱芜猪、鲁莱黑猪、莱芜杂交二元母猪的死亡率较低,分别为0、0、0.6%,但是国外品种母猪的死亡率则高达54%,说明我国地方猪的抗病能力要高于外种猪,暗示中外猪种的免疫能力可能存在差异。民猪与莱芜黑猪同属黄淮海黑猪,是我国优良的地方品种,同样具有抗病力强的特点[32]。本试验从全局层面阐述了中外猪种不同SLAⅠ类基因多样性,对抗病力较强的地方猪——民猪和大白猪SLAⅠ类基因的SNP以及发生错义突变的SNP进行统计分析,发现民猪的数量都要高于大白猪,这可能使多肽链的氨基酸种类和序列发生改变。本试验还对单倍型进行分析,发现民猪具有更多长度较短的单倍型块,并且平均单倍型个数要高于大白猪,表明民猪的单倍型复杂程度更高,排列的方式更多,具有丰富的多样性[33]。
MHCⅠ类分子都含两条独立的多肽链,一条为MHC编码的α或重链,另一条为非MHC编码的β链。MHCⅠ类分子包含4个独立区:氨基末端细胞外肽结合区、细胞外免疫球蛋白样区、穿膜区和胞浆区。SLAⅠ类基因具有高度丰富的多态序列区域主要集中在编码区的第2和第3外显子[34],其主要编码SLAⅠ类分子的α1和α2区域[35](氨基末端细胞外肽结合区),而α3区域则是高度保守的。编码SLA抗原的基因具有丰富的多态性,多等位基因决定了多种SLA分子,其可结合不同的蛋白质抗原肽。刘建欣和郑昌学[36]的研究发现,MHC基因复合体多态性位点是由基因重复产生的。新的位点形成以后,不再受原功能限制,因而可以产生新的功能。Ando等[37-38]在SLA-1、SLA-2、SLA-3外显子1、2、3中发现了等位基因多样化,并且导致其编码的SLAⅠ类分子的α1和α2区的氨基酸呈现高度多态性。核苷酸多样性是衡量群体遗传多样性的重要指标,该值越高表示群体遗传变异程度越高,对不同的环境具有很强的适应能力[39]。刘德武等[40]对广东蓝塘猪、大花白猪及4个引入品种的群体遗传结构进行分析,结果发现,地方品种的群体内遗传变异要高于引入品种。本研究对民猪和大白猪SLA-1、SLA-2、SLA-3的核苷酸多样性进行分析,民猪3个基因的核苷酸多样性均高于大白猪。结果表明,民猪的遗传多样性优于大白猪。可能是因为不同猪种所生长的环境不同,从而具有不同的抵抗能力,导致具有不同的遗传多态性[41]。
MHC多态性明显表现出适应性意义,如抗病性和免疫应答能力增强,对环境适应性能力的提高等[27]。SLAⅠ类分子中参与抗原肽结合的主要是抗原识别位点(ARSs),由α1和α2区两个α螺旋结构及一个β片层结构构成。本试验中,民猪和大白猪经典SLAⅠ类基因在3个基因位点都高度杂合,SLA-1、SLA-2、SLA-3基因外显子2、3序列上的碱基突变表现出了多态性,具有极为丰富的变异。在民猪和大白猪中所检测到的SLA-1、SLA-2、SLA-3等位基因各有2个,表现出等位基因多样化,民猪碱基突变的数量均多于大白猪,编码的氨基酸位于抗原结合位点的数量也均多于大白猪,这可能是民猪的抗病力比大白猪强的原因之一。同样,这些变异位点也可以为猪抗病能力的分子标记辅助选择育种提供新的候选分子标记。
MHCⅠ类分子几乎表达于所有有核细胞表面[42],其多态性位点主要分布在与肽结合的区域,使得MHCⅠ类分子具有不同的抗原递呈特征。本研究对SLA-1、SLA-2、SLA-3基因编码产物氨基酸的组成、疏水性预测分析的结果对其蛋白质的稳定性、构象和功能的研究具有重要意义,是进一步研究该基因作为疾病候选基因可能性的基础。对于SLA-1、SLA-3基因,两个猪种编码产物均为不稳定蛋白,且大白猪的不稳定性要高于民猪;对于SLA-2,两个猪种编码产物均为稳定蛋白。两个猪种的SLA-1、SLA-2、SLA-3氨基酸均表现为亲水性,编码产物均为可溶性蛋白,并且民猪的SLA-1、SLA-2、SLA-3氨基酸亲水性都要比大白猪强,民猪3个基因编码产物具有较高的亲水性可能是由于较高的氨基酸变异率造成的。
4 结论本研究通过对民猪和大白猪SLAⅠ类基因的研究,发现民猪具有更强的多态性。同时,本研究对经典SLAⅠ类基因的外显子2和3进行分析,发现民猪的碱基突变以及位于抗原结合位点上氨基酸突变的数量都要多于大白猪。结果表明,民猪的SLAⅠ类基因具有更加丰富的多态性。本研究结果为分析不同猪种SLAⅠ类基因的遗传多态性提供了理论基础。
| [1] | WANG J Y, WANG Y P, WANG H Z, et al. Transcriptomic analysis identifies candidate genes and gene sets controlling the response of porcine peripheral blood mononuclear cells to Poly Ⅰ: C Stimulation[J]. G3 (Bethesda), 2016, 6(5): 1267–1275. DOI: 10.1534/g3.116.028290 |
| [2] | LUNNEY J K, CHEN H B. Genetic control of host resistance to porcine reproductive and respiratory syndrome virus (PRRSV) infection[J]. Virus Res, 2010, 154(1-2): 161–169. DOI: 10.1016/j.virusres.2010.08.004 |
| [3] | YANG H. Livestock development in China: animal production, consumption and genetic resources[J]. J Anim Breed Genet, 2013, 130(4): 249–251. DOI: 10.1111/jbg.12045 |
| [4] | LE M T, CHOI H, LEE H, et al. SLA-1 genetic diversity in pigs: extensive analysis of copy number variation, heterozygosity, expression, and breed specificity[J]. Sci Rep, 2020, 10(1): 743. DOI: 10.1038/s41598-020-57712-5 |
| [5] |
奚衍洋, 赵云蛟, 钱爱东. 猪SLA免疫学相关研究进展[J]. 黑龙江畜牧兽医, 2018(3): 70–72.
XI Y Y, ZHAO Y J, QIAN A D. Immunological progress of swine major histocompatibility complex[J]. Heilongjiang Animal Science and Veterinary Medicine, 2018(3): 70–72. (in Chinese) |
| [6] | PARK J Y, KIM H S, SEO S H. Characterization of interaction between porcine reproductive and respiratory syndrome virus and porcine dendritic cells[J]. J Microbiol Biotechnol, 2008, 18(10): 1709–1716. |
| [7] | WANG X, EATON M, MAYER M, et al. Porcine reproductive and respiratory syndrome virus productively infects monocyte-derived dendritic cells and compromises their antigen-presenting ability[J]. Arch Virol, 2007, 152(2): 289–303. DOI: 10.1007/s00705-006-0857-1 |
| [8] |
訾臣, 刘璐, 包文斌. SLA-DQA基因在F18大肠杆菌抗性和敏感性断奶仔猪间的差异表达分析[J]. 畜牧兽医学报, 2012, 43(4): 516–520.
ZI C, LIU L, BAO W B. The differential expression of SLA-DQA gene between weaning piglets resistant or sensitive to Escherichia coli F18[J]. Acta Veterinaria et Zootechnica Sinica, 2012, 43(4): 516–520. (in Chinese) |
| [9] |
包文斌, 吴圣龙, 曹晶晶, 等. 19个猪种SLA-DQB基因外显子2多态性分析[J]. 畜牧兽医学报, 2009, 40(10): 1550–1554.
BAO W B, WU S L, CAO J J, et al. Polymorphism analysis on exon 2 of SLA-DQB gene in 19 pig populations[J]. Acta Veterinaria et Zootechnica Sinica, 2009, 40(10): 1550–1554. DOI: 10.3321/j.issn:0366-6964.2009.10.020 (in Chinese) |
| [10] | HAMMER S E, HO C S, ANDO A, et al. Importance of the Major histocompatibility complex (swine leukocyte antigen) in swine health and biomedical research[J]. Annu Rev Anim Biosci, 2020, 8: 171–198. DOI: 10.1146/annurev-animal-020518-115014 |
| [11] | RENARD C, VAIMAN M, CHIANNILKULCHAI N, et al. Sequence of the pig major histocompatibility region containing the classical class Ⅰ genes[J]. Immunogenetics, 2001, 53(6): 490–500. DOI: 10.1007/s002510100348 |
| [12] | CHARDON P, RENARD C, VAIMAN M. The major histocompatibility complex in swine[J]. Immunol Rev, 1999, 167(1): 179–192. DOI: 10.1111/j.1600-065X.1999.tb01391.x |
| [13] | FROMMER L, FLESCH B K, KNIG J, et al. Amino acid polymorphisms in Hla class Ⅱ differentiate between thyroid and polyglandular autoimmunity[J]. J Cli Endocrinol Metab, 2020, 105(6): 1737–1747. DOI: 10.1210/clinem/dgz164 |
| [14] |
韦思婷, 程一飞, 刘丽霞. 猪SLA基因多态性研究进展[J]. 猪业科学, 2019, 36(4): 102–103.
WEI S T, CHENG Y F, LIU L X. Research progress of porcine SLA gene polymorphism[J]. Swine Industry Science, 2019, 36(4): 102–103. DOI: 10.3969/j.issn.1673-5358.2019.04.030 (in Chinese) |
| [15] | HUANG Z Y, YU Y Y, DONG S, et al. Major histocompatibility complex class ⅡA and ⅡB genes of loach (Misgurnus anguillicaudatus): Molecular cloning and expression analysis in response to bacterial and parasitic challenge[J]. Aquaculture, 2019, 500: 359–369. DOI: 10.1016/j.aquaculture.2018.10.043 |
| [16] | CORTÁZAR-CHINARRO M, MEYER-LUCHT Y, LAURILA A, et al. Signatures of historical selection on MHC reveal different selection patterns in the moor frog (Rana arvalis)[J]. Immunogenetics, 2018, 70(7): 477–484. DOI: 10.1007/s00251-017-1051-1 |
| [17] | KIM T, HUNT H D, PARCELLS M S, et al. Two class Ⅰ genes of the chicken MHC have different functions: BF1 is recognized by NK cells while BF2 is recognized by CTLs[J]. Immunogenetics, 2018, 70(9): 599–611. DOI: 10.1007/s00251-018-1066-2 |
| [18] | MORGAN R A, KARL J A, BUSSAN H E, et al. Restricted MHC class Ⅰ A locus diversity in olive and hybrid olive/yellow baboons from the Southwest National Primate Research Center[J]. Immunogenetics, 2018, 70(7): 449–458. DOI: 10.1007/s00251-018-1057-3 |
| [19] | YU S, WU J, BAI J, et al. Polymorphic analysis of peptide binding domain of major histocompatibility complex class Ⅰ in domestic ducks[J]. Pol J Vet Sci, 2019, 22(2): 415–422. |
| [20] | LI Z B, ZHANG N, MA L Z, et al. Distribution of ancient α1 and α2 domain lineages between two classical MHC class Ⅰ genes and their alleles in grass carp[J]. Immunogenetics, 2019, 71(5-6): 395–405. DOI: 10.1007/s00251-019-01111-2 |
| [21] | KIEMNEC-TYBURCZY K M, TRACY K E, LIPS K R, et al. Genetic variation and selection of MHC class Ⅰ loci differ in two congeneric frogs[J]. Genetica, 2018, 146(2): 125–136. DOI: 10.1007/s10709-018-0016-0 |
| [22] | SAVAGE A E, MULDER K P, TORRES T, et al. Lost but not forgotten: MHC genotypes predict overwinter survival despite depauperate MHC diversity in a declining frog[J]. Conservat Genet, 2018, 19(2): 309–322. DOI: 10.1007/s10592-017-1001-3 |
| [23] |
陶久臣. 东北林蛙MHC基因的多态性及其遗传进化[D]. 哈尔滨: 哈尔滨师范大学, 2019.
TAO J C. Polmorphism and genetic evolution of MHC class genes in Rana Dybowskii[D]. Harbin: Harbin Normal University, 2019. (in Chinese) |
| [24] | LIANG W, LI Z H, WANG P, et al. Differences of immune responses between Tongcheng (Chinese local breed) and Large White pigs after artificial infection with highly pathogenic porcine reproductive and respiratory syndrome virus[J]. Virus Res, 2016, 215: 84–93. DOI: 10.1016/j.virusres.2016.02.004 |
| [25] | XING J Y, XING F, ZHANG C H, et al. Genome-wide gene expression profiles in lung tissues of pig breeds differing in resistance to porcine reproductive and respiratory syndrome virus[J]. PLoS One, 2014, 9(1): e86101. DOI: 10.1371/journal.pone.0086101 |
| [26] | JIANG C L, XING F, XING J Y, et al. Different expression patterns of PRRSV mediator genes in the lung tissues of PRRSV resistant and susceptible pigs[J]. Dev Comp Immunol, 2013, 39(1-2): 127–131. DOI: 10.1016/j.dci.2012.01.003 |
| [27] |
莫斯科. 中国合作猪SLAⅠ类和Ⅱ类分子的克隆及基因结构与多态性分析[D]. 兰州: 甘肃农业大学, 2009.
MO S K. Cloning and analysis of genomic structure and genetic polymorphism of swine lymphocyte antigen class Ⅰ and Ⅱ molecules in Chinese Hezuo pig[D]. Lanzhou: Gansu Agricultural University, 2009. (in Chinese) |
| [28] | DAVIDOVICH O, KIMMEL G, SHAMIR R. GEVALT: An integrated software tool for genotype analysis[J]. BMC Bioinform, 2007, 8(1): 36. DOI: 10.1186/1471-2105-8-36 |
| [29] | KIMMEL G, SHAMIR R. GERBIL: Genotype resolution and block identification using likelihood[J]. Proc Natl Acad Sci U S A, 2005, 102(1): 158–162. DOI: 10.1073/pnas.0404730102 |
| [30] | FILIPSKI A, MURILLO O, FREYDENZON A, et al. Prospects for building large timetrees using molecular data with incomplete gene coverage among species[J]. Mol Biol Evol, 2014, 31(9): 2542–2550. DOI: 10.1093/molbev/msu200 |
| [31] | ROZAS J, FERRER-MATA A, SÁNCHEZ-DELBARRIO J C, et al. DnaSP 6:DNA sequence polymorphism analysis of large data sets[J]. Mol Biol Evol, 2017, 34(12): 3299–3302. DOI: 10.1093/molbev/msx248 |
| [32] |
张冬杰, 刘娣. 民猪种质资源特点及研究现状[J]. 黑龙江农业科学, 2019(2): 48–50.
ZHANG D J, LIU D. Germplasm resources characteristics and research status of min pig[J]. Heilongjiang Agricultural Sciences, 2019(2): 48–50. (in Chinese) |
| [33] | FENG S H, FANG Q, BARNETT R, et al. The genomic footprints of the fall and recovery of the crested ibis[J]. Curr Biol, 2019, 29(2): 340–349.e7. DOI: 10.1016/j.cub.2018.12.008 |
| [34] | KOSCH T A, EIMES J A, DIDINGER C, et al. Characterization of MHC class ⅠA in the endangered southern corroboree frog[J]. Immunogenetics, 2017, 69(3): 165–174. DOI: 10.1007/s00251-016-0965-3 |
| [35] |
边若菲, 徐笑, 刘玉芬, 等. 嗜水气单胞菌胁迫下东北林蛙MHCⅠ基因在不同组织的表达[J]. 生物工程学报, 2020, 36(7): 1323–1333.
BIAN R F, XU X, LIU Y F, et al. Expression of MHC Ⅰ genes in different tissues of Rana dybowskii under the stress of Aeromonas hydrophila[J]. Chinese Journal of Biotechnology, 2020, 36(7): 1323–1333. (in Chinese) |
| [36] |
刘建欣, 郑昌学.
现代免疫学: 免疫的细胞和分子基础[M]. 北京: 清华大学出版社, 2002.
LIU J X, ZHENG C X. Modern immunology: the cellular and molecular basis of immunity[M]. Beijing: Tsinghua University Press, 2002. (in Chinese) |
| [37] | ANDO A, KAWATA H, SHIGENARI A, et al. Genetic polymorphism of the swine major histocompatibility complex (SLA) class Ⅰ genes, SLA-1, -2 and -3[J]. Immunogenetics, 2003, 55(9): 583–593. DOI: 10.1007/s00251-003-0619-0 |
| [38] |
陶璇. SLAⅠ类经典基因多态性及正选择检验分析[D]. 成都: 四川农业大学, 2008.
TAO X. Polymorphisms Analysis and detecting positive selection of SLA classic class Ⅰ genes[D]. Chengdu: Sichuan Agricultural University, 2008. (in Chinese) |
| [39] | DUNNER S, CHECA M L, GUTIERREZ J P, et al. Genetic analysis and management in small populations: the Asturcon pony as an example[J]. Genet Sel Evol, 1998, 30(4): 397–405. DOI: 10.1186/1297-9686-30-4-397 |
| [40] |
刘德武, 杨关福, 李加琪, 等. 用RAPD标记分析6个品种猪的群体遗传结构[J]. 畜牧兽医学报, 2002, 33(1): 18–22.
LIU D W, YANG G F, LI J Q, et al. Analysing genetic construction of six pig breeds using RAPD marker[J]. Acta Veterinaria et Zootechnica Sinica, 2002, 33(1): 18–22. DOI: 10.3321/j.issn:0366-6964.2002.01.004 (in Chinese) |
| [41] |
张国华. 合作猪SLA-DQA第2、4外显子多态性分析[D]. 兰州: 甘肃农业大学, 2014.
ZHANG G H. Analyzing polymorphism of Hezuo swine SLA-DQA gene exon 2, 4[D]. Lanzhou: Gansu Agricultural University, 2014. (in Chinese) |
| [42] |
杨汉春.
动物免疫学[M]. 2版. 北京: 中国农业大学出版社, 2003.
YANG H C. Animal immunology[M]. 2nd ed. Beijing: China Agricultural University Press, 2003. (in Chinese) |



