2. 南京农业大学动物科技学院, 南京 210095;
3. 江苏省家禽遗传育种重点实验室, 扬州 225125
2. College of Animal Science and Technology, Nanjing Agricultural University, Nanjing 210095, China;
3. Key Laboratory for Poultry Genetics and Breeding of Jiangsu Province, Yangzhou 225125, China
线粒体DNA(mitochondrial DNA,mtDNA)为动物核外遗传物质,发生变异的机率相对稳定,无遗传重组,具有从母系起源上确定品种间遗传关系的特点,通常能够全面反映种群内和种群间的遗传变异[1],已成为探讨种群遗传结构、系统发育进化及种质鉴定研究的重要研究对象[2-4]。线粒体控制区又称D-环区(D-loop区),是一段非编码区,其进化速率较线粒体其它区域快5~10倍,是研究亲缘关系较近群体的起源进化和种质鉴定理想的分子标记[5-8],目前已成功应用于猪[9-11]、牛[12-14]、羊[15-17]、马[18-20]、驴[21-23]、鹿[24]和鸽[25]等多个物种的遗传多样性和系统发育研究。近年来,在对我国地方鸡种遗传多样性和母系起源方面的研究显示,线粒体D-loop区作为目标位点已逐渐成为研究热点[26-28]。黄勋和等[29]基于线粒体DNA D-loop序列研究了广东省和邻省共12个地方鸡品种间的遗传距离和系统关系,系统评估了其遗传变异水平并追溯了其母系起源。贾晓旭等[30]基于线粒体DNA控制区序列研究了我国华东地区11个地方鸡品种的遗传多样性与亲缘关系,并构建了其与红色原鸡系统发生的邻接树,探讨了11个品种的母系起源。
随着对线粒体基因组序列研究的关注热度越来越高,在探讨基于线粒体DNA的物种遗传多样性和起源进化的同时,对线粒体单倍型(世系)的划分也有相关研究。Liu等[31]以线粒体控制区第一高变区为遗传标记,深入研究了家鸡的母系起源,揭示世界家鸡和红色原鸡由9个母系世系构成,分别为A、B、C、D、E、F、G、H和I型。研究显示,94.84%的家鸡属于A、B、C、E、F、G和I型,其中A和B型普遍存在于东亚和东南亚地区,C型为东亚特有,E型普遍存在于南亚和欧美地区,F和G型仅分布在中国云南地区,I型主要分布在中国云南地区。Miao等[32]构建了基于家鸡线粒体全基因组序列的系统发育树,详细界定了各单倍型类群,并对世界各地家鸡样本mtDNA控制区序列进行了重新分析,确定了世界家鸡及其近缘种红色原鸡可分为A、B、C、D、E、F、G、H、I、W和X等世系。然而,这些研究的关注点基本集中于物种的遗传多样性和起源进化,即在不同单倍型的来源方面取得了诸多成就。但是很少有人关注不同单倍型究竟去了哪里,即对杂交后代(配套系)中单倍型的分布情况研究少之又少,迄今为止,对线粒体优势单倍型在后代的分布和传递情况以及与鸡生产性能相关性研究甚少,还未见相关报道。
本研究拟通过研究不同杂交组合鸡群体中线粒体控制区遗传多样性特点和单倍型分布特征,探讨线粒体优势单倍型在不同世代间的传递规律,并通过与GenBank数据库中红色原鸡序列的聚类分析探究其遗传起源,为肉鸡品种选育和溯源以及资源化开发利用提供理论依据和技术支撑。
1 材料与方法 1.1 试验材料以固始鸡、藏鸡、隐性白羽鸡、正交F1代、反交F1代以及F2代等6个鸡群共387只个体为试验材料(表 1),鸡种来源于国家级地方鸡种基因库(江苏),翅静脉采血,ACD抗凝。其中正交F1代为固始鸡(♂)×隐性白羽鸡(♀)、反交F1代为隐性白羽鸡(♂)×固始鸡(♀)、F2代为藏鸡(♂)×正交F1代(♀)。
|
|
表 1 6个鸡群体采样数 Table 1 The sample size of 6 chicken populations |
1.2.1 总DNA的提取 采用常规酚/氯仿法[33]提取血样基因组DNA,利用0.8%琼脂糖(Promega,美国)凝胶电泳检测DNA提取效果。
1.2.2 引物合成 利用Primer Premier 5.0软件根据GenBank发布的红色原鸡线粒体控制区全序列(序列号: NC_007235)设计特异性引物1对,PF:5′-AAACACCCAAACTCACTAAC-3′;PR:5′- CACTGGGATGCGGATACTTGC-3′,预期扩增产物全长为1 586 bp。引物由生工生物工程(上海)股份有限公司合成。
1.2.3 PCR扩增和测序 扩增体系50 μL:2×PCR Master Mix(大连宝生物公司)25 μL,10 μmol·L-1上、下游引物(上海生工生物工程公司)各1.5 μL,DNA模板2 μL,灭菌双蒸水20 μL。扩增条件:95 ℃预变性5 min;95 ℃变性30 s,56 ℃退火60 s,72 ℃延伸2 min,共32个循环;72 ℃延伸10 min。扩增产物采用1.0%琼脂糖(Promega,美国)凝胶电泳检测,之后选择扩增效果良好的产物交由生工生物工程(上海)股份有限公司完成测序,所有序列均采用双向一代Sanger测序。
1.3 数据处理与分析采用DNAStar 5.02软件SeqMan程序拼接序列,剪去多余片段,然后与参考序列进行逐碱基比对。利用MEGA 4.0软件计算遗传距离,构建基于双参数遗传距离的系统发育树(neighbor-jointing, NJ)[34]。利用DnaSP 5.10软件统计突变位点总数(total number of mutations)、单倍型数(number of haplotype)、单倍型多样度(haplotype diversity, Hd)、核苷酸多样度(nucleotide diversity, Pi)以及平均核苷酸差异(average number of nucleotide differences, K)等遗传多样性信息[35]。利用Network 10.1软件构建中介网络关系图(median- joining)(www.fluxus-technology.com)。
2 结果 2.1 线粒体控制区序列分析统计分析6个群体387个个体PCR产物测序结果,发现所研究鸡样本线粒体控制区全序列大小为1 231 bp,与参考序列NC_007237比对显示,所有个体均在859 bp处存在C碱基缺失。多态性分析显示,387条序列共检测到28个单核苷酸突变位点,其中单一多态位点3处,分别位于211、321和927 bp处;其余突变位点均为简约信息位点。突变类型以转换为主,其中T-C转换20处,A-G转换8处。碱基组成分析显示,T、C、A和G碱基含量分别为33.5%、26.6%、26.5%和13.4%,A+T含量为60.0%,G+C含量为40.0%。
2.2 线粒体控制区单倍型分析6个群体单倍型分析显示,28个突变位点共组成19种单倍型,可以划分为A、B、C、E 4个单倍型群(分支),其中A单倍型群包括7个单倍型共116个个体,B单倍型群包括4个单倍型共36个个体,C单倍型群包括5个单倍型共132个个体,E单倍型群包括3个单倍型共103个个体。各单倍型数量及在群体间的分布情况见表 2。其中,单倍型C1出现频率最高,在5个群体中共出现122个个体;其次是单倍型E1,在3个群体中共出现92次;第三是单倍型A1,在4个群体中共出现72次;这3种单倍型共计286个个体,占样品总数的73.9%。
|
|
表 2 6个群体线粒体D-loop区单倍型数量分布 Table 2 The number of mtDNA D-loop region haplotypes in 6 chicken populations |
进一步分析显示,固始鸡(GS)主要有A、C单倍型,占比分别为53.42%和46.58%,反交F1代(YG)与GS一样,主要有A、C单倍型,占比分别为50.75%和49.25%。正交F1代(GY)、F2代和隐性白羽鸡(YXB)的A、B、C、E四种单倍型占比相近,E单倍型为这3个群体的优势单倍型,占比分别为48.84%、50.00%和48.89%。结果见表 3。
|
|
表 3 不同群体D-loop区全序列单倍型汇总 Table 3 Summary of haplotypes in different populations based on complete sequence of D-loop region |
6个鸡群体遗传多样性分析结果见表 4,平均核苷酸差异(K)分布在4.192~6.655之间,其中最高为正交F1代(GY),最低为藏鸡(Z);核苷酸多样度(Pi)分布在0.003 40~0.005 41之间,其中最高为GY,最低为Z;单倍型多样度(Hd)分布在0.496~0.729之间,其中最高为GY,最低为Z。反交F1代(YG)与固始鸡(GS)遗传多样性相近,GY和F2代与YXB遗传多样性相近。
|
|
表 4 6个鸡群体线粒体控制区的遗传多样性 Table 4 The genetic diversity of mtDNA control region in 6 chicken populations |
6个鸡群体平均遗传距离结果见表 5,种群内遗传距离范围为0.003 4~0.005 5,最小为藏鸡(Z),最大为正交F1代(GY)。种间遗传距离大小体现了鸡种之间亲缘关系的远近,最大为Z与隐性白羽鸡(YXB)、GY、F2之间(均为0.009 1);最小为固始鸡(GS)与反交F1代(YG)之间(0.004 3);YXB、GY以及F2代两两之间遗传距离相近。
|
|
表 5 基于Kimura双参数模型计算的6个群体平均遗传距离 Table 5 The mean genetic distance of the 6 chicken populations calculated by Kimura-2-parameter model |
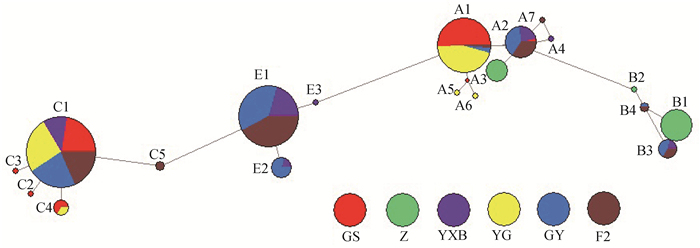
对6个群体387个个体19种单倍型构建了NJ系统进化树,结果如图 1所示,19种单倍型明显地被分成了A、B、C和E等4个支系,分别含有116、36、132和103条序列。用Network10.1软件构建的网络关系图也证实了这一点,如图 2所示,中介网络图主要有4个进化枝,分别以A1、B1、C1和E1为中心节点,这4种单倍型在各自的单倍型类群里出现的频率最高。

|
图 1 基于NJ法构建的6个鸡群体线粒体控制区不同单倍型系统发育树 Fig. 1 Neighbor-joining tree among different haplotypes of 6 chicken populations based on mitochondrial D-loop region complete sequence |
|
图 2 6个鸡群体mtDNA控制区19种单倍型Network图 Fig. 2 Median-joining (MJ) network showing genetic relationships among 19 haplotypes for mtDNA D-loop region of 6 chicken populations |
同时,结合本研究中387条序列19种单倍型和从GenBank数据库下载的19条红色原鸡线粒体控制区全长序列,采用K2P模型构建了系统发育树,如图 3所示,单倍型A、B、C和E在发育树中形成了4个大分枝,2个原鸡海南亚种个体GGJ1和GGJ2单独形成一枝。7种A单倍型和4种B单倍型仅与原鸡滇南亚种交叉聚为一枝,其中A单倍型群均与滇南亚种GGS5(GU261695)聚为一枝;B单倍型群均与滇南亚种GGS1(NC007235)和GGS8(GU261704)2条序列交叉聚为一枝。3种E单倍型均与红色原鸡印度亚种(GGM1、GGM3和GGM4)交叉聚为一枝。5种C单倍型与印尼亚种(GGB)、指名亚种(GGG1、GGG2)、印度亚种(GGM2)以及滇南亚种(GGS10、GGS3、GGS9)等4种红色原鸡亚种7条序列交叉聚为一枝。

|
GGG. 原鸡指名亚种;GGS. 原鸡滇南亚种;GGM. 原鸡印度亚种;GGJ. 原鸡海南亚种;GGB. 原鸡印尼亚种 GGG. Gallus gallus gallus; GGS. Gallus gallus spadiceus; GGM. Gallus gallus murghi; GGJ. Gallus gallus jabouillei; GGB. Gallus gallus bankiva 图 3 基于mtDNA控制区序列采用NJ法构建的系统发育树 Fig. 3 Neighbor-joining tree based on mtDNA D-loop region sequences |
本研究中,6个群体387个个体测序结果显示,线粒体控制区全序列大小为1 231 bp,所有个体均在859 bp处有1个C碱基缺失,共检测到多态位点28个,发现单倍型19种,按照鸡线粒体DNA单倍型分类通用标准[31],可分为A、B、C和E共4个单倍型群(分支),每个分支分别有116、36、132和103个个体。由构建的NJ系统进化树和Network中介网络图均可以看出,387个个体明显地被分成了A、B、C和E等4个支系,其中A1、B1、C1和E1分别为各个支系的主要单倍型。
进一步分析显示,反交F1代与固始鸡一样,主要有A、C两种单倍型,他们的占比非常接近,固始鸡A、C两种单倍型占比分别为53.42%和46.58%,反交F1代占比分别为50.75%和49.25%。正交F1代和F2代与隐性白羽鸡一样,均含有A、B、C和E共4种单倍型,3个群体4种单倍型占比相近,均是以E单倍型为优势单倍型,其中隐性白羽鸡的占比为48.89%,正交F1代和F2代E单倍型占比分别为48.84%和50.00%。在本研究中,试验鸡主要采用自由交配的方式,因此后代的几种单倍型比例均与母系的单倍型比例相似。但在实际生产或进化过程中由于自然选择和人工选择的干预,或者基因序列的突变,往往造成某些单倍型的遗失或改变,进而影响后代的单倍型比例。虽然一直以来,研究者认为mtDNA遵循母系遗传方式遗传,但也有研究显示,动物mtDNA存在重组现象,且最可能的途径是父系渗漏[36],在人类线粒体遗传研究中发现存在双亲线粒体DNA遗传的可能性[37],但在其他物种中还未发现双亲遗传的证据。本试验结果显示,在固始鸡与隐性白羽鸡的杂交传代过程中,线粒体D-loop区在世代传递过程中仍遵循着严格的母系遗传特点。
线粒体E分支为印度半岛、中东和欧洲大陆地方鸡种的主要单倍型,研究显示,E单倍型在中快速型肉鸡以及高产蛋鸡中普遍存在,在中国少数地方鸡种中也能检测到,但是所占比例均较低,至今为止未发现占绝对优势(比例在80%以上)的品种[38]。本研究也证实了这一点,在对固始鸡和臧鸡研究时发现,所采集的111个样本中均未发现E单倍型个体,说明这两个地方鸡种在长期的进化过程中受外来鸡种入侵机会较小,可能跟当地交通不便或者高山等天然屏障有关,从而保护了该品种的独有特性。有研究显示,利用线粒体标记可以进行分子辅助育种,早在1985年,Bell等[39]研究认为,细胞质效应对奶牛的产奶量、乳脂率等生产性能有影响,且假设这种影响是mtDNA的遗传效应引起的。Toelle等[40]通过研究杜洛克猪和约克夏猪的细胞质母系效应,发现对初生重、断奶重和背膘厚度等有重要影响。钱明娟等[41]采用线粒体基因组DNA超纯提取和多态性扩增的方法,开发了一套水稻线粒体基因组分子标记,并用于水稻不育系品种鉴定和细胞质进化研究。虽然目前有关线粒体分子标记在鸡育种方面的研究还未见相关报道,然而基于线粒体E单倍型在中快速型商品代肉鸡培育过程中的重要贡献以及线粒体母系遗传的独有特性,在今后的育种工作中,可以结合线粒体单倍型特征和育种目标,加快培育出满足消费者需求的品种(配套系)。
3.2 6个鸡群体mtDNA控制区遗传多样性单倍型多样度(Hd)和核苷酸多样度(Pi)是衡量一个群体mtDNA变异程度的重要指标,通常情况下Hd和Pi值越大,群体遗传多样性越丰富,选择潜力也越大[42-43]。本研究中,藏鸡Hd、Pi值以及K值均较低,说明藏鸡遗传多样性相对较为贫乏,选择潜力相对较小,质量性状趋于稳定。藏鸡主要采用保护区和基因库保护,据资料显示,自2002年开始已经过多个世代选育,目前群体的遗传特性和生产性能均保持相对稳定[44]。进一步分析显示,固始鸡遗传多样性程度与以其为母本的F1代(反交F1代)相近,而隐性白羽鸡遗传多样性程度与以其为母本的F1代(正交F1代)以及以F1代母鸡为母本的F2代相近。可见线粒体控制区的变异程度也是遵循着母系遗传特征世代传递的。
种间遗传距离大小体现了不同鸡种之间亲缘关系的远近,本研究中,Kiumura双参数距离结果显示,固始鸡与以其为母本的F1代(反交F1代)之间遗传距离最小,藏鸡与隐性白羽鸡、正交F1代以及F2代之间遗传距离最大。藏鸡和正交F1代分别为F2代群体的父本和母本,研究显示,藏鸡与F2代之间的遗传距离为0.009 1,而正交F1代与F2代之间的遗传距离为0.005 3,可见相对于父本,母本与其子代之间的亲缘关系更近,而随着线粒体的世代传递,母本与下一代的亲缘关系基本不变,本试验中,母本隐性白羽鸡、以隐性白羽鸡为母本的F1代(正交F1代)以及以F1代母鸡为母本的F2代两两之间遗传距离非常相近。
3.3 6个鸡群体系统发生关系迄今为止,家鸡究竟起源于哪种红色原鸡还没有明确定论。Fumihito等[45]的研究提出,生活在泰国及其周边的原鸡指名亚种(Gallus gallus gallus)是家鸡的唯一祖先。而Kanginakudru等[46]研究表明,生活在印度及其周边的原鸡滇南亚种(Gallus gallus spadiceus)和印度亚种(Gallus gallus murghi)在家鸡进化过程中贡献相当。本研究将所发现的19种单倍型与从NCBI下载的19条红色原鸡线粒体控制区全长序列联合分析,构建了K2P模型系统发育树,结果显示,A分支中的7种单倍型和B分支中的4种单倍型均仅与原鸡滇南亚种聚为一枝,A、B单倍型为我国地方鸡种的主要单倍型[13],推测原鸡滇南亚种在我国地方鸡种长期驯化形成过程中母系贡献较大。E分支中3种单倍型均与原鸡印度亚种聚为一枝,E单倍型在南亚和西方商品鸡中普遍存在,我国快长型鸡种在培育过程中可以引入这些外来鸡种血液,从而提高肉鸡生长速度或者蛋鸡产蛋性能。C分支中5种单倍型与原鸡指名亚种、滇南亚种、印度亚种以及印尼亚种(Gallus gallus bankiva)等4种红色原鸡亚种交叉聚为一枝,母系起源颇为丰富,推测拥有C单倍型的鸡种在其形成过程中可能受到了多种红色原鸡血液的入侵,然后经过长期的驯化逐渐形成了具有本身独特个性的群体。
4 结论本研究在6个鸡群体387个个体线粒体控制区全序列共发现了28个突变位点和19种单倍型;在固始鸡与隐性白羽鸡的杂交过程中,线粒体控制区在鸡种中遵循着严格的母系遗传,不同杂交组合和不同世代中后代与其母本的遗传多样性、单倍型数以及单倍型比例非常接近;19种单倍型共分为A、B、C和E共4个分支,A、B分支均主要起源于原鸡滇南亚种,E分支主要起源于原鸡印度亚种,C分支母系起源较为丰富,包括原鸡印度亚种、滇南亚种、指名亚种和印尼亚种。
| [1] | KUSZA S, ZAKAR E, BUDAI C, et al. Mitochondrial DNA variability in Gyimesi Racka and Turcana sheep breeds[J]. Acta Biochim Pol, 2015, 62(1): 273–280. |
| [2] | ARMSTRONG E, IRIARTE A, MARTINEZ A M, et al. Genetic diversity analysis of the Uruguayan Creole cattle breed using microsatellites and mtDNA markers[J]. Genet Mol Res, 2013, 12(2): 1119–1131. DOI: 10.4238/2013.April.10.7 |
| [3] | ARORA R, YADAV H S, MISHRA B P. Mitochondrial DNA diversity in Indian sheep[J]. Livest Sci, 2013, 153(1-3): 50–55. DOI: 10.1016/j.livsci.2013.02.006 |
| [4] |
李雪娟, 黄原, 雷富民. 山鹧鸪属鸟类线粒体基因组的比较及系统发育研究[J]. 遗传, 2014, 36(9): 912–920.
LI X J, HUANG Y, LEI F M. Comparative and phylogenomic analyses on mitochondrial genomes of Arborophila species[J]. Hereditas (Beijing), 2014, 36(9): 912–920. (in Chinese) |
| [5] | MALTSEV A N, STAKHEEV V V, BOGDANOV A S, et al. Phylogenetic relationships of intraspecific forms of the house mouse Mus musculus: Analysis of variability of the control region (D-loop) of mitochondrial DNA[J]. Dokl Biol Sci, 2015, 465(1): 285–288. DOI: 10.1134/S0012496615060058 |
| [6] | TAKASU M, ISHIHARA N, TOZAKI T, et al. Genetic diversity of maternal lineage in the endangered Kiso horse based on polymorphism of the mitochondrial DNA D-loop region[J]. J Vet Med Sci, 2014, 76(11): 1451–1456. DOI: 10.1292/jvms.14-0231 |
| [7] | GUO H W, LI C, WANG X N, et al. Genetic diversity of mtDNA D-loop sequences in four native Chinese chicken breeds[J]. Br Poult Sci, 2017, 58(5): 490–497. DOI: 10.1080/00071668.2017.1332403 |
| [8] |
邹辉, 韦玲静, 黄杰, 等. 基于线粒体CO I基因和D-Loop区序列的7个鲤群体遗传差异分析[J]. 南方农业学报, 2020, 51(5): 1209–1216.
ZOU H, WEI L J, HUANG J, et al. Genetic differences among seven carp populations based on mitochondrial CO I gene and D-Loop region sequences[J]. Journal of Southern Agriculture, 2020, 51(5): 1209–1216. DOI: 10.3969/j.issn.2095-1191.2020.05.028 (in Chinese) |
| [9] |
李正杰, 周先坤, 王浥尘, 等. 阿坝藏族羌族自治州若尔盖地区藏猪mtDNA D-Loop的遗传多样性分析[J]. 畜牧兽医学报, 2019, 50(12): 2387–2399.
LI Z J, ZHOU X K, WANG Y C, et al. Genetic diversity of Tibetan pig Mitochondrial DNA D-loop in Ruoergai area of A'ba Tibetan and Qiang Autonomous Prefecture[J]. Acta Veterinaria et Zootechnica Sinica, 2019, 50(12): 2387–2399. DOI: 10.11843/j.issn.0366-6964.2019.12.003 (in Chinese) |
| [10] |
赵忠海, 周迪, 李辉, 等. 利用mtDNA D-loop区研究贵州猪种的遗传多样性与起源[J]. 农业生物技术学报, 2019, 27(7): 1206–1214.
ZHAO Z H, ZHOU D, LI H, et al. Genetic diversity and origin of Guizhou Pig (Sus scrofa domesticus) breeds analyzed by mtDNA D-loop region[J]. Journal of Agricultural Biotechnology, 2019, 27(7): 1206–1214. (in Chinese) |
| [11] |
李新建, 薛亚辉, 王明宇, 等. 河南地方猪mtDNA D-loop序列遗传多样性及系统进化分析[J]. 河南农业大学学报, 2020, 54(3): 452–461.
LI X J, XUE Y H, WANG M Y, et al. Genetic diversity and phylogenetic relationships of Henan native pig breeds based on the sequence of mtDNA D-loop region[J]. Journal of Henan Agricultural University, 2020, 54(3): 452–461. (in Chinese) |
| [12] |
胡丹, 钟金城, 柴志欣. 新疆塔什库尔干牦牛mtDNA Cytb基因和D-Loop区遗传多样性及系统进化分析[J]. 家畜生态学报, 2018, 39(11): 11–16.
HU D, ZHONG J C, CHAI Z X. Genetic diversity and phyletic evolution on mtDNA Cytb gene and D-loop region of taxkorgan yak[J]. Acta Ecologae Animalis Domastici, 2018, 39(11): 11–16. DOI: 10.3969/j.issn.1673-1182.2018.11.003 (in Chinese) |
| [13] |
李静, 郭丽君, 王莉, 等. 喀喇昆仑-帕米尔地区牦牛mtDNA D-Loop区遗传多样性及系统发育分析[J]. 中国畜牧兽医, 2020, 47(8): 2481–2492.
LI J, GUO L J, WANG L, et al. Analyses of genetic diversity and phylogeny of mtDNA D-loop from yak in Karakoram-Pamir area[J]. China Animal Husbandry & Veterinary Medicine, 2020, 47(8): 2481–2492. (in Chinese) |
| [14] |
齐国强, 昝林森, 张桂香, 等. 中国部分地方水牛品种mtDNA D-loop区遗传多样性与起源研究[J]. 畜牧兽医学报, 2008, 39(1): 7–11.
QI G Q, ZAN L S, ZHANG G X, et al. Mitochondrial DNA D-loop genetic diversity and origin of some Chinese domestic buffalo breeds[J]. Acta Veterinaria et Zootechnica Sinica, 2008, 39(1): 7–11. DOI: 10.3321/j.issn:0366-6964.2008.01.002 (in Chinese) |
| [15] | ALJUBOURI T R S, AL-SHUHAIB M B S. Genotyping of mitochondrial D-loop sequences in three breeds of sheep[J]. Biologia, 2021, 76: 203–211. DOI: 10.2478/s11756-020-00543-6 |
| [16] |
梁瑞圆, 陈晓勇, 孙洪新, 等. 五个地方绵羊种群mtDNA D-loop区系统进化及遗传多样性分析[J]. 华北农学报, 2018, 33(1): 135–142.
LIANG R Y, CHEN X Y, SUN H X, et al. Phylogenety and genetic diversity of five local sheep breeds on mtDNA D-loop[J]. Acta Agriculturae Boreali-Sinica, 2018, 33(1): 135–142. (in Chinese) |
| [17] |
李芳玉, 黄光云, 王自豪, 等. 都安山羊和隆林山羊mtDNA D-loop区遗传多态性研究[J]. 中国畜牧杂志, 2020, 56(8): 69–72.
LI F Y, HUANG G Y, WANG Z H, et al. Genetic polymorphism of mtDNA D-loop Region in Du'an Goat and Longlin Goat[J]. Chinese Journal of Animal Science, 2020, 56(8): 69–72. (in Chinese) |
| [18] | DELL A C, CURRY M C, YARNELL K M, et al. Mitochondrial D-loop sequence variation and maternal lineage in the endangered Cleveland Bay horse[J]. PLoS One, 2020, 15(12): e0243247. DOI: 10.1371/journal.pone.0243247 |
| [19] |
周凡莉, 黄卫平, 金素钰, 等. 建昌马mtDNA D-loop区遗传多样性及系统进化分析[J]. 中国畜牧兽医, 2020, 47(1): 63–73.
ZHOU F L, HUANG W P, JIN S Y, et al. Genetic diversity and evolutionary analysis of mtDNA D-loop region in Jianchang horse[J]. China Animal Husbandry & Veterinary Medicine, 2020, 47(1): 63–73. (in Chinese) |
| [20] |
张涛, 路宏朝. 宁强矮马线粒体DNA D-loop区的遗传多样性[J]. 中国农业科学, 2012, 45(8): 1587–1594.
ZHANG T, LU H Z. Genetic diversity of mitochondrial DNA D-loop sequences in Ningqiang pony[J]. Scientia Agricultura Sinica, 2012, 45(8): 1587–1594. DOI: 10.3864/j.issn.0578-1752.2012.08.014 (in Chinese) |
| [21] |
李慧锋, 赵婧微, 陈员玉, 等. 晋南驴mtDNA D-loop区部分序列系统进化分析与体型测定[J]. 山西农业大学学报: 自然科学版, 2020, 40(4): 121–128.
LI H F, ZHAO J W, CHEN Y Y, et al. Phylogenetic analysis of partial mtDNA D-Loop sequence in Jinnan donkey and their body size measurement[J]. Journal of Shanxi Agricultural University: Natural Science Edition, 2020, 40(4): 121–128. (in Chinese) |
| [22] | XIA X, YU J, ZHAO X, et al. Genetic diversity and maternal origin of Northeast African and South American donkey populations[J]. Anim Genet, 2019, 50(3): 266–270. DOI: 10.1111/age.12774 |
| [23] | COZZI M C, VALIATI P, CHERCHI R, et al. Mitochondrial DNA genetic diversity in six Italian donkey breeds (Equus Asinus)[J]. Mitochondrial DNA Part A, 2018, 29(3): 409–418. DOI: 10.1080/24701394.2017.1292505 |
| [24] | JU Y, LIU H M, HE J M, et al. Genetic diversity of Aoluguya Reindeer based on D-loop region of mtDNA and its conservation implications[J]. Gene, 2020, 733: 144271. DOI: 10.1016/j.gene.2019.144271 |
| [25] | LEE J C I, TSAI L C, LIAO S P, et al. Evaluation of the polymorphic D-loop of Columba livia in forensic applications[J]. Electrophoresis, 2010, 31(23-24): 3889–3894. DOI: 10.1002/elps.201000414 |
| [26] |
王荣琼, 张自芳, 黄静, 等. 腾冲雪鸡mtDNA D-loop区遗传多样性分析[J]. 云南农业大学学报: 自然科学, 2021, 36(1): 75–81.
WANG R Q, ZHANG Z F, HUANG J, et al. Genetic diversity of mtDNA D-loop region in Tengchong White Chicken[J]. Journal of Yunnan Agricultural University: Natural Science, 2021, 36(1): 75–81. (in Chinese) |
| [27] |
李兴才, 潘成勇, 李辉, 等. 香炉山鸡线粒体DNA D-Loop区遗传多样性及系统进化研究[J]. 中国畜牧兽医, 2019, 46(9): 2633–2641.
LI X C, PAN C Y, LI H, et al. Study on genetic diversity and system evolution of mtDNA D-loop region in Xianglushan chicken[J]. China Animal Husbandry & Veterinary Medicine, 2019, 46(9): 2633–2641. (in Chinese) |
| [28] |
唐修君, 贾晓旭, 樊艳凤, 等. 基于mtDNA控制区遗传变异分析大骨鸡遗传多样性及系统进化[J]. 扬州大学学报: 农业与生命科学版, 2019, 40(1): 67–71.
TANG X J, JIA X X, FAN Y F, et al. The genetic diversity and phyletic evolution of Dagu chicken based on the genetic variation of mtDNA D-loop region sequence[J]. Journal of Yangzhou University: Agricultural and Life Science Edition, 2019, 40(1): 67–71. (in Chinese) |
| [29] |
黄勋和, 余哲琪, 翁茁先, 等. 广东省地方鸡线粒体遗传多样性与母系起源[J]. 生物多样性, 2018, 26(3): 238–247.
HUANG X H, YU Z Q, WENG Z X, et al. Mitochondrial genetic diversity and maternal origin of Guangdong indigenous chickens[J]. Biodiversity Science, 2018, 26(3): 238–247. (in Chinese) |
| [30] |
贾晓旭, 唐修君, 樊艳凤, 等. 华东地区地方鸡品种mtDNA控制区遗传多样性[J]. 生物多样性, 2017, 25(5): 540–548.
JIA X X, TANG X J, FAN Y F, et al. Genetic diversity of local chicken breeds in East China based on mitochondrial DNA D-loop region[J]. Biodiversity Science, 2017, 25(5): 540–548. (in Chinese) |
| [31] | LIU Y P, WU G S, YAO Y G, et al. Multiple maternal origins of chickens: out of the Asian jungles[J]. Mol Phylogenet Evol, 2006, 38(1): 12–19. DOI: 10.1016/j.ympev.2005.09.014 |
| [32] | MIAO Y W, PENG M S, WU G S, et al. Chicken domestication: an updated perspective based on mitochondrial genomes[J]. Heredity, 2013, 110(3): 277–282. DOI: 10.1038/hdy.2012.83 |
| [33] |
萨姆布鲁克J, 拉塞尔D W. 分子克隆实验指南[M]. 黄培堂, 译. 3版. 北京: 科学出版社, 2002: 461-512.
SAMBROOK J, RUSSELL D W. Molecular cloning[M]. HUANG P T, trans. 3rd ed. Beijing: Science Press, 2002: 461-512. (in Chinese) |
| [34] | TAMURA K, DUDLEY J, NEI M, et al. MEGA4:molecular evolutionary genetics analysis (MEGA) software version 4.0[J]. Mol Biol Evol, 2007, 24(8): 1596–1599. DOI: 10.1093/molbev/msm092 |
| [35] | ROZAS J, SÁNCHEZ-DELBARRIO J C, MESSEGUER X, et al. DnaSP, DNA polymorphism analyses by the coalescent and other methods[J]. Bioinformatics, 2003, 19(18): 2496–2497. DOI: 10.1093/bioinformatics/btg359 |
| [36] |
于晓丽, 黄原. 动物线粒体DNA重组的研究进展[J]. 动物学杂志, 2008, 43(2): 145–149.
YU X L, HUANG Y. Animal mitochondrial DNA recombination[J]. Chinese Journal of Zoology, 2008, 43(2): 145–149. DOI: 10.3969/j.issn.0250-3263.2008.02.027 (in Chinese) |
| [37] | RIUS R, COWLEY M J, RILEY L, et al. Biparental inheritance of mitochondrial DNA in humans is not a common phenomenon[J]. Genet Med, 2019, 21(12): 2823–2826. DOI: 10.1038/s41436-019-0568-0 |
| [38] | OSMAN S A M, YONEZAWA T, NISHIBORI M. Origin and genetic diversity of Egyptian native chickens based on complete sequence of mitochondrial DNA D-loop region[J]. Poult Sci, 2016, 95(6): 1248–1256. DOI: 10.3382/ps/pew029 |
| [39] | BELL B R, MCDANIEL B T, ROBISON O W. Effects of cytoplasmic inheritance on production traits of dairy cattle[J]. J Dairy Sci, 1985, 68(8): 2038–2051. DOI: 10.3168/jds.S0022-0302(85)81066-3 |
| [40] | TOELLE V, MCDANIEL B, ROBISON O. Cytoplasmic effects in swine[J]. J Anim Sci, 1986, 63: 203. |
| [41] |
钱明娟, 谢红卫, 李绍清, 等. 水稻线粒体SCAR分子标记的开发与应用[J]. 分子植物育种, 2016, 14(10): 2671–2679.
QIAN M J, XIE H W, LI S Q, et al. Development and application of rice mitochondrial SCAR molecular markers[J]. Molecular Plant Breeding, 2016, 14(10): 2671–2679. (in Chinese) |
| [42] | SMITH M A, WOODLEY N E, JANZEN D H, et al. DNA barcodes reveal cryptic host-specificity within the presumed polyphagous members of a genus of parasitoid flies (Diptera: Tachinidae)[J]. Proc Natl Acad Sci U S A, 2006, 103(10): 3657–3662. DOI: 10.1073/pnas.0511318103 |
| [43] |
潘建飞, 史兆国, 成述儒, 等. 甘肃主要马群体遗传多样性及系统发育研究[J]. 农业生物技术学报, 2014, 22(2): 210–218.
PAN J F, SHI Z G, CHENG S R, et al. The study of genetic diversity and Phylogenetic evolution in indigenous horses (Equus caballus) of Gansu[J]. Journal of Agricultural Biotechnology, 2014, 22(2): 210–218. DOI: 10.3969/j.issn.1674-7968.2014.02.010 (in Chinese) |
| [44] |
国家畜禽遗传资源委员会.
中国畜禽遗传资源志-家禽志[M]. 北京: 中国农业出版社, 2011: 124-368.
National Committee of Animal Genetic Resources. Animal genetic resources in China Poultry[M]. Beijing: China Agriculture Press, 2011: 124-368. (in Chinese) |
| [45] | FUMIHITO A, MIYAKE T, SUMI S, et al. One subspecies of the red junglefowl (Gallus gallus gallus) suffices as the matriarchic ancestor of all domestic breeds[J]. Proc Natl Acad Sci U S A, 1994, 91(26): 12505–12509. DOI: 10.1073/pnas.91.26.12505 |
| [46] | KANGINAKUDRU S, METTA M, JAKATI R D, et al. Genetic evidence from Indian red jungle fowl corroborates multiple domestication of modern day chicken[J]. BMC Evol Biol, 2008, 8: 174. DOI: 10.1186/1471-2148-8-174 |



