2. 贵州大学 贵州省动物遗传育种与繁殖重点实验室, 贵阳 550025;
3. 山东滨州沃华生物工程有限公司, 滨州 256600
2. Guizhou Province Key Laboratory of Animal Genetics, Breeding and Reproduction, Guizhou University, Guiyang 550025, China;
3. Shandong Binzhou Wohua Biological Engineering Co. LTD., Binzhou 256600, China
肿瘤坏死因子受体相关因子6(tumor necrosis factor(TNF) receptor-associated factor 6, TRAF6)是一个具有典型环指结构域的E3泛素连接酶[1],它作为一种细胞内信号转导通路上的关键接头蛋白,可通过激活下游的细胞核因子κB(nuclear factor-kB, NF-κB)、激活因子蛋白-1(activator protein-1, AP-1)、PI3K/Akt和JNK/p38以及干扰素调节因子等信号通路影响细胞的增殖、分化和凋亡,并且调控细胞的炎症反应、氧化应激和骨代谢等生物学过程[2-3]。研究表明,TRAF6蛋白在人和不同动物的组织中广泛表达,其中在肺、肝、脾、胸腺和法氏囊中高表达,在脑、心和腿肌中低表达[4-5]。Jin等[6]研究发现,TRAF6基因在新城疫病毒(newcastle disease virus, NDV)未感染鸡的肺和脾中呈现高表达,而在NDV感染鸡后的免疫器官(法氏囊、脾和胸腺)中表现为高表达,这表明TRAF6蛋白在抵抗NDV感染中有着重要作用。研究人员利用基因敲除小鼠试验进一步证实,TRAF6蛋白还在动物的天然免疫和获得性免疫,以及淋巴结、脾、胸腺和中枢神经系统的发育等方面发挥重要作用[7]。此外,TRAF6蛋白还是病毒感染细胞后的靶标分子,如仙台病毒和水泡性口炎病毒能分别以蛋白酶依赖或不依赖的方式降解TRAF6蛋白,抑制NF-κB的激活和I型干扰素以及促炎性细胞因子的产生,进而促进病毒在细胞中的复制[8-9]。
肿瘤坏死因子受体相关因子相互作用的具有叉形头相关结构域的蛋白(TNF receptor associated factor(TRAF)-interacting protein with a forkhead-associated(FHA)domain, TIFA)是一种在白细胞介素1(interleukin 1, IL-1)信号通路中将TRAF6蛋白连接到IL-1受体相关激酶1的蛋白分子[10]。研究表明,TIFA蛋白与先天性免疫相关,它通过调节TRAF6蛋白的泛素化和寡聚化激活NF-κB信号通路,促进细胞因子的产生[11]。例如,在细菌感染人胚肾细胞过程中,α蛋白激酶1 (alpha-protein kinase 1,ALPK1) 的N-端结构域可以直接结合细菌脂多糖合成的前体糖分子ADP-heptose,通过蛋白构象变化诱导C-端激酶结构域活化,进而磷酸化TIFA使其发生寡聚化,并进一步通过与TRAF6蛋白的相互作用激活下游NF-κB信号通路[12-13]。本课题组最近研究发现,在NDV感染过程中,病毒的M蛋白可以通过剂量依赖的方式降低幼仓鼠肾细胞中TIFA和TRAF6蛋白的表达,进而抑制NF-κB信号通路介导的细胞因子产生,促进NDV复制[14]。因此,TIFA和TRAF6蛋白相互作用所介导的NF-κB信号通路在抵抗微生物感染方面具有重要作用,逐渐得到研究人员的重视和关注。
目前,TRAF6和TIFA蛋白相互作用的报道仅在非洲爪蟾[15]和人[16]可见。由于鸡与人和非洲爪蟾TRAF6和TIFA蛋白的同源性较低,鸡TRAF6和TIFA蛋白是否也具有相互作用尚不清楚。因此,本研究首先扩增鸡TRAF6和TIFA基因,构建其重组真核表达载体pCMV-HA-TRAF6和pEGFP-C1-TIFA,并进行鸡TRAF6和TIFA蛋白的序列分析;将构建的重组真核表达载体pCMV-HA-TRAF6和pEGFP-C1-TIFA共转染细胞,利用荧光共定位和免疫共沉淀(co-immunoprecipitation, Co-IP)试验验证鸡TRAF6和TIFA蛋白的相互作用。本研究将为后续进一步探究鸡TRAF6和TIFA蛋白相互作用参与的NF-κB信号通路调控鸡相关病毒复制的作用机制奠定工作基础。
1 材料与方法 1.1 菌株与细胞真核表达载体pEGFP-C1、pCMV-HA购自Invitrogen公司;鸡胚成纤维细胞(DF-1)、人胚胎肾细胞(HEK-293T)和大肠杆菌DH5α感受态细胞由本实验室保存。
1.2 主要试剂TRIzol、2×Taq PCR starMix、T4 DNA连接酶购自Invitrogen公司;卡那霉素、氨苄青霉素、DAPI购自Sigma公司;cDNA第一链合成试剂盒、限制性内切酶Sal Ⅰ、Kpn Ⅰ、Xho Ⅰ、EcoR Ⅰ、DNA Marker购自Thermo Fisher公司;质粒小提试剂盒购自Omega公司;FuGENEⓇHD Transfection Reagent购自Promega公司;anti-HA和anti-GFP鼠单克隆抗体购自Proteintech公司;RIPA(弱)细胞裂解液、Protein A+G琼脂球、预染蛋白Marker、HRP标记山羊抗小鼠IgG(H+L)、Cy3标记山羊抗小鼠IgG(H+L)、SDS-PAGE上样缓冲液(5×)、极超敏ECL化学发光试剂盒购自碧云天生物技术公司;DMEM高糖培养基、胎牛血清购自Gibco公司。
1.3 引物的设计与合成根据GenBank中鸡TRAF6基因登录号(XM_015287208.2)和TIFA基因登录号(XM_015276339.2),利用Primer Premier 5.0软件设计扩增鸡TRAF6和TIFA基因ORF序列的特异性引物(表 1)。引物由擎科(成都)生物技术有限公司合成。
|
|
表 1 构建重组真核表达载体所用引物信息 Table 1 The information of primers used for recombinant plasmid construction |
使用TRIzol法从DF-1细胞中提取总RNA并进行反转录,以设计合成的特异性引物分别扩增鸡TRAF6和TIFA基因的CDS区。PCR反应体系:cDNA模板1.0 μL,2×Taq PCR starMix 10.0 μL,上、下游引物(10 μmol·L-1)各1.0 μL,加ddH2O加至20.0 μL。PCR扩增程序:94 ℃预变性2 min;94 ℃ 30 s,60 ℃ 30 s,72 ℃ 90 s,25个循环;72 ℃延伸5 min;12 ℃保存。将获得的PCR扩增产物用1%琼脂糖凝胶电泳进行检测。
1.5 鸡TRAF6和TIFA基因重组真核表达载体的构建分别使用Sal Ⅰ、Kpn Ⅰ和Xho Ⅰ、EcoR Ⅰ限制性内切酶对真核表达载体pCMV-HA和pEGFP-C1以及TRAF6和TIFA基因的胶回收产物进行双酶切,经1%琼脂糖凝胶电泳检测后进行切胶纯化,回收目的片段后进行连接并转化大肠杆菌DH5α感受态细胞。挑取单菌落扩大培养,提取质粒并进行双酶切鉴定。将鉴定正确的重组真核表达载体pCMV-HA-TRAF6和pEGFP-C1-TIFA送擎科(成都)生物技术有限公司测序。
1.6 鸡TRAF6和TIFA基因的生物信息学分析利用生物信息学在线软件ProtParam(http://us.expasy.org/tools/protparam.html/)、SOPMA(http://npsapbil.ibcp.fr/cgi-bin/npsa_automat.pl?page=npsa_sopma.html)和SWISS-MODEL(http://swissmodel.ex-pasy.org/)分别对鸡TRAF6和TIFA基因编码的蛋白质进行蛋白质二级结构和三级结构分析;从GenBank下载不同物种TRAF6和TIFA蛋白序列,用MegAlign软件进行氨基酸同源性分析;用NCBI服务器上的Protein数据库和MegAlign软件对鸡TRAF6和TIFA蛋白氨基酸序列的功能结构域保守性进行预测分析;用PSORTⅡ(https://psort.hgc.jp/form2.html)软件进行蛋白质亚细胞定位分析;用在线软件STRING(https://version11.string-db.org/cgi/input.pl?sessionId=TqSW5v1qFKp0&input_page_show_search=on)对TRAF6和TIFA蛋白相互作用的细胞蛋白进行预测。
1.7 HEK-293T细胞的培养和转染将HEK-293T细胞用含10%胎牛血清、100 U·mL-1青霉素、100 μg·mL-1链霉素的DMEM培养基置于37 ℃、含5% CO2的细胞培养箱中培养。转染前1 d,按5×105个·孔-1的量将消化后的HEK-293T细胞铺在35 mm细胞培养皿中培养过夜,待细胞完全贴壁生长至80%左右,将纯化的空载体或重组真核表达载体和脂质体混合后分别共转染细胞(转染组分为3组,分别为pEGFP-C1和pCMV-HA-TRAF6;pCMV-HA和pEGFP-C1-TIFA;pEGFP-C1-TIFA和pCMV-HA-TRAF6),轻轻吹打混匀后室温静置25 min,然后加入细胞培养皿中,放入37 ℃、含5% CO2的细胞培养箱中继续培养。
1.8 重组蛋白的荧光观察重组真核表达载体转染HEK-293T细胞后24 h,PBS洗涤细胞3次,依次加入适量4%多聚甲醛固定、0.25% Triton X-100破膜、含10%小牛血清的PBS封闭;PBS洗涤3遍,加入anti-HA鼠多克隆抗体于37 ℃作用2 h;PBS洗涤3遍,加入Cy3标记山羊抗小鼠IgG (H+L)于37 ℃作用1 h;PBS洗涤3遍,加入DAPI染细胞核10 min,最后用倒置荧光显微镜观察荧光。将获得的荧光图片用Photoshop CS3软件进行Merge处理,观察重组蛋白的共定位。
1.9 免疫共沉淀(Co-IP)试验将重组真核表达载体转染HEK-293T细胞后36 h,用PBS清洗细胞3次,加入适量RIPA(弱)细胞裂解液(含PMSF)裂解细胞,4 ℃ 12 000 r·min-1离心10 min,收集上清,加入2 μg anti-GFP鼠单克隆抗体,4 ℃作用过夜;加入40 μL protein A+G琼脂球继续作用3 h;用预冷的RIPA洗涤protein A+G琼脂球,重复3次后加入20 μL SDS-PAGE上样缓冲液煮沸5 min,离心收集上清,对样品进行SDS-PAGE和转膜,最后用anti-HA鼠单克隆抗体进行Western blot,检测蛋白之间是否存在相互作用。反之,以anti-HA鼠单克隆抗体进行免疫沉淀,用anti-GFP鼠单克隆抗体进行Western blot检测。
2 结果 2.1 鸡TRAF6和TIFA基因的RT-PCR扩增以DF-1细胞总RNA反转录合成的cDNA为模板,使用特异性引物分别扩增鸡TRAF6和TIFA基因的CDS区,PCR产物经1% 琼脂糖凝胶电泳检测,结果获得与预期大小相符的TRAF6 (1 657 bp,图 1A)和TIFA(586 bp,图 1B)基因的目的条带,但特异性引物含酶切位点和保护碱基的长度,分别为19和22 bp,因此,最终的CDS区长度分别为1 638和564 bp。
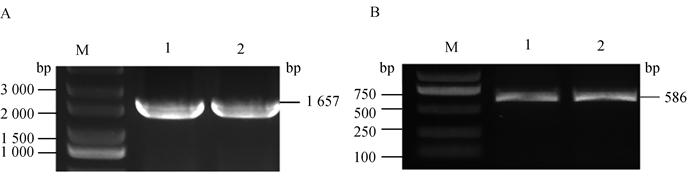
|
A. 鸡TRAF6基因PCR扩增结果(M. DNA相对分子质量标准; 1~2. 鸡TRAF6基因);B. 鸡TIFA基因PCR扩增结果(M. DNA相对分子质量标准; 1~2. 鸡TIFA基因) A. The PCR amplification results of chicken TRAF6 gene (M. DL5000 DNA marker; 1-2. Chicken TRAF6 gene); B. The PCR amplification results of chicken TIFA gene (M. DL5000 DNA marker; 1-2. Chicken TIFA gene) 图 1 鸡TRAF6和TIFA基因的PCR电泳结果 Fig. 1 The results of PCR electrophoresis of chicken TRAF6 and TIFA genes |
将获得的鸡TRAF6和TIFA基因片段分别亚克隆至真核表达载体pCMV-HA和pEGFP-C1,用于构建重组真核表达载体pCMV-HA-TRAF6和pEGFP-C1-TIFA。双酶切鉴定结果显示,重组真核表达载体pCMV-HA-TRAF6酶切后获得3 782 bp的载体条带和1 657 bp的目的基因条带(图 2A),而重组真核表达载体pEGFP-C1-TIFA酶切后则获得约4 731 bp的载体条带和586 bp的目的基因条带(图 2B)。测序结果显示,与GenBank中登录的鸡TRAF6和TIFA基因序列相比,送测的9个样中鸡TRAF6基因CDS区第148位核苷酸均存在碱基突变(T→C),导致苯丙氨基酸(F)变为亮氨基酸(L),但F和L均为非极性疏水性氨基酸,推测该位点本身就存在突变;而TIFA基因第265位核苷酸发生一个碱基突变(T→C),但没有引起氨基酸的改变。
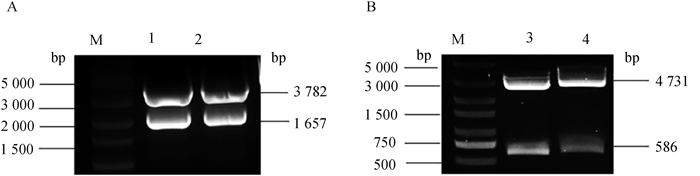
|
M. DNA相对分子质量标准:1~2. 重组真核表达载体pCMV-HA-TRAF6双酶切产物;3~4. 重组真核表达载体pEGFP-C1-TIFA双酶切产物 M. DL5000 DNA marker; 1-2. Enzyme digestion products of recombinant vector pCMV-HA-TRAF6; 3-4. Enzyme digestion products of recombinant vector pEGFP-C1-TIFA 图 2 重组真核表达载体的双酶切鉴定 Fig. 2 Identification of the recombinant plasmids by double restriction endonuclease digestion |
2.3.1 鸡TRAF6和TIFA蛋白的理化性质、二级结构和三级结构预测 利用生物信息学在线软件ProtParam、SOPMA和SWISS-MODEL对鸡TRAF6和TIFA蛋白进行理化性质、二级结构和三级结构分析。结果显示,鸡TRAF6蛋白共编码545个氨基酸,分子量约为62 ku,理论pI为6.14;鸡TIFA蛋白共编码187个氨基酸,分子量约为22 ku,理论pI为5.18。鸡TRAF6蛋白含有丰富的二级结构,其中无规则卷曲占47.08%、α-螺旋占38.14%、延伸链占11.86%、β-转角占2.92%;鸡TIFA蛋白二级结构中无规则卷曲占51.87%、α-螺旋占27.81%、延伸链占16.58%、β-转角占3.74%。蛋白质三级结构预测结果表明,鸡TRAF6和TIFA蛋白的三级结构(图 3)与二级结构相符,主要由无规则卷曲和α-螺旋构成。

|
图 3 鸡TRAF6(A)和TIFA(B)蛋白的三级结构预测 Fig. 3 Tertiary structure prediction of chicken TRAF6(A) and TIFA(B) proteins |
2.3.2 鸡TRAF6和TIFA蛋白氨基酸同源性分析 用MegAlign软件将鸡与人、鼠、牛、猪和非洲爪蟾的TRAF6和TIFA蛋白进行氨基酸同源性分析,结果表明,鸡与人和其他哺乳动物TRAF6和TIFA蛋白的同源性分别为76.4%~80.3% 和49.7%~53.3%,而与非洲爪蟾的同源性分别为69.4% 和49.7%(图 4A、B),其中鸡与牛TRAF6蛋白的同源性最高(80.3%),与非洲爪蟾的同源性最低(69.4%),而鸡与牛TIFA蛋白的同源性最高(53.3%),与猪和非洲爪蟾的同源性最低(49.7%)。

|
数字1~9表示不同物种;表格右上角数字表示氨基酸同源性,表格左下角数字表示变异度 Number 1-9 indicate the different species; Data in the upper right corner of the tables are amino acid homology and data in the bottom left corner of the tables are divergence 图 4 不同物种TRAF6(A)和TIFA(B)蛋白氨基酸同源性分析结果 Fig. 4 Amino acid homology analysis of TRAF6 (A) and TIFA(B) proteins in different species |
2.3.3 鸡TRAF6和TIFA蛋白功能结构域保守性分析 用NCBI服务器上的Protein数据库和MegAlign软件对鸡TRAF6和TIFA蛋白氨基酸序列的功能结构域保守性进行预测和分析。功能结构域分析显示,鸡TRAF6蛋白中存在3个明显的功能结构域,主要包括RING结构域、zinc-finger结构域和MATH结构域,分别在69~ 107、204~360和378~505位氨基酸区域。TIFA蛋白存在1个FHA结构域,位于47~104个氨基酸区域。分别对鸡、人和非洲爪蟾的TRAF6和TIFA蛋白功能结构域进行保守性分析,结果发现(图 5),与非洲爪蟾和人TRAF6蛋白相比,鸡TRAF6蛋白的RING结构域氨基酸变异位点主要位于92(G/A)位氨基酸,zinc-finger结构域氨基酸变异位点主要位于325(Q/E)和340(V/I)位氨基酸,MATH结构域氨基酸变异位点主要位于394(R/K)、398(M/I)、414(L/M)、428(F/Y)、488(H/P)位氨基酸;与非洲爪蟾和人TIFA蛋白相比,鸡TIFA蛋白的FHA结构域变异位点主要位于47~48(MA/VV)、75~77(FYR/LFK)位氨基酸。以上结果说明,鸡和人以及非洲爪蟾TRAF6和TIFA蛋白功能结构域氨基酸位点的保守性较低。

|
方框表示氨基酸变异位点 The boxes indicate the amino acid mutation sites 图 5 鸡TRAF6(A)和TIFA(B)蛋白功能结构域氨基酸位点变异分析 Fig. 5 The amino acid site variation analysis in the functional domains of chicken TRAF6 (A) and TIFA (B) proteins |
2.3.4 鸡TRAF6和TIFA蛋白亚细胞定位分析 运用在线软件PSORTⅡ预测鸡TRAF6和TIFA蛋白的亚细胞定位,结果表明,鸡TRAF6蛋白在细胞核中占82.6%、细胞质占13.0%,表明鸡TRAF6蛋白主要定位在细胞核。TIFA蛋白在细胞核中占56.5%、细胞质占17.4%、线粒体占17.4%,表明鸡TIFA蛋白主要定位在细胞核。
2.3.5 鸡TRAF6和TIFA蛋白相互作用蛋白的预测 运用在线软件STRING对鸡TRAF6和TIFA蛋白可能存在的互作蛋白进行预测,结果显示(图 6),与TRAF6和TIFA蛋白可能存在互作的蛋白有IL1R1、IL1RL2、IRAK4、UBE2N、TICAM1、TAB2、TAB1、MAP3K7、IRF7、TLR4、MYD88、TRAF3等。其中,IL1R1和IL1RL2同属于IL-1家族受体,IL1R1在许多细胞因子诱导的炎症反应和宿主防御中发挥重要作用[17-18],IL1RL2是炎症反应中重要的衔接分子[19]。TLR4可以通过MYD88和TRAF6来调控NF-κB信号通路参与炎症反应,TLR4/MYD88/NF-κB信号通路通过调控促炎因子的产生,从而诱导心肌组织的炎症反应[20]。IRF7是I型干扰素的重要调节因子,在抵抗病毒感染中发挥着重要作用,另外,研究表明,IRF7的过表达会增加人巨噬细胞中免疫缺陷病毒1型(human immunodeficiency virus-1,HIV-1)的复制,在NF-κB信号通路中TRAF6的下调也会导致巨噬细胞中HIV-1复制增强[21]。
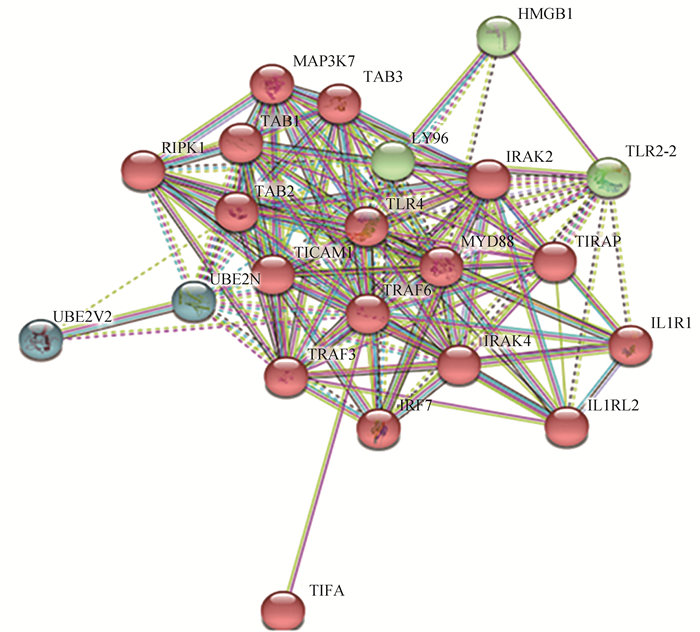
|
图 6 与TRAF6和TIFA相互作用蛋白的预测 Fig. 6 Prediction of proteins interacting with TRAF6 and TIFA proteins |
将鸡TRAF6和TIFA基因重组真核表达载体共转染HEK-293T细胞后24 h,对其进行荧光观察。荧光共定位结果显示,当pCMV-HA-TRAF6和pEGFP-C1-TIFA共转染细胞后,重组蛋白EGFP-TIFA和HA-TRAF6主要定位在细胞核;当pEGFP-C1和pCMV-HA-TRAF6共转染细胞后,EGFP标签蛋白和重组蛋白HA-TRAF6在细胞核和细胞质均存在;而当pEGFP-C1-TIFA和pCMV-HA共转染细胞后,重组蛋白EGFP-TIFA和HA标签蛋白同样定位在细胞核和细胞质(图 7)。
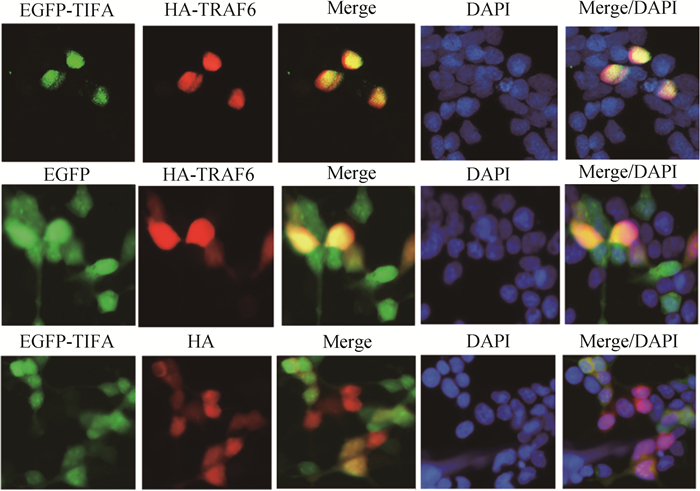
|
图 7 鸡TRAF6和TIFA重组蛋白在HEK-293T细胞中的共定位(200×) Fig. 7 Co-localization of chicken TRAF6 and TIFA recombinant proteins in HEK-293T cells(200×) |
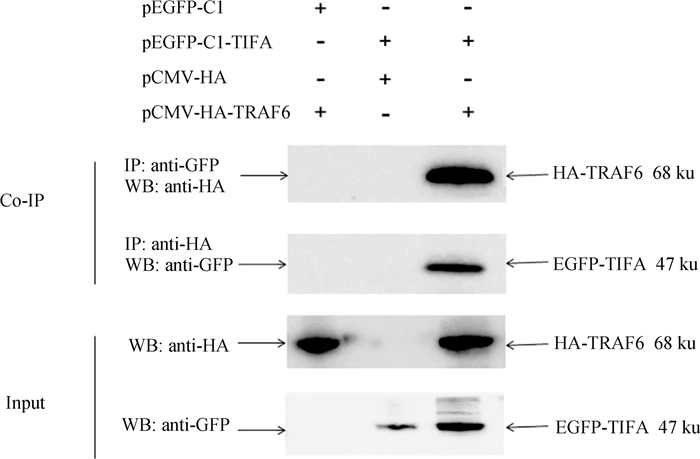
为验证鸡TRAF6和TIFA蛋白在细胞内是否存在相互作用,将重组真核表达载体pMCV-HA-TRAF6和pEGFP- C1-TIFA以及空载体pMCV-HA和pEGFP-C1分别进行组合共转染HEK-293T细胞,转染细胞后36 h收集细胞裂解上清进行Co-IP试验。Western blot检测结果显示,重组蛋白HA-TRAF6能与EGFP-TIFA发生相互作用,而重组蛋白HA-TRAF6和EGFP标签蛋白以及重组蛋白EGFP-TIFA和HA标签蛋白均不发生相互作用(图 8)。
|
图 8 Co-IP验证鸡TRAF6和TIFA蛋白的相互作用 Fig. 8 Identification of the interaction between chicken TRAF6 and TIFA proteins by Co-IP assay |
TRAF6作为重要的多功能信号接头分子,在免疫系统中发挥重要作用[22]。研究表明,TRAF6蛋白含有RING、zinc-finger和MATH结构域[1],其中MATH结构域具有高度保守性,可以介导TRAF蛋白分子间同源三聚体的形成以及与不同受体蛋白MATH结构域或胞质中含有该结构域的蛋白相结合[23];而RING和zinc-finger结构域在信号传导通路中发挥泛素连接酶的作用,可以激活下
游信号通路和催化底物的泛素化[24]。本研究进行了鸡、人和非洲爪蟾TRAF6蛋白功能结构域氨基酸位点的变异分析,结果发现,鸡和人及非洲爪蟾TRAF6蛋白中存在多个氨基酸位点变异,这些变异的氨基酸位点可能会导致鸡TRAF6蛋白在细胞中的免疫调控机制不同。而NF-κB作为一种核转录因子,当细胞受到病毒和细菌感染,NF-κB通过TARF6细胞因子刺激后可被激活,增强基因的转录。研究表明,TRAF6与NF-κB受体激活因子(receptor activator of nuclear factor-κB, RANK)相结合,参与IL-1及脂多糖等因子介导的信号通路,激活NF-κB信号通路[25]。近年来,关于TRAF6上游蛋白分子介导的TRAF6/NF-κB信号通路相关报道越来越多,如TLR4/TRAF6/NF-κB、IRAK1/TRAF6/NF-κB和IL-17RA/ TRAF6/NF-κB等[26-28]。已有研究表明,广州管圆线虫引起的曲霉菌感染可以通过下调IL-17RA/TRAF6/NF-κB信号通路来降低TRAF6的表达,从而促进曲霉菌的感染[28]。志贺菌感染可以抑制宿主的TRAF6/NF-κB信号通路,从而抑制炎症细胞因子的产生来促进志贺菌的复制[29]。另外,Zhang等[30]研究表明,博尔纳病毒1感染下调了人小胶质细胞中IRAK1和TRAF6蛋白的表达,并抑制NF-κB信号途径,从而在宿主细胞中降低细胞因子的产生,促进病毒的复制。这些研究结果表明,TRAF6蛋白介导的NF-κB信号通路在研究病毒和细菌感染方面有重要意义。
TIFA是由Takatsuna等[10]通过酵母菌双杂交技术首次筛选并被鉴定为一种与TRAF6相互作用的蛋白。在结构上,TIFA具有N末端叉形头相关结构域(the forkhead-associated,FHA)和C末端有1个共同的TRAF6结合基序[31]。其中,FHA结构域已经被证实可以与磷酸苏氨酸和磷酸丝氨酸结合,其主要功能与细胞周期调控有关,以及在细胞生长、DNA损伤修复和信号转导等过程中都发挥关键作用[32-33]。本研究对TIFA蛋白功能结构域氨基酸位点变异分析发现,鸡和人及非洲爪蟾TIFA蛋白中存在多个氨基酸位点变异,说明TIFA蛋白功能结构域在哺乳动物中的保守性较低,而鸡TIFA蛋白功能结构域氨基酸位点的变异可能会导致其功能的改变。Huang等[16]研究发现,相对于人正常的TIFA与TRAF6蛋白结构域之间的弱结合,将人TIFA蛋白的C末端第174位氨基酸S突变为Q和第179位氨基酸M突变为D可以增强TIFA与TRAF6蛋白TRAF结构域之间的相互作用。对鸡TIFA蛋白氨基酸序列分析发现,鸡TIFA蛋白第174位氨基酸为Q,因此,该位点的变化可能会增强鸡TIFA与TRAF6蛋白之间的相互作用。
Inoue等[15]在非洲爪蟾中首次证实,TIFA蛋白C末端的TRAF6结合基序结合TRAF6蛋白后可以激活NF-κB信号通路,而携带TRAF6结合位点突变的TIFA无法激活NF-κB[10]。另外,研究发现,人TRAF6蛋白的TRAF结构域和TIFA蛋白的FHA结构域之间的相互作用是介导NF-κB激活的重要因素[16],并且TIFA/TRAF6/NF-κB信号通路的活化也是产生炎性细胞因子的关键通路[13-14]。骆磊[34]研究发现,TRAF6及其下游基因在NDV感染的鸡胚成纤维细胞或SPF鸡免疫器官中均表现为显著上调表达,可能在鸡抵抗NDV感染过程中发挥作用,但是TIFA/TRAF6/NF-κB信号通路在鸡相关病毒感染方面的研究还未见报道。近年来,ALPK1控制TIFA/TRAF6/NF-κB依赖的先天免疫抵抗细菌感染的研究也逐渐被报道[35-37]。例如,鼠伤寒沙门菌和志贺菌感染细胞后会降低ALPK1的产生,并且诱导TIFA和TRAF6蛋白在感染的细胞中形成寡聚体,进而抑制NF-κB的活化以及细胞因子的产生[37]。鉴于TRAF6和TIFA蛋白相互作用在NF-κB信号通路调控的免疫应答中发挥的重要作用,开展TIFA/TRAF6/NF-κB信号通路调控鸡相关病毒复制作用机制的研究具有重要意义。本研究通过对鸡TRAF6和TIFA蛋白的同源性分析发现,鸡与人和其他哺乳动物及其非洲爪蟾的TRAF6和TIFA蛋白之间的同源性差异较大,并且它们功能结构域的氨基酸并不保守。另外,虽然鸡TRAF6和TIFA重组蛋白单独表达时定位在细胞核和细胞质,但是两者共表达时却共同定位在细胞核。进一步通过Co-IP证实,鸡TRAF6和TIFA蛋白存在相互作用,说明两者主要在细胞核中发生相互作用。以上研究将为后续深入探讨与鸡TRAF6和TIFA蛋白相互作用的细胞蛋白以及两者互作调控鸡相关病毒复制的作用机制奠定重要基础。
4 结论本研究成功构建了鸡TRAF6和TIFA基因的重组真核表达载体pCMV-HA-TRAF6和pEGFP-C1-TIFA;虽然鸡与人和其他哺乳动物以及非洲爪蟾TRAF6和TIFA蛋白的序列同源性及功能结构域保守性均较低,但是利用荧光共定位和Co-IP试验证实,鸡TRAF6和TIFA蛋白能在细胞核中发生相互作用。
| [1] | DOU Y, TIAN X D, ZHANG J, et al. Roles of TRAF6 in central nervous system[J]. Curr Neuropharmacol, 2018, 16(9): 1306–1313. DOI: 10.2174/1570159X16666180412094655 |
| [2] | SCHNETZKE U, FISCHER M, KUHN A K, et al. The E3 ubiquitin ligase TRAF6 inhibits LPS-induced AKT activation in FLT3-ITD-positive MV4-11 AML cells[J]. J Cancer Res Clin Oncol, 2013, 139(4): 605–615. DOI: 10.1007/s00432-012-1362-4 |
| [3] | JEONG S, CHO I R, AN W G, et al. STP-A11, an oncoprotein of herpesvirus saimiri augments both NF-κB and AP-1 transcription activity through TRAF6[J]. Exp Mol Med, 2007, 39(1): 56–64. DOI: 10.1038/emm.2007.7 |
| [4] | GUO Y X, XU Y, KANG X L, et al. Molecular cloning and functional analysis of TRAF6 from Yangzhou great white goose Anser anser[J]. Dev Comp Immunol, 2019, 101(1): 103435. DOI: 10.1016/j.dci.2019.103435 |
| [5] |
王晓芬. TRAF6在家蝇免疫及生殖中的作用[D]. 保定: 河北大学, 2019.
WANG X F. The role of TRAF6 in the immunity and reproduction of house flies[D]. Baoding: Hebei University, 2019. (in Chinese) |
| [6] | JIN J, RAN J S, YANG C W, et al. Molecular characterization, expression, and functional analysis of chicken TRAF6[J]. Genet Mol Res, 2017, 16(1). DOI: 10.4238/gmr/6019138 |
| [7] | KOBAYASHI N, KADONO Y, NAITO A, et al. Segregation of TRAF6-mediated signaling pathways clarifies its role in osteoclastogenesis[J]. EMBO J, 2001, 20(6): 1271–1280. DOI: 10.1093/emboj/20.6.1271 |
| [8] | YOSHIDA R, TAKAESU G, YOSHIDA H, et al. TRAF6 and MEKK1 play a pivotal role in the RIG-I-like helicase antiviral pathway[J]. J Biol Chem, 2008, 283(52): 36211–36220. DOI: 10.1074/jbc.M806576200 |
| [9] | LEE N R, BAN J S, LEE N J, et al. Activation of RIG-I-mediated antiviral signaling triggers autophagy through the MAVS-TRAF6-Beclin-1 signaling axis[J]. Front Immunol, 2018, 9: 2096. DOI: 10.3389/fimmu.2018.02096 |
| [10] | TAKATSUNA H, KATO H, GOHDA J, et al. Identification of TIFA as an adapter protein that links tumor necrosis factor receptor-associated factor 6 (TRAF6) to interleukin-1 (IL-1) receptor-associated kinase-1 (IRAK-1) in IL-1 receptor signaling[J]. J Biol Chem, 2003, 278(14): 12144–12150. DOI: 10.1074/jbc.M300720200 |
| [11] | EA C K, SUN L J, INOUE J I, et al. TIFA activates IκB kinase (IKK) by promoting oligomerization and ubiquitination of TRAF6[J]. Proc Natl Acad Sci U S A, 2004, 101(43): 15318–15323. DOI: 10.1073/pnas.0404132101 |
| [12] | GARCÍA-WEBER D, ARRIEUMERLOU C. ADP-heptose: a bacterial PAMP detected by the host sensor ALPK1[J]. Cell Mol Life Sci, 2021, 78(1): 17–29. DOI: 10.1007/s00018-020-03577-w |
| [13] | GAUDET R G, GUO C X, MOLINARO R, et al. Innate recognition of intracellular bacterial growth is driven by the TIFA-dependent cytosolic surveillance pathway[J]. Cell Rep, 2017, 19(7): 1418–1430. DOI: 10.1016/j.celrep.2017.04.063 |
| [14] | DUAN Z Q, YUAN C, HAN Y F, et al. TMT-based quantitative proteomics analysis reveals the attenuated replication mechanism of Newcastle disease virus caused by nuclear localization signal mutation in viral matrix protein[J]. Virulence, 2020, 11(1): 607–635. DOI: 10.1080/21505594.2020.1770482 |
| [15] | INOUE J I, YAGI S, ISHIKAWA K, et al. Identification and characterization of Xenopus laevis homologs of mammalian TRAF6 and its binding protein TIFA[J]. Gene, 2005, 358: 53–69. DOI: 10.1016/j.gene.2005.05.016 |
| [16] | HUANG W C, LIAO J H, HSIAO T C, et al. Binding and enhanced binding between key immunity proteins TRAF6 and TIFA[J]. ChemBioChem, 2019, 20(2): 140–146. DOI: 10.1002/cbic.201800436 |
| [17] | LI Z, LIU X M, LI A Y, et al. Teleost type 2 interleukin-1 receptor (IL-1R2) from the spotted halibut (Verasper variegatus): 3D structure and a role in immune response[J]. Mol Biol (Mosk), 2019, 53(2): 256–266. DOI: 10.1134/S0026893319020109 |
| [18] | XU Q Q, XU P, ZHOU J W, et al. Cloning and expression analysis of two pro-inflammatory cyto-kines, IL-1β and its receptor, IL-1R2, in the Asian swamp eel Monopterus albus[J]. Mol Biol (Mosk), 2016, 50(5): 671–683. DOI: 10.1134/S0026893316030122 |
| [19] | TOMUSCHAT C, O'DONNELL A M, COYLE D, et al. Altered expression of IL36γ and IL36 receptor (IL1RL2) in the colon of patients with Hirschsprung's disease[J]. Pediatr Surg Int, 2017, 33(2): 181–186. DOI: 10.1007/s00383-016-4011-1 |
| [20] | ZHANG R B, LU M, ZHANG S, et al. Renoprotective effects of Tilianin in diabetic rats through modulation of oxidative stress via Nrf2-Keap1 pathway and inflammation via TLR4/MAPK/NF-κB pathways[J]. Int Immunopharmacol, 2020, 88: 106967. DOI: 10.1016/j.intimp.2020.106967 |
| [21] | SIROIS M, ROBITAILLE L, ALLARY R, et al. TRAF6 and IRF7 control HIV replication in macrophages[J]. PLoS One, 2011, 6(11): e28125. DOI: 10.1371/journal.pone.0028125 |
| [22] | PARK H H. Structure of TRAF family: current understanding of receptor recognition[J]. Front Immunol, 2018, 9: 1999. DOI: 10.3389/fimmu.2018.01999 |
| [23] | BISWAS R, CHOWDHURY N, BISWAS S, et al. Structure based virtual screening of natural products to disrupt the structural integrity of TRAF6 C-terminal domain homotrimer[J]. J Mol Graph Model, 2019, 93: 107428. DOI: 10.1016/j.jmgm.2019.08.005 |
| [24] | MOHANTY P, AGRATA R, HABIBULLAH B I, et al. Deamidation disrupts native and transient contacts to weaken the interaction between UBC13 and RING-finger E3 ligases[J]. eLife, 2019, 8: e49223. DOI: 10.7554/eLife.49223 |
| [25] | ZHI X, WANG L P, CHEN H W, et al. L-tetrahydropalmatine suppresses osteoclastogenesis in vivo and in vitro via blocking RANK-TRAF6 interactions and inhibiting NF-κB and MAPK pathways[J]. J Cell Mol Med, 2020, 24(1): 785–798. DOI: 10.1111/jcmm.14790 |
| [26] | HE X M, ZHENG Y Q, LIU S Z, et al. MiR-146a protects small intestine against ischemia/reperfusion injury by down-regulating TLR4/TRAF6/NF-κB pathway[J]. J Cell Physiol, 2018, 233(3): 2476–2488. DOI: 10.1002/jcp.26124 |
| [27] | JI L N, HOU X L, LIU W H, et al. Paeoniflorin inhibits activation of the IRAK1-NF-κB signaling pathway in peritoneal macrophages from lupus-prone MRL/lpr mice[J]. Microb Pathog, 2018, 124: 223–229. DOI: 10.1016/j.micpath.2018.08.051 |
| [28] | FENG Y, ZHENG C J, ZHOU Z P, et al. IL-17A neutralizing antibody attenuates eosinophilic meningitis caused by Angiostrongylus cantonensis by involving IL-17RA/TRAF6/NF-κB signaling[J]. Exp Cell Res, 2019, 384(1): 111554. DOI: 10.1016/j.yexcr.2019.111554 |
| [29] | SANADA T, KIM M, MIMURO H, et al. The Shigella flexneri effector OspI deamidates UBC13 to dampen the inflammatory response[J]. Nature, 2012, 483(7391): 623–626. DOI: 10.1038/nature10894 |
| [30] | ZHANG X, GUO Y J, XU X Y, et al. miR-146a promotes Borna disease virus 1 replication through IRAK1/TRAF6/NF-κB signaling pathway[J]. Virus Res, 2019, 271: 197671. DOI: 10.1016/j.virusres.2019.197671 |
| [31] | HUANG C C F, WENG J H, WEI T Y W, et al. Intermolecular binding between TIFA-FHA and TIFA-pT mediates tumor necrosis factor alpha stimulation and NF-κB activation[J]. Mol Cell Biol, 2012, 32(14): 2664–2673. DOI: 10.1128/MCB.00438-12 |
| [32] | ALMAWI A W, MATTHEWS L A, GUARNÉ A. FHA domains: phosphopeptide binding and beyond[J]. Prog Biophys Mol Biol, 2017, 127: 105–110. DOI: 10.1016/j.pbiomolbio.2016.12.003 |
| [33] | PENNELL S, WESTCOTT S, ORTIZ-LOMBARDÍA M, et al. Structural and functional analysis of phosphothreonine-dependent FHA domain inter-actions[J]. Structure, 2010, 18(12): 1587–1595. DOI: 10.1016/j.str.2010.09.014 |
| [34] |
骆磊. 鸡TRAF3、TRAF6基因的克隆、表达及其在鸡抗病毒免疫应答过程中的作用研究[D]. 雅安: 四川农业大学, 2014.
LUO L. Molecular cloning, tissue distribution of chicken TRAF-3 and TRAF-6 genes, and their roles in chicken's ativiral immune response[D]. Ya'an: Sichuan Agricultural University, 2014. (in Chinese) |
| [35] | YING L, FERRERO R L. Role of NOD1 and ALPK1/TIFA signalling in innate immunity against Helicobacter pylori infection[J]. Curr Top Microbiol Immunol, 2019, 421: 159–177. DOI: 10.1007/978-3-030-15138-6_7 |
| [36] | XUE Y S, MAN S M. ALPK1:innate attraction to the sweetness of bacteria[J]. Cell Res, 2018, 28(12): 1125–1126. DOI: 10.1038/s41422-018-0100-0 |
| [37] | MILIVOJEVIC M, DANGEARD A S, KASPER C A, et al. ALPK1 controls TIFA/TRAF6-dependent innate immunity against heptose-1, 7-bisphosphate of gram-negative bacteria[J]. PLoS Pathog, 2017, 13(2): e1006224. DOI: 10.1371/journal.ppat.1006224 |



