2. 江苏省家禽科学研究所 科技创新中心, 扬州 225125
2. Science and Technology Innovation Center, Poultry Institute of Jiangsu Province, Yangzhou 225125, China
由于近交而引起的后代群体适合度下降的现象被称为近交衰退[1],近交衰退是物种资源保护尤其是濒危物种保护过程中备受关注的问题之一。群体遗传学提出了两个解释近交衰退遗传基础的经典假说: 显性假说和超显性假说。显性假说认为,近交过程中,基因组纯合性的增加导致了有害隐性等位基因的暴露;超显性假说提出,在整个基因组中,杂合子一般优于纯合子,近交增加了纯合子出现的概率,因此会表现出衰退[2]。近些年研究发现,这两种假说对于某些近交衰退现象却无法完全解释[2-6],比如近交衰退程度会随着环境压力的变化而变化的不确定现象等[7-8]。DNA表观修饰也会受到环境的影响,进而影响到基因的表达和表型的可塑性[9-11]。并且已有研究表明,近交衰退与DNA表观遗传修饰密切相关[12-14]。表观遗传变化是发生在基因组DNA中的重要变异,它在不直接改变DNA编码序列的情况下影响着基因的表达[15-16]。
DNA甲基化是目前研究最广泛的表观遗传修饰方式之一[17],其参与多种生物学过程[18-19],在调控基因表达和维持基因组稳定性方面起着关键的作用[20]。脊椎动物基因组DNA甲基化主要发生在CpG胞嘧啶上,并且它的调控多为通过CpG岛(CpG island)多个CpG位点的甲基化共同作用来完成,而不是通过对某单个CpG位点的甲基化或去甲基化来激活或失活基因[21]。与CpG岛邻近(~2 kb)的低CpG密度区域被定义为CpG岛岸(CpG island shore),这些区域的甲基化与转录的激活也密切相关。匡颖等[22]研究表明,近交系小鼠生长发育缓慢可能与H19基因上游区域被甲基化进而引起该基因的异常表达有关;蒋曹德[23]以梅山猪、大白猪群体为亲本构建了两个自繁群体与两个杂交群体,发现群体内自繁的子代甲基化水平略高于杂交子代群体,且两个群体间某些性状差异可能与某特定位点甲基化差异相关[23];Venney等[24]研究表明,三文鱼的近交衰退与其CK-1、GTIIBS和hsp70三个特定基因的甲基化变化有关;杨晶淼等[25]研究报告了马氏珠母贝的近交与杂交家系的生长性状和甲基化水平均存在显著性差异。植物上,关于山萝卜[14]和茄科植物[26]的两项研究均表明,DNA甲基化与近交衰退之间存在着密切的关系,并且近交个体比远交个体存在更为广泛的全基因组DNA甲基化现象。尽管有这些研究,但是人们对DNA甲基化,尤其是脊椎动物CpG岛及其附近区域甲基化在近交衰退中的调控作用还没有完全深刻地理解。
家禽繁殖性能在家禽生产、育种和品种保护中具有极其重要的地位,尤其在地方鸡遗传资源活体保护过程中,群体繁殖性能近交衰退现象时有发生,严重影响着保种群的规模和世代的延续。本课题组在前期的研究中,已成功组建了狼山鸡高近交组(分子近交系数FIS> 0.15)和低近交组(分子近交系数FIS<0.04),且观察到狼山鸡高近交组繁殖性能发生了显著衰退现象[27],然而,关于繁殖性能近交衰退个体DNA甲基化变化,尤其是CpG岛及其附近重要调控区域甲基化变化情况尚未进行深入探索。基于此,本研究利用新一代测序技术—全基因组重亚硫酸盐测序(whole-genome bisulfite sequencing,WGBS)技术检测和分析狼山鸡高、低近交组性腺轴组织(包括卵巢和下丘脑)全基因组甲基化水平变化情况,着重筛选CpG岛及其附近区域内存在的差异甲基化区域(differential methylation region,DMR),并探讨其在狼山鸡繁殖性能近交衰退中可能发挥的调控作用,研究结果将为今后家禽育种和物种资源保护工作提供理论参考依据。
1 材料与方法 1.1 试验动物和样品采集选取江苏省家禽科学研究所国家级地方鸡种基因库保存的地方鸡品种—狼山鸡为试验素材,基于前期狼山鸡高、低近交组母鸡繁殖性状(包括开产日龄、300天产蛋数、开产体重和开产蛋重)的表型值记录,选取高近交组中繁殖性能显著衰退的个体3只和低近交组中繁殖性能处于群体均值附近的个体3只,快速屠宰并采集其下丘脑和卵巢组织,迅速投入液氮中,然后转移至-80 ℃冰箱中保存备用。
1.2 DNA提取和WGBS建库测序使用德国凯杰公司提供的组织DNA快速提取试剂盒提取各样本基因组DNA, 并使用安捷伦2100分光光度计进行检测。DNA样品经超声片段化后进行亚硫酸氢盐转化,使用DNA甲基化测序文库构建试剂盒(斯威夫特生物科技,美国),将单链DNA片段进行接头连接,对DNA片段进行8个循环PCR扩增:95 ℃预变性2 min;8个循环(95 ℃变性30 s,65 ℃退火20 s,72 ℃延伸45 s);最后72 ℃延伸性7 min。扩增反应体系包括预混反应体系25 μL(低浓度EDTA TE 10 μL, Buffer R1 10 μL, 反应物R2 4 μL,R3酶1 μL),DNA纯化产物和探针引物25 μL,共计50 μL。对扩增产物进行纯化和完整性检测,最后使用Illumina HiSeq 4000测序平台对测序文库进行双末端测序,每个文库测序深度达30×。
1.3 数据过滤质控和甲基化胞嘧啶(mC)位点识别使用Cutadapt[28]软件去除测序Reads中含有测序接头、污染、低质量碱基和未知碱基的Reads,序列质量进一步使用FastQC (http://www.bioinformatics.babraham.ac.uk/projects/fastqc/)进行验证。通过质控的Reads被比对到鸡的参考基因组中,使用Samtool[29]软件对比对上的Reads进一步去重复。对于每个胞嘧啶位点,其甲基化水平取决于覆盖到mC位点的Reads数与总Reads数的比值。使用二项分布检验鉴别错误检出率(false-discovery rate,FDR)≤0.01的mC位点,所有mC位点测序覆盖度需≥3。根据mC位点上、下游碱基种类,将mC位点分为3种类型:mCG、mCHG和mCHH,其中H代表腺苷(A)、胞嘧啶(C)或胸腺嘧啶(T)。
1.4 DMR检测与CpG岛区DMR相关基因功能富集分析使用R语言包MethylKit[30]进行DMR检测,采用滑窗方法(1 000 bp窗口,500 bp步长)扫描基因组,检测高、低近交组中存在差异甲基化的区域,对每个窗口进行Fisher精确检验,并使用FDR对多次检验的P值进行校正。FDR < 0.05且甲基化水平差异倍数至少为2倍的窗口被鉴定作为DMR。采用emboss[31-32]进行CpG岛鉴定,将CpG岛定义为长度至少100 bp、GC含量大于50%,且CpG二核苷酸的出现率(观测值/期望值)大于60%的基因组区域。对CpG岛区(CpG岛及其上、下游2 kb区域)甲基化信息进行统计分析,筛选位于CpG岛区的DMR。为探索这些DMR的功能,对包含这些DMR的基因(称为DMR相关基因或差异甲基化基因)进行GO和KEGG数据库功能注释和富集分析,P < 0.05的GO条目和Pathway信号通路为显著富集的条目和通路。
1.5 亚硫酸氢盐测序PCR(bisulfite sequencing PCR, BSP)方法验证WGBS分析结果为验证WGBS测序分析结果的可靠性,从卵巢中随机选取2个DMR相关基因(CDC27、SRD5A1),对其CpG岛区域甲基化水平进行验证。利用Methylation Primer Express V1.0软件设计扩增引物,引物信息见表 1。选用WGBS相同的DNA样品,每个样品取1 mg,用EZ DNA Methylation-GoldTM试剂盒进行亚硫酸氢盐转化。利用设计的引物扩增转化后的DNA目标区域,PCR产物纯化后进行TA载体克隆,每个基因挑选10~15个克隆子进行Sanger测序,对测序结果采用BiQ Analyzer软件分析。
|
|
表 1 BSP验证试验引物信息 Table 1 Primers information of BSP experiment |
采用IBM SPSS Statistics 20.0软件统计分析高、低近交组各繁殖性状,通过独立样本t(two-sample t-test)检验方法估计两组间各性状差异显著性,当P < 0.05时差异显著,当P < 0.01时差异极显著;根据测序Reads中覆盖到mC位点的Reads数与总Reads数的比值,计算全基因组甲基化水平;根据clean reads中唯一比对的reads,运用R语言psych包对各样本进行主成分(principle components analysis, PCA)分析。
2 结果 2.1 狼山鸡高、低近交组繁殖性能差异比较分析狼山鸡高、低近交组间各繁殖性状的差异,发现高、低近交组在开产日龄、300天产蛋数和开产体重3个性状上均存在显著或极显著差异(P < 0.05或P < 0.01)(表 2),而在开产蛋重上差异不显著(P>0.05)。
|
|
表 2 高、低近交组狼山鸡各繁殖性状差异 Table 2 Differences in reproductive traits between the two chicken groups |
测序样本总共12个,包含4个组,每组3个生物学重复,每个样本测序深度≥30×,产生约154.9 Mb clean reads,平均69.14% 的clean reads可以唯一比对到参考基因组中,这些clean reads用于后续的分析。为证明样本采集的准确性和可靠性,对其进行主成分分析(PCA),结果表明,来自同一组的3个生物学重复相距较近(图 1),表明试验样本采集以及测序数据较为准确、可靠。
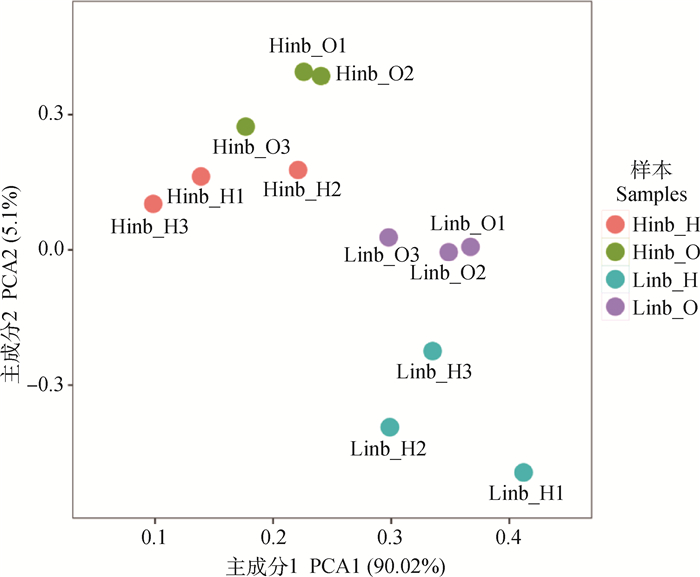
|
Hinb_H. 高近交组下丘脑;Hinb_O. 高近交组卵巢;Linb_H. 低近交组下丘脑;Linb_O. 低近交组卵巢 Hinb_H. and Linb_H. Represent hypothalamus of strongly and weakly inbred chickens, respectively; Hinb_O. and Linb_O. Represent ovaries of strongly and weakly inbred chickens, respectively 图 1 样本主成分分析 Fig. 1 Principal components analysis of the samples |
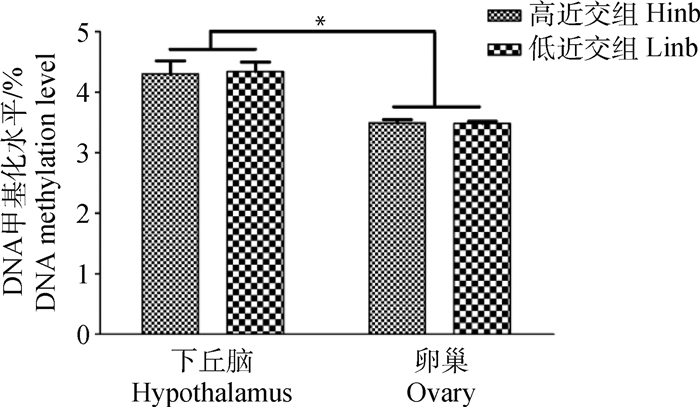
根据测序获得reads中覆盖到mC位点的reads数以及reads总数,计算并分析了狼山鸡高近交组和低近交组中卵巢和下丘脑全基因组DNA甲基化水平,结果发现,狼山鸡高近交组卵巢和下丘脑全基因组甲基化水平分别为3.49%和4.31%,低近交组卵巢和下丘脑全基因组甲基化水平分别为3.48%和4.35%。相同组织,狼山鸡高近交组与低近交组全基因组甲基化水平差异不显著(P>0.05),然而两种组织间比较发现,下丘脑基因组整体甲基化水平显著高于卵巢组织(P < 0.05,图 2)。
|
“*”表示差异显著(P < 0.05) "*"indicate significant difference(P < 0.05) 图 2 狼山鸡高、低近交组卵巢和下丘脑全基因组甲基化水平 Fig. 2 Whole-genome methylation levels of ovaries and hypothalamus from strongly and weakly inbred Langshan chickens |
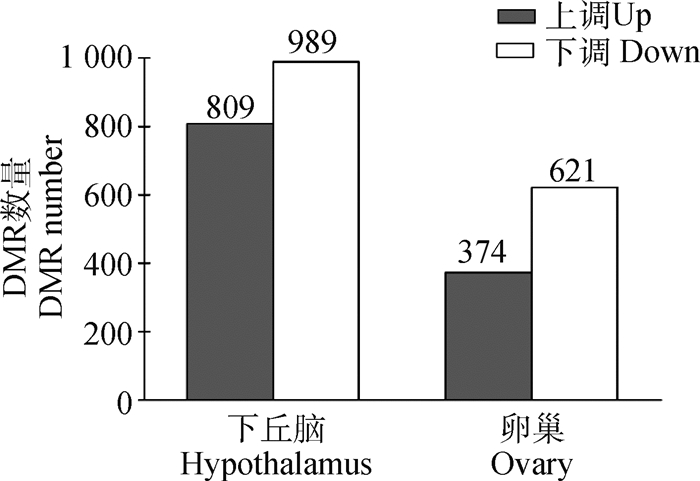
差异甲基化区域(DMR)是表观遗传变化的一个重要标志,涉及到许多生物学过程调控。为探索与狼山鸡繁殖性能近交衰退相关的差异甲基化区域, 本研究筛选高近交组和低近交组间甲基化水平差异大于等于2倍,且FDR < 0.05的区域作为差异甲基化区域。结果在两组狼山鸡的下丘脑和卵巢中分别检测到5 948和4 593个差异甲基化区域,其中分别有1 798和995个差异甲基化区域位于CpG岛区。下丘脑CpG岛区检测到的差异甲基化区域中,有809个上调,989个下调;卵巢CpG岛区的995个差异甲基化区域中,有374个上调,621个下调(图 3)。
|
图 3 狼山鸡高、低近交组卵巢和下丘脑基因组CpG岛区差异甲基化区域分析 Fig. 3 Analysis of differential methylation regions in CpG islands and their nearby regions in the genomes of hypothalamus and ovary between the strongly and weakly inbred Langshan chickens |
脊椎动物CpG岛区DNA甲基化在基因转录、沉默等过程中起重要调控作用,因此,对下丘脑和卵巢基因组CpG岛区差异甲基化区域进行注释分析发现,下丘脑和卵巢基因组CpG岛区差异甲基化区域分别注释到了1 020和552个基因,这些基因称为DMR相关基因或差异甲基化基因。
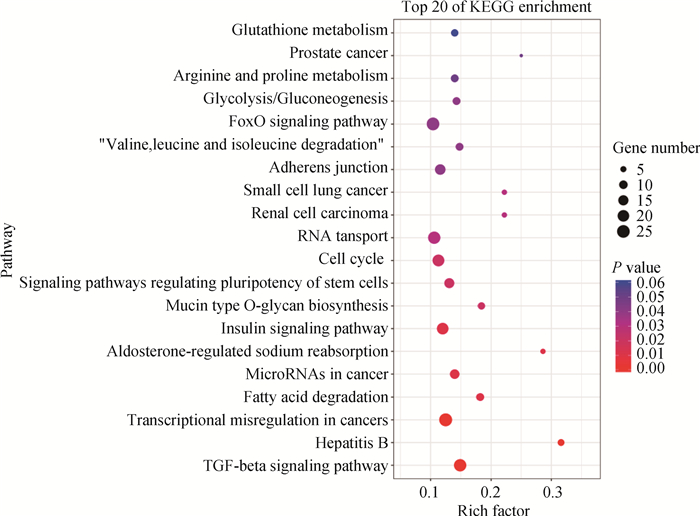
2.4 下丘脑基因组CpG岛区差异甲基化基因功能注释分析对下丘脑基因组CpG岛区差异甲基化基因进行GO和KEGG注释和功能富集分析发现,分别有920和686个基因被注释到了GO和KEGG数据库。GO富集分析结果显示,有141个GO条目被这些基因显著富集(P < 0.05),这些GO条目主要涉及到信号转导、神经系统发育、转录调控、蛋白激酶活性等生物学过程,另外,卵母细胞成熟调控以及生殖系统发育相关的GO条目也被显著富集到。KEGG富集分析表明,这些差异甲基化基因显著富集于19条KEGG信号通路中(P < 0.05),包括转化生长因子β信号通路、乙型肝炎、脂肪酸代谢、胰岛素信号通路、调控多能性干细胞信号通路、糖酵解/糖异生、氨基酸代谢等信号通路(图 4),其中Smad7a、SMAD6、BMP2、TGFB2、MAPK6等繁殖相关基因包含在这些通路中。
|
图 4 下丘脑基因组CpG岛区差异甲基化基因KEGG功能富集分析 Fig. 4 KEGG enrichment analysis of genes with differentially methylated CpG island in hypothalamus |
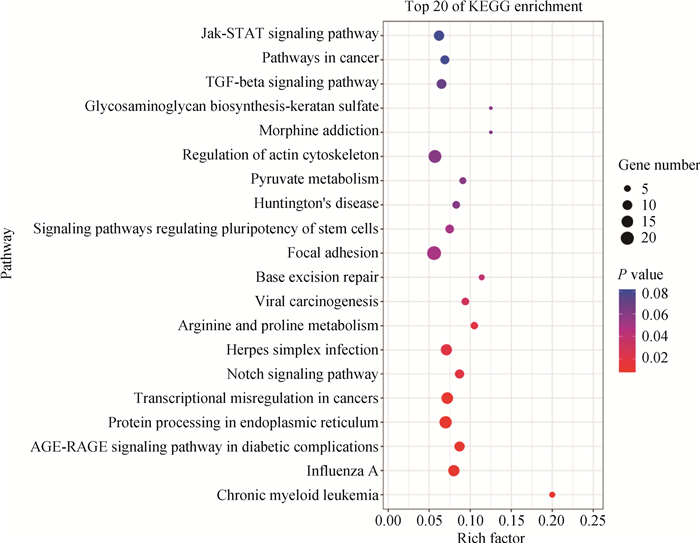
对卵巢基因组CpG岛区差异甲基化基因进行GO和KEGG注释和功能富集分析发现,分别有500和369个基因被注释到了GO和KEGG数据库。GO富集分析结果表明,有105个GO条目被这些基因显著富集(P < 0.05),其中涉及到神经系统发育、细胞增殖、离子转运、转录调控等(P < 0.05)。KEGG富集分析结果显示,12条信号通路被显著富集(P < 0.05),包括慢性骨髓白血病、流感A、精氨酸和脯氨酸代谢、黏着连接、调控多能性干细胞信号通路等(图 5)。另外,还发现一些与卵子发育和性激素分泌相关的通路被富集到,如黄体酮介导的卵母细胞成熟、卵母细胞减数分裂、GnRH信号通路、雌激素信号通路等,其中包含CDC27、ADCY8、AKT3等10个差异甲基化基因(表 3)。
|
图 5 卵巢基因组CpG岛区差异甲基化基因KEGG功能富集分析 Fig. 5 KEGG enrichment analysis of genes with differentially methylated CpG island in ovary |
|
|
表 3 卵巢中富集的繁殖相关通路和CpG岛区差异甲基化基因 Table 3 Enriched pathways related to reproduction and genes with differentially methylated CpG island in ovary |
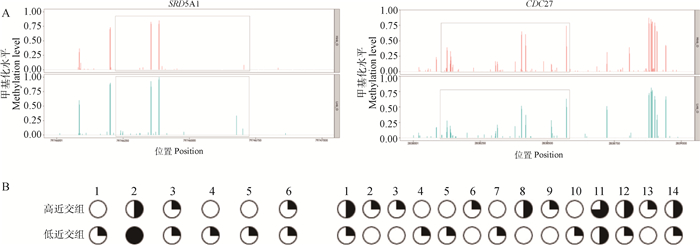
为验证全基因组重亚硫酸盐测序分析结果的可靠性,随机挑选2个CpG岛区差异甲基化区域(Chr27:2838112-2838584和Chr2:79746219-79746715),这两个差异甲基化区域分别位于CDC27和SRD5A1基因的CpG岛中。采用BSP—克隆测序法对这两个区域甲基化情况进行检测,结果显示,在SRD5A1和CDC27基因的验证区域分别检测到了6和14个甲基化CpG位点(图 6B),且这些位点的平均甲基化水平在高近交组与低近交组间的变化趋势与全基因组重亚硫酸盐测序结果(图 6A)相一致,表明了全基因组重亚硫酸盐测序分析结果的准确性和可靠性。
|
图A中红色峰代表高近交组的甲基化水平,蓝色峰为低近交组的甲基化水平,灰色框中的区域为BSP验证区域;图B中每个圆点代表一个甲基化位点,各圆点中黑色部分的占比代表各甲基化位点的甲基化水平 In figure A, red and blue peaks represent methylation levels of strongly and weakly inbred chicken groups, respectively, the regions in grey boxes are the verified regions by BSP. In figure B, each dot represents a methylation site, and the proportion of black part in each dot represents the methylation level of each methylation site 图 6 BSP—克隆测序法对SRD5A1和CDC27基因CpG岛区差异甲基化区域验证分析 Fig. 6 Verification of differentially methylated regions in CpG islands of SRD5A1 and CDC27 genes using BSP—clone sequencing method |
DNA甲基化尤其是脊椎动物CpG岛区的甲基化在调控基因转录以及转录后生物学过程中发挥重要作用[21],大量研究表明,DNA甲基化与动植物的近交衰退现象密切相关[10-12]。本研究基于课题组前期狼山鸡高、低近交组的成功组建,以及狼山鸡高近交组个体出现的繁殖性能近交衰退现象,通过全基因组重亚硫酸盐测序技术开展狼山鸡高近交组与低近交组间性腺轴组织DNA甲基化差异比较分析,试图揭示DNA甲基化在狼山鸡繁殖性能近交衰退中的调控作用。
通过比较分析狼山鸡高、低近交组卵巢和下丘脑基因组整体甲基化水平差异,发现高近交组和低近交组间同一组织基因组整体甲基化水平差异不显著(P>0.05),然而,将基因组划分成许多个小的区域,分析各区域高、低近交组间的甲基化水平差异时,却发现了大量的上调或下调的差异甲基化区域,可能是由于这些区域上、下调趋势正好相反,导致整个基因组甲基化水平在高、低近交组间差异不显著。差异甲基化区域是表观遗传变化的一个重要标志,被看作为参与基因转录水平调控的功能性区域,尤其是脊椎动物CpG岛区DNA甲基化的变化一直以来受到研究者们的极大关注[33-36]。本研究在下丘脑和卵巢基因组CpG岛区分别检测到了1 798和995个差异甲基化区域,这些差异甲基化区域分别注释到了1 020和552个基因,为了进一步探索这些差异甲基化区域的功能,对差异甲基化区域相关基因进行了功能注释和富集分析,发现了大量差异甲基化基因富集在神经系统发育、信号转导和离子转运等神经内分泌调节相关生物学过程GO条目,这些生物学过程与鸡的繁殖性能密切相关。KEGG功能富集分析显示,卵巢中一些差异甲基化基因富集在了卵子发育和性激素分泌相关的通路,如黄体酮介导的卵母细胞成熟、卵母细胞减数分裂、GnRH信号通路、雌激素信号通路等,包含CDC27、ADCY8、AKT3等10个差异甲基化基因,推测这些基因CpG岛区的差异甲基化在狼山鸡繁殖性能近交衰退中可能发挥重要调控作用[37]。其中,CDC27基因CpG岛区甲基化差异情况进一步得到了BSP-克隆测序方法的验证,CDC27是编码细胞周期进程所必需的一种蛋白质[38-39],被认为与细胞增殖和细胞分裂相关[40],本研究中,该基因CpG岛区甲基化水平在狼山鸡高近交组中显著上调,推测该基因CpG岛区甲基化上调影响了其转录表达,进而影响了卵母细胞分裂和成熟,导致狼山鸡高近交组繁殖性能衰退。
另外,在下丘脑和卵巢差异甲基化基因显著富集的通路中,发现多条通路与氨基酸、脂肪和糖代谢相关,推测狼山鸡的高度近交可能影响了其基础代谢,进而可能间接影响到其生殖系统发育,导致其繁殖性能下降。一些与疾病相关的信号通路也被下丘脑和卵巢差异甲基化基因显著富集到,如乙型肝炎、慢性骨髓白血病、流感A等,表明狼山鸡高度近交后,其抗病力或免疫力方面也受到了影响,可能需要后续进一步关注和研究。
4 结论狼山鸡高、低近交组间基因组整体DNA甲基化水平差异不显著(P>0.05),但全基因组范围内仍检测到大量差异甲基化区域,且发现大量差异甲基化基因富集到繁殖相关的生物学过程和通路中,如CDC27、ADCY8、AKT3等差异甲基化基因,推测这些基因CpG岛区甲基化在狼山鸡繁殖性能近交衰退过程中发挥重要作用。本研究结果为今后深入研究鸡繁殖性能近交衰退调控机制提供了有力线索,为物种资源保护工作提供了科学依据。
| [1] | WILLOUGHBY J R, IVY J A, LACY R C, et al. Inbreeding and selection shape genomic diversity in captive populations: Implications for the conservation of endangered species[J]. PLoS One, 2017, 12(4): e0175996. DOI: 10.1371/journal.pone.0175996 |
| [2] | KRISTENSEN T N, PEDERSEN K S, VERMEULEN C J, et al. Research on inbreeding in the 'omic' era[J]. Trends Ecol Evol, 2010, 25(1): 44–52. DOI: 10.1016/j.tree.2009.06.014 |
| [3] | ARMBRUSTER P, REED D H. Inbreeding depression in benign and stressful environments[J]. Heredity (Edinb), 2005, 95(3): 235–242. DOI: 10.1038/sj.hdy.6800721 |
| [4] | CHEPTOU P O, DONOHUE K. Environment-dependent inbreeding depression: its ecological and evolutionary significance[J]. New Phytol, 2011, 189(2): 395–407. DOI: 10.1111/j.1469-8137.2010.03541.x |
| [5] | FOX C W, REED D H. Inbreeding depression increases with environmental stress: an experimental study and meta-analysis[J]. Evolution, 2011, 65(1): 246–258. DOI: 10.1111/j.1558-5646.2010.01108.x |
| [6] | REED D H, FOX C W, ENDERS L S, et al. Inbreeding-stress interactions: evolutionary and conservation consequences[J]. Ann N Y Acad Sci, 2012, 1256: 33–48. DOI: 10.1111/j.1749-6632.2012.06548.x |
| [7] | ENDERS L S, NUNNEY L. Sex-specific effects of inbreeding in wild-caught Drosophila melanogaster under benign and stressful conditions[J]. J Evol Biol, 2010, 23(11): 2309–2323. DOI: 10.1111/j.1420-9101.2010.02085.x |
| [8] | PILAKOUTA N, JAMIESON S, MOORAD J A, et al. Parental care buffers against inbreeding depression in burying beetles[J]. Proc Natl Acad Sci U S A, 2015, 112(26): 8031–8035. DOI: 10.1073/pnas.1500658112 |
| [9] | BOSSDORF O, RICHARDS C L, PIGLIUCCI M. Epigenetics for ecologists[J]. Ecol Lett, 2008, 11(2): 106–115. |
| [10] |
丁勇, 许超, 吴季辉, 等. 表观遗传学研究进展[J]. 中国科学: 生命科学, 2017, 47(1): 3–15.
DING Y, XU C, WU J H, et al. Recent progress in epigenetics[J]. Scientia Sinica Vitae, 2017, 47(1): 3–15. (in Chinese) |
| [11] |
甘麦邻, 杨大洪, 谭娅, 等. 环境因素引起的哺乳动物跨代DNA甲基化修饰现象[J]. 畜牧兽医学报, 2017, 48(12): 2225–2231.
GAN M L, YANG D H, TAN Y, et al. The study of influence of environment on transgenerational inheritance of DNA methylation in mammals[J]. Acta Veterinaria et Zootechnica Sinica, 2017, 48(12): 2225–2231. (in Chinese) |
| [12] | HOWARD J T, PRYCE J E, BAES C, et al. Invited review: Inbreeding in the genomics era: Inbreeding, inbreeding depression, and management of genomic variability[J]. J Dairy Sci, 2017, 100(8): 6009–6024. DOI: 10.3168/jds.2017-12787 |
| [13] | CHEPTOU P O, DONOHUE K. Epigenetics as a new avenue for the role of inbreeding depression in evolutionary ecology[J]. Heredity (Edinb), 2013, 110(3): 205–206. DOI: 10.1038/hdy.2012.66 |
| [14] | VERGEER P, WAGEMAKER N, OUBORG N J. Evidence for an epigenetic role in inbreeding depression[J]. Biol Lett, 2012, 8(5): 798–801. DOI: 10.1098/rsbl.2012.0494 |
| [15] | HONG Y, HONG S H, OH Y M, et al. Identification of lung cancer specific differentially methylated regions using genome-wide DNA methylation study[J]. Mol Cell Toxicol, 2018, 14(3): 315–322. DOI: 10.1007/s13273-018-0034-0 |
| [16] |
晏强, 周献青, 薛雯, 等. 表观遗传DNA甲基化和组蛋白修饰与疾病关系的研究进展[J]. 医学综述, 2017, 23(16): 3160–3163, 3169.
YAN Q, ZHOU X Q, XUE W, et al. Advances in the relationship between epigenetic DNA methylation and histone modification with diseases[J]. Medical Recapitulate, 2017, 23(16): 3160–3163, 3169. (in Chinese) |
| [17] | ELHAMAMSY A R. DNA methylation dynamics in plants and mammals: overview of regulation and dysregulation[J]. Cell Biochem Funct, 2016, 34(5): 289–298. DOI: 10.1002/cbf.3183 |
| [18] | YANG K L, LIANG L L, RAN F L, et al. The DmtA methyltransferase contributes to Aspergillus flavus conidiation, sclerotial production, aflatoxin bio-synthesis and virulence[J]. Sci Rep, 2016, 6(1): 23259. DOI: 10.1038/srep23259 |
| [19] |
赵亚涵, 郝海生, 杜卫华, 等. 新鲜、玻璃化冷冻牛卵母细胞体外受精囊胚全基因组甲基化模式初探[J]. 畜牧兽医学报, 2019, 50(6): 1179–1188.
ZHAO Y H, HAO H S, DU W H, et al. Study on whole genome methylation pattern of in vitro fertilized blastocysts from fresh and vitrified bovine oocytes[J]. Acta Veterinaria et Zootechnica Sinica, 2019, 50(6): 1179–1188. (in Chinese) |
| [20] | MEHTA A, DOBERSCH S, ROMERO-OLMEDO A J, et al. Epigenetics in lung cancer diagnosis and therapy[J]. Cancer Metastasis Rev, 2015, 34(2): 229–241. DOI: 10.1007/s10555-015-9563-3 |
| [21] | MENG H, CAO Y, QIN J, et al. DNA methylation, its mediators and genome integrity[J]. Int J Biol Sci, 2015, 11(5): 604–617. DOI: 10.7150/ijbs.11218 |
| [22] |
匡颖, 邓涛, 王龙, 等. 近交系ES鼠胚胎发育及印迹异常的研究[C]//中国遗传学会"发育、遗传和疾病"研讨会论文汇编集. 乌鲁木齐: 中国遗传学会, 2007.
KUANG Y, DENG T, WANG L, et al. Study on embryonic development and imprinting abnormalities of inbred ES mice[C]//Chinese Genetic Society Seminar Papers on "Development, Heredity and Disease". 2007. (in Chinese) |
| [23] |
蒋曹德. 猪DNA甲基化与杂种表现的关系及遗传多样性分析[D]. 武汉: 华中农业大学, 2004.
JIANG C D. Relationship between DNA methylation and hybrid performances and pigs genetic diversity analysis[D]. Wuhan: Huazhong Agricultural University, 2004. (in Chinese) |
| [24] | VENNEY C J, JOHANSSON M L, HEATH D D. Inbreeding effects on gene-specific DNA methylation among tissues of Chinook salmon[J]. Mol Ecol, 2016, 25(18): 4521–4533. DOI: 10.1111/mec.13777 |
| [25] |
杨晶淼, 蔡炜裕, 罗少杰, 等. 马氏珠母贝(Pinctada fucata martensii)近交家系与其杂交子代DNA甲基化差异分析[J]. 基因组学与应用生物学, 2018, 37(5): 1926–1932.
YANG J M, CAI W Y, LUO S J, et al. Variation analysis of DNA methylation in inbred and hybrid families of pearl oyster (Pinctada fucata martensii)[J]. Genomics and Applied Biology, 2018, 37(5): 1926–1932. (in Chinese) |
| [26] | NAKAMURA S, HOSAKA K. DNA methylation in diploid inbred lines of potatoes and its possible role in the regulation of heterosis[J]. Theor Appl Genet, 2010, 120(2): 205–214. DOI: 10.1007/s00122-009-1058-6 |
| [27] | HAN W, XUE Q, LI G H, et al. Genome-wide analysis of the role of DNA methylation in inbreeding depression of reproduction in Langshan chicken[J]. Genomice, 2020, 112(4): 2677–2687. DOI: 10.1016/j.ygeno.2020.02.007 |
| [28] | MARTIN M. Cutadapt removes adapter sequences from high-throughput sequencing reads[J]. EMBnet J, 2011, 17(1): 10–12. DOI: 10.14806/ej.17.1.200 |
| [29] | ETHERINGTON G J, RAMIREZ-GONZALEZ R H, MACLEAN D. bio-samtools 2:a package for analysis and visualization of sequence and alignment data with SAMtools in Ruby[J]. Bioinformatics, 2015, 31(15): 2565–2567. DOI: 10.1093/bioinformatics/btv178 |
| [30] | AKALIN A, KORMAKSSON M, LI S, et al. MethylKit: a comprehensive R package for the analysis of genome-wide DNA methylation profiles[J]. Genome Biol, 2012, 13(10): R87. DOI: 10.1186/gb-2012-13-10-r87 |
| [31] | IOSHIKHES I P, ZHANG M Q. Large-scale human promoter mapping using CpG islands[J]. Nat Genet, 2000, 26(1): 61–63. DOI: 10.1038/79189 |
| [32] | RICE P, LONGDEN I, BLEASBY A. EMBOSS: the european molecular biology open software suite[J]. Trends Genet, 2000, 16(6): 276–277. DOI: 10.1016/S0168-9525(00)02024-2 |
| [33] | CHAPMAN A G, COTTON A M, KELSEY A D, et al. Differentially methylated CpG island within human XIST mediates alternative P2 transcription and YY1 binding[J]. BMC Genet, 2014, 15(1): 89. DOI: 10.1186/s12863-014-0089-4 |
| [34] |
郭添福, 张志燕, 陈冬, 等. 不同性别大白猪肌肉全基因组高分辨率单碱基甲基化差异分析[J]. 畜牧兽医学报, 2018, 49(11): 2326–2339.
GUO T F, ZHANG Z Y, CHEN D, et al. High resolution and single base genome-wide methylation variance analysis of muscle of large white pigs with different sexes[J]. Acta Veterinaria et Zootechnica Sinica, 2018, 49(11): 2326–2339. (in Chinese) |
| [35] |
杜小龙, 王麒, 张乐超, 等. 山羊DCT基因启动子区甲基化水平、SNP与毛色特征关系研究[J]. 畜牧兽医学报, 2019, 50(2): 271–279.
DU X L, WANG Q, ZHANG L C, et al. Study on the relationship between methylation level, SNP in promoter region of DCT gene and hair color in goat[J]. Acta Veterinaria et Zootechnica Sinica, 2019, 50(2): 271–279. (in Chinese) |
| [36] |
胡慧艳, 贾青, 侯胜奎, 等. 猪DKK1基因启动子区的克隆及其活性分析[J]. 畜牧兽医学报, 2017, 48(6): 1150–1157.
HU H Y, JIA Q, HOU S K, et al. Cloning and activity analysis of the promoter region of swine DKK1 Gene[J]. Acta Veterinaria et Zootechnica Sinica, 2017, 48(6): 1150–1157. (in Chinese) |
| [37] | CHAMBERS S, TARASOV K, AON M A, et al. Metabolic enzyme acetylation is elicited in response to stress induced by cardiac specific overexpression of human ADCY8[J]. FASEB J, 2020, 34(S1): 1. |
| [38] | LAMB J R, MICHAUD W A, SIKORSKI R S, et al. Cdc16p, Cdc23p and Cdc27p form a complex essential for mitosis[J]. EMBO J, 1994, 13(18): 4321–4328. DOI: 10.1002/j.1460-2075.1994.tb06752.x |
| [39] | XIN Y F, NING S L, ZHANG L, et al. CDC27 facilitates gastric cancer cell proliferation, invasion and metastasis via twist-induced epithelial-mesenchymal transition[J]. Cell Physiol Biochem, 2018, 50(2): 501–511. DOI: 10.1159/000494164 |
| [40] | ABBASPOURKHARYEKI M, ANVEKAR N J, RAMACHANDRA N B. The possible role of point mutations and activation of the CDC27 gene in progression of multiple myeloma[J]. Meta Gene, 2020, 26: 100761. DOI: 10.1016/j.mgene.2020.100761 |



