2. 福建傲农生物科技集团股份有限公司, 漳州 363000;
3. 厦门银祥集团有限公司 肉食品安全生产技术国家重点实验室, 厦门 361100;
4. 汕头大学理学院, 广东省海洋生物技术重点实验室, 汕头 515063
2. Fujian Aonong Biological Science and Technology Group Co. Ltd., Zhangzhou 363000, China;
3. State Key Laboratory of Food Safety Technology for Meat Products, Xiamen Yinxiang Group Co. Ltd., Xiamen 361100, China;
4. Guangdong Provincial Key Laboratory of Marine Biotechnology, College of Science, Shantou University, Shantou 515063, China
在畜禽选育中,育种值估计准确性是影响畜禽遗传进展的重要因素之一。基因组选择(genomic selection,GS)能有效提高育种值估计准确性,已在奶牛育种中取得了较大效益[1],并逐渐推广到猪[2]、鸡[3]、鱼[4]等畜禽及水产品种选育中。在实施基因组选择时,参考群规模和标记密度是影响基因组选择准确性的重要因素[5-6]。由于基因分型成本较高,常通过构建具有高密度SNP芯片数据或全基因组序列数据的参考群体,根据不同方法对低密度SNP芯片进行基因型填充,以获得高质量的基因型数据[7-9]。目前,该技术已广泛应用于奶牛[10]、猪[11]、鸡[12]等畜禽育种中。
通过基因型填充获取高质量高密度的基因型数据是提高基因组选择准确性的有效方式之一[13]。然而,基因型填充的准确性受诸多因素影响,如基因型填充软件[14]、参考群规模[15]、参考群体和目标群体之间的关系[16]等。其中,参考群筛选和构建方式尤为重要,参考群规模越大,基因型填充准确性越高,然而基因分型成本也随之增加[17]。研究表明,当参考群体与目标群体间的亲缘关系较近时,两者之间的单倍型长度和数量都会增加,使填充更准确[18-19]。因此,如何筛选关键个体构建参考群至关重要,能够保证基因型填充准确性从而降低成本。
本研究拟使用矮小黄羽肉鸡资源群体作为研究对象,采用不同方法筛选关键个体构建参考群,将低密度标记的验证群填充至高密度标记,通过比较不同参考群筛选方法的基因型填充准确性及其在基因组预测中的应用效果,探究构建参考群最佳的策略,为基因型填充技术在遗传育种中的应用提供参考。
1 材料与方法 1.1 试验群体本研究所用群体来自广东温氏南方家禽育种有限公司提供的矮小型黄羽肉鸡N301系第25世代03批次,共1 600羽个体,公母各半,全部由30只公鸡和360只母鸡产生。结合系谱信息和性状记录的完整性,选取15羽亲本公鸡及435羽子代公鸡送往上海伯豪生物技术有限公司(Shanghai Biotechnology Corporation)进行基因分型,基因分型采用鸡600K SNP芯片(Affymetrix Axion HD genotyping array),最终共检测到559 898个SNPs位点。同时测定435羽子代公鸡45、56、70、84、91日龄体重。
1.2 低密度芯片数据模拟为保证SNP芯片的质量及基因型填充,本研究使用Plink v1.90[20]去除未定位到参考基因组染色体上的SNP位点后剩余552 335个SNPs,使用Beagle4.0(r1399)[21]对缺失位点进行填充;使用Plink对填充后的数据进行质量控制,剔除小等位基因频率小于0.005的SNP位点,剩余464 119个SNPs。60K芯片数据生成采用随机提取和相等排序间隔提取两种方式,共进行5次重复。随机提取方式是使用R v4.0.1[22]软件设立3组随机数种子,从464 119个SNPs中随机选取46 412个SNPs生成,共3次重复。相等排序间隔提取方式是从根据染色体与物理位置排序后的600K芯片中每隔10个SNPs取1个SNP,从第2、7个SNP开始,共2次重复。
1.3 关键群体筛选方法为探究不同参考群筛选方法对基因型填充准确性的影响,本研究主要涉及表 1中的5种参考群筛选方法。
|
|
表 1 不同参考群筛选方法 Table 1 The different methods for choosing reference population |
为探究不同版本Beagle软件及系谱使用对基因型填充准确性的影响,本研究根据MCA、RELA、KIN、RAN方法筛选前50个关键群体作为参考群,分别使用Beagle4.0+系谱信息、Beagle4.0、Beagle5.1(18May20.d20)[21, 23]将60K SNP芯片数据填充至600K SNP芯片数据,比较不同版本Beagle软件及系谱使用基因型填充等位基因一致性比率。
为探究不同参考群筛选方法对基因型填充准确性的影响,本研究分别根据RELA、MCA、KIN、RAN方法筛选的前15个关键个体和15个共同祖先作为参考群,使用Beagle4.0与系谱信息对目标群体进行填充,软件使用默认参数,将60K SNP芯片数据填充至600K SNP芯片数据,比较不同筛选关键群体方法进行基因型填充等位基因一致性比率。
为探究参考群规模对基因型填充准确性的影响,本研究分别根据MCA、KIN、RAN方法筛选的前15、25、50个关键个体作为参考群,使用Beagle4.0与系谱信息将60K SNP芯片数据填充至600K SNP芯片数据,比较使用不同规模参考群进行基因型填充等位基因一致性比率。
为探究填充芯片对基因组预测准确性的影响,本研究分别根据MCA、RAN方法筛选的前15、25、50个关键个体作为参考群,使用Beagle4.0与系谱信息将60K SNP芯片数据填充至600K SNP芯片数据,比较填充芯片数据与真实芯片数据基因组预测准确性与基因组预测无偏性。
1.5 基因型填充准确性的评估本研究使用两种验证标准来衡量基因型填充准确性。一种是等位基因一致性比率,另一种是基因组预测准确性和无偏性。
1.5.1 等位基因一致性比率 等位基因一致性比率为正确填充基因型所占百分比,其具体操作为:将60K SNP芯片填充至600K SNP芯片数据后,使用R软件比较每个个体填充基因型与真实基因型间的正确率,并将其平均值作为基因型填充准确性的评判依据。
1.5.2 基因组预测准确性和无偏性 为比较填充芯片数据与真实芯片数据在基因组预测中的应用效果,本研究采用基因组最佳线性无偏预测(genomic best linear unbiased prediction,GBLUP)进行预测,具体公式如下:
| $ {y_i} = u + {a_i} + {e_i}, $ |
其中,yi为校正后的表型值;u为表型均值;ai为加性效应值,ai~N(0,Gσa2);ei为残差,ei~N(0,Iσe2)。
基因组预测准确性与无偏性为基因组估计育种值(GEBV)和校正表型间的相关系数与回归系数。其具体操作为:使用R软件中的lm函数对子代435羽个体45、56、70、84、91日龄体重等原始表型值进行年-季节固定效应校正,将残差作为校正后的表型值用于交叉验证。使用R软件中的rrBLUP包[24]计算群体基因组估计育种值,通过5*10的交叉验证计算填充芯片数据与真实芯片数据基因组预测准确性与无偏性。
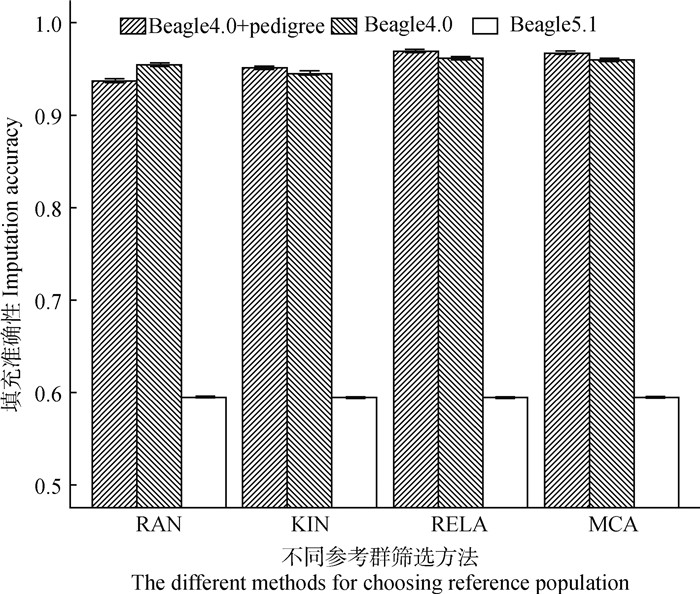
2 结果 2.1 Beagle版本与系谱信息对基因型填充准确性的影响根据MCA、RELA、KIN、RAN方法筛选前50个关键个体作为参考群,使用不同版本Beagle与系谱信息进行基因型填充准确性估计,如图 1所示。从图 1可知,使用Beagle5.1进行基因型填充的等位基因一致性比率最低,为0.594~0.595。使用Beagle4.0进行基因型填充,未使用系谱信息时,MCA、RELA、KIN等方法进行基因型填充的等位基因一致性比率为0.961、0.963、0.947;使用系谱信息时,MCA、RELA、KIN等方法进行基因型填充的等位基因一致性比率为0.970、0.971、0.953。提供系谱信息可以提高MCA、RELA、KIN等方法的等位基因一致性比率,但会导致RAN方法等位基因一致性比率降低,使其从0.956降低至0.939。
|
图 1 不同Beagle版本和系谱信息使用与否的基因型填充准确性 Fig. 1 Genotype imputation accuracy by different Beagle versions and pedigree information |
表 2展示了不同参考群筛选方法筛选的关键个体交叉数量。可以看出,使用CA、MCA、RELA、KIN等方法筛选的参考群至少有10个共同个体,而RAN方法筛选出的15个关键个体与其它方法筛选出的关键个体无共同个体。
|
|
表 2 不同参考群筛选方法所筛选关键个体交叉表 Table 2 Individual crosstab selected by different reference population selection methods |
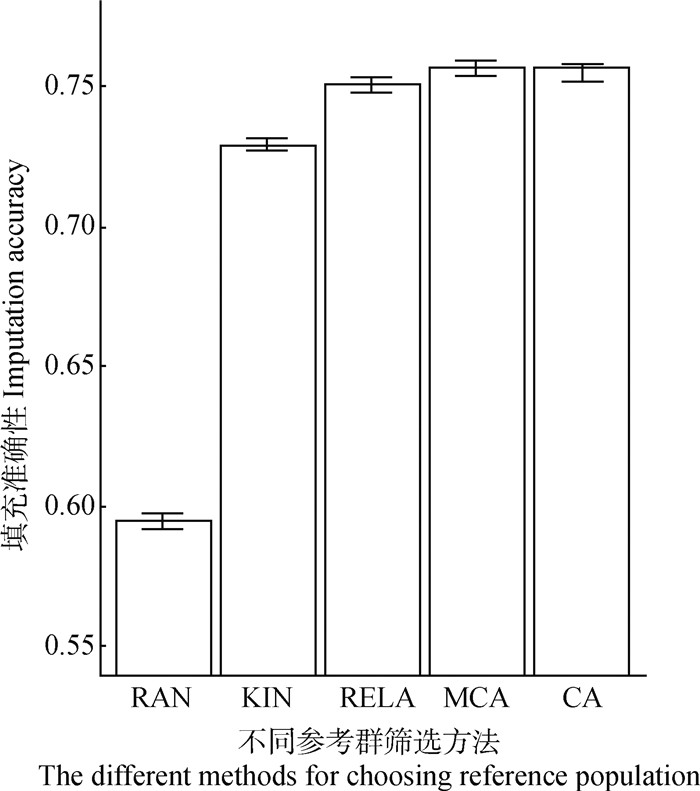
根据RELA、MCA、KIN、RAN方法筛选的前15关键个体和15个共同祖先作为参考群,使用Beagle4.0与系谱信息对目标群体进行基因型填充,不同参考群筛选方法基因型填充准确性如图 2所示。由图 2可知,MCA方法筛选参考群进行基因型填充的等位基因一致性比率最高为0.757,其次是CA、RELA方法为0.755、0.751。此外,MCA、RELA、CA 3种方法的等位基因一致性比率差别较小,RAN方法等位基因一致性比率最低为0.595。
|
图 2 不同参考群筛选方法的基因型填充准确性 Fig. 2 Genotype imputation accuracy of different reference population choosing methods |
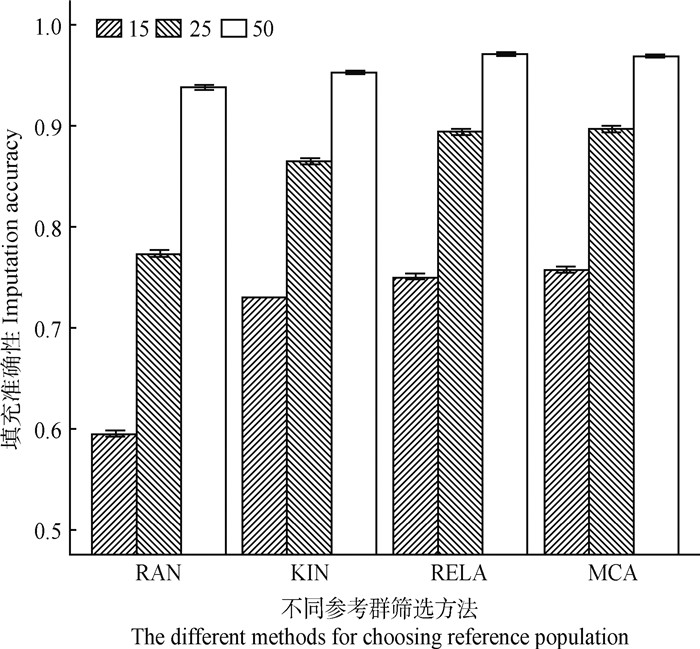
根据MCA、RELA、KIN、RAN方法筛选的前15、25、50个关键个体作为参考群,使用Beagle4.0与系谱信息对目标群体进行填充,不同参考群规模对基因型填充准确性的影响如图 3所示。由图 3可以看出,MCA、RELA、KIN、RAN方法筛选的前15、25、50个关键个体作为参考群,基因型填充的等位基因一致性比率为0.595~0.757、0.773~0.897、0.939~0.971。随着参考群规模增加,等位基因一致性比率也随之增加。但随着参考群规模增大(从15增加至25再增加至50),等位基因一致性比率的提升幅度下降,从0.150降低至0.100。当参考群规模较小时,MCA与RELA方法筛选参考群的等位基因一致性比率保持明显优势,但是随着参考群规模的增加,MCA与RELA方法筛选参考群的等位基因一致性比率优势降低。
|
图 3 不同参考群规模的基因型填充准确性 Fig. 3 Genotype imputation accuracy of different reference population sizes |
根据MCA、RAN方法筛选的前15、25、50个关键个体作为参考群,使用Beagle4.0与系谱信息进行基因填充,不同填充芯片与真实芯片数据基因组预测准确性如表 3、表 4所示。可以看出,真实芯片预测无偏性一般表现为最佳。随着参考群规模增加,填充基因组预测准确性与无偏性同真实芯片基因组预测准确性与无偏性相比差别越小。相对于RAN方法,使用MCA方法筛选关键个体进行基因型填充的基因组预测准确性与真实芯片数据的预测结果更为接近。
|
|
表 3 基因型填充的基因组预测准确性 Table 3 Genome prediction accuracy of genotype imputation |
|
|
表 4 基因型填充的基因组预测无偏性 Table 4 Genome prediction unbiased of genotype imputation |
本研究使用Beagle4.0、Beagle4.0+系谱信息、Beagle5.1等软件进行基因型填充,其中Beagle4.0+ 系谱信息填充效果最佳,其次为Beagle4.0,而Beagle5.1填充效果最差。Pook等[25]对比了Beagle4.0、Beagle5.1未使用系谱信息时双单倍型玉米基因型填充效果,结果发现Beagle5.1填充错误率最低,这与本研究结果存在较大差异,可能是由于其试验群体基因型都为纯合位点且群体较大,而本研究选取的群体是由15羽亲本公鸡与435羽子代群体构成,杂合位点较高且群体较小。Whalen和Hickey[26]使用Beagle4.1、Beagle5.1在18 349个个体的模拟猪家系中使用350、10 000、33 000、46 000个标记进行填充,Beagle4.1填充准确性为0.995、0.944、0.969、0.327,Beagle5.1填充准确性为0.626、0.909、0.939、0.219,当标记数目为46 000时,与本研究结果类似。这可能与Beagle5.1采用综合单倍型有关,在输入目标单倍型时,仅使用参考单倍型的子集[27-28]。本研究仅使用Beagle软件进行测试,根据不同使用需求可以选择不同软件进行基因型填充[29],如FImpute[17]、Impute5[14]等。
本研究结果表明,参考群规模一定时,不同参考群筛选方法基因型填充准确性不同。使用CA、MCA、RELA法筛选参考群进行基因型填充的准确性较好,使用RAN法筛选参考群进行基因型填充的准确性较差。Druet等[19]发现,RELA方法填充效果优于RAN法,特别是在MAF较低的位点,这与本研究结果类似。Yu等[18]发现,MCA方法填充效果优于RELA、KIN、RAN方法。当使用Beagle4.0+系谱信息进行基因型填充时,对筛选构建的参考群填充效果更佳。这是因为通过参考群筛选方法筛选关键个体可最大限度的代表群体的遗传变异,同时使参考群体与低密度基因型目标群体有更强的亲缘关系[15, 18, 30]。所以使用共同祖先与亲本信息进行基因型填充,也可获取高质量的填充基因型数据[31]。本研究仅采用系谱亲缘关系矩阵进行参考群筛选,在拥有稀疏基因型信息的情况下,可以考虑使用基因型亲缘关系矩阵进行关键个体筛选[32]。同时,当仅存在部分个体有低密度基因型的情况下,可以使用一步法建立H矩阵[33-34],进行关键个体筛选。
随着参考群规模增大,基因型填充准确性增高。但随着关键个体参考群的增大(从15增加至25再增加至50),基因型填充准确性的提升幅度下降。Wang等[35]使用3K、50K芯片对2 246头安格斯牛进行基因型填充,参考群体比例分别为总群体的1%、10%、20%、50%,填充准确性分别为64%、71%、75%、75%。Ghoreishifar等[36]使用水牛群体在保持目标群体数目不变的情况下增加参考群规模,填充准确性在参考群从小到中等规模变化时提升较快,从中等到大规模时提升较慢。这与本研究结果相似,随着参考群规模增加,单倍型推断及匹配更加准确,基因型填充准确性也随之增加,这可能是因为较多的参考群个体能够提供更多的单倍型。但是随着参考群规模的进一步扩大提升幅度会逐渐降低[30, 37],因此,在保证基因型填充准确性足够分析的情况下,可以适当控制参考群规模,以降低检测成本。
不同填充芯片与真实芯片数据基因组预测准确性相比,基因型填充一致性比率越大越接近真实芯片预测结果,真实芯片预测无偏性一般表现最佳。然而当参考群规模较小时,其基因型填充一致性比率较低,而预测准确性却表现较佳。这可能是因为使用了系谱信息进行填充,导致填充后的结果与系谱预期更符合,从而导致预测准确性更高。
小等位基因频率[38]、目标群体与参考群体芯片密度[38]、填充软件、参考群组成、参考群体大小等因素都会影响基因型填充准确性,然而在实际生产中对于小等位基因频率难以进行控制,目标群体与参考群体也基本使用定制芯片。本研究通过探究参考群筛选方法及规模对基因型填充准确性的影响,发现使用Beagle4.0+系谱信息有更好的填充效果。使用MCA法筛选参考群进行基因型填充准确性最高,但当系谱关系清晰时使用共同祖先与亲本信息进行基因型填充也可以获取高质量的高密度芯片。参考群规模增加,基因型填充准确性也随之增加,但过多的增加参考群所带来的的收益较低。将公共动植物基因型填充数据库中的个体添加到参考群体中,以此增加参考群数目也可以有效增加基因型填充准确性[39-40]。
4 结论综上所述,可以通过参考群筛选方法构建参考群以及控制参考群规模,以保证基因型填充和基因组预测准确性并节省成本,本研究可为基因型填充在畜禽遗传育种中的应用提供技术参考。
| [1] |
WIGGANS G R, VANRADEN P M, COOPER T A. The genomic evaluation system in the United States: past, present, future[J]. J Dairy Sci, 2011, 94(6): 3202-3211. DOI:10.3168/jds.2010-3866 |
| [2] |
WEISHAAR R, WELLMANN R, CAMARINHA-SILVA A, et al. Selecting the hologenome to breed for an improved feed efficiency in pigs—a novel selection index[J]. J Anim Breed Genet, 2020, 137(1): 14-22. DOI:10.1111/jbg.12447 |
| [3] |
GAO N, TENG J Y, PAN R Y, et al. Accuracy of whole genome prediction with single-step GBLUP in a Chinese yellow-feathered chicken population[J]. Livest Sci, 2019, 230: 103817. DOI:10.1016/j.livsci.2019.103817 |
| [4] |
ZENGER K R, KHATKAR M S, JONES D B, et al. Genomic selection in aquaculture: application, limitations and opportunities with special reference to marine shrimp and pearl oysters[J]. Front Genet, 2019, 9: 693. DOI:10.3389/fgene.2018.00693 |
| [5] |
DAETWYLER H D, VILLANUEVA B, WOOLLIAMS J A. Accuracy of predicting the genetic risk of disease using a genome-wide approach[J]. PLoS One, 2008, 3(10): e3395. DOI:10.1371/journal.pone.0003395 |
| [6] |
HAYES B J, BOWMAN P J, CHAMBERLAIN A J, et al. Invited review: genomic selection in dairy cattle: progress and challenges[J]. J Dairy Sci, 2009, 92(2): 433-443. DOI:10.3168/jds.2008-1646 |
| [7] |
DAS S, FORER L, SCHÖNHERR S, et al. Next-generation genotype imputation service and methods[J]. Nat Genet, 2016, 48(10): 1284-1287. DOI:10.1038/ng.3656 |
| [8] |
SOLLERO B P, HOWARD J T, SPANGLER M L. The impact of reducing the frequency of animals genotyped at higher density on imputation and prediction accuracies using ssGBLUP1[J]. J Anim Sci, 2019, 97(7): 2780-2792. DOI:10.1093/jas/skz147 |
| [9] |
FERNANDES JÚNIOR G A, CARVALHEIRO R, DE OLIVEIRA H N, et al. Imputation accuracy to whole-genome sequence in Nellore cattle[J]. Genet Sel Evol, 2021, 53(1): 27. DOI:10.1186/s12711-021-00622-5 |
| [10] |
VAN BINSBERGEN R, BINK M C, CALUS M P, et al. Accuracy of imputation to whole-genome sequence data in Holstein Friesian cattle[J]. Genet Sel Evol, 2014, 46(1): 41. DOI:10.1186/1297-9686-46-41 |
| [11] |
VAN DEN BERG S, VANDENPLAS J, VAN EEUWIJK F A, et al. Imputation to whole-genome sequence using multiple pig populations and its use in genome-wide association studies[J]. Genet Sel Evol, 2019, 51(1): 2. DOI:10.1186/s12711-019-0445-y |
| [12] |
YE S, YUAN X, HUANG S, et al. Comparison of genotype imputation strategies using a combined reference panel for chicken population[J]. Animal, 2019, 13(6): 1119-1126. DOI:10.1017/S1751731118002860 |
| [13] |
VANRADEN P M. Symposium review: how to implement genomic selection[J]. J Dairy Sci, 2020, 103(6): 5291-5301. DOI:10.3168/jds.2019-17684 |
| [14] |
RUBINACCI S, DELANEAU O, MARCHINI J. Genotype imputation using the positional burrows wheeler transform[J]. PLoS Genet, 2020, 16(11): e1009049. DOI:10.1371/journal.pgen.1009049 |
| [15] |
ROS-FREIXEDES R, WHALEN A, GORJANC G, et al. Evaluation of sequencing strategies for whole-genome imputation with hybrid peeling[J]. Genet Sel Evol, 2020, 52(1): 18. DOI:10.1186/s12711-020-00537-7 |
| [16] |
YE S P, YUAN X L, LIN X R, et al. Imputation from SNP chip to sequence: a case study in a Chinese indigenous chicken population[J]. J Anim Sci Biotechnol, 2018, 9: 30. DOI:10.1186/s40104-018-0241-5 |
| [17] |
SARGOLZAEI M, CHESNAIS J P, SCHENKEL F S. A new approach for efficient genotype imputation using information from relatives[J]. BMC Genomics, 2014, 15(1): 478. DOI:10.1186/1471-2164-15-478 |
| [18] |
YU X J, WOOLLIAMS J A, MEUWISSEN T H. Prioritizing animals for dense genotyping in order to impute missing genotypes of sparsely genotyped animals[J]. Genet Sel Evol, 2014, 46(1): 46. DOI:10.1186/1297-9686-46-46 |
| [19] |
DRUET T, MACLEOD I M, HAYES B J. Toward genomic prediction from whole-genome sequence data: impact of sequencing design on genotype imputation and accuracy of predictions[J]. Heredity (Edinb), 2014, 112(1): 39-47. DOI:10.1038/hdy.2013.13 |
| [20] |
PURCELL S, NEALE B, TODD-BROWN K, et al. PLINK: a tool set for whole-genome association and population-based linkage analyses[J]. Am J Hum Genet, 2007, 81(3): 559-575. DOI:10.1086/519795 |
| [21] |
BROWNING S R, BROWNING B L. Rapid and accurate haplotype phasing and missing-data inference for whole-genome association studies by use of localized haplotype clustering[J]. Am J Hum Genet, 2007, 81(5): 1084-1097. DOI:10.1086/521987 |
| [22] |
R Core Team. R: a language and environment for statistical computing[J]. Vienna, Austria: R Foundation for Statistical Computing, 2020. |
| [23] |
BROWNING B L, ZHOU Y, BROWNING S R. A one-penny imputed genome from next-generation reference panels[J]. Am J Hum Genet, 2018, 103(3): 338-348. DOI:10.1016/j.ajhg.2018.07.015 |
| [24] |
ENDELMAN J B. Ridge regression and other kernels for genomic selection with R package rrBLUP[J]. Plant Genome, 2011, 4(3): 250-255. DOI:10.3835/plantgenome2011.08.0024 |
| [25] |
POOK T, MAYER M, GEIBEL J, et al. Improving imputation quality in BEAGLE for crop and livestock data[J]. G3 (Bethesda), 2020, 10(1): 177-188. DOI:10.1534/g3.119.400798 |
| [26] |
WHALEN A, HICKEY J M. AlphaImpute2: fast and accurate pedigree and population based imputation for hundreds of thousands of individuals in livestock populations[J/OL]. bioRxiv, 2020, doi: 10.1101/2020.09.16.299677.
|
| [27] |
HOWIE B N, DONNELLY P, MARCHINI J. A flexible and accurate genotype imputation method for the next generation of genome-wide association studies[J]. PLoS Genet, 2009, 5(6). |
| [28] |
LIU E Y, LI M Y, WANG W, et al. MaCH-admix: genotype imputation for admixed populations[J]. Genet Epidemiol, 2013, 37(1): 25-37. DOI:10.1002/gepi.21690 |
| [29] |
WANG X, SU G S, HAO D, et al. Comparisons of improved genomic predictions generated by different imputation methods for genotyping by sequencing data in livestock populations[J]. J Anim Sci Biotechnol, 2020, 11: 3. DOI:10.1186/s40104-019-0407-9 |
| [30] |
PAUSCH H, AIGNER B, EMMERLING R, et al. Imputation of high-density genotypes in the Fleckvieh cattle population[J]. Genet Sel Evol, 2013, 45(1): 3. DOI:10.1186/1297-9686-45-3 |
| [31] |
TSAIRIDOU S, HAMILTON A, ROBLEDO D, et al. Optimizing low-cost genotyping and imputation strategies for genomic selection in atlantic salmon[J]. G3 (Bethesda), 2020, 10(2): 581-590. DOI:10.1534/g3.119.400800 |
| [32] |
BICKHART D M, HUTCHISON J L, NULL D J, et al. Reducing animal sequencing redundancy by preferentially selecting animals with low-frequency haplotypes[J]. J Dairy Sci, 2016, 99(7): 5526-5534. DOI:10.3168/jds.2015-10347 |
| [33] |
LEGARRA A, AGUILAR I, MISZTAL I. A relationship matrix including full pedigree and genomic information[J]. J Dairy Sci, 2009, 92(9): 4656-4663. DOI:10.3168/jds.2009-2061 |
| [34] |
MEUWISSEN T H E, LUAN T, WOOLLIAMS J A. The unified approach to the use of genomic and pedigree information in genomic evaluations revisited[J]. J Anim Breed Genet, 2011, 128(6): 429-439. DOI:10.1111/j.1439-0388.2011.00966.x |
| [35] |
WANG H Y, WOODWARD B, BAUCK S, et al. Imputation of missing SNP genotypes using low density panels[J]. Livest Sci, 2012, 146(1): 80-83. DOI:10.1016/j.livsci.2011.12.010 |
| [36] |
GHOREISHIFAR S M, MORADI-SHAHRBABAK H, MORADI-SHAHRBABAK M, et al. Accuracy of imputation of single-nucleotide polymorphism marker genotypes for water buffaloes (Bubalus bubalis) using different reference population sizes and imputation tools[J]. Livest Sci, 2018, 216: 174-182. DOI:10.1016/j.livsci.2018.08.009 |
| [37] |
BUTTY A M, SARGOLZAEI M, MIGLIOR F, et al. Optimizing selection of the reference population for genotype imputation from array to sequence variants[J]. Front Genet, 2019, 10: 510. DOI:10.3389/fgene.2019.00510 |
| [38] |
邓天宇, 杜立新, 王立贤, 等. 基因型填充策略研究[J]. 畜牧兽医学报, 2020, 51(9): 2068-2078. DENG T Y, DU L X, WANG L X, et al. Study on the strategies of genotype imputation[J]. Acta Veterinaria et Zootechnica Sinica, 2020, 51(9): 2068-2078. (in Chinese) |
| [39] |
YANG W Q, YANG Y B, ZHAO C C, et al. Animal-ImputeDB: a comprehensive database with multiple animal reference panels for genotype imputation[J]. Nucleic Acids Res, 2020, 48(D1): D659-D667. DOI:10.1093/nar/gkz854 |
| [40] |
GAO Y J, YANG Z Q, YANG W Q, et al. Plant-ImputeDB: an integrated multiple plant reference panel database for genotype imputation[J]. Nucleic Acids Res, 2021, 49(D1): D1480-D1488. DOI:10.1093/nar/gkaa953 |
(编辑 郭云雁)



