2. 华南农业大学生命科学学院 广东省农业生物蛋白质功能与调控重点实验室, 广州 510642;
3. 华南农业大学兽医学院, 广州 510642
2. Guangdong Provincial Key Laboratory of Protein Function and Regulation in Agricultural Organisms, College of Life Sciences, South China Agricultural University, Guangzhou 510642, China;
3. College of Veterinary Medicine, South China Agricultural University, Guangzhou 510642, China
富含半胱氨酸的肠蛋白(cysteine-rich intestinal protein,CRIP)是LIM(Lin-1、Isl1、Mec3)蛋白家族的一个亚家族,包含一个保守的LIM结构域以及一个富含半胱氨酸和组氨酸的基序[1]。迄今为止,CRIP2的研究主要集中在肿瘤和细胞凋亡方面[2-4]。CRIP2是CRIP家族的成员,与其他成员序列具有一定的相似性,CRIP2基因已在许多哺乳动物中被克隆,但在猪中尚缺少相关研究。与其他成员相比,它缺乏DNA结合能力,但在蛋白质的每个末端包含两个LIM结构域[5]。在组织表达分布上也与其他成员相似,广泛分布于心、卵巢、脑、骨骼肌、脾、前列腺、小肠、胰腺、睾丸和神经元神经节[5]。研究指出,CRIP2与肠道免疫系统重要调节剂NF-κB紧密相关,并在肿瘤发展中起着抑制剂的作用[6-7],但CRIP2在免疫系统中的作用到目前为止尚无相关报道。
由于猪胃肠炎临床表现和致病易感性与人相似,猪模型被广泛应用于人类胃肠道的临床相关模型[8]。肠道炎症的暴发与病原菌刺激产生的有害信号通路和胃肠上皮的破坏有关[9],肠道感染主要病原菌包括铜绿假单胞菌[10]、肠致病大肠杆菌[11]、肠毒性大肠杆菌[12]、金黄色葡萄球菌[13]、肠沙门菌[14]、衣原体[15]和粪肠球菌[16]。
尽管许多哺乳动物的CRIP2序列已被克隆,其在猪中的基因序列至今仍未被报道,poCRIP2在免疫系统特别是肠道黏膜免疫方面的功能尚不明确。本研究利用广泛用于肠道感染机制研究的IPEC-J2细胞系探究poCRIP2在猪胃肠道炎症中的作用[17],识别poCRIP2基因的全长序列,并在此基础上研究poCRIP2的有关特征,同时,对其组织表达模式进行分析,并建立有效的poCRIP2三维结构模型来解释其功能,以了解poCRIP2在细菌感染过程中的免疫作用及其与NF-κB通路的关系。
1 材料与方法 1.1 主要试剂RNAiso plus购于TaKaRa公司;ReverTra AceⓇ qPCR RT Kit(Toyobo,Japan);pMDTM18-T载体克隆试剂盒(TaKaRa, 大连, 中国);IPEC-J2,大肠杆菌MG1655、产肠毒素大肠杆菌196(ETEC196)、金黄色葡萄球菌29213(S. aureus 29213)和铜绿假单胞菌PAO1(P. aeruginosa PAO1),本实验室鉴定保存。
1.2 样品采集对3头4日龄长白猪仔猪进行屠宰取样,将心、脑、肝、脾、肺、肾、气管、小肠、胃、背最长肌、脑、附睾共12个组织样品快速液氮冷冻提取RNA。按照华南农业大学动物伦理委员会(IAEC)批准的方案(SCAU-AEC-2010-0416),在受控环境中饲养仔猪。所有研究均经广东省科技厅批准。
1.3 总RNA提取与cDNA合成根据RNAiso plus说明书,用TRIzol法提取总RNA(Li and Trick,2005),测量OD260 nm和OD280 nm吸光度,确定提取RNA的浓度和纯度。使用ReverTra AceⓇ qPCR RT Kit(Toyobo,Japan)制备猪组织的cDNA第一链:将总RNA在65 ℃下预热5 min,然后在冰上加入2 μL 5×RT缓冲液、0.5 μL RT酶混合液、0.5 μL Primer混合液和无核酸酶水,总体积为10 μL。反应体系在37 ℃下孵育30 min,在98 ℃下加热5 min,-80 ℃保存备用。
1.4 poCRIP2基因的RT-PCR扩增本研究采用电子克隆方法获得poCRIP2的全长序列。首先,在GenBank中搜索猪表达序列标签(EST)序列数据库,得到3个与人或家鼠CRIP2基因高度同源的EST片段(GenBank注册号:DT324610.1、BP144826.1和CN156214.1)。然后在CExpress程序中,将所有EST序列拼接,得到推定的全长poCRIP2。然后利用Primer Premier6软件设计了一对引物,用于克隆poCRIP2的全长序列。利用引物进行RT-PCR克隆,从猪心cDNA文库中分离出poCRIP2全长序列。反转录-聚合酶链反应(RT-PCR)方法采用KOD FX DNA聚合酶(TOYOBO, Osaka, Japan)。反应体系包含:2.5 μL cDNA、10 μL 2x PCR缓冲液的KOD FX、0.4 mmol·L-1 dNTP、0.3 μmol·L-1的各引物和0.5 U KOD FX。PCR程序如下:94 ℃下初始变性2 min,随后98 ℃下10 s、65 ℃下30 s、68 ℃下1 min 20 s进行40个循环,最后68 ℃下延伸10 min。随后,用1%琼脂糖凝胶电泳检测。使用Taq DNA聚合酶为PCR产物添加dATP尾,将DNA片段克隆到pMD-18T载体上,由Invitrogen公司测序。
1.5 poCRIP2组织表达分析本研究检测CRIP2基因在猪体内的组织分布,利用Primer Premier5.0和NCBI Primer BLAST程序设计了特异性引物对,参考现有计算方法[18],采用管家基因β-肌动蛋白(β-actin)对结果进行归一化处理,其引物见表 1。用商业试剂盒(Toyobo,Japan)进行基因表达分析,其程序为为:95 ℃初始变性3 min,95 ℃变性、退火、延伸45个循环15 s,60 ℃延伸1 min,在Bio-Rad CFX96系统(Bio-Rad,Hercules,CA,USA)中进行反应,poCRIP2在不同组织中的表示为2-ΔΔCt。将poCRIP2的Ct值归一化为β-肌动蛋白(β-actin),得到ΔCt值,并将肝的ΔCt设为对照组。所有试验均设置了3个重复。
|
|
表 1 本研究中使用到的引物 Table 1 The primer sequences used in this study |
采用表 2所列的各种生物信息学方法对poCRIP2的特性进行分析。用于多序列比对和系统发育树构建的poCRIP2蛋白如表 3所示。
|
|
表 2 生物信息学分析软件 Table 2 Bioinformatics analysis software |
|
|
表 3 本研究使用的其他物种的CRIP2氨基酸序列 Table 3 CRIP2 amino acid of other species used in this study |
利用ROBETTA服务器(Protein Homology/Analogy Recognition Engine)(http://www.robetta.org),以与推导poCRIP2序列高度同源的小鼠LIM-homeodomain protein islet 1(Isl1)(PDB:4 JCJ)为模板,进行poCRIP2的三维结构模拟。用PROCHECK(http://services.mbi.ucla.edu/PROCHECK/)和ProSA (https://prosa.services.came.sbg.ac.at/prosa.php)对模型进行了分析。应用Procheck对蛋白质折叠进行立体化学和拓扑分析,并应用ProSA对蛋白质折叠进行评价。并使用PyMOL程序(www.pymol.org)对建模结果进行评估,该程序也同时用于分析poCRIP2的保守结构域。
1.8 微生物与IPEC-J2培养用多种细菌(表 4)体外感染IPEC-J2,包括大肠杆菌MG1655、产肠毒素大肠杆菌196(ETEC196)、金黄色葡萄球菌29213(S. aureus 29213)和铜绿假单胞菌PAO1(P. aeruginosa PAO1),以上主要生长在Luria-Bertani(LB)培养基中,而粪肠球菌FA2-2(E. faecalis FA2-2)主要生长在AC培养基中。感染前,所有细菌使用新鲜磷酸盐缓冲溶液(PBS,pH7.4)冲洗3次以去除培养基,并在DMEM/F12中调节密度至1×108CFU·mL-1。
|
|
表 4 本试验使用到的菌株 Table 4 Bacteria strains used in this experiment |
本实验室培养的IPEC-J2细胞为猪肠道上皮细胞系,对其进行感染实验,细胞生长在Dulbecco’s MEM营养液混合液F12(DMEM-F12)(1∶1)和10%(v/v) FCS中,37 ℃,5% CO2条件下进行培养。在感染前,先用0.25%胰蛋白酶-EDTA的胰蛋白酶消化重悬所有细胞,然后接种到24孔板,每孔2×105个细胞,无抗生素。
1.9 IPEC-J2感染在感染试验中,使用各类菌株或新鲜PBS(对照)按固定的感染倍数(MOI,multiplicity of infection,细菌与细胞的比例)处理24孔板培养的IPEC-J2。所有用于感染试验的菌株在攻毒时均设定为20 MOI。24孔板中的IPEC-J2均用相同体积的细菌或PBS处理6 h。最后,按前述方法提取各组总RNA。用公式2-ΔΔCt测定目的基因(poCRIP2、NF-κB、IL- 6)和β-肌动蛋白(β-actin)的表达,ΔCt代表试验组与PBS组的差异。每个试验均包含3个重复组。
1.10 统计分析RT-qPCR的所有数据均展示为3个独立试验的平均值,并使用Origin 8软件进行数据统计分析。通过单向方差分析(ANOVA)或t检验确定试验组与对照组之间的差异。当P < 0.05时,差异被认为具有统计学显著性;此外,P < 0.01则被认为具有高度显著性。
2 结果 2.1 poCRIP2基因的克隆与特征分析为了获得poCRIP2基因全长cDNA序列,利用人(NM_001270837.1)、家鼠(NM_024223.2)和挪威大鼠(NM_022501.1)等其他物种的cDNA序列对NCBI网站EST数据库进行了匹配。获得了3个猪EST序列(DT324610.1、BP144826.1和CN156214.1)。根据EST序列,克隆了poCRIP2的全长DNA片段,并进行了1%琼脂糖凝胶电泳检测(图 1)。利用KOD FX DNA聚合酶和商用试剂盒将该DNA片段与pMD-18T载体连接。然后对重组质粒pMD-18T-CRIP2进行测序,得到了poCRIP2的cDNA序列,长度为1 118 bp,已提交至GenBank,注册号为KU933587。通过ORF Finder软件分析,poCRIP2的开放阅读框长627 bp,A+T=36.20%,C+G=63.80%。翻译起始位点(ATG)位于36 bp处,TAG停止密码子位于639 bp处。
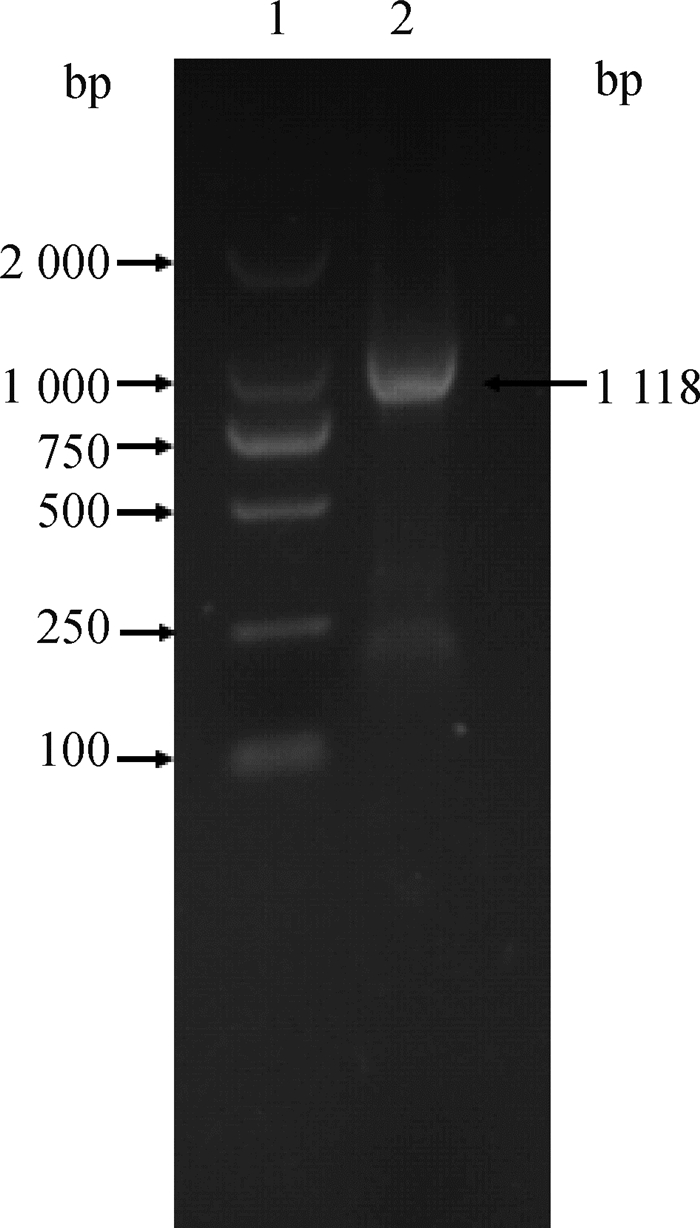
|
1.DNA相对分子质量标准DL2000;2.poCRIP2基因扩增产物 1.DNA relative molecular quality standard DL2000; 2.poCRIP2 gene amplification product 图 1 poCRIP2基因扩增结果 Fig. 1 The results of poCRIP2 gene amplification |
通过Bio Edit软件预测poCRIP2的蛋白序列,可见与其他物种的CRIP2蛋白具有相同的长度,均为208个氨基酸。预测结果显示(图 2),poCRIP2由31个强碱性(+)氨基酸、20个强酸性(-)氨基酸、47个疏水氨基酸和53个极性氨基酸组成。利用ProtParam服务器对poCRIP2的等电点和分子量进行了计算,计算结果分别为9.02和22 629.88 u。由Lasergene 9.0软件分析得,在pH为7.0时,poCRIP2的电荷量为11.986。ProtParam software服务器预测结果显示,以水为参比,poCRIP2在280 nm处的消光系数约为25 285 M-1·cm-1。在消除所有Cys残基后,消光系数约减少到24 105 M-1·cm-1。根据Prot Param服务器提供的结果,可以预测poCRIP2为稳定蛋白,脂肪族氨基酸指数为47.36,亲水性均值(GRAVY)为-0.670,表明POCRIP2可能是一种亲水蛋白。
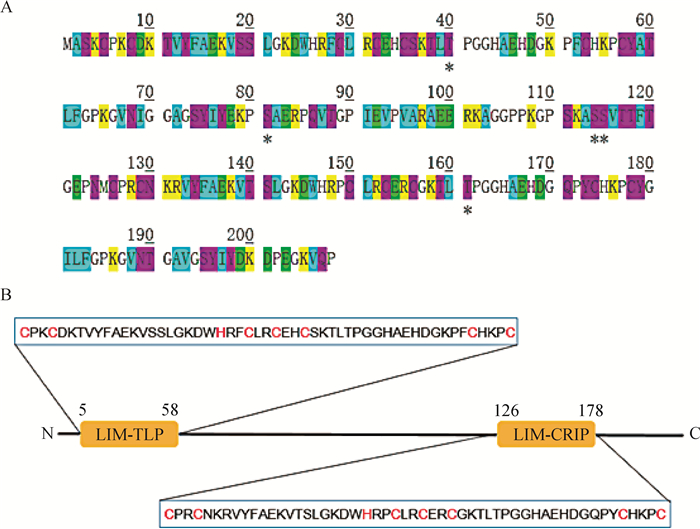
|
A. poCRIP2肽链上的氨基酸组成和磷酸化位点。黄色字母代表强碱性(+)氨基酸,绿色字母代表强酸性(-)氨基酸,蓝色字母代表疏水性氨基酸,粉色字母代表极性氨基酸,带星号的字母代表可能的磷酸化位点。B. poCRIP2中包含的保守域。两个保守域,LIM-TLP和LIM-CRP,在序列中用黄色标出。而保守域中的Zn结合位点也在序列中以红色字母标出 A. Amino acid composition and phosphorylation sites on the peptide chain of poCRIP2. The yellow letters represent strongly basic (+) amino acids, the green letters represent strongly acidic (-) amino acids, the blue letters represent hydrophobic amino acids, the pink letters represent polar amino acids, and the letters with asterisks represent possible phosphorylation sites. B. Conserved domain contained in poCRIP2. Two conserved domains, LIM-TLP and LIM-CRP, are marked in yellow in the sequence. The Zn binding site in the conserved domain is also marked with red letters in the sequence 图 2 poCRIP2的序列特征 Fig. 2 Sequence characteristics of poCRIP2 |
利用poCRIP2的氨基酸序列与NCBI蛋白数据进行匹配,并在其他物种中找到几个类似的序列。通过ClustalW软件分析和DNAMAN7[21]软件进行可视化,可见poCRIP2与人、小鼠、大鼠、牛、斑马鱼等其他物种的氨基酸序列基本一致。由图 3A可知,poCRIP2与以下物种的特征相似性较高:斑马鱼73.79%,大鼠98.08%,人94.23%,小鼠93.75%。由于poCRIP2与其它物种之间的氨基酸序列差异有限,poCRIP2在猪中也具有类似的生理生化功能。

|
A. 黑色字母代表100%的相似度,粉色字母代表 75%以上的相似度,蓝色字母代表 50%以上的相似度,黄色字母代表 33%以上的相似度;B. 系统发育树 A. Black letters represent 100% similarity, pink letters represent 75% or more similarity, blue letters represent 50% or more similarity, yellow letters represent 33% or more similarity; B. Phylogenetic tree 图 3 poCRIP2与其他物种氨基酸序列的比对和系统发育比较 Fig. 3 Alignment and phylogenetic comparison of the amino acid sequence of poCRIP2 with other species |
为了弄清poCRIP2蛋白与其他物种CRIP2蛋白之间的进化关系,利用MEGA 7.0软件,基于NJ法,以1 000个引导重复的方式建立系统发育树。和斑马鱼相比,猪、人和小鼠隶属于同一祖先(图 3B)。而猪和牛的CRIP2具有高度的相似性,并从一个分支上分离出来。几乎在同一阶段,人和类人猿也发生了分支并且也具有高度相似性。因此,本研究建立的进化树可以代表真实的生物进化。
2.3 poCRIP2的结构与功能注释分别用TMHMM[22]和Signal4.0服务器[23]对poCRIP2的跨膜域和信号肽进行预测。结果表明,poCRIP2可能不含跨膜区和信号肽,提示poCRIP2可能是一种胞内蛋白。iLoc-Animal服务器[24]的亚细胞定位预测结果表明,poCRIP2可能位于胞浆。因此,poCRIP2可能主要控制细胞浆内的信号通路,这与Wei等[25]报道的小鼠CRIP2的结果相似。
利用NCBI网站上的保守域服务器对poCRIP2的保守域进行扫描,结果如图 2B所示。poCRIP2有两个LIM锌结合域,即LIM-TLP域和LIM-CRP域,这两个域与锌原子配位结合。这两个结构域具有高度相似的结构,因为它们包含保守的锌结合残基C-X2-C-X17-H-X2-C-X2-C-X2-C-X17-C-X3-C,该结构域同样存在于如牛、人、大鼠、小鼠、斑马鱼等物种的CRIP2蛋白中(如图 3A)。LIM结构域在器官或肿瘤发育中被广泛研究的同时,其在免疫调节中的作用也逐渐受到关注。
2.4 三维结构建模将poCRIP2的氨基酸序列上传到ROBETTA[26]服务器,预测其三维结构模型。采用多个度量对模型进行分析(图 4A、B),验证poCRIP2模型的可靠性。Ramachandran图(图 5A)由PROCHECK服务器绘制。结果表明, 100 %的残基位于允许的区域内,而在较大的允许区域内仅包含0.4 %的残基。随后通过ProSA-web检查了模型结果(图 5B),在Z-score图中显示了一个完美有序的结构,Z-score为-4.37,在常见类似大小蛋白的有效范围内。此外,还在ProSA-web中绘制了残基分数图,结果表明(图 5B),模型中的残基对于poCRIP2的天然结构可能在很大程度上是阴性的,不影响poCRIP2的活性分析。因此,本研究预测的poCRIP2三维结构是可靠的。

|
在模型中,锌离子用白色小球表示,锌离子的结合位点在模型中也用黄色或绿色标识。A. poCRIP2的骨架结构;B. poCRIP2的表面结构展示,蓝色区域代表LIM-TLP域,红色区域代表LIM-CRP域;C. poCRIP2蛋白中LIM-TLP保守域的三维结构模型;D. poCRIP2蛋白中LIM-CRP保守域的三维结构模型 In the model, zinc ions are represented by white balls, and the binding sites of zinc ions are also marked in yellow or green in the model. A. The skeleton structure of poCRIP2; B. The surface structure of poCRIP2, the blue area represents LIM-TLP domain, the red area represents the LIM-CRP domain; C. The 3D structure model of the LIM-TLP conserved domain in the poCRIP2 protein; D. The 3D structure model of the LIM-CRP conserved domain in the poCRIP2 protein 图 4 poCRIP2的三维结构模型 Fig. 4 The 3D structure model of poCRIP2 |
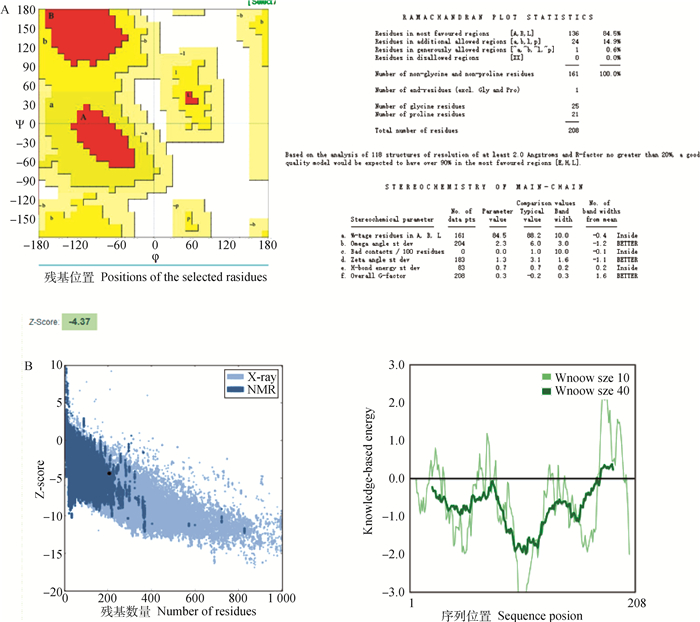
|
A. Ramachandran图由PROCHECK软件绘制。在左侧,它显示了该图的概要。在预测模型中,几乎所有残基都在允许的区域内,并且该模型只有0.14%的残基具有一般评分。B. ProSA-Web模型结果评估。在左图中,通过ProSA-Web进行poCRIP2的Z评分,结果表明预测的模型可以识别为天然蛋白质结构。右图残基得分图也在ProSA-Web中完成,结果显示,在模型中可以找到合适的结构 A. Ramachandran graph is drawn by PROCHECK software. On the left, it shows the outline of the graph. In the prediction model, almost all residues are in the allowable region, and only 0.14% of the residues in the model have a general score. B. Evaluation of the results of the ProSA-Web model. In the left picture, the Z score of poCRIP2 was performed through ProSA-Web, and the results indicate that the predicted model can be recognized as a natural protein structure. The residue score map on the right is also completed in ProSA-Web, the results show that a suitable structure can be found in the model 图 5 poCRIP2三维结构模型的评估结果 Fig. 5 Evaluation results of poCRIP2 3D structure model |
poCRIP2的二级结构(图 6)由SOPMA服务器完成[27],结果显示,poCRIP2包含5.77 %的α螺旋,主要位于N末端,有23.08%的延伸链(β折叠),几乎遍布所有骨架。同时还分布有12.02 % β转角和59.13 %的无规卷曲,结果与图 4A和4B所示的三维结构相似,红色表示的β折叠和无规卷曲是poCRIP2的主要成分,α螺旋主要存在于N末端。利用保守结构域的三维结构模型,不难发现β折叠是蛋白结构的重要部分,分布在该结构域两侧的α螺旋也可能在蛋白功能中发挥重要作用。在图 4C和4D中显示了LIM-TLP和LIM-CRP中的锌离子结合位点,每4个锌离子结合位点可以捕获1个锌离子,两种结构域分别可以得到两个锌离子。尽管结合位点主要位于无规卷曲中,但也需要由β折叠和α螺旋构建的构象。因此,poCRIP2中包含的功能性结构域大部分通过β折叠和α-螺旋结构实现。

|
图 6 使用SOPM服务器自优化预测方法预测的poCRIP2的二级结构 Fig. 6 The secondary structure of poCRIP2 predicted by the SOPM server self-optimizing prediction method |
以往对人或小鼠的研究表明,CRIP2在附睾、卵巢、脑、脾、小肠、心、睾丸等组织中表达较高。本研究以猪为研究对象,采用RT-qPCR法分析了猪CRIP2的组织分布,使用管家基因β-actin对RNA样品进行归一分析,以肝的Ct为对照,结果如图 7,可见poCRIP2在所有组织样本中均有表达,其中在附睾、肝、肾、皮肤、心和气管中有较高的表达,其在气管中的表达水平是肝、肾、脑和附睾的10倍。但在小肠、胃、肺、肌肉和脾中表达较低。这说明,除脾外,poCRIP2的组织分布与人相似。
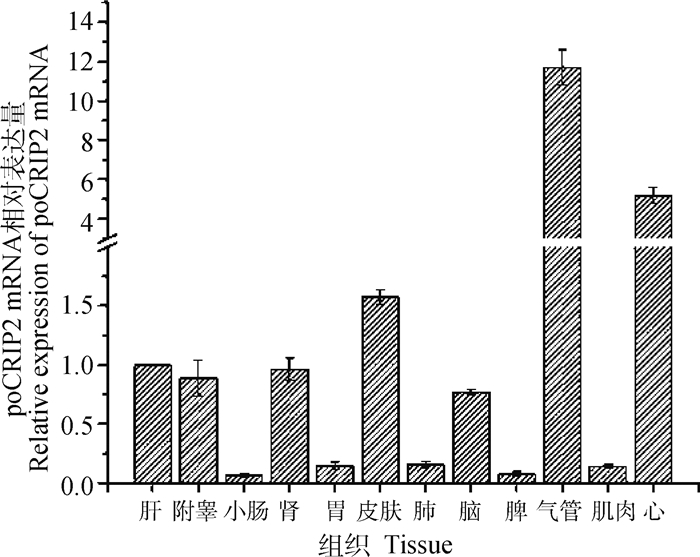
|
图 7 poCRIP2基因的组织表达 Fig. 7 Tissue expression of poCRIP2 gene |
本研究以IPEC-J2作为肠道免疫模型,研究poCRIP2与细菌感染的关系(图 8)。由图 7可知,poCRIP2在肠道中表达水平较低。用革兰阴性菌(MG1655,ETEC 196和PAO1)处理时,poCRIP2表达水平是PBS对照组的1~2倍,差异显著(P < 0.05,图 8A),结果无法揭示共生细菌和致病细菌之间的差异。当用革兰阳性细菌处理细胞时,与金黄色葡萄球菌29213和粪肠球菌FA 2-2共孵育的组中的表达水平与PBS对照组相比没有显著差异,这说明在革兰阳性细菌组中,共生细菌或致病细菌之间也没有差异,但表明poCRIP2可能参与了肠道中的革兰阴性细菌感染。

|
*、**表示组间的显著性差异,* P < 0.05,** P < 0.01;A. 在攻毒试验中poCRIP2的表达水平;B. 在攻毒试验中NF-κB和IL-6的表达水平 *, **indicate significant difference between groups, * P < 0.05, ** P < 0.01; A. The expression level of poCRIP2 in the challenge experiment; B. The expression level of NF-κB and IL-6 in the challenge experiment 图 8 IPEC-J2细胞株中在各类菌株攻毒6 h后靶基因的表达水平 Fig. 8 The expression levels of target genes in the IPEC-J2 cell line after 6 hours of challenge with various strains |
由于NF-κB/p65亚基主要参与肠道炎症,故本研究也对NF-κB的表达水平进行了研究。如图 8B,在革兰阴性菌(MG1655、ETEC196和PAO1)刺激的细胞中NF-κB的表达水平比对照组高约3~4倍,差异显著(P < 0.01)。使用革兰阳性细菌粪肠杆菌FA2-2处理后,细胞NF-κB的表达水平也显著升高。IL-6表达水平检测结果表明,无论革兰阴性菌还是革兰阳性菌,用致病菌(ETEC、FA2-2和PAO1)处理的细胞中IL-6的表达水平全部一定程度地升高,进一步验证了NF-κB通路在细菌感染过程中被激活。NF-κB结果表明,在革兰阴性菌组中,poCRIP2表达较高,同时存在NF-κB的激活;然而对于革兰阳性菌组,NF-κB激活的同时poCRIP2并无显著变化。推测,poCRIP2可能与NF-κB通路无关,在肠道感染中起着新的作用。
3 讨论肠道环境中存在着数千种细菌、共生细菌或致病菌,肠道免疫功能复杂。胃肠炎是人和猪的一种常见疾病。它多数由致病菌刺激的损伤性信号通路引起。由于细菌种类的多样性,很难找到解释肠道感染或肠道炎症的共同机制。由于猪模型最近被广泛用作人类临床相关模型,研究猪肠道的感染机制对人类具有重大参考价值。
富含半胱氨酸蛋白2(CRIP2)是LIM结构域蛋白家族的CRIP型亚家族。几乎所有的CRIPs蛋白在生物体的生理功能和疾病发展中都具有多种功能。但迄今为止,CRIPs家族在猪、马、羊等家畜中仍未被克隆。养猪业是重要的经济产业,同时,猪也是重要的人类临床模型。因此,本研究以获得poCRIP2的编码序列为目的,采用电子克隆方法,得到poCRIP2的全长序列。通过结构分析,poCRIP2编码了208个氨基酸,同时分析了等电点、分子量、氨基酸组成、保守区和信号肽等理化性质。poCRIP2的理化性质与人类相似。在此基础上,本试验还研究了poCRIP2的组织分布,其在皮肤、心、肝和气管中高度表达,而在其他组织,如肌肉、肺、小肠和脾中表达程度较低,结果与人和小鼠的趋势相似,因此,猪的poCRIP2可以很好地代表人CRIP2的作用。
本研究首次对poCRIP2的三维结构进行了分析。利用ROBETTA服务器,构建了poCRIP2的三维结构,并通过PROCHECH和Pro SA验证了该结构的有效性。在SOPMA服务器上,poCRIP2的三维结构也与poCRIP2的第二级结构接近。通过Phymol程序处理,分析了poCRIP2的保守区:LIM-TLP和LIM-CRP。LIM-TLP结构域和LIM-CRP结构域具有相似的氨基酸序列。令人惊讶的是,LIM-TLP结构域和LIM-CRP结构域的三维结构也非常相似,并且主要位于α-螺旋周围。而保守域中的重要位点,即锌离子结合位点,也建立在β折叠和α螺旋上。因此,poCRIP2中的功能结构主要由β折叠和α-螺旋结构实现。
本研究是首份关于CRIP2免疫功能的报导,到目前为止,关于CRIP2的研究主要针对肿瘤[28],尚未有关于免疫的报道。已证明CRIP1在免疫中起重要作用,但该报道主要在鱼类而不是哺乳动物中进行。同时,CIRP2与NF-κB密切相关[3]。因此,本研究在克隆了poCRIP2的序列后,对poCRIP2在肠道免疫中的作用进行了深入研究,使用了肠道免疫的通用模型——IPEC-J2细胞系作为研究材料[29]。本研究发现,首先,当IPEC-J2细胞被正常细菌(大肠杆菌MG1655)感染时,poCRIP2高度表达,但其表达水平与其他细菌组没有显著差异,例如金黄色葡萄球菌ATCC29213。其次,poCRIP2在IPEC-J2中能被革兰阴性菌显著上调,但不能被革兰阳性菌上调,这是迄今为止的一个重要发现,由于poCRIP2在肠道中的表达水平很低,但能被革兰阴性菌显著上调,且与鱼类CRIP1相似,也能被革兰阳性菌调控,推测poCRIP2是以一种新的机制参与肠道免疫。
由于NF-κB/p65亚基主要参与炎症反应,通常由LPS或LTA诱导,因此,肠感染期间NF-κB途径的激活也是可预期的[30]。人CRIP2是NF-κB通路的阻遏剂[5],本研究发现,poCRIP2高度表达时,NF-κB通路也是在同一组中被激活(被革兰阴性细菌攻击),这表明,poCRIP2通过非NF-κB依赖的其他途径参与了肠道的免疫。
综上所述,本研究成功获得了猪CRIP2基因的全长序列,poCRIP2的组织表达模式表明,poCRIP2与人CRIP2相似。同时,本研究建立了可靠的三维结构模型,并且基于该模型,推测poCRIP2中包含的功能主要由β折叠和α-螺旋结构实现。本研究首次报道了CRIP2在免疫方面的功能,更为重要的是,研究发现革兰阴性菌感染肠道后,poCRIP2能被显著上调。因此,poCRIP2应不依赖于NF-κB通路,在肠道免疫中起其他作用。到目前为止,关于CRIP在肠道免疫中的作用的信息还很少,因此,本研究不仅将揭开poCRIP2免疫调节功能的面纱,而且还将为增强人类肠道免疫健康提供新的切入点。
4 结论本研究识别了从猪心获得的poCRIP2 cDNA基因的全长序列为1 118 bp,poCRIP2蛋白与人和小鼠的同源性分别为94.23%和93.75%,它还含有两个保守区(LIM-TLP和LIM-CRP)。组织表达模式研究表明,poCRIP2与人CRIP2相似,在所有组织中均有表达,但在小肠、肺、肌肉等组织中表达较低;根据建立的三维结构模型,推测poCRIP2中包含的功能主要由β折叠和α-螺旋结构实现,且革兰阴性菌感染肠道后poCRIP2能被显著上调,poCRIP2应不依赖于NF-κB通路,在肠道免疫中起其他作用。
| [1] |
CHEN L, LONG X L, DUAN S Y, et al. CSRP2 suppresses colorectal cancer progression via p130Cas/Rac1 axis-meditated ERK, PAK, and HIPPO signaling pathways[J]. Theranostics, 2020, 10(24): 11063-11079. DOI:10.7150/thno.45674 |
| [2] |
SUN X J, ZHANG R L, LLIN X Y, et al. Wnt3a regulates the development of cardiac neural crest cells by modulating expression of cysteine-rich intestinal protein 2 in rhombomere 6[J]. Circ Res, 2008, 102(7): 831-839. DOI:10.1161/CIRCRESAHA.107.166488 |
| [3] |
ZHOU L, WANG Y H, ZHOU M J, et al. HOXA9 inhibits HIF-1α-mediated glycolysis through interacting with CRIP2 to repress cutaneous squamous cell carcinoma development[J]. Nat Commun, 2018, 9(1): 1480. DOI:10.1038/s41467-018-03914-5 |
| [4] |
NAIN Z, RANA H K, LIÒ P, et al. Pathogenetic profiling of COVID-19 and SARS-like viruses[J]. Brief Bioinform, 2021, 22(2): 1175-1196. DOI:10.1093/bib/bbaa173 |
| [5] |
CHEUNG A K L, KO J M Y, LUNG H L, et al. Cysteine-rich intestinal protein 2 (CRIP2) acts as a repressor of NF-κB-mediated proangiogenic cytokine transcription to suppress tumorigenesis and angiogenesis[J]. Proc Natl Acad Sci U S A, 2011, 108(20): 8390-8395. DOI:10.1073/pnas.1101747108 |
| [6] |
NEURATH M F, BECKER C, BARBULESCU K. Role of NF-κB in immune and inflammatory responses in the gut[J]. GUT, 1998, 43(6): 856-860. DOI:10.1136/gut.43.6.856 |
| [7] |
KONG H J, MOON J H, HAN Y H, et al. PoCRIP1, Paralichthys olivaceus cysteine-rich intestinal protein 1:molecular characterization, expression analysis upon Edwardsiella tarda challenge and a possible role in the immune regulation[J]. Fish Shellfish Immunol, 2011, 30(3): 917-922. DOI:10.1016/j.fsi.2011.01.017 |
| [8] |
ZHANG Q S, WIDMER G, TZIPORI S. A pig model of the human gastrointestinal tract[J]. Gut Microbes, 2013, 4(3): 193-200. DOI:10.4161/gmic.23867 |
| [9] |
NUNES T, BERNARDAZZI C, DE SOUZA H S. Cell death and inflammatory bowel diseases: apoptosis, necrosis, and autophagy in the intestinal epithelium[J]. Biomed Res Int, 2014, 2014: 218493. |
| [10] |
LAUGHLIN R S, MUSCH M W, HOLLBROOK C J, et al. The key role of Pseudomonas aeruginosa PA-I lectin on experimental gut-derived sepsis[J]. Ann Surg, 2000, 232(1): 133-142. DOI:10.1097/00000658-200007000-00019 |
| [11] |
CLEARY J, LAI L C, SHAW R K, et al. Enteropathogenic Escherichia coli (EPEC) adhesion to intestinal epithelial cells: role of bundle-forming pili (BFP), EspA filaments and intimin[J]. Microbiology (Reading), 2004, 150(Pt3): 527-538. |
| [12] |
GHOSAL A, CHATTERJEE N S, CHOU T, et al. Enterotoxigenic Escherichia coli infection and intestinal thiamin uptake: studies with intestinal epithelial Caco-2 monolayers[J]. Am J Physiol Cell Physiol, 2013, 305(11): C1185-C1191. DOI:10.1152/ajpcell.00276.2013 |
| [13] |
LI Y, LIU Y H, LI Z J, et al. Staphylococcus aureus infection of intestinal epithelial cells induces human umbilical cord-derived mesenchymal stem cell migration[J]. Int Immunopharmacol, 2013, 15(1): 176-181. DOI:10.1016/j.intimp.2012.10.016 |
| [14] |
LYCZAK J B. Commensal bacteria increase invasion of intestinal epithelium by Salmonella enterica serovar Typhi[J]. Infect Immun, 2003, 71(11): 6610-6614. DOI:10.1128/IAI.71.11.6610-6614.2003 |
| [15] |
RANK R G, YERUVA L. Hidden in plain sight: chlamydial gastrointestinal infection and its relevance to persistence in human genital infection[J]. Infect Immun, 2014, 82(4): 1362-1371. DOI:10.1128/IAI.01244-13 |
| [16] |
STECK N, HOFFMANN M, SAVA I G, et al. Enterococcus faecalis metalloprotease compromises epithelial barrier and contributes to intestinal inflammation[J]. Gastroenterology, 2011, 141(3): 959-971. DOI:10.1053/j.gastro.2011.05.035 |
| [17] |
BROSNAHAN A J, BROWN D R. Porcine IPEC-J2 intestinal epithelial cells in microbiological investigations[J]. Vet Microbiol, 2012, 156(3-4): 229-237. DOI:10.1016/j.vetmic.2011.10.017 |
| [18] |
SCHMITTGEN T D, LIVAK K J. Analyzing real-time PCR data by the comparative CT method[J]. Nat Protoc, 2008, 3(6): 1101-1108. DOI:10.1038/nprot.2008.73 |
| [19] |
ZHU L, BI H K, MA J C, et al. The two functional enoyl-acyl carrier protein reductases of Enterococcus faecalis do not mediate triclosan resistance[J]. mBio, 2013, 4(5): e00613-13. |
| [20] |
ZHU L, LIN J S, MA J C, et al. Triclosan resistance of Pseudomonas aeruginosa PAO1 is due to FabV, a triclosan-resistant enoyl-acyl carrier protein reductase[J]. Antimicrob Agents Chemother, 2010, 54(2): 689-698. DOI:10.1128/AAC.01152-09 |
| [21] |
龚腾芳, 贺峻琳, 谭磊, 等. 湖南省蛇源裂头蚴线粒体nad4和nad5基因遗传多样性分析[J]. 中国血吸虫病防治杂志, 2021, 33(1): 35-39. GONG T F, HE J L, TAN L, et al. Genetic diversity of Sparganum isolates from snakes in Hunan Province based on mitochondrial nad4 and nad5 genes[J]. Chinese Journal of Schistosomiasis Control, 2021, 33(1): 35-39. (in Chinese) |
| [22] |
MÖLLER S, CRONING M D R, APWEILER R. Evaluation of methods for the prediction of membrane spanning regions[J]. Bioinformatics, 2001, 17(7): 646-653. DOI:10.1093/bioinformatics/17.7.646 |
| [23] |
PETERSEN T N, BRUNAK S, VON HEIJNE G, et al. SignalP 4.0:discriminating signal peptides from transmembrane regions[J]. Nat Methods, 2021, 8(10): 785-786. |
| [24] |
LIN W Z, FANG J A, XIAO X, et al. iLoc-Animal: a multi-label learning classifier for predicting subcellular localization of animal proteins[J]. Mol BioSyst, 2013, 9(4): 634-644. DOI:10.1039/c3mb25466f |
| [25] |
WEI T C, LIN H Y, LU C C, et al. Expression of Crip2, a LIM-domain-only protein, in the mouse cardiovascular system under physiological and pathological conditions[J]. Gene Expr Patterns, 2011, 11(7): 384-394. DOI:10.1016/j.gep.2011.05.001 |
| [26] |
SONG Y F, DIMAIO F, WANG R Y R, et al. High-resolution comparative modeling with rosettaCM[J]. Structure, 2013, 21(10): 1735-1742. DOI:10.1016/j.str.2013.08.005 |
| [27] |
GEOURJON C, DELÉAGE G. SOPMA: significant improvements in protein secondary structure prediction by consensus prediction from multiple alignments[J]. Bioinformatics, 1995, 11(6): 681-684. DOI:10.1093/bioinformatics/11.6.681 |
| [28] |
LO P H Y, KO J M Y, YU Z Y, et al. The LIM domain protein, CRIP2, promotes apoptosis in esophageal squamous cell carcinoma[J]. Cancer Lett, 2012, 316(1): 39-45. DOI:10.1016/j.canlet.2011.10.020 |
| [29] |
ARCE C, RAMIREZ-BOO M, LUCENA C, et al. Innate immune activation of swine intestinal epithelial cell lines (IPEC-J2 and IPI-2I) in response to LPS from Salmonella typhimurium[J]. Comp Immunol Microbiol Infect Dis, 2010, 33(2): 161-174. DOI:10.1016/j.cimid.2008.08.003 |
| [30] |
KOCH L, FROMMHOLD D, BUSCHMANN K, et al. LPS-and LTA-induced expression of IL-6 and TNF-α in neonatal and adult blood: role of MAPKs and NF-κB[J]. Mediators Inflamm, 2014, 2014: 283126. |
(编辑 范子娟)



