猪急性腹泻综合征冠状病毒(swine acute diarrhea syndrome coronavirus,SADS-CoV)是2017年在中国广东省首次发现的一种可引起仔猪感染并发病的重要病原体[1],感染的仔猪表现为呕吐,腹泻,脱水直至死亡。5日龄以下的仔猪死亡率高达90%,育肥猪症状轻微,只引起轻度腹泻,在感染后2 d可恢复健康。组织病理学分析,感染仔猪表现为空肠和回肠明显的病变,肠绒毛萎缩变短,十二指肠切片仅显示轻微的病理变化[2]。SADS-CoV的基因序列与猪场附近洞穴内发现的蝙蝠冠状病毒(HKU2-CoV)相似率高达90%以上,暗示SADS-CoV可能由蝙蝠跨种传播给猪[1]。SADS-CoV感染仔猪的临床症状与PEDV、TGEV、PDCoV感染相似,目前,只能通过实验室检测方法进行鉴别诊断。因此,迫切需要建立一种灵敏可靠的诊断方法鉴定SADS-CoV感染。常规PCR虽然操作简单,但灵敏度不高。逆转录环介导恒温扩增技术(RT-LAMP)可以实现对SADS-CoV的检测,但不能用于定量分析样本中病毒拷贝数[3]。荧光定量PCR技术发展迅速,具有精确度高、灵敏性强、重复性好的优点[4-5],目前已广泛应用于各种动物和人类病原的检测。本研究以SADS-CoV高度保守的N基因序列作为靶标,建立SADS-CoV荧光定量PCR检测方法,并对感染的细胞和攻毒仔猪的临床样品进行检测,为SADS-CoV流行病学调查和基础研究提供可靠快捷的技术。
1 材料与方法 1.1 病毒与细胞SADS-CoV由华南农业大学马静云教授惠赠,PEDV、TGEV、PDCoV、PRRSV均由本实验室分离鉴定并保存。临床检测样品为实验室保存的口服SADS-CoV攻毒组和SPF新生仔猪对照组的组织。IPI-2I细胞和IPEC-J2细胞由本实验室保存。
1.2 主要试剂RNAiso Plus(Total RNA提取试剂),pMD18-T载体、T4 DNA连接酶、One Step TB Green® PrimeScriptTM RT-PCR Kit II、DH5α感受态细胞、PrimeSTAR Max DNA Polymerase和DNA凝胶回收试剂盒均购自宝生物工程(大连)有限公司;质粒小提试剂盒购自杭州Axygen公司;细胞总RNA提取试剂盒购自博日公司;病毒RNA提取试剂盒和琼脂糖购自天根生化科技有限公司;DNA Marker购自赛默飞公司。
1.3 引物设计与合成根据GenBank上发表的SADS-CoV的N基因的核苷酸序列(序列号:JX312064),通过MEGA4 (Molecular Evolutionary Genetics Analysis Version 4.0)软件比对序列,选择一段相对保守序列的区域,利用Primer Premier 5.0软件进行分析,设计特异性qPCR引物。上游引物序列为SADS-N-qF:5′-CCCCTAAACCGGCTCGTAA-3′,下游引物序列为SADS-N-qR:5′-CAGAATTAGGAACACGC-TTCCA-3′,引物由吉林省库美生物工程有限公司合成。
1.4 病毒RNA的提取及反转录临床组织样品取100 mg组织用剪镊剪碎并用液氮进行研磨,然后通过TRIzol法提取病毒RNA。细胞内RNA利用细胞总RNA提取试剂盒提取。收集细胞上清内RNA利用病毒RNA提取试剂盒提取。提取的RNA采用TaKaRa(中国大连)的PrimeScriptTM Ⅱ 1 st Strand cDNA Synthesis Kit获得cDNA。
1.5 重组质粒标准品的制备以SADS-CoV的cDNA为模板,进行PCR扩增。PCR反应体系为25 μL:2×PrimeSTAR Max DNA Polymerase为12.5 μL;上下游引物各0.5 μL;DNA模板1 μL;ddH2O 10.5 μL。反应程序:94 ℃预变性5 min;98 ℃变性10 s,55 ℃退火30 s,72 ℃延伸30 s,30个循环;最后72 ℃延伸10 min。采用1%浓度的琼脂凝胶电泳对PCR产物进行验证。PCR扩增完成后验证扩增片段大小为449 bp,将扩增后的目的片段按照凝胶回收试剂盒说明书回收,并将其克隆至pMD18-T载体,随后转化感受态细胞DH5α。筛选阳性重组质粒送库美生物工程有限公司测序,测序正确后的重组质粒pMD18-T-SADS-qN作为阳性质粒标准品,测定浓度并计算其拷贝数。
1.6 荧光定量PCR方法的建立1.6.1 荧光定量PCR反应扩增体系 经过条件优化确定反应体系为20 μL,上下游引物确定最佳引物浓度为10 mmol·L-1,RNA模板量为2 μL,2× One Step TB Green RT-PCR Buffer为10 μL,PrimeScript one Step Enzyme Mix为2 μL,无RNA酶水补至20 μL。反应条件:42 ℃反转录5 min,95 ℃变性10 s;95 ℃预变性5 s,60 ℃退火/延伸34 s,共40个循环。
1.6.2 标准曲线的建立及敏感性试验 利用以上优化的反应体系,使用灭菌ddH2O将质粒标准品进行10倍倍比稀释(3.31×107~3.31×101拷贝·μL-1) 后作为模板进行实时荧光定量PCR扩增反应,反应结束后由系统自动绘制生成标准曲线。
1.6.3 特异性试验 分别以SADS-CoV、PEDV、TGEV、PDCoV、PRRSV阳性质粒为模板通过建立的反应体系进行实时荧光定量PCR扩增反应,同时设立阴性对照,以此来评价该方法的特异性。
1.6.4 重复性试验 利用建立的荧光定量PCR检测方法,以相同拷贝数质粒标准品为模板进行组内重复试验;以10倍倍比稀释好的7个梯度(3.31× 107~3.31×101拷贝·μL-1)标准品为模板进行荧光定量PCR扩增,每个稀释度重复检测3次,进行组间重复试验;并通过计算变异系数(CV),分析荧光定量PCR的组内和组间重复性。
1.7 SADS-CoV感染IPI-2I和IPEC-J2细胞后荧光定量PCR检测1.7.1 SADS-CoV感染IPI-2I和IPEC-J2细胞病毒生长曲线测定 当细胞密度长至90%左右时接种SADS-CoV(MOI=1),对照组为DMEM,37 ℃感作1 h后,用DMEM洗涤细胞3次,然后加入维持液,37 ℃恒温CO2培养箱培养。维持液为DMEM中按照2.5 μg·mL-1的量加入不含EDTA的胰酶。分别于感染后2、12、24、36、48 h收集细胞上清,按照病毒RNA提取试剂盒提取病毒RNA。利用已经建立的荧光定量PCR进行病毒含量的测定。
1.7.2 不同剂量SADS-CoV感染IPI-2I和IPEC-J2细胞荧光定量PCR检测 当细胞密度长至90%左右时,分别采用MOI为0.1和1的SADS-CoV感染细胞,对照组为DMEM,37 ℃感作1 h后,用DMEM洗涤细胞3次,然后加入维持液,37 ℃恒温CO2培养箱培养。在感染后24 h收集细胞,反复冻融3次,按照细胞总RNA提取试剂盒提取RNA。利用已经建立的荧光定量PCR进行病毒含量的测定。
1.8 仔猪攻毒组织样品的检测将6头1日龄的SPF仔猪随机分为两组,试验组仔猪口服5 mL 104 TCID50SADS-CoV,对照组仔猪口服5 mL DMEM,定期观察仔猪临床症状。在感染病毒后36 h出现急性腹泻,采集对照组和试验组仔猪的各个组织脏器,分别称量100 mg各个组织,用液氮进行研磨捣碎,随后提取各个组织的RNA,并按照优化的反应条件进行检测。
2 结果 2.1 质粒标准品的制备以SADS-CoV的基因组为模板,利用所设计的特异性引物PCR扩增后获得预期大小的片段(见图 1)。随后将目的片段回收纯化与pMD18-T载体连接、转化,挑取典型单菌落提取质粒。对构建的重组质粒进行测序,结果显示成功构建重组质粒,命名为pMD18-T-SADS-qN。测其浓度为114 ng·μL-1,换算成拷贝数为3.31×1010拷贝·μL-1。
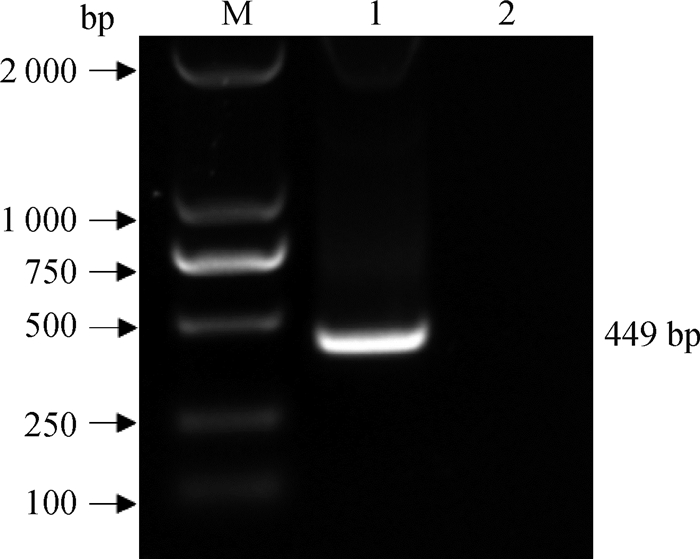
|
M. DL2000 DNA相对分子质量标准;1. 扩增的SADS-CoV N目的片段;2. 阴性对照 M. DL2000 DNA marker; 1. PCR product of SADS-CoV N; 2. Negative control 图 1 SADS-CoV N基因PCR扩增结果 Fig. 1 PCR product of SADS-CoV N |
将标准质粒进行10倍倍比稀释后,选取其中7个稀释度利用优化的荧光定量PCR反应条件进行扩增,得到不同浓度模板的扩增曲线。结果显示,该方法在3.31×101~3.31×107拷贝·μL-1模板量时,呈良好的线性关系,相关系数(R2)为0.997,斜率为-3.318,标准曲线方程:y=-3.318x+37.246(图 2A)。本试验建立的qPCR对阳性重组质粒的扩增产物熔解曲线显示,在熔解温度Tm为(80.0±0.1)℃时出现了唯一的特异性峰,无引物二聚体和非特异性扩增产物(图 2B)。
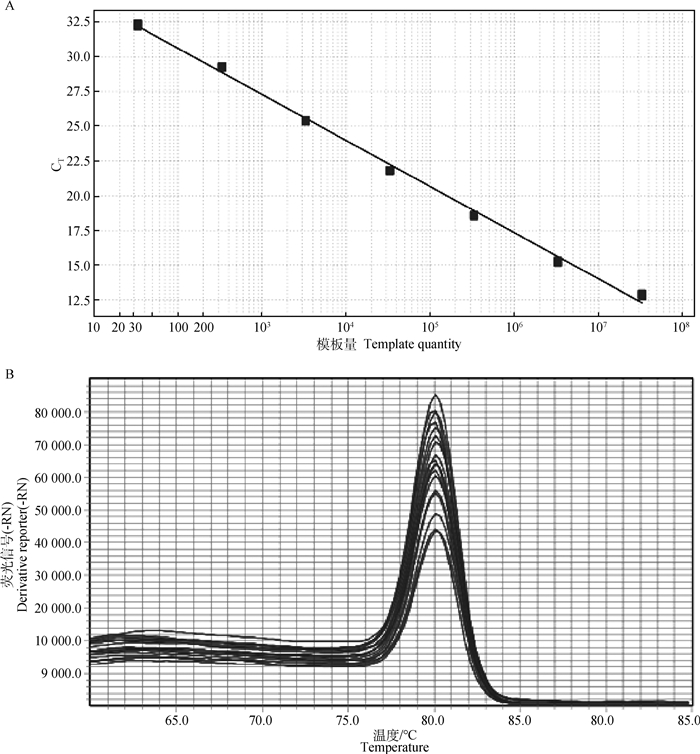
|
图 2 荧光定量PCR标准品标准曲线(A)与熔解曲线(B) Fig. 2 Standard curve(A) and melting curve(B) of qPCR |
以SADS-CoV、PEDV、TGEV、PDCoV和PRRSV阳性质粒为模板通过建立的反应体系进行实时荧光定量PCR扩增反应,结果显示除SADS-CoV阳性对照外,其他病毒检测结果均为阴性(图 3)。随后将SADS-CoV扩增产物进行测序比对验证,结果显示测序结果与参考序列一致,表明该方法具有良好的特异性。

|
图 3 荧光定量PCR特异性试验扩增曲线 Fig. 3 Amplification plot of the specificity assay |
将SADS-CoV阳性标准品倍比稀释后的样品作为模板,同时采用上述已建立好的荧光定量PCR方法和普通PCR方法对稀释好的模板进行灵敏性对比检测。结果显示,实时荧光定量PCR可检出最低限度为3.31×101拷贝·μL-1的标准品(图 4A),而普通PCR方法检测的最低限度为3.31×103拷贝·μL-1的标准品(图 4B),由此可见建立的荧光定量PCR方法的敏感性是普通PCR的100倍。

|
A.荧光定量PCR检测结果(1~7. 3.31×101~3.31×107拷贝·μL-1);B.普通PCR检测结果(M. DNA分子质量标准;1~7. 3.31×107~3.31×101拷贝·μL-1) A. Result of real-time PCR(1-7. 3.31×101-3.31×107copies·μL-1); B. Result of common PCR (M. DL2000 DNA marker; 1-7. 3.31×107~3.31×101copies·μL-1) 图 4 荧光定量PCR敏感性试验 Fig. 4 Sensitivity test of the qPCR |
采用上述建立的荧光定量PCR方法,分别做组内重复试验和组间重复试验,通过统计学方法计算变异系数,结果见表 1。结果显示,组内变异系数为0.16%~0.64%,组间变异系数为0.11%~0.72%,变异系数均小于1%,表明建立的荧光定量PCR具有良好的重复性。
|
|
表 1 荧光定量PCR重复性试验结果 Table 1 The result of qPCR repeatability test |
将MOI为1的SADS-CoV感染IPI-2I和IPEC-J2细胞,分别于感染后不同时间点(hpi)收集细胞上清,提取病毒RNA,利用已建立的检测方法绘制病毒生长曲线。结果如图 5显示,SADS-CoV感染细胞后2 h病毒含量较低,在12~36 h病毒含量迅速增长,36 h后病毒含量变化不大维持在较高水平。
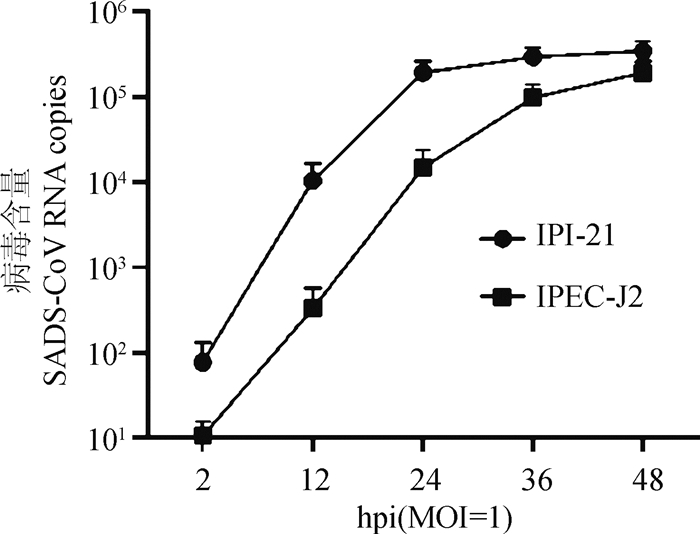
|
图 5 SADS-CoV在IPI-2I和IPEC-J2细胞中一步生长曲线 Fig. 5 The one step growth curve of SADS-CoV in IPI-2I and IPEC-J2 cellls |
将不同剂量的SADS-CoV感染IPI-2I和IPEC-J2细胞,在感染后24 h收集细胞,反复冻融3次,提取细胞总RNA,利用已建立的荧光定量PCR方法检测病毒的含量。结果如图 6显示,SADS-CoV的mRNA转录水平呈现剂量依赖性增加,当MOI为1时,IPI-2I和IPEC-J2细胞病毒含量分别为106.7、105.3拷贝·mL-1。表明建立的荧光定量PCR可用于细胞中病毒含量的检测,且可得到准确的病毒拷贝数。
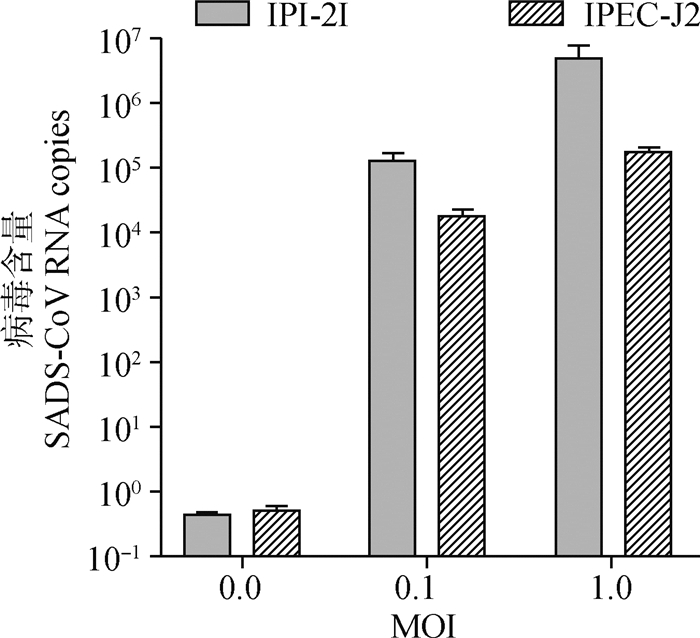
|
图 6 荧光定量PCR检测不同MOI的SADS-CoV感染IPI-2I和IPEC-J2细胞的情况 Fig. 6 Infection of IPI-2I and IPEC-J2 with different MOI of SADS-CoV examined by qPCR |
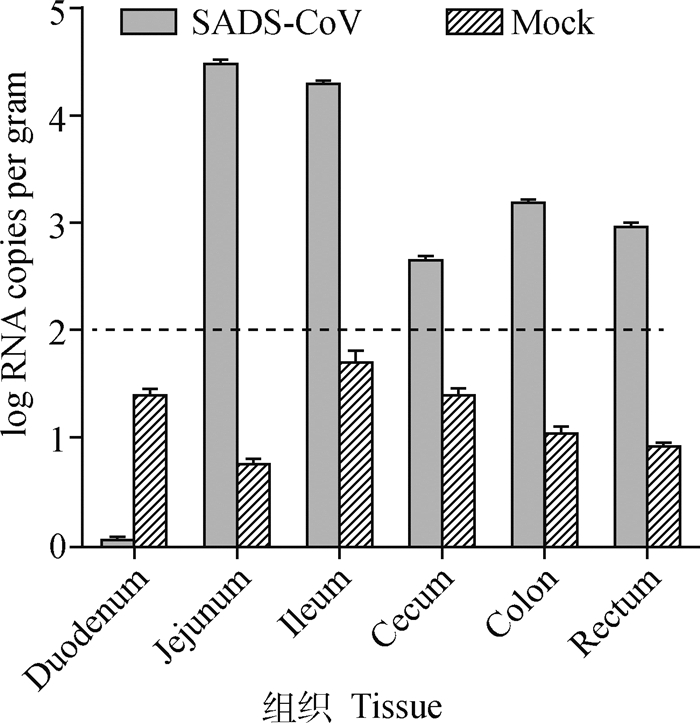
采集对照组和攻毒组仔猪的各个肠道组织进行荧光定量PCR检测,结果显示,仔猪在感染36 h后,空肠和回肠病毒含量达到104.2~104.5拷贝·g-1(图 7)。盲肠、结肠和直肠病毒含量高主要因为在粪便中含有大量的病毒,病毒随粪便在各个肠段分布,在组织处理时未能将粪便中的病毒排除。而对照组并未检测到病毒的存在。随后经测序进一步验证,结果显示攻毒组测序结果与参考序列一致,确定为SADS-CoV N基因序列。所建立的方法检测组织的最低检测度为1×102拷贝·g-1,低于最低检测线为阴性。综合以上,表明建立的荧光定量PCR可用于临床样品的检测。
|
图 7 荧光定量PCR检测不同组织中病毒RNA Fig. 7 Viral RNA in different tissues was detected by qRCR |
SADS-CoV为2017年新发现的猪冠状病毒,给我国广东省养猪业的发展带来了巨大威胁。SADS-CoV、PEDV、TGEV、PDCoV、猪轮状病毒(PRoV)五种病毒都可引起猪腹泻、呕吐,临床症状相似,且存在着混合感染的现象,给临床诊断带来了很大困扰,因此亟需建立可靠的实验室鉴别诊断方法。目前针对该病毒的检测方法包括病毒分离、间接ELISA、普通的PCR和荧光定量RT-PCR、血清中和试验、环介导等温扩增技术(LAMP)等[2, 6]。荧光定量PCR检测方法由于其高灵敏性、高特异性,重复性好、操作简单等优点,目前被应用于多种病原微生物的检测,如新型冠状病毒[7]、HIV[8]、人和动物多种冠状病毒[9]等。对于SADS-CoV的检测,Wang等[3]针对SADS-CoV N基因建立了一种简单快速的RT-LAMP检测方法,具有较高的特异性和灵敏性,但是此方法无法对病毒的感染情况进行定量分析。Zhou等[10]针对SADS-CoV N基因设计特异性的引物和探针,建立了基于TaqMan的实时RT-PCR检测方法。该方法过于昂贵,且需要特殊的配套仪器,用于临床样品检测成本较高。本研究针对SADS-CoV N基因保守区域,设计荧光定量PCR检测引物,经PCR扩增后构建该基因标准品。随后进行荧光定量PCR条件优化,绘制标准曲线呈现良好的线性关系,熔解曲线峰值单一。建立的荧光定量PCR方法特异性好,只能特异性检测出SADS-CoV,而不能检测出PEDV、TGEV、PDCoV、PRRSV等其他病毒。组内和组间的重复性变异系数均小于1%,表明建立的方法重复性好。敏感性试验结果显示本研究建立的荧光定量PCR检测方法的灵敏度高于普通PCR方法100倍。而Ma等[11]针对SADS-CoV的M基因保守区域建立的RT-qPCR检测方法的敏感性比普通PCR高10倍。表明本研究建立的荧光定量PCR方法敏感性更好。
此外,SADS-CoV可以在多种细胞系内复制,包括人的细胞系[12]和鼠的细胞系[13],但SADS-CoV感染仔猪主要表现空肠和回肠的病理变化。IPI-2I细胞为猪的回肠上皮细胞,IPEC-J2细胞为猪的空肠细胞,两种细胞均为SADS-CoV感染的靶细胞[14]。探究SADS-CoV在靶细胞上的感染情况,需要测定病毒的生长曲线,而传统生长曲线的测定主要选用TCID50或病毒蚀斑试验测定病毒滴度,TCID50在结果判定与统计过程中,人为因素对结果影响较大。病毒蚀斑试验可以有效反映样品中病毒滴度,但对细胞要求较高,而且并非所有病毒都能形成典型的病毒蚀斑。因此,作者利用建立的方法对SADS-CoV感染的IPI-2I和IPEC-J2细胞不同时间点病毒含量进行检测,绘制SADS-CoV在这两种细胞的生长曲线图。定量描述病毒生长规律的曲线为一步生长曲线,分为潜伏期、突破期和平稳期[15]。试验结果显示,SADS-CoV呈“S型”曲线增长,感染后2 h病毒含量较少,12~36 h病毒呈对数增长,36 h后病毒进入稳定期,生长速度变慢,病毒含量维持较高水平。检测不同剂量SADS-CoV的病毒含量试验结果显示,同样MOI在不同细胞上病毒拷贝数不同,当MOI为1时,IPI-2I和IPEC-J2细胞分别为106.7、105.3拷贝·mL-1。这种差异可能与病毒在不同细胞间复制动力学不同相关,还需进一步研究。本研究也对攻毒仔猪病毒在特定器官中的分布进行检测,发现病毒主要在空肠和回肠内定殖,病毒含量为104.2~104.5拷贝·g-1。本试验结果与之前报道的结果相符[1]。
4 结论建立的SADS-CoV SYBR Green荧光定量PCR检测方法特异性高、敏感性强、重复性好,不仅可用于对SADS-CoV的诊断和监测,还能对检测样品进行病毒定量。为SADS-CoV的早期监测和流行病学调查提供了有效手段,为今后开展SADS-CoV的研究奠定了良好基础。
| [1] |
ZHOU P, FAN H, LAN T, et al. Fatal swine acute diarrhoea syndrome caused by an HKU2-related coronavirus of bat origin[J]. Nature, 2018, 556(7700): 255-258. DOI:10.1038/s41586-018-0010-9 |
| [2] |
PAN Y F, TIAN X Y, QIN P, et al. Discovery of a novel swine enteric alphacoronavirus (SeACoV) in southern China[J]. Vet Microbiol, 2017, 211: 15-21. DOI:10.1016/j.vetmic.2017.09.020 |
| [3] |
WANG H N, CONG F, ZENG F W, et al. Development of a real time reverse transcription loop-mediated isothermal amplification method (RT-LAMP) for detection of a novel swine acute diarrhea syndrome coronavirus (SADS-CoV)[J]. J Virol Methods, 2018, 260: 45-48. DOI:10.1016/j.jviromet.2018.06.010 |
| [4] |
MACKAY I M, ARDEN K E, NITSCHE A. Real-time PCR in virology[J]. Nucleic Acids Res, 2002, 30(6): 1292-1305. DOI:10.1093/nar/30.6.1292 |
| [5] |
GINZINGER D G. Gene quantification using real-time quantitative PCR: an emerging technology hits the mainstream[J]. Exp Hematol, 2002, 30(6): 503-512. DOI:10.1016/S0301-472X(02)00806-8 |
| [6] |
GONG L, LI J, ZHOU Q F, et al. A new Bat-HKU2-like coronavirus in Swine, China, 2017[J]. Emerg Infect Dis, 2017, 23(9): 1607-1609. DOI:10.3201/eid2309.170915 |
| [7] |
MUNGMUNGPUNTIPANTIP R, WIWANITKIT V. RT-PCR Tests for COVID-19:observation on use and cost[J]. Clin Lab, 2020, 66. |
| [8] |
LIU Z Y, LI W, XU M, et al. Development of an in-house multiplex nested RT-PCR method for detecting acute HIV-1 infection in high risk populations[J]. Curr HIV Res, 2015, 13(4): 315-324. DOI:10.2174/1570162X13666150424154657 |
| [9] |
HU H, JUNG K, WANG Q H, et al. Development of a one-step RT-PCR assay for detection of pancoronaviruses (α-, β-, γ-, and δ-coronaviruses) using newly designed degenerate primers for porcine and avian fecal samples[J]. J Virol Methods, 2018, 256: 116-122. DOI:10.1016/j.jviromet.2018.02.021 |
| [10] |
ZHOU L, SUN Y, WU J L, et al. Development of a TaqMan-based real-time RT-PCR assay for the detection of SADS-CoV associated with severe diarrhea disease in pigs[J]. J Virol Methods, 2018, 255: 66-70. DOI:10.1016/j.jviromet.2018.02.002 |
| [11] |
MA L, ZENG F W, CONG F, et al. Development of a SYBR green-based real-time RT-PCR assay for rapid detection of the emerging swine acute diarrhea syndrome coronavirus[J]. J Virol Methods, 2019, 265: 66-70. DOI:10.1016/j.jviromet.2018.12.010 |
| [12] |
EDWARDS C E, YOUNT B L, GRAHAM R L, et al. Swine acute diarrhea syndrome coronavirus replication in primary human cells reveals potential susceptibility to infection[J]. Proc Natl Acad Sci U S A, 2020, 117(43): 26915-26925. DOI:10.1073/pnas.2001046117 |
| [13] |
YANG Y L, QIN P, WANG B, et al. Broad cross-species infection of cultured cells by bat HKU2-related swine acute diarrhea syndrome coronavirus and identification of its replication in murine dendritic cells in vivo highlight its potential for diverse interspecies transmission[J]. J Virol, 2019, 93(24): e01448-19. |
| [14] |
WANG X L, FANG L R, LIU S D, et al. Susceptibility of porcine IPI-2I intestinal epithelial cells to infection with swine enteric coronaviruses[J]. Vet Microbiol, 2019, 233: 21-27. DOI:10.1016/j.vetmic.2019.04.014 |
| [15] |
KROPINSKI A M. Practical advice on the one-step growth curve[M]//CLOKIE M, KROPINSKI A, LAVIGNE R, et al. Bacteriophages. Methods in Molecular Biology. New York, NY: Humana Press, 2018, 1681: 41-47.
|
(编辑 白永平)



