2. 江苏省动物重要疫病与人兽共患病防控协同创新中心, 扬州 225009;
3. 宁夏农林科学院动物科学研究所, 银川 750002
2. Jiangsu Co-innovation Center for Prevention and Control of Important Animal Infectious Diseases and Zoonoses, Yangzhou 225009, China;
3. Institute of Animal Science, Ningxia Academy of Agricultural and Forestry Sciences, Yinchuan 750002, China
牛病毒性腹泻-黏膜病(bovine viral diarrhea-mucosal disease,BVD-MD),简称牛病毒性腹泻(BVD),由牛病毒性腹泻病毒(bovine viral diarrhea virus,BVDV)感染导致,主要发生于牛,主要特征包括发热、腹泻、呼吸道症状、出血综合征、黏膜糜烂、孕牛流产等[1]。
BVDV属于黄病毒科(Flaviviridae)、瘟病毒属(Pestivirus),毒株无血清型差别,可根据抗原性或基因组差异分为BVDV-1、BVDV-2、BVDV-3(Hobi-like瘟病毒)等不同的基因型[2-3];每一基因型可能包含不同的基因亚型(BVDV-1a~BVDV-1u、BVDV-2a~BVDV-2d等)[4]。在接种细胞,毒株可表现为致细胞病变型(cytopathic,CP)或非致细胞病变型(noncytopathic,NCP)两种不同的生物型[5]。二者均可导致牛发生急性感染,但只有NCP型BVDV可在妊娠期母牛经胎盘感染40~120日龄胎儿并造成持续性感染犊牛产出[6]。
持续感染牛生长缓慢,也可表现为毛发、皮肤以及骨骼神经系统等先天性缺陷,通常继发感染1型牛疱疹病毒、3型牛副流感病毒、呼吸道合胞体病毒、传染性鼻气管炎病毒、牛呼吸道冠状病毒,罹患呼吸道疾病综合征[7]。在持续感染牛体内,NCP型BVDV可以通过重组宿主细胞mRNA、病毒基因组重复复制、重组、缺失、突变等多种机制变异转型为CP型,引起致死性的黏膜病[5]。因此,鉴定并剔除持续感染牛在BVD防控中具有重要意义。目前,在我国除云南、贵州、重庆、广东、海南、香港和台湾外,各地均有BVD的报道,流行毒株主要有BVDV-1a~BVDV-1d、BVDV-1m~BVDV-1q、BVDV-1u、BVDV-1v、BVDV-2a、BVDV-2b等基因亚型以及BVDV-3基因型[8-17],但关于导致牛发生持续感染的毒株分离鉴定、与同群急性感染牛毒株的抗原变异等信息知之甚少。本研究从宁夏回族自治区银川市某示范区持续感染高产奶牛分离获得BVDV毒株,测定了毒株的基因组序列并对其进行了遗传演化分析。
1 材料与方法 1.1 材料BVDV抗原检测试剂盒(HerdCheck BVDV antigen/Serum Plus)购自北京爱德士元亨生物科技有限公司;CP型对照毒株BVDV-AV69购自中国兽医药品监察所;FITC标记的猪抗BVDV多克隆抗体购自美国VMRD公司;牛外周血淋巴细胞分离液试剂盒为天津市灏洋生物制品科技有限责任公司产品;无BVDV污染的牛肾细胞(MDBK)由本实验室保存;MEM细胞培养基、胰蛋白酶和马血清均购自HyClone公司。RNA提取试剂RNAiso Plus、一步法RNA RT-PCR试剂盒PrimeScript One Step RT-PCR Kit Ver.2、E. coli DH5α感受态细胞、质粒提取试剂盒、DNA分子质量标准(DL2000)均为宝生物工程(大连)有限公司产品;pGEM T-easy载体试剂盒为Promega公司产品。EDTA抗凝血240份,采自宁夏回族自治区银川市某一高产奶牛示范区内240头3~6月龄犊牛。
1.2 抗原检测根据爱德士BVDV抗原检测试剂盒说明书,在反应板每孔加入50 μL检测抗体,然后分别加入双孔阴性对照各50 μL、双孔阳性对照各50 μL、待检全血50 μL,按指定步骤进行ELISA操作,最终用酶标仪在450 nm波长读取每个反应孔的A450 nm值。根据公式“S-N=样品A450 nm-阴性对照平均A450 nm”计算每个反应孔样品的修正OD值(corrected optical density,COD)。当样品COD值大于0.300时,判定为阳性。BVDV抗原检测阳性的牛,隔离2周后再次无菌采集抗凝血以试剂盒复检BVDV抗原,复检阳性者判为持续感染牛。
1.3 RT-PCR检测取病毒抗原阳性抗凝血100 μL,参照RNA提取试剂RNAiso Plus使用说明,提取总RNA。利用一步法RNA RT-PCR试剂盒与通用引物5′UTRF(5′-CTAGCCATGCCCTTAGTAGGACTA-3′)和5′UTRR(5′-CAACTCCATGTGCCATGTACAGCA-3′)进行RT-PCR扩增5′UTR区(293 bp),同时,利用引物E1-382F(5′-TGGTGGCCTTATGAGAC-3′)和E2-569R(5′-ATACAAGTCCAGTTTCCTCC-3′)扩增E2蛋白N端编码区(773 bp),反应程序:50 ℃反转录30 min;94 ℃预变性2 min;94 ℃ 30 s,55 ℃ 30 s,72 ℃ 50 s,35个循环;72 ℃延伸10 min。电泳切胶回收目的DNA片段,委托西安擎科泽西生物技术有限责任公司进行序列测定,然后进行BLAST(http://blast.ncbi.nlm.nih.gov/)工具在线比对,分析病毒的基因型。
1.4 病毒分离利用牛外周血淋巴细胞分离液,从复检阳性的5 mL牛抗凝血分离外周血淋巴细胞,悬浮于1 mL MEM细胞培养基,反复冻融3次后制备裂解液[18],接种至MDBK单层细胞。加入MEM维持液后,置37 ℃含5% CO2的培养箱培养,连续观察96 h,收集细胞培养物,盲传6代后,于-80 ℃保存。
1.5 免疫荧光鉴定以BVDV-AV69株为阳性对照,未接种病毒的MDBK细胞为阴性对照,利用200 μL培养物上清接种12孔细胞培养板中的MDBK单层细胞。根据FITC标记的猪抗BVDV多克隆抗体操作说明,在培养48 h后,利用免疫荧光检测细胞培养物中的BVDV抗原。
1.6 病毒基因组扩增、克隆及序列测定利用RNAiso Plus提取BVDV细胞培养物总RNA,利用一步法RNA RT-PCR试剂盒以9对引物扩增9段重叠的BVDV cDNA片段(表 1)。经琼脂糖电泳、切胶回收目的片段,与pGEM T-easy载体连接并转化E. coli DH5α感受态细胞。挑取单个菌落进行PCR鉴定,培养初步鉴定阳性的克隆, 并提取质粒送西安擎科泽西生物技术有限责任公司进行序列测定。
|
|
表 1 引物序列 Table 1 The sequences of the primers |
利用SeqMan软件对测序结果进行拼接以获得病毒基因组序列。参照UniProtKB数据库中BVDV参考毒株SD-15(登录号为Q01499),分析毒株基因结构。应用BioEdit对获得的病毒基因组与参考毒株的核苷酸序列进行同源性分析,以重组检测软件RDP4分析毒株基因组可能的重组位点。利用Mega7.0软件包的Neighbor-Joining方法,构建系统进化树,分析毒株的分类地位。
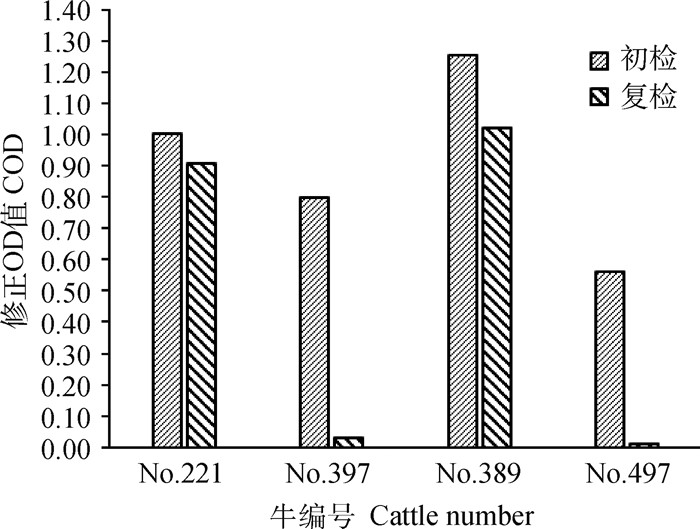
2 结果 2.1 抗原检测利用抗原检测试剂盒检测抗凝血中的BVDV抗原,结果如图 1所示,从240份样品共检出4份为BVD抗原阳性,复检判定两头犊牛(No. 221、No.389)为BVDV持续感染牛。
|
图 1 牛EDTA抗凝血BVDV抗原检测结果 Fig. 1 Detection of BVDV antigen from EDTA anticoagulated bovine blood |
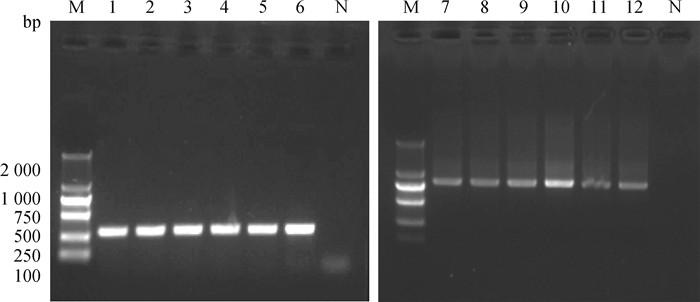
利用初检病毒抗原阳性(4份)和复检阳性(2份)的抗凝血提取总RNA,分别利用5′UTR通用引物和E2蛋白N端编码区引物进行RT-PCR检测,结果均扩增获得预期目的条带(图 2)。测序所得BVDV 5′UTR核苷酸只有1~2个碱基的差异,一致性为98.9%~100%,与GenBank中收录的ZM-95株(登录号:AF526381)、SD-15株(登录号:KR866116)等BVDV-1m代表性毒株一致性较高(94.37%~97.19%)。利用Blast分析测定获得的E2蛋白N端编码区1~507位核苷酸序列,与SD-15株和ZM-95株的核苷酸一致性较高(89.47%~91.21%)。
|
M. DL2000 DNA相对分子质量标准;N.阴性对照;1~6. 5′UTR扩增产物(293 bp);7~12.E2蛋白N端编码区扩增产物(773 bp) M. DL2000 DNA marker; N. Negative control; 1-6. The RT-PCR product(293 bp)of 5′UTR; 7-12. The RT-PCR product (773 bp)of N terminus coding region of E2 图 2 BVDV基因片段RT-PCR扩增结果 Fig. 2 Amplified results of BVDV gene fragments by RT-PCR |
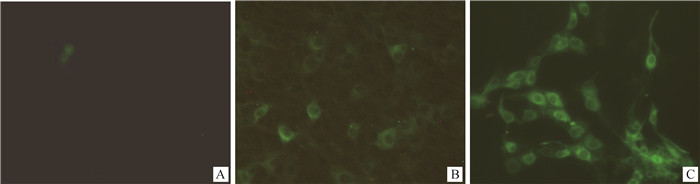
利用病毒抗原含量较高的复检阳性牛(No. 389)抗凝血制备淋巴细胞裂解液,接种MDBK细胞盲传至第6代仍未出现细胞病变,但第6代细胞裂解液接种MDBK细胞48 h利用免疫荧光鉴定可观察到绿色荧光(图 3),说明成功获得NCP型BVDV分离株,命名为NX2019/01。
|
A. MDBK细胞阴性对照;B. NX2019/01株接种MDBK细胞;C. AV69接种MDBK细胞 A. MDBK cells as negative control; B. MDBK cells infected with NX2019/01 isolate; C. MDBK cells infected with AV69 strain 图 3 免疫荧光法鉴定NX2019/01分离株(200×) Fig. 3 Identification of the NX2019/01 isolate by immunofluorescence assay (200×) |
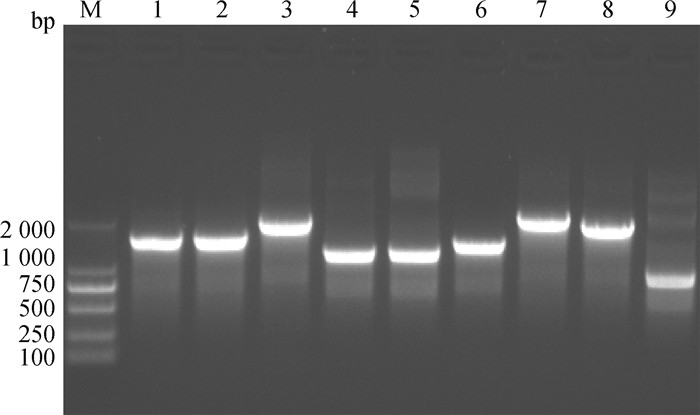
利用9对引物进行RT-PCR分段扩增NX2019/01分离株基因组,电泳表明获得与预期大小相符的9个目的片段(图 4)。将目的片段克隆测序拼接后获得NX2019/01分离株的基因组序列(12 107 nt)。已将该序列提交GenBank,登录号为MN623291。
|
M. DL2000 DNA相对分子质量标准;1~9. 1~9引物对(参见表 1)RT-PCR扩增产物 M. DL2000 DNA marker; 1-9. RT-PCR product amplified with primer pairs 1-9 (see Table 1) 图 4 RT-PCR扩增NX2019/01分离株基因组 Fig. 4 The RT-PCR amplification of the genome of NX2019/01 isolate |
NX2019/01株基因组长12 107 bp(5′UTR和3′UTR部分测定),其ORF长11 703 nt,编码3 898个氨基酸。毒株编码区组成包括N基因(504 nt)、C基因(312 nt)、Erns基因(681 nt)、E1基因(585 nt)、E2基因1 122 nt)、P7(210 nt)基因、NS2(1 359 nt)基因、NS3(2 049 nt)基因、NS4A基因(192 nt)、NS4B基因(1 041 nt)、NS5A基因(1 488 nt)、NS5B基因(2 160 nt)。BLAST结果显示,NX2019/01基因组与GenBank收录的1m亚型分离株的基因组核苷酸一致性较高,包括SD-15(93.84%)、ZM-95(93.62%)、XC(92.49%)、LN-1(92.17%);其次为1o亚型毒株IS26/01ncp(92.17%)、1q亚型(3株,83.15%~83.25%)。NX2019/01分离株与BVDV-1其他亚型毒株的基因组核苷酸一致性较低(125株,79.16%~81.90%),略高于BVDV-2(139株,71.75%~72.57%)和BVDV-3(20株,70.40%~73.20%)。
2.6 基因变异分析与其他参考毒株比对,NX2019/01 5′UTR相对保守,3′UTR与1m、1o及部分1q亚型毒株相似性均较高(表 2)。Blast分析表明,NX2019/01株5′UTR与国内前期报道的1m亚型毒株的相似性为93.23%~98.24%,表明该亚型毒株在我国具有较高的多态性。NX2019/01株各蛋白裂解位点与同亚型毒株的相似度高于1o、1q、1a、2a亚型以及BVDV-3毒株,表明其他亚型毒株完整基因重组的可能性较小(图 5)。与同源性最高的1m亚型毒株比较,NX2019/01株E1基因与牛源毒株的相似性(91.9%~94.8%)高于猪源毒株ZM-95(91.6%),但Erns基因与ZM-95株相似性最高(94.8%),E2基因与猪源毒株ZM-95的相似性(89.9%)也略高于牛源毒株SD-15(89.3%)LN-1(87.3%)、XC(88.8%)。与NX2019/01分离株相比,示范区内急性感染牛(No. 397、No. 497)体内病毒E2蛋白N端编码区核苷酸发生突变,其中,第26位(C/T)、第102位(A/G)、第171位(G/A)、第199位(G/T)突变导致E2蛋白结构域Ⅰ(第1—91位氨基酸)内第9位(S→L)和第67位(A→S)氨基酸变异;而第372位(G/A)核苷酸突变为同义突变,未导致E2蛋白结构域Ⅱ(第92—169位)氨基酸的变化。
|
|
表 2 NX2019/01分离株与参考毒株基因片段的核苷酸相似性 Table 2 Percentage of nucleotide similarity between individual gene fragments of NX2019/01 isolate and reference strains |

|
图 5 NX2019/01分离株与参考毒株蛋白裂解位点比较分析(方框中为BVDV-1m亚型毒株) Fig. 5 Comparative analysis of the protein cleavage site among NX2019/01 isolate and referenced viral strains(strains of BVDV-1m are in the box) |
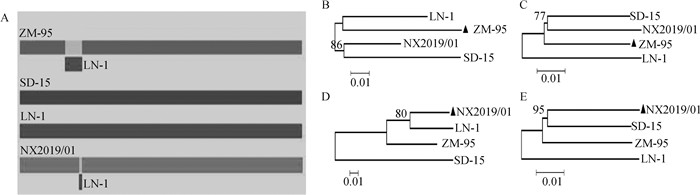
利用MEGA7.0软件对NX2019/01株基因组序列与参考毒株基因组序列进行比对,并构建系统进化树。结果显示,NX2019/01分离株与我国牛源分离株SD-15、LN1和XC以及猪源分离株ZM-95同属于BVDV-1m亚型。在氨基酸水平,NX2019/01株与SD-1株的相似性为96.0%,略高于ZM-95株(95.7%)、LN-1株(95.6%)以及XC株(95.4%)。利用全基因组、5′UTR以及E2基因构建进化树一致表明NX2019/01与SD15株、ZM-95株在进化上密切相关,但以N基因进行进化分析显示,3个毒株关联程度相对较低(图 6)。

|
图 6 基于基因组(A)、E2基因(B)、5′UTR(C)和N基因(D)的NX2019/01分离株的系统发育分析 Fig. 6 phylogenetic analysis of NX2019/01 based on nucleotide sequences of the genome (A), E2 gene (B), 5′UTR (C), and N gene (D) |
利用RDP4软件进行重组分析,发现NX2019/01株、ZM-95株存在亚型内基因重组的信号,推测二者基因组可能由主要亲本SD-15株与次要亲本LN-1株重组形成(图 7A)。ZM-95株分离鉴定早于SD-15株和LN-1株,因此,笔者推测,在早期可能存在与SD-15、LN-1高度相似的毒株流行。NX2019/01株基因组重组信号位于E2基因第179—288位核苷酸区段,重组区段核苷酸序列与LN-1株相似性为91.74%,高于XC株(89.91%)、ZM-95株(88.18%)以及SD-15株(78.30%),而非重组区段与SD-15株相似性(93.85%)高于ZM-95株(93.74%)、XC株(92.33%)以及LN-1株(92.10%)。ZM-95株基因组重组信号位于E1基因第168位—E2基因第332位,重组区段核苷酸序列与LN-1株相似性最高,为89.72%,高于XC株(89.59%)和SD-15株(87.85%),而非重组区段与SD-15株相似性(94.01%)均高于XC株(92.64%)和LN-1株(92.56%)。利用重组区段和非重组区段分别构建系统进化树,与重组分析结果一致(图 7B~E)。这些结果表明,NX2019/01株、ZM-95株在进化上与SD-15株、LN-1株或早期流行的高度相似毒株存在密切关联。
|
A.利用RDP4软件对1m亚型BVDV毒株进行同源重组分析;B. ZM-95株重组区段DNA序列的系统发育分析;C. ZM-95株非重组区段基因组DNA序列的系统发育分析;D. NX2019/01株重组区段DNA序列的系统发育分析;D. NX2019/01株非重组区段基因组DNA序列的系统发育分析 A. Homologous recombination analysis of 1m sub-genotypes by RDP4 software; B. Phylogenetic analysis of ZM-95 strain based on DNA sequence of the recombinant fragment; C. Phylogenetic analysis of ZM-95 strain based on genome sequence without the recombinant fragment; D. Phylogenetic analysis of NX2019/01 strain based on DNA sequence of the recombinant fragment; E. Phylogenetic analysis of NX2019/01 strain based on genome sequence without the recombinant fragment 图 7 NX2019分离株的同源重组分析 Fig. 7 Homologous recombination analysis of NX2019/01 isolate |
通过胎盘屏障垂直传播并感染胎儿导致持续感染,是BVDV维持生存的一种重要策略。宿主持续感染BVDV时由于免疫抑制而降低对毒株的选择压力,可能导致病毒突变体的产生[19],因此,对持续感染毒株的研究具有重要意义。本研究通过对间隔两周双份血样进行抗原检测,筛选获得BVDV持续感染牛2例,持续感染率(0.83%)与欧洲牛群持续感染率(1%~2%)以及美国牛群持续感染率(< 1%)类似[20],但抗原检出率(2.5%)低于同区前期的报道[21],可能与该示范区生物安全水平较高有关。免疫荧光鉴定表明分离获得的毒株NX2019/01为NCP生物型,与可形成持续感染的表型一致。NX2019/01株与我国ZM-95株、SD-15株、LN-1株以及XC株同源性高,但毒株抗原基因存在一定的差异。ZM-95株于1995年分离于内蒙古哲盟地区疑似猪瘟病料,是最早报道的BVDV-1m亚型的代表性毒株[22]。日本于1996年也从我国进口的奶牛分离获得类似的毒株[23]。目前,BVDV-1m是我国牛群和猪群流行最普遍的主要BVDV亚型[4, 8, 10, 24],在江苏地区羊群[25]和西北地区双峰驼[26]也有报道,但有关我国宁夏和西北地区该亚型毒株的基因信息较少,比较分析5′UTR、N基因等部分基因片段只能初步推断BVDV-1m毒株存在较高的多态性。
研究表明,持续感染牛在导致群体特异性毒株的出现中发挥重要作用。持续感染牛可通过免疫监视维持体内持续存在病毒群落的抗原稳定性,但特定牛群持续感染毒株的演化包含在其他孕畜再次形成持续感染的过程,是造成病毒基因变异的关键环节。Neill等[27]研究发现在持续感染过程中BVDV基因编码区核苷酸的突变速率远高于急性感染,且突变多发生于抗原基因E2编码区,导致了1b亚型毒株子代病毒抗原性改变。他们进一步研究证实,BVDV感染孕畜导致胎儿持续感染时,子代病毒变异主要发生于BVDV感染孕畜的环节,其次为BVDV经母体感染胎儿和持续感染小牛产出后期[28]。在本研究中,笔者发现在急性感染牛BVDV抗原基因E2 N端编码区存在2个核苷酸的错义突变,导致两头急性感染牛血液中的病毒在E2结构域Ⅰ出现不同的氨基酸变异,但未在持续感染牛隔离前后不同病毒发现E2基因变异导致抗原区氨基酸的改变,可能与筛选获得病例较少、对持续感染牛采样间隔较短有一定关系。
同源重组在RNA病毒演化进程中扮演着重要的角色,有利于修复病毒基因组损伤、增加病毒对宿主和环境的适应性。据推测,BVDV分离株NADL、SD1、Oregon、ILLNC、BVDV-2-890、799cp、839cp等存在不同基因亚型毒株的基因重组现象[29-30],但未见关于同亚型BVDV重组的报道。在本研究中,笔者首次通过重组预测、序列比对以及进化树分析,结果一致表明在NX2019/01株和ZM-95株均存在同基因型毒株重组现象,提示NX2019/01株、ZM-95株与SD-15株以及LN-1株或早期流行的高度相似毒株在进化上存在密切关联,但BVDV-1m亚型重组毒株浮现的机制,还有待进一步阐明。
4 结论分离获得了牛源BVDV-1m亚型NX2019/01株,以获得的全基因组序列厘定了BVDV-1m亚型毒株的进化关系,并首次发现同亚型BVDV毒株基因同源重组,信息有助于后续全面研究BVDV毒株在我国的演化规律。
| [1] | BAKER J C. The clinical manifestations of bovine viral diarrhea infection[J]. Vet Clin North Am Food Anim Pract, 1995, 11(3): 425–445. DOI: 10.1016/S0749-0720(15)30460-6 |
| [2] | RIDPATH J F, BOLIN S R, DUBOVI E J. Segregation of bovine viral diarrhea virus into genotypes[J]. Virology, 1994, 205(1): 66–74. |
| [3] | LIU L H, XIA H Y, WAHLBERG N, et al. Phylogeny, classification and evolutionary insights into pestiviruses[J]. Virology, 2009, 385(2): 351–357. |
| [4] | YEŞILBAČ K, ALPAY G, BECHER P. Variability and global distribution of subgenotypes of bovine viral diarrhea virus[J]. Viruses, 2017, 9(6): 128. DOI: 10.3390/v9060128 |
| [5] | PETERHANS E, BACHOFEN C, STALDER H, et al. Cytopathic bovine viral diarrhea viruses (BVDV):emerging pestiviruses doomed to extinction[J]. Vet Res, 2010, 41(6): 44. DOI: 10.1051/vetres/2010016 |
| [6] | HARDING M J, CAO X M, SHAMS H, et al. Role of bovine viral diarrhea virus biotype in the establishment of fetal infections[J]. Am J Vet Res, 2002, 63(10): 1455–1463. DOI: 10.2460/ajvr.2002.63.1455 |
| [7] | RIDPATH J. The contribution of infections with bovine viral diarrhea viruses to bovine respiratory disease[J]. Vet Clin North Am Food Anim Pract, 2010, 26(2): 335–348. DOI: 10.1016/j.cvfa.2010.04.003 |
| [8] | XUE F, ZHU Y M, LI J, et al. Genotyping of bovine viral diarrhea viruses from cattle in China between 2005 and 2008[J]. Vet Microbiol, 2010, 143(2-4): 379–383. DOI: 10.1016/j.vetmic.2009.11.010 |
| [9] | LANG Y F, GAO S D, DU J Z, et al. Polymorphic genetic characterization of E2 gene of bovine viral diarrhea virus in China[J]. Vet Microbiol, 2014, 174(3-4): 554–559. DOI: 10.1016/j.vetmic.2014.10.018 |
| [10] | DENG M L, JI S K, FEI W T, et al. Prevalence study and genetic typing of bovine viral diarrhea virus (BVDV) in four bovine species in China[J]. PLoS One, 2015, 10(4): e0121718. DOI: 10.1371/journal.pone.0121718 |
| [11] | ZHU L Q, REN M, LIN Y Q, et al. Identification of a bovine viral diarrhea virus 2 isolated from cattle in China[J]. Acta Virol, 2009, 53(2): 131–134. DOI: 10.4149/av_2009_02_131 |
| [12] | WANG W, SHI X C, CHEN C Y, et al. Genetic characterization of a noncytopathic bovine viral diarrhea virus 2b isolated from cattle in China[J]. Virus Genes, 2014, 49(2): 339–341. |
| [13] | SHI H, KAN Y, YAO L, et al. Identification of natural infections in sheep/goats with HoBi-like pestiviruses in China[J]. Transbound Emerg Dis, 2016, 63(5): 480–484. DOI: 10.1111/tbed.12551 |
| [14] |
陈新诺, 肖敏, 阮文强, 等. 川藏地区牦牛牛病毒性腹泻病毒分子流行病学调查及分离鉴定[J]. 畜牧兽医学报, 2018, 49(3): 606–613.
CHEN X N, XIAO M, RUAN W Q, et al. Molecular epidemiological investigation and isolation of bovine viral diarrhea virus in yak in Sichuan-Tibet Plateau region[J]. Acta Veterinaria et Zootechnica Sinica, 2018, 49(3): 606–613. (in Chinese) |
| [15] |
朱远茂, 杨立新, 马磊, 等. 牛病毒性腹泻病毒LJ36/14的分离与鉴定[J]. 中国预防兽医学报, 2016, 38(12): 949–952.
ZHU Y M, YANG L X, MA L, et al. Isolation and identification of bovine viral diarrhea virus strain LJ36/14[J]. Chinese Journal of Preventive Veterinary Medicine, 2016, 38(12): 949–952. (in Chinese) |
| [16] |
邓宇, 孙春清, 张宏彪, 等. 1株猪源牛病毒性腹泻病毒的分离与鉴定[J]. 畜牧兽医学报, 2012, 43(3): 416–423.
DENG Y, SUN C Q, ZHANG H B, et al. Isolation and identification of a pig bovine viral diarrhea virus[J]. Acta Veterinaria et Zootechnica Sinica, 2012, 43(3): 416–423. (in Chinese) |
| [17] |
李艳萍, 王立群, 李慧霞, 等. 一株源于商品胎牛血清的BVDV-2型的分离鉴定及基因组序列分析[J]. 畜牧兽医学报, 2020, 51(2): 320–328.
LI Y P, WANG L Q, LI H X, et al. Identification and complete genome sequencing analysis of a cytopathic BVDV-2 from commercial fetal bovine serum[J]. Acta Veterinaria et Zootechnica Sinica, 2020, 51(2): 320–328. (in Chinese) |
| [18] |
李海涛, 苗利光, 刘艳环, 等. 牛病毒性腹泻病活疫苗BVDV2/JZ05-1株毒力返强试验[J]. 特产研究, 2012, 34(1): 6–7, 26.
LI H T, MIAO L G, LIU Y H, et al. Study on pathogenicity of live BVDV2/JZ05-1 vaccine[J]. Special Wild Economic Animal and Plant Research, 2012, 34(1): 6–7, 26. DOI: 10.3969/j.issn.1001-4721.2012.01.002 (in Chinese) |
| [19] | BROCK K V. The persistence of bovine viral diarrhea virus[J]. Biologicals, 2003, 31(2): 133–135. DOI: 10.1016/S1045-1056(03)00029-0 |
| [20] | WALZ P H, GROOMS D L, PASSLER T, et al. Control of bovine viral diarrhea virus in ruminants[J]. J Vet Intern Med, 2010, 24(3): 476–486. |
| [21] |
王晓亮, 王玉梅, 张玉玲, 等. 宁夏部分地区牛病毒性腹泻的血清学检测[J]. 中国动物检疫, 2016, 33(2): 17–19.
WANG X L, WANG Y M, ZHANG Y L, et al. Serological investigation of bovine viral diarrhea in some regions of Ningxia[J]. China Animal Health Inspection, 2016, 33(2): 17–19. DOI: 10.3969/j.issn.1005-944X.2016.02.008 (in Chinese) |
| [22] |
王新平, 涂长春, 李红卫, 等. 从疑似猪瘟病料中检出牛病毒性腹泻病毒[J]. 中国兽医学报, 1996, 16(4): 341–345.
WANG X P, TU C C, LI H W, et al. Pigs naturally infected by bovine diarrhea virus present signs resembling hog cholera[J]. Chinese Journal of Veterinary Science, 1996, 16(4): 341–345. (in Chinese) |
| [23] | YAMAMOTO T, KOZASA T, AOKI H, et al. Genomic analyses of bovine viral diarrhea viruses isolated from cattle imported into Japan between 1991 and 2005[J]. Vet Microbiol, 2008, 127(3-4): 386–391. DOI: 10.1016/j.vetmic.2007.08.020 |
| [24] | DENG Y, SUN C Q, CAO S J, et al. High prevalence of bovine viral diarrhea virus 1 in Chinese swine herds[J]. Vet Microbiol, 2012, 159(3-4): 490–493. DOI: 10.1016/j.vetmic.2012.04.023 |
| [25] | MAO L, LI W L, YANG L L, et al. Primary surveys on molecular epidemiology of bovine viral diarrhea virus 1 infecting goats in Jiangsu province, China[J]. BMC Vet Res, 2016, 12(1): 181. DOI: 10.1186/s12917-016-0820-7 |
| [26] | GAO S D, LUO J H, DU J Z, et al. Serological and molecular evidence for natural infection of Bactrian camels with multiple subgenotypes of bovine viral diarrhea virus in Western China[J]. Vet Microbiol, 2013, 163(1-2): 172–176. DOI: 10.1016/j.vetmic.2012.12.015 |
| [27] | NEILL J D, NEWCOMER B W, MARLEY S D, et al. Genetic change in the open reading frame of bovine viral diarrhea virus is introduced more rapidly during the establishment of a single persistent infection than from multiple acute infections[J]. Virus Res, 2011, 158(1-2): 140–145. DOI: 10.1016/j.virusres.2011.03.024 |
| [28] | NEILL J D, NEWCOMER B W, MARLEY S D, et al. Greater numbers of nucleotide substitutions are introduced into the genomic RNA of bovine viral diarrhea virus during acute infections of pregnant cattle than of non-pregnant cattle[J]. Virol J, 2012, 9(1): 150. DOI: 10.1186/1743-422X-9-150 |
| [29] | JONES L R, WEBER E L. Homologous recombination in bovine pestiviruses:phylogenetic and statistic evidence[J]. Infect Genet Evol, 2004, 4(4): 335–343. |
| [30] | KAMEYAMA K, SAKODA Y, TAMAI K, et al. Genetic recombination at different points in the Npro-coding region of bovine viral diarrhea viruses and the potentials to change their antigenicities and pathogenicities[J]. Virus Res, 2006, 116(1-2): 78–84. DOI: 10.1016/j.virusres.2005.08.016 |



