肠道是动物消化吸收的重要场所,也是重要的免疫器官,对维持机体整体健康发挥着极其重要的作用。肠道内栖息着数量庞大的微生物,且这些微生物是维持肠道健康必不可少的因素[1-4]。近年来,基于宏基因组学探究肠道微生物组成的研究揭示了肠道微生物中存在着数量丰富的病毒,而病毒组的主要成分是以细菌为特异性宿主的噬菌体[5-6]。噬菌体分为温和噬菌体和烈性噬菌体,温和噬菌体在感染细菌后将基因组整合到细菌的基因组中,随着细菌基因组的复制而复制,并不裂解细菌,只有受到某些特定因素诱导时才会进入裂解周期[7];而烈性噬菌体在感染细菌后能够将其裂解,致使细菌死亡,随后释放子代噬菌体。近年,大量研究表明,肠道内的噬菌体与细菌存在着动态平衡的关系[8-10],且烈性噬菌体能够保护宿主抵抗外来病原菌,维持肠道微生态稳态[11]。
产肠毒素大肠杆菌(enterotoxigenic Escherichia coli, ETEC)是引起新生仔猪及断奶仔猪腹泻的主要病原菌之一,一直以来给养猪业带来巨大的经济损失[12-13],目前,多采用抗生素进行防控。但抗生素药物残留、细菌耐药性等问题日趋严重,噬菌体作为一种能够杀死细菌但对真核细胞无害的病毒,受到人们越来越多的关注[14]。目前,已有不少研究通过噬菌体疗法治疗ETEC感染,并取得了显著的治疗效果[15-16]。然而,猪肠道内本身存在的大量大肠杆菌噬菌体在ETEC入侵机体时发挥着怎样的作用还不太清楚。此前本实验室从健康猪肠道分离到的57株大肠杆菌噬菌体,发现有12株可裂解ETEC,本试验探究这些噬菌体的宿主谱以及对ETEC的裂解能力,以期进一步了解肠道噬菌体在维持机体健康中发挥的作用,并为防治ETEC感染提供新的思路。
1 材料与方法 1.1 试验材料1.1.1 菌株与噬菌体 本试验所用的12株大肠杆菌噬菌体以及用于宿主谱鉴定的89株大肠杆菌(其中包括5株ETEC)均为南京农业大学消化道微生物实验室从健康猪肠道内容物、肠黏膜或粪样中分离得到,ETEC的具体信息见表 1(其他用于宿主谱鉴定的大肠杆菌菌株信息见OSID开放科学数据与内容)。
|
|
表 1 5株产肠毒素大肠杆菌菌株信息 Table 1 The information of 5 Enterotoxigenic Escherichia coli strains |
1.1.2 主要试剂及培养基 SM缓冲液、PBS缓冲液、液体LB培养基、半固体LB培养基及固体培养基均由南京农业大学消化道微生物实验室常规配制。
1.2 试验方法1.2.1 噬菌体的扩增 采用双层平板法扩增:将12株噬菌体与各自的宿主菌混合后加入经溶解并冷却至45 ℃的半固体培养基,混匀后,立即倒入已凝固的固体琼脂平板上制成双层平板,待琼脂凝固后,37 ℃培养6~8 h。然后将3 mL SM缓冲洗脱平板上的噬菌体,经0.22 μm微孔滤膜过滤后备用。
1.2.2 噬菌体宿主谱的测定 以本实验室此前分离得到的89株大肠杆菌(包括5株ETEC)为指示菌,用点滴法[17]、双层平板法和液体培养法鉴定纯化后噬菌体的宿主谱。
点滴法:用无菌棉签沾取菌液,均匀涂抹在固体琼脂平板上,待平板自然干燥后,取10 μL纯化并经0.22 μm微孔滤膜过滤的噬菌体滤液滴在涂抹过菌液的平板上,37 ℃培养6~8 h,若平板上出现圆形空斑,则判定噬菌体可裂解该菌株。
双层平板法:以点滴法检测为阳性的菌为待测菌,取100 μL梯度稀释的噬菌体(103~109 PFU·mL-1)与培养至对数期的待测菌混合后铺双层平板,37 ℃培养6~8 h,若出现噬菌斑,则判定噬菌体可感染该菌株;若噬菌体浓度为109 PFU·mL-1时,仍无法出现噬菌斑,则视为疑似无法感染菌株,待进一步进行液体培养确认。
液体培养法:将噬菌体与培养至对数期的疑似无法感染的菌株按MOI=(1 000:1)~(1:100)混合至新鲜液体LB培养基中,37 ℃培养6~8 h,检测培养液噬菌体终浓度,若培养后噬菌体浓度没有升高,则确认该菌株无法感染。
1.2.3 抑菌试验 噬菌体的体外抑菌效力参照Wang等[18]的方法进行,稍作改动:将噬菌体与培养至对数生长期的菌株按照100:1及10:1的比例混匀,加入装有10 mL液体LB培养基的试管中,立即置于37 ℃、120 r·min-1振荡培养,每隔1 h测定OD600 nm的吸光值,得到细菌的生长曲线。以不加噬菌体的细菌作为对照,试验重复3次。
1.2.4 T4样噬菌体鉴定 参照Xu等[19]的方法提取噬菌体基因组,并置于-20 ℃保存备用。利用T4样噬菌体的主要衣壳蛋白基因(gene23)[20]为目标片段进行PCR扩增,引物序列:上游5′-TGTTATIGGTATGGTICGICGTGCTAT-3′,下游5′-TGAAGTTACCTTCACCACGACCGG-3′,PCR扩增产物用1%琼脂糖凝胶电泳进行检测。
1.2.5 噬菌体的电镜观察 参照Amarillas等[17]的方法,将浓缩后滴度为1010~1012 PFU·mL-1的噬菌体悬液滴在铜网上,作用15 min,从侧面用滤纸吸干多余液体,加1滴2%磷钨酸(PTA,pH7.2)于铜网,然后置于干燥滤纸上,自然干燥后进行透射电镜观察。
2 结果 2.1 噬菌体的宿主谱本试验结果表明,不同方法测得的噬菌体宿主谱并不一致(表 2),点滴法测得的宿主谱广于双层平板法,而且用108 PFU噬菌体颗粒铺双层平板无法出现噬菌斑的菌株用液体培养法同样无法扩增该噬菌体,笔者视这些菌株为噬菌体无法成功感染的菌株。点滴法得到的宿主谱表明本试验的12株噬菌体均能够裂解1株或多株ETEC,其中能裂解ETEC102的有7株(S144_2、S35、S38_1、L191、S86_1、L86、S172),能裂解ETEC103的有4株(S144_2、S35、L86、S172),能够裂解ETEC104的有8株(S143_1、S143_2、S144_2、S35、S37_1、S244_2、L42、L86),能够裂解ETEC105的有5株(S35、L191、S86_1、L86、S172)。但是此12株噬菌体在用ETEC菌株作双层平板法检测时均无法观测到噬菌斑,用液体培养法噬菌体浓度也未升高,说明这些噬菌体能够裂解ETEC,但是无法通过ETEC释放子代噬菌体,即无法感染。
|
|
表 2 用点滴法、双层平板法检测12株大肠杆菌噬菌体的宿主谱 Table 2 Host range of 12 E.coli phages |
通过测定噬菌体对4株ETEC菌株的裂解效力,发现不同噬菌体之间以及噬菌体对不同ETEC菌株的裂解效力有所不同。其中,噬菌体S143_1在MOI为100和10时均呈现出很强的裂解能力,能抑制ETEC104生长(图 1)。
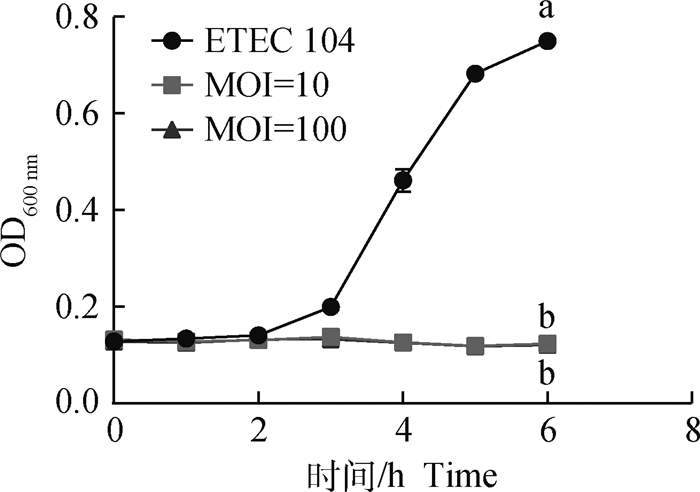
|
由于MOI为10和100时细菌生长曲线的OD值一直为最低检测值左右,图中两条曲线重叠。所标字母相异表示6 h时不同处理组差异显著(P < 0.05),所标字母相同表示6 h时不同处理组差异不显著(P>0.05)。下同 The 2 curves (MOI=10 and 100) are indistinguishable as a consequence of the OD value of 2 curves close approximation to the limit detection value. Different letters means significant difference between the treatments at 6h(P < 0.05), same letter means no significant difference between treatments at 6h(P>0.05). The same as below 图 1 噬菌体S143_1的抑菌曲线 Fig. 1 Bacteriostatic curves of phage S143_1 |
有3株噬菌体S143_2、S144_2及S86_1在MOI为100和10时都能够在一定程度上抑制ETEC104生长,且MOI为100时抑菌效果更好(图 2)。另外,有3株噬菌体S172、L86及S35只有在MOI为100时有一定的裂解效力,在MOI为10时无法抑制相应的ETEC生长(图 3)。其余噬菌体在MOI为100和10时均无法抑制ETEC生长。

|
图 2 噬菌体S143_2、S144_2及S86_1的抑菌曲线 Fig. 2 Bacteriostatic curves of phage S143_2, S144_2 and S86_1 |
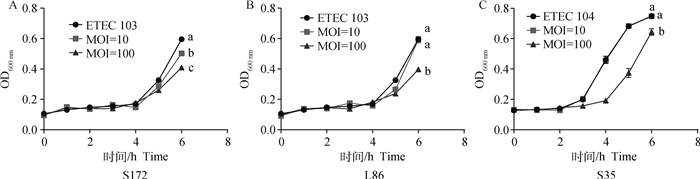
|
图 3 噬菌体S172、L86及S35的抑菌曲线 Fig. 3 Bacteriostatic curves of phage S172, L86 and S35 |
由于革兰阴性细菌能够被噬菌体裂解, 但是噬菌体无法增殖的现象多见于T偶数噬菌体中,尤其是T4噬菌体[21-22],因此,首先对本试验的12株噬菌体是否为T4样噬菌体进行了鉴定。提取噬菌体基因组,检测T4样噬菌体的保守基因主要衣壳蛋白基因(gene 23),结果显示,有8株噬菌体均为T4样噬菌体,而S143_1、S172、S86_1和L42等4株噬菌体不属于T4样噬菌体(图 4)。随后,对电泳结果呈阴性的4株噬菌体进行透射电镜鉴定(图 5),发现这4株噬菌体均属于有尾噬菌体目肌尾噬菌体科。
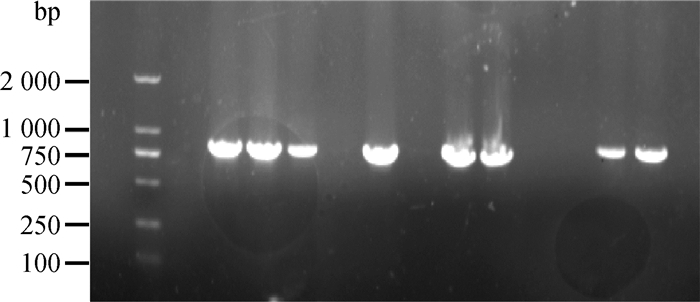
|
从左至右样品顺序:DL 2000 DNA相对分子质量标准、阴性对照、S143_2、S144_2、S35、S143_1、S37_1、L42、L191、S38_1、S86_1、S172、S244_2、L86 Samples from left to right: DL 2000 DNA maker, negative control, S143_2, S144_2, S35, S143_1, S37_1, L42, L191, S38_1, S86_1, S172, S244_2, L86 图 4 T4样噬菌体主要衣壳蛋白基因鉴定结果 Fig. 4 Identification of gene 23 of T4-like phage |
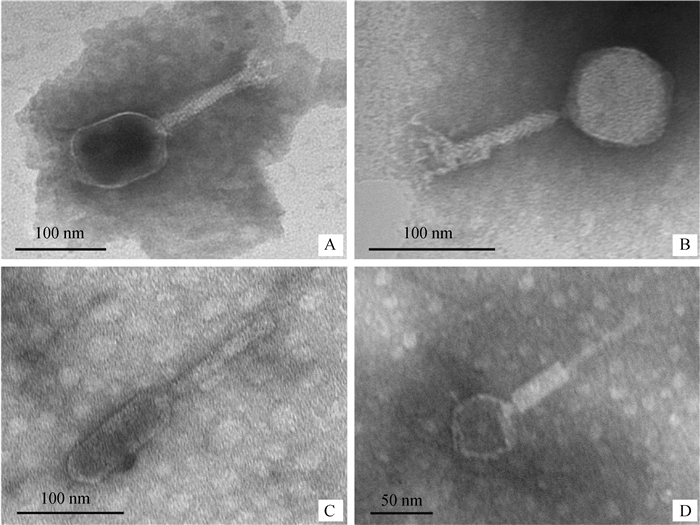
|
A. S143_1; B. S172; C. S86_1; D. L42 图 5 噬菌体S143_1、S172、S86_1和L42的电镜形态 Fig. 5 TEM of S143_1、S172、S86_1 and L42 |
对噬菌体宿主谱的测定通常用点滴法或双层平板培养法,但是相关研究表明,点滴法测定出的宿主谱往往要比双层平板培养法得到的结果更广,并且两种方法得到的结果之间并无相关性[23-26]。本试验得到的结果也表明,用点滴法与双层平板法得到的宿主谱并不一致,因此,确定噬菌体的宿主谱时单一的用点滴法或平板法无法准确判断,须将点滴法和双层平板培养法结合使用来判定结果。
点滴法测定的宿主谱比用双层平板法广,是由于噬菌体可以裂解某些细菌但并不能在这些细菌细胞内产生子代噬菌体。导致噬菌体裂解细菌可能的原因:1)用于扩增噬菌体的大肠杆菌本身会产生大肠杆菌素(colicin),抑制指示菌的生长从而形成空斑[27]。然而将用于扩增噬菌体的菌株单独培养后取菌液上清重复点滴试验,并未观测到空斑,说明本试验使用的大肠杆菌并未产生大肠杆菌素;2)噬菌体扩增液中含有裂解酶(lysin),但裂解酶对革兰阳性菌效果较好,对革兰阴性菌的作用效果较差[28],因此,本试验中观测到的现象并非是裂解酶的作用;3)外溶菌作用(lysis from without),即过多的噬菌体同时吸附到细菌表面,噬菌体在细胞壁上打孔致使细菌死亡。该现象主要发生在革兰阴性菌上,需要较高的MOI[29],并且目前报道的该现象多见于T偶数噬菌体,尤其是T2和T4噬菌体[21, 22]。本试验中的S143_2、S144_2、L86和S35均为T4样噬菌体,且均是在MOI为100时裂解效果优于MOI为10时,因此,推测这4株噬菌体裂解细菌时发生外溶菌作用。另外,4株T4样噬菌体在MOI为10和100时均无抑菌效果,推测可能需要更高的MOI才能发生外溶菌作用。4)噬菌体感染流产(abortive infection),即细菌通过多种机制导致噬菌体在细菌细胞内生物合成受阻[30-31]。本试验中有4株噬菌体不是T4噬菌体,这4株噬菌体电镜结果显示,除了S143_1不确定其是否为T2噬菌体外,其余3株(S172、S86_1、L42)均不是T偶数样噬菌体,因此,推测这3株噬菌体可能是发生了感染流产导致噬菌体裂解细菌却不产生子代噬菌体。
研究表明,噬菌体能够黏附到黏膜表面的黏液中,并且黏附到黏液层的噬菌体能够通过裂解细菌显著减少细菌对上皮的黏附和侵袭[11, 32],而肠道内存在数量巨大的噬菌体,说明肠道内的噬菌体可能起着重要的生物屏障作用。本研究的12株噬菌体均分离自健康猪肠道,其中,7株噬菌体在高MOI时对ETEC有抑制作用,虽然均为体外研究结果,体内抑菌效果需要进一步开展研究确认,但可以根据体外抑菌结果推测肠道内存在这些噬菌体的个体在受到ETEC侵袭时可能具有更好的抵抗力。当动物的消化道环境较稳定时,并不易受到病原菌侵袭,但是当仔猪断奶后,日粮组成、消化道形态结构、肠道内营养物质等诸多因素发生改变,使得仔猪肠道抵抗力差,易受到潜在的病原菌侵袭[33-35]。因此,仔猪在断奶前,如果其肠道内存在能够裂解病原菌的噬菌体,那么仔猪断奶后产生腹泻的可能性便会降低。事实上,笔者在养殖场上确实观察到这样的现象,同一窝断奶仔猪,有些发生腹泻有些则不腹泻,不腹泻的仔猪除了其自身免疫能力和抗应激能力较强外,也可能是由于其肠道内有定植的噬菌体提供了保护作用。尽管本试验的大肠杆菌噬菌体均不能通过ETEC增殖,但是由于肠道内存在其他大肠杆菌可作为这些噬菌体的宿主,这些噬菌体可增殖至较高浓度后通过外溶菌作用致使ETEC死亡。笔者的研究只是针对了ETEC这一种致病菌,而肠道内存在种类繁多的细菌及其噬菌体,很可能肠道内的其他噬菌体也同样存在上述现象,这些噬菌体共同作用,为机体提供一定程度的保护作用。
4 结论从猪肠道内分离到12株大肠杆菌噬菌体,虽然无法感染ETEC,但仍可裂解ETEC,其中,7株在MOI为10或100时可有效地抑制ETEC生长。
| [1] | HOOPER L V, MIDTVEDT T, GORDON J I. How host-microbial interactions shape the nutrient environment of the mammalian intestine[J]. Annu Rev Nutr, 2002, 22: 283–307. DOI: 10.1146/annurev.nutr.22.011602.092259 |
| [2] | BARTHEL M, HAPFELMEIER S, QUINTANILLA-MARTÍNEZ L, et al. Pretreatment of mice with streptomycin provides a Salmonella enterica serovar typhimurium colitis model that allows analysis of both pathogen and host[J]. Infect Immun, 2003, 71(5): 2839–2858. DOI: 10.1128/IAI.71.5.2839-2858.2003 |
| [3] | IVANOV I I, DE LLANOS FRUTOS R, MANEL N, et al. Specific microbiota direct the differentiation of IL-17-producing t-helper cells in the mucosa of the small intestine[J]. Cell Host Microbe, 2008, 4(4): 337–349. DOI: 10.1016/j.chom.2008.09.009 |
| [4] | REEVES A E, KOENIGSKNECHT M J, BERGIN I L, et al. Suppression of Clostridium difficile in the gastrointestinal tracts of germfree mice inoculated with a murine isolate from the family Lachnospiraceae[J]. Infect Immun, 2012, 80(11): 3786–3794. DOI: 10.1128/IAI.00647-12 |
| [5] | EDWARDS R A, ROHWER F. Viral metagenomics[J]. Nat Rev Microbiol, 2005, 3(6): 504–510. DOI: 10.1038/nrmicro1163 |
| [6] | SACHSENRÖDER J, TWARDZIOK S O, SCHEUCH M, et al. The general composition of the faecal virome of pigs depends on age, but not on feeding with a probiotic bacterium[J]. PLoS One, 2014, 9(2): e88888. DOI: 10.1371/journal.pone.0088888 |
| [7] | WEINBAUER M G. Ecology of prokaryotic viruses[J]. FEMS Microbiol Rev, 2004, 28(2): 127–181. DOI: 10.1016/j.femsre.2003.08.001 |
| [8] | THINGSTAD T F. Elements of a theory for the mechanisms controlling abundance, diversity, and biogeochemical role of lytic bacterial viruses in aquatic systems[J]. Limnol Oceanogr, 2000, 45(6): 1320–1328. DOI: 10.4319/lo.2000.45.6.1320 |
| [9] | LIM E S, ZHOU Y J, ZHAO G Y, et al. Early life dynamics of the human gut virome and bacterial microbiome in infants[J]. Nat Med, 2015, 21(10): 1228–1234. DOI: 10.1038/nm.3950 |
| [10] | SHARON I, MOROWITZ M J, THOMAS B C, et al. Time series community genomics analysis reveals rapid shifts in bacterial species, strains, and phage during infant gut colonization[J]. Genome Res, 2013, 23(1): 111–120. DOI: 10.1101/gr.142315.112 |
| [11] | BARR J J, AURO R, FURLAN M, et al. Bacteriophage adhering to mucus provide a non-host-derived immunity[J]. Proc Natl Acad Sci U S A, 2013, 110(26): 10771–10776. DOI: 10.1073/pnas.1305923110 |
| [12] | NOAMANI B N, FAIRBROTHER J M, GYLES C L. Virulence genes of O149 enterotoxigenic Escherichia coli from outbreaks of postweaning diarrhea in pigs[J]. Vet Microbiol, 2003, 97(1-2): 87–101. DOI: 10.1016/j.vetmic.2003.08.006 |
| [13] | GRESSE R, CHAUCHEYRAS-DURAND F, FLEURY M A, et al. Gut microbiota dysbiosis in postweaning piglets:understanding the keys to health[J]. Trends Microbiol, 2017, 25(10): 851–873. DOI: 10.1016/j.tim.2017.05.004 |
| [14] | DALMASSO M, HILL C, ROSS R P. Exploiting gut bacteriophages for human health[J]. Trends Microbiol, 2014, 22(7): 399–405. DOI: 10.1016/j.tim.2014.02.010 |
| [15] | LEE C Y, KIM S J, PARK B C, et al. Effects of dietary supplementation of bacteriophages against enterotoxigenic Escherichia coli (ETEC) K88 on clinical symptoms of post-weaning pigs challenged with the ETEC pathogen[J]. J Anim Physiol Anim Nutr, 2017, 101(1): 88–95. DOI: 10.1111/jpn.12513 |
| [16] | CHA S B, YOO A N, LEE W J, et al. Effect of bacteriophage in enterotoxigenic Escherichia coli (ETEC) infected pigs[J]. J Vet Med Sci, 2012, 74(8): 1037–1039. DOI: 10.1292/jvms.11-0556 |
| [17] | AMARILLAS L, RUBÍ-RANGEL L, CHAIDEZ C, et al. Isolation and characterization of phiLLS, a novel phage with potential biocontrol agent against multidrug-resistant Escherichia coli[J]. Front Microbiol, 2017, 8: 1355. DOI: 10.3389/fmicb.2017.01355 |
| [18] | WANG Z F, ZHENG P P, JI W H, et al. SLPW:a virulent bacteriophage targeting methicillin-resistant staphylococcus aureus in vitro and in vivo[J]. Front Microbiol, 2016, 7: 934. |
| [19] | XU J T, CHEN M M, HE L C, et al. Isolation and characterization of a T4-like phage with a relatively wide host range within Escherichia coli[J]. J Basic Microb, 2016, 56(4): 405–421. DOI: 10.1002/jobm.201500440 |
| [20] | TéTART F, DESPLATS C, KUTATELADZE M, et al. Phylogeny of the major head and tail genes of the wide-ranging T4-type bacteriophages[J]. J Bacteriol, 2001, 183(1): 358–366. DOI: 10.1128/JB.183.1.358-366.2001 |
| [21] | YOUNG R Y. Bacteriophage lysis:mechanism and regulation[J]. Microbiol Rev, 1992, 56(3): 430–481. DOI: 10.1128/MMBR.56.3.430-481.1992 |
| [22] | ARISAKA F, KANAMARU S, LEIMAN P, et al. The tail lysozyme complex of bacteriophage T4[J]. Int J Biochem Cell Biol, 2003, 35(1): 16–21. DOI: 10.1016/S1357-2725(02)00098-5 |
| [23] | MIRZAEI M K, NILSSON A S. Isolation of phages for phage therapy:a comparison of spot tests and efficiency of plating analyses for determination of host range and efficacy[J]. PLoS One, 2015, 10(5): e0118557. |
| [24] | KUTTER E. Phage host range and efficiency of plating[M]//CLOKIE M R J, KROPINSKI A M. Bacteriophages. Totowa: Humana Press, 2009: 141-149. |
| [25] | XIE Y C, WAHAB L, GILL J J. Development and validation of a microtiter plate-based assay for determination of bacteriophage host range and virulence[J]. Viruses, 2018, 10(4): 189. |
| [26] | LETAROV A V, KULIKOV E E. Determination of the bacteriophage host range: culture-based approach[M]//AZEREDO J, SILLANKORVA S. Bacteriophage Therapy. New York: Humana Press, 2018: 75-84. |
| [27] | CASCALES E, BUCHANAN S K, DUCHé D, et al. Colicin biology[J]. Microbiol Mol Biol Rev, 2007, 71(1): 158–229. |
| [28] | LOESSNER M J. Bacteriophage endolysins-current state of research and applications[J]. Curr Opin Microbiol, 2005, 8(4): 480–487. DOI: 10.1016/j.mib.2005.06.002 |
| [29] | ABEDON S T. Lysis from without[J]. Bacteriophage, 2011, 1(1): 46–49. |
| [30] | HYMAN P, ABEDON S T. Bacteriophage host range and bacterial resistance[J]. Adv Appl Microbiol, 2010, 70: 217–248. DOI: 10.1016/S0065-2164(10)70007-1 |
| [31] | DE JONGE P A, NOBREGA F L, BROUNS S J J, et al. Molecular and evolutionary determinants of bacteriophage host range[J]. Trends Microbiol, 2019, 27(1): 51–63. |
| [32] | ALMEIDA G M F, LAANTO E, ASHRAFI R, et al. Bacteriophage adherence to mucus mediates preventive protection against pathogenic bacteria[J]. mBio, 2019, 10(6): e01984–19. |
| [33] | LALLÈS J P, BOSI P, SMIDT H, et al. Nutritional management of gut health in pigs around weaning[J]. Proc Nutr Soc, 2007, 66(2): 260–268. DOI: 10.1017/S0029665107005484 |
| [34] | SCHOKKER D, ZHANG J, VASTENHOUW S A, et al. Long-lasting effects of early-life antibiotic treatment and routine animal handling on gut microbiota composition and immune system in pigs[J]. PLoS One, 2015, 10(2): e0116523. DOI: 10.1371/journal.pone.0116523 |
| [35] | MCLAMB B L, GIBSON A J, OVERMAN E L, et al. Early weaning stress in pigs impairs innate mucosal immune responses to enterotoxigenic E. coli challenge and exacerbates intestinal injury and clinical disease[J]. PLoS One, 2013, 8(4): e59838. DOI: 10.1371/journal.pone.0059838 |



