2. 华中农业大学 农业微生物学国家重点实验室, 武汉 430070
2. State Key Laboratory of Agricultural Microbiology, Huazhong Agricultural University, Wuhan 430070, China
猪流感由猪流感病毒(swine influenza virus,SIV)引发,该病毒属正黏病毒科,是单股负链RNA病毒,该病的传染性极强,发病率几近100%[1-3]。由于流感病毒感染宿主时的受体选择性,猪也被称为流感病毒的“混合器”,多种流感病毒感染猪后容易发生重配导致新型毒株的产生,因此对猪流感进行防控,能极大地降低流感对人的危害[4-6]。流感病毒的生命周期大致可以分为吸附、侵入、复制、组装和出芽5个阶段,在整个生命周期中都需要借助宿主因子完成其生命过程,因此,找到流感病毒复制的关键宿主因子具有极其重要的意义[7-9]。
随着近些年CRISPR/Cas9技术的流行,该系统早已应用于各种物种及细胞的基因编辑改造中,从而获得抗病物种[10-11]。Peng等[12]通过CRISPR/Cas9技术对柑橘中疾病易感基因CsLOB1的启动子进行编辑赋予了柑橘对溃疡病的抗性;Xie等[13-14]结合CRISPR/Cas9和RNAi技术在猪Rosa26基因座中插入抗病毒小发夹RNA(shRNA),最后通过体细胞核移植生产了抗经典猪瘟病毒的转基因猪;Burkard等[15]利用CRISPR/Cas9技术得到了对猪繁殖与呼吸综合征病毒具有完全抗性的缺少CD163 SRCR5结构域的基因编辑猪。另外,一些实验室利用CRISPR/Cas9系统筛选到了流感病毒复制的关键宿主因子。Han等[16]通过CRISPR/Cas9在A549细胞上筛选出了流感病毒复制的必需基因SLC35A1,该文章指出SLC35A1敲除后通过影响唾液酸的转运影响了病毒的增殖;Heaton等[17]利用CRISPR SAM系统在人基因组范围筛选出了抑制流感病毒增殖的宿主基因B4GALNT2,其主要是通过影响α-2, 3唾液酸的功能影响了病毒的吸附,进而影响病毒的增殖。实验室前期利用猪肾细胞CRISPR/Cas9全基因组敲除文库筛选到了当前流行的欧亚禽系H1N1亚型猪流感病毒复制相关的宿主基因,通过验证发现ALG5是猪流感病毒复制过程的重要基因。
ALG5基因编码的蛋白质为磷酸十二烷基磷酸酯-葡萄糖基转移酶(dolichyl-phosphate beta-glucosyltransferase),主要功能是向寡甘露糖核心中添加葡萄糖残基,并有效地将寡甘露糖核心转移至新生糖蛋白,参与寡甘露糖核心的糖基化[18-19]。A型流感病毒表面蛋白的翻译后修饰对于规避宿主防御和支持病毒生命周期意义重大[20],而ALG5作为一个糖基化激酶[21],可能参与到了A型流感病毒表面蛋白的翻译后修饰过程。为研究ALG5在SIV复制过程中的功能,同时为了更接近自然感染状态,本研究利用CRISPR/Cas9技术在NPTr上构建了ALG5基因敲除细胞系,并构建了ALG5真核表达的质粒,从而验证了ALG5在SIV复制中的作用,为阐述SIV的复制过程和致病机制提供理论依据。
1 材料与方法 1.1 材料1.1.1 细胞、病毒及质粒 人胚胎肾细胞(HEK293T,293T)、犬肾细胞(MDCK)由本实验室保存;稳定表达Cas9蛋白的新生猪气管上皮细胞(NPTr)由本实验室制备;流感病毒毒株A/swine/Hubei/221/2016(H1N1)简称HB、A/swine/Henan/F26/2017(H1N1)简称F26由本实验室保存;p3×Flag-CMV-14(Flag)由北京大学蒋争凡教授馈赠;psPAX2、pMD2.G均由华中农业大学罗锐副教授馈赠;LentiGuide-puro由华中农业大学谢胜松副教授馈赠。
1.1.2 主要试剂 DNA marker、Solution Ⅰ购自大连宝生物公司;Taq-T DNA聚合酶购自北京全式金公司;BsmB Ⅰ购自NEB公司;TPCK胰酶、polybrene购自美国Sigma公司;lipofectamine 8000购自碧云天公司;Puromycin购于Selleck公司;细胞基因组DNA提取试剂盒购自北京天根生化科技有限公司;无内毒素大量质粒提取试剂盒购自美国OMEGA公司;兔抗ALG5多克隆抗体(A17693)购自中国武汉ABclonal公司;兔抗流感病毒蛋白NP多克隆抗体GTX125989购自美国GeneTex公司;鼠抗GAPDH单克隆抗体PMK043F购自北京普瑞美斯生物科技有限公司;鼠二抗、兔二抗均购自北京博奥龙免疫技术有限公司;所用引物及sgRNA均由北京TSingKe公司(武汉)合成。
1.2 方法1.2.1 sgRNA重组质粒的构建 选定猪ALG5基因(Genbank:NC_010453.5)第二个外显子上两个合适的靶向位点,通过网站https://crispr.cos.uni-heidelberg.de/index.html?tdsourcetag=s_pctim_aiomsg设计两对sgRNA(表 1)。合成的sgRNA引物稀释至100 μmol·L-1进行退火杂交,退火程序为37℃ 30 min,95℃ 5min,以0.1 ℃·s-1的速度缓慢降温至25℃,退火引物稀释200倍。同时将LentiGuide-puro用BsmB Ⅰ在55℃条件下酶切30~60 min,将酶切产物进行琼脂糖凝胶电泳可观察到两条带,大小分别为2 000和8 000 bp左右,回收相对分子质量大的条带。回收产物与退火引物用Solution I于16 ℃水浴连接1~2 h。连接产物转化到DH5α感受态细胞中,经挑菌测序正确的样品使用无内毒素大量质粒提取试剂盒提取质粒。
|
|
表 1 相关引物序列 Table 1 Primer sequences |
1.2.2 ALG5基因敲低多克隆细胞系的构建 质粒按pMD2.G:psPAX2:重组质粒(LentiGuide-puro)=1:3:4的比例借助lipo 8000(1 μg质粒:2 μL lipo 8000)共转染入293T细胞中,72 h收取细胞上清,并用0.45 μm滤器过滤,-80 ℃储存。NPTr细胞于6孔板长至30%细胞密度时感染慢病毒,按照慢病毒原液:完全培养基(10%FBS的DMEM)=1:1的比例,加入终浓度为8 μg·mL-1的polybrene,24 h后更换新鲜完全培养基,长满单层后, 用含Puromycin的完全培养基进行筛选,存活的细胞扩大培养,冻存细胞并收取部分细胞样品。设计含有sgRNA靶向区域的基因组序列扩增引物(表 1)。将收取的细胞样品使用基因组DNA提取试剂盒提取基因组,用表 1中验证突变引物扩增含有gRNA的序列,凝胶电泳回收,胶回收产物与pMD-18T载体连接。将连接产物经转化、挑菌、测序,分析所插入的外源基因的序列是否在gRNA位置及附近发生插入、缺失或突变。
1.2.3 ALG5基因敲除单克隆细胞株的构建 将多克隆细胞系用有限稀释法挑选ALG5基因敲除单克隆细胞株,待单细胞孔中形成细胞集落,消化细胞转移至24孔板中扩大培养,最终将细胞冻存。收集野生型细胞和各个单克隆细胞株的细胞样,哺乳动物细胞裂解液裂解细胞,制成蛋白样品,用蛋白染色液G250测定蛋白浓度进行蛋白定量,通过SDS-PAGE电泳将蛋白转印至NC膜上,1%BSA室温封闭1 h,目的条带(37 ku)孵育兔抗ALG5(1:1 000),内参用鼠抗GAPDH(1:3 000),二抗分别孵育羊抗兔IgG抗体(1:5 000)和羊抗鼠IgG抗体(1:5 000),通过化学发光成像仪检测ALG5是否敲除。
1.2.4 细胞活力鉴定 96孔细胞板接入等细胞数的野生型细胞和基因敲除细胞,分别于12、24和36 h在避光条件下向每孔加入10 μL CCK-8试剂,37 ℃ CO2培养箱孵育1 h,用酶标仪测定在450 nmol·L-1处的吸光度。计算细胞存活率,细胞存活率=[(As-Ab)/(Ac-Ab)]×100%,As:试验孔(ALG5基因敲除细胞孔+CCK-8);Ac:对照孔(野生型细胞孔+CCK-8);Ab:空白孔(培养基+CCK-8)。
1.2.5 ALG5敲除对猪流感病毒复制的影响 将野生型细胞和基因敲除细胞铺于12孔细胞板长满单层,用无菌PBS洗两遍,将HB毒株以MOI=0.01的剂量感染细胞,37 ℃ CO2培养箱孵育1 h,弃去每孔病毒液,用无菌PBS洗两遍后, 换成0.25 μg·mL-1 TPCK的无血清DMEM培养基,分别于12、24和36 h收取上清和细胞样,细胞上清测定病毒的滴度并计算TCID50,细胞样通过Western blot检测流感病毒NP蛋白表达量的变化。
1.2.6 ALG5过表达对猪流感病毒复制的影响 将ALG5基因克隆到p3×Flag-cmv-14载体上:在NCBI上找到猪的ALG5基因的CDS区,用软件Primer 5设计引物(表 1)后通过PCR扩增、酶切、连接、转化、挑菌、测序、摇菌大提质粒后获得Flag-ALG5质粒。野生型细胞铺于12孔细胞板长至60%~80%的细胞密度时,用转染试剂lipo 8000将质粒Flag和Flag-ALG5(1.5 μg·孔-1)分别转入NPTr细胞中。转染后24 h感染HB毒株,分别于12、24和36 h收取上清和细胞样,测定病毒的滴度,并计算TCID50,Western blot检测NP蛋白表达量的变化。
1.2.7 SIV感染对内源性ALG5表达的影响 野生型NPTr细胞感染HB毒株,分别于0、6、12和24 h收取细胞样并进行蛋白定量,通过Western blot检测NP和内源性ALG5蛋白表达量的变化。
1.2.8 ALG5敲除对其他猪流感毒株复制的影响 F26毒株按MOI=0.01的剂量分别感染野生型与敲除细胞系,1 h后换成含0.25 μg·mL-1 TPCK的无血清DMEM培养基,36 h后收取上清,测定病毒的滴度并计算TCID50。
2 结果 2.1 ALG5基因敲低多克隆细胞系的构建为了验证sgRNA引物是否连接到了LentiGuide-puro载体上,笔者将重组质粒进行测序,结果显示(图 1A~B)两个ALG5-sgRNA均构建成功。为了初步验证设计的两对sgRNA的敲除效率,笔者将基因编辑质粒ALG5-sgRNA-1和ALG5-sgRNA-2分别进行慢病毒包装并感染NPTr细胞,Puromycin筛选出NPTr-ALG5-1和NPTr-ALG5-2细胞系,分别提取基因组用表 1验证突变引物进行PCR扩增、T克隆、转化后,分别挑取5个菌落进行测序。结果显示,NPTr-ALG5-2细胞系的单菌落没有检测到插入或缺失,而NPTr-ALG5-1细胞系的5个单菌落测序有4个发生了插入或缺失(图 1C)。因此,笔者选用NPTr-ALG5-1细胞系挑选ALG5基因敲除单克隆细胞株。

|
A.ALG5-sgRNA-1测序结果;B.ALG5-sgRNA-2测序结果(阴影部分为sgRNA序列);C.ALG5-sgRNA-1敲除效率检测 A. ALG5-sgRNA-1 sequencing results; B. ALG5-sgRNA-2 sequencing results (the shaded part is for sgRNA sequence); C. Detection of ALG5-sgRNA-1 knockout efficiency 图 1 ALG5基因敲低多克隆细胞系的构建 Fig. 1 Construction of ALG5 gene knockdown polyclonal cell line |
NPTr-ALG5-1细胞系通过有限稀释法筛选表达Puromycin的单克隆细胞株并进一步培养,筛选到了6株单克隆细胞,通过Western blot检测6株单克隆细胞的ALG5蛋白水平(图 2A)。结果显示,2号单克隆细胞株效果最好,并进一步检测到该株细胞ALG5完全敲除(图 2B),最终获得了NPTr细胞上ALG5基因敲除的单克隆细胞系ΔALG5。通过对敲除细胞系的细胞活性进行检测,结果显示,在12、24和36 h基因敲除细胞系与野生型细胞活力相当(图 2C),表明在NPTr细胞上敲除ALG5基因后对细胞的活力没有影响。

|
A. Western blot检测6株单克隆细胞株ALG5的敲除水平(框内为选择的2号单克隆细胞);B. Western blot进一步检测2号单克隆细胞中ALG5的敲除水平;C. ALG5基因敲除细胞与野生型细胞的细胞活性 A. Knockout levels of ALG5 in 6 monoclonal cell lines detected by Western blot (the box is the selected monoclonal cell number 2.); B. Knockout levels of ALG5 in monoclonal cell 2 were further detected by Western blot; C. Cellular viability of ALG5 knockout cells and wild-type cells 图 2 ALG5基因敲除单克隆细胞系的构建 Fig. 2 Construction of ALG5 gene knockout monoclonal cell line |
为了检测ALG5基因敲除是否影响了猪流感病毒的复制,笔者将HB毒株感染敲除细胞与野生型细胞后,12、24和36 h测定病毒的滴度(图 3A),同时检测NP的表达量(图 3B),结果表明,ALG5基因敲除能显著抑制猪流感病毒的复制,ALG5可能是猪流感病毒复制过程中的一个关键蛋白。

|
A.通过TCID50结果检测ALG5敲除后对HB毒株复制的影响(** P < 0.01,*** P < 0.001);B. Western blot检测ALG5敲除对NP表达量的影响;hpi.感染后时间 A. The effect of ALG5 knockout on replication of HB strains was tested by TCID50 results (** P < 0.01, *** P < 0.001); B. The Effect of ALG5 knockout on the expression level of NP detected by Western blot; hpi. Hours post infection 图 3 ALG5敲除对SIV复制的影响 Fig. 3 The Effect of ALG5 knockout on SIV replication |
为了进一步验证ALG5对病毒的增殖是否起正调控作用,笔者构建了ALG5的真核表达质粒,并且该质粒可以在NPTr细胞上表达(图 4A),将Flag空载和Flag-ALG5分别转染NPTr细胞,转染后24 h感染HB毒株,于12、24和36 h收取细胞上清和细胞样,测定病毒滴度计算TCID50(图 4B),同时检测NP蛋白的表达量(图 4C),结果表明,ALG5过表达促进了猪流感病毒的复制。

|
A. Western blot检测Flag-ALG5质粒在NPTr细胞上的表达;B.通过TCID50结果检测ALG5过表达对HB毒株复制的影响(* P < 0.05,*** P < 0.001);C.Western blot检测ALG5过表达对NP表达量的影响; hpi.感染后时间 A. The expression of Flag-ALG5 plasmid on NPTr cells was tested by Western blot; B. The effect of ALG5 overexpression on the replication of HB strains was tested by TCID50 results (* P < 0.05, *** P < 0.001); C. The effect of ALG5 overexpression on the expression level of NP was detected by Western blot; hpi. Hours post infection 图 4 ALG5过表达对HB毒株复制的影响 Fig. 4 The Effect of ALG5 overexpression on replication of HB strains |
之前的结果证明了ALG5敲除与过表达都影响了病毒的复制,为了验证病毒复制是否影响了ALG5的蛋白表达量,将野生型NPTr感染猪流感病毒后,通过Western blot检测内源性ALG5蛋白表达量的变化。结果表明(图 5),病毒感染后,伴随病毒的大量增殖,ALG5的蛋白表达量上调,病毒可能是通过调节ALG5的蛋白水平来促进自身的增殖。

|
图 5 HB毒株感染对内源性ALG5表达的影响 Fig. 5 The effect of HB strain infection on the expression of endogenous ALG5 |
为了验证ALG5敲除也能影响其他猪流感病毒株的复制,选用实验室保存的一株猪流感病毒株F26,将其分别感染野生型与敲除细胞系,测定36 h的病毒滴度并计算TCID50,结果表明(图 6),ALG5敲除可以抑制F26毒株的复制,无毒株特异性。
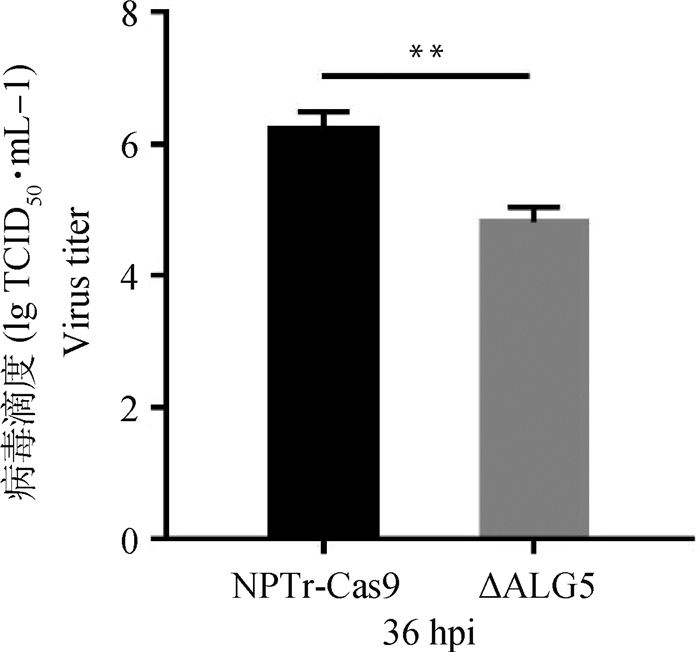
|
**. P < 0.01;hpi.感染后时间 **. P < 0.01; hpi. Hours post infection 图 6 ALG5敲除对猪流感毒株F26复制的影响 Fig. 6 The Effect of ALG5 knockout on replication of swine flu strain F26 |
本研究根据实验室前期全基因组文库筛选的结果,选择ALG5深入研究与猪流感病毒复制的关系。我们用CRISPR/Cas9基因编辑技术在稳定表达Cas9蛋白的NPTr细胞上建立了ALG5基因敲除细胞系,发现ALG5基因敲除并没有影响细胞活性。ALG5基因敲除抑制猪流感病毒复制,过表达促进猪流感病毒的复制,并且病毒感染后使细胞中ALG5的表达量上调,最后,证实了ALG5敲除也可以抑制猪流感病毒株F26的复制,无毒株特异性。
ALG5作为一个糖基化激酶,主要定位在内质网膜上,编码的蛋白质参与蛋白质的N-连接糖基化[22-24]。流感病毒的HA、NA和M2蛋白合成后的糖基化修饰对蛋白本身的功能意义重大[25-28]。猜想ALG5蛋白可能参与了病毒HA、NA或M2合成后的糖基化修饰过程,ALG5敲除后,病毒亚基的糖基化修饰过程受阻,蛋白质的折叠和成熟则会受到影响[29-31],内质网相关蛋白降解(ERAD)系统会将错误折叠的蛋白质降解[32-33],流感病毒的蛋白合成受阻,从而影响了病毒的增殖[34]。本研究证实了ALG5敲除也可以抑制猪流感毒株F26的复制,下一步要验证ALG5基因敲除是否也可以影响其他流感毒株的复制,并深入研究ALG5的糖基化功能,找到ALG5发挥糖基化功能的结构域,同时分析HA、NA和M2的糖基化修饰是否与ALG5有关,进一步找到猪流感病毒亚基上的糖基化位点,阐明ALG5影响猪流感病毒复制的具体机制。
猪流感具有高度传染性,同时,猪作为流感病毒“混合器”能够不断产生新型重配病毒,增加人感染流感的可能性,因此,预防和控制猪流感病毒的流行具有重要的意义[35-37]。本研究利用当前流行的CRISPR/Cas9基因编辑技术验证了ALG5基因敲除对猪流感病毒复制的影响,发现了ALG5是猪流感病毒复制过程中的一个重要宿主因子,为研究猪流感病毒的复制过程和致病机制奠定了基础。
4 结论根据实验室前期PK-15-GeCKO全基因组文库筛选的结果,研究ALG5与SIV复制的关系。结果显示,ALG5敲除的NPTr单克隆细胞系与野生型细胞的细胞活性无显著差异,ALG5基因敲除显著抑制SIV的增殖,过表达促进SIV的增殖,SIV感染NPTr使ALG5的蛋白表达量上调,表明ALG5是猪流感病毒复制过程一个重要的正调控蛋白。ALG5基因敲除后,也可以抑制猪流感病毒株F26的复制,无毒株特异性。
| [1] | MORENS D M, TAUBENBERGER J K. A possible outbreak of swine influenza, 1892[J]. Lancet Infect Dis, 2014, 14(2): 169–172. |
| [2] | GHEDIN E, SENGAMALAY N A, SHUMWAY M, et al. Large-scale sequencing of human influenza reveals the dynamic nature of viral genome evolution[J]. Nature, 2005, 437(7062): 1162–1166. DOI: 10.1038/nature04239 |
| [3] | MCHARDY A C, ADAMS B. The role of genomics in tracking the evolution of influenza A virus[J]. PLoS Pathog, 2009, 5(10): e1000566. DOI: 10.1371/journal.ppat.1000566 |
| [4] | NELSON M I, WOROBEY M. Origins of the 1918 pandemic:revisiting the swine "Mixing Vessel" hypothesis[J]. Am J Epidemiol, 2018, 187(12): 2498–2502. DOI: 10.1093/aje/kwy150 |
| [5] | WANG C, FORST C V, CHOU T W, et al. Cell-to-cell variation in defective virus expression and effects on host responses during influenza virus infection[J]. mBio, 2020, 11(1): e02880–19. |
| [6] | HALLER O, KOCHS G. Mx genes:host determinants controlling influenza virus infection and trans-species transmission[J]. Hum Genet, 2019. DOI: 10.1007/s00439-019-02092-8 |
| [7] | DOU D, REVOL R, ÖSTBYE H, et al. Influenza a virus cell entry, replication, virion assembly and movement[J]. Front Immunol, 2018, 9: 1581. DOI: 10.3389/fimmu.2018.01581 |
| [8] | KÖNIG R, STERTZ S, ZHOU Y Y, et al. Human host factors required for influenza virus replication[J]. Nature, 2010, 463(7282): 813–817. DOI: 10.1038/nature08699 |
| [9] | GAMBLIN S J, SKEHEL J J. Influenza hemagglutinin and neuraminidase membrane glycoproteins[J]. J Biol Chem, 2010, 285(37): 28403–28409. DOI: 10.1074/jbc.R110.129809 |
| [10] | JIANG F G, DOUDNA J A. CRISPR-Cas9 structures and mechanisms[J]. Annu Rev Biophys, 2017, 46: 505–529. DOI: 10.1146/annurev-biophys-062215-010822 |
| [11] | LI B, CLOHISEY S M, CHIA B S, et al. Genome-wide CRISPR screen identifies host dependency factors for influenza A virus infection[J]. Nat Commun, 2020, 11(1): 164. DOI: 10.1038/s41467-019-13965-x |
| [12] | PENG A H, CHEN S C, LEI T G, et al. Engineering canker-resistant plants through CRISPR/Cas9-targeted editing of the susceptibility gene CsLOB1 promoter in citrus[J]. Plant Biotechnol J, 2017, 15(12): 1509–1519. DOI: 10.1111/pbi.12733 |
| [13] | XIE Z C, PANG D X, YUAN H M, et al. Genetically modified pigs are protected from classical swine fever virus[J]. PLoS Pathog, 2018, 14(12): e1007193. DOI: 10.1371/journal.ppat.1007193 |
| [14] | XIE Z C, JIAO H P, XIAO H N, et al. Generation of pRSAD2 gene knock-in pig via CRISPR/Cas9 technology[J]. Antiviral Res, 2020, 174: 104696. DOI: 10.1016/j.antiviral.2019.104696 |
| [15] | BURKARD C, LILLICO S G, REID E, et al. Precision engineering for PRRSV resistance in pigs:macrophages from genome edited pigs lacking CD163 SRCR5 domain are fully resistant to both PRRSV genotypes while maintaining biological function[J]. PLoS Pathog, 2017, 13(2): e1006206. DOI: 10.1371/journal.ppat.1006206 |
| [16] | HAN J L N, PEREZ J T, CHEN C, et al. Genome-wide CRISPR/Cas9 screen identifies host factors essential for influenza virus replication[J]. Cell Rep, 2018, 23(2): 596–607. DOI: 10.1016/j.celrep.2018.03.045 |
| [17] | HEATON B E, KENNEDY E M, DUMM R E, et al. A CRISPR activation screen identifies a pan-avian influenza virus inhibitory host factor[J]. Cell Rep, 2017, 20(7): 1503–1512. DOI: 10.1016/j.celrep.2017.07.060 |
| [18] | PALAMARCZYK G, DRAKE R, HALEY B, et al. Evidence that the synthesis of glucosylphosphodolichol in yeast involves a 35-kDa membrane protein[J]. Proc Natl Acad Sci U S A, 1990, 87(7): 2666–2670. DOI: 10.1073/pnas.87.7.2666 |
| [19] | LOMBARD J. The multiple evolutionary origins of the eukaryotic N-glycosylation pathway[J]. Biol Direct, 2016, 11: 36. DOI: 10.1186/s13062-016-0137-2 |
| [20] | WU N C, WILSON I A. A perspective on the structural and functional constraints for immune evasion:insights from influenza virus[J]. J Mol Biol, 2017, 429(17): 2694–2709. DOI: 10.1016/j.jmb.2017.06.015 |
| [21] | MAURER M A, MEYER L, BIANCHI M, et al. Glycosylation of human IgA directly inhibits influenza a and other sialic-acid-binding viruses[J]. Cell Rep, 2018, 23(1): 90–99. |
| [22] | HAECKER A, BERGMAN M, NEUPERT C, et al. Wollknäuel is required for embryo patterning and encodes the Drosophila ALG5 UDP-glucose:dolichyl-phosphate glucosyltransferase[J]. Development, 2008, 135(10): 1745–1749. DOI: 10.1242/dev.020891 |
| [23] | SHAIK K S, PABST M, SCHWARZ H, et al. The Alg5 ortholog Wollknöuel is essential for correct epidermal differentiation during Drosophila late embryogenesis[J]. Glycobiology, 2011, 21(6): 743–756. DOI: 10.1093/glycob/cwq213 |
| [24] | SINGH Y, WANG T H, GERINGER S A, et al. Regenerative glycosylation[J]. J Org Chem, 2018, 83(1): 374–381. DOI: 10.1021/acs.joc.7b02768 |
| [25] | YORK I A, STEVENS J, ALYMOVA I V. Influenza virus N-linked glycosylation and innate immunity[J]. Biosci Rep, 2019, 39(1): BSR20171505. DOI: 10.1042/BSR20171505 |
| [26] | WU C Y, LIN C W, TSAI T I, et al. Influenza A surface glycosylation and vaccine design[J]. Proc Natl Acad Sci U S A, 2017, 114(2): 280–285. DOI: 10.1073/pnas.1617174114 |
| [27] | ZHAO D M, LIANG L B, WANG S, et al. Glycosylation of the hemagglutinin protein of H5N1 influenza virus increases its virulence in mice by exacerbating the host immune response[J]. J Virol, 2017, 91(7): e02215–16. |
| [28] | DE GREEF J C, SLVTTER B, ANDERSON M E, et al. Protective role for the N-terminal domain of α-dystroglycan in influenza A virus proliferation[J]. Proc Natl Acad Sci U S A, 2019, 116(23): 11396–11401. DOI: 10.1073/pnas.1904493116 |
| [29] | KOSIK I, INCE W L, GENTLES L E, et al. Correction:influenza A virus hemagglutinin glycosylation compensates for antibody escape fitness costs[J]. PLoS Pathog, 2018, 14(6): e1007141. DOI: 10.1371/journal.ppat.1007141 |
| [30] | CHEREPANOVA N, SHRIMAL S, GILMORE R. N-linked glycosylation and homeostasis of the endoplasmic reticulum[J]. Curr Opin Cell Biol, 2016, 41: 57–65. DOI: 10.1016/j.ceb.2016.03.021 |
| [31] | HAJBA L, SZEKRÉNYES A, BORZA B, et al. On the glycosylation aspects of biosimilarity[J]. Drug Discov Today, 2018, 23(3): 616–625. DOI: 10.1016/j.drudis.2018.01.009 |
| [32] | ALBERT S, WIETRZYNSKI W, LEE C W, et al. Direct visualization of degradation microcompartments at the ER membrane[J]. Proc Natl Acad Sci U S A, 2020, 117(2): 1069–1080. DOI: 10.1073/pnas.1905641117 |
| [33] | MA W R, MAYR C. A membraneless organelle associated with the endoplasmic reticulum enables 3'UTR-mediated protein-protein interactions[J]. Cell, 2018, 175(6): 1492–1506. DOI: 10.1016/j.cell.2018.10.007 |
| [34] | LIU W L, BAI T, GUO J L, et al. The S128N mutation combined with an additional potential N-linked glycosylation site at residue 133 in hemagglutinin affects the antigenicity of the human H7N9 virus[J]. Emerg Microbes Infect, 2016, 5: e66. |
| [35] | DADONAITE B, GILBERTSON B, KNIGHT M L, et al. The structure of the influenza A virus genome[J]. Nat Microbiol, 2019, 4(11): 1781–1789. DOI: 10.1038/s41564-019-0513-7 |
| [36] | RAJAO D S, VINCENT A L, PEREZ D R. Adaptation of human influenza viruses to swine[J]. Front Vet Sci, 2019, 5: 347. DOI: 10.3389/fvets.2018.00347 |
| [37] | ANDREASEN V, GOG J R. Pease (1987):The evolutionary epidemiology of influenza A[J]. Theor Popul Biol, 2020. DOI: 10.1016/j.tpb.2019.12.006 |



