2. 西藏农牧科学院畜牧兽医研究所, 拉萨 850009;
3. 重庆市长寿区畜牧兽医局品种改良站, 重庆 401220
2. Institute of Animal Science, Tibet Academy of Agricultural & Animal Husbandry Science, Lhasa 850009, China;
3. Breed Improvement Station, Animal Husbandry & Veterinary Bureau of Changshou District, Chongqing 401220, China
藏山羊(Capra hircus)是中国最古老的羊品种之一,最早记录于公元前3 300~2 000年[1]。由于长期以来所处的复杂地形、多样的海拔高度和极端的气候环境等生态条件,藏山羊具备了独特的低氧适应遗传特性[2]。相比之下,低海拔山羊及其他物种如果处于低氧环境中,由于氧供应不足会产生高原反应,甚至导致心力衰竭[3]。最早关于藏山羊高原适应性的开拓性研究始于二十年前,主要通过解剖、生化和生理层面去理解[4-5]。与低海拔山羊相比,藏山羊的血红蛋白(Hb)浓度较高,心和肺体积较大,心率较低[6],表明藏山羊已经很好地适应了高海拔地区适应解剖和生理特征的进化。至今为止,高海拔适应性的遗传分子机制仍有很多未知。通过研究藏山羊,可以为低氧适应和缺氧相关疾病的遗传机制研究提供参考[7]。
研究表明,动物线粒体DNA(mitochondrial DNA, mtDNA)对自然选择敏感,与环境适应性相关[8-9]。目前,mtDNA多态性与高原适应性的关联研究已经在许多物种中进行了探索,包括人[10]、草原毛虫[11]、藏猪[12]、骆驼[13]、鸡[8]、牛[14-15]和藏绵羊[16]等。近年来,通过全基因组,mtDNA标记和微卫星标记研究表明,藏山羊与低海拔绒山羊群体产生了高水平遗传分化[17-20]。mtDNA基因编码区能合成13个多肽,这些多肽与5个大型多亚基复合物(complexes I-V)一起参与了氧化磷酸化反应(oxidative phosphorylation, OXPHOS)[21],从而产生ATP供能并维持体温[22]。由于OXPHOS是能量代谢必不可少的保守机制,因此纯化选择主导着mtDNA的进化。值得注意的是,尽管mtDNA基因编码区处于严格的纯化选择中,但分子的适应性仍然可能导致保守区域产生非同义突变,从而使转录的氨基酸理化性质发生根本变化[23-25]。
mtDNA基因ATP6(ATP synthase F0 subunit 6)和Cytb(cytochrome b)均参与OXPHOS反应。由ATP6编码的ATP合成酶亚单位6在跨膜的质子梯度存在下由ADP产生ATP,质子梯度是由呼吸链的电子转运复合物产生的,而Cytb编码的细胞色素b参与呼吸链,产生与ATP合成偶联的电化学势。mtDNA基因中,ATP6与Cytb基因对自然选择最为敏感[26],关键部位氨基酸置换通常会改变蛋白质功能[27]。Wang等[28]检测到ATP6和Cytb序列的阳性选择位点与藏泥鳅在极端环境中的适应性相关。目前,在ATP6和Cytb中发现了多个氨基酸变化,它们改变了氧合反应效率,表明自然选择如气候[26]等在形成mtDNA多态性中具有重要作用。基于此推测,不同于低海拔山羊群体,藏山羊OXPHOS过程具有潜在的高海拔环境适应性突变。
本研究通过对藏山羊(Tibetan goat, TG)与低海拔山羊(lowland goat, LG)261条ATP6和Cytb核苷酸组成、遗传多样性、单倍型网络图以及蛋白结构域功能影响的比较分析,探究藏山羊高海拔适应性的分子机制,将增加对动物缺氧适应性机制的了解。
1 材料与方法 1.1 样本采集根据藏山羊的主要分布地区,从西藏8个地区采集藏山羊血样组织共计157个(平均海拔>3 500 m,设为TG,表 1)。使用颈静脉采血方法,3.9%ACD抗凝,随后放置-20 ℃冰箱冻存。
|
|
表 1 藏山羊样品采集信息 Table 1 Sample information of Tibetan goats |
1.2.1 基因组DNA提取、ATP6和Cytb基因序列扩增及测序 采用DNA提取试剂盒(TIANamp Genomic DNA Kit,北京)提取基因组DNA。基于藏山羊线粒体基因组参考序列(序列号:KJ940969),使用Primer Premier 5.0软件设计以下两对引物扩增mtDNA基因ATP6和Cytb序列:ATP6,AF:5′-GATATGCCACAACTAGACA-3′,AR:5′-CGGCTGGAGTATGATGACC-3′;Cytb,CF:5′-AATAGGCGAAGGTTTTGAA-3′,CR:5′-GCTTTGGGTGCTGATAGTG-3′。本研究中,PCR反应体系总体积为30 μL:10×缓冲液15 μL,DNA(2.5 ng·μL-1)1 μL,正、反向引物(10 pmol·μL-1)各1 μL,超纯水12 μL。PCR扩增条件均为94℃预变性5 min,35个循环(95 ℃ 30 s,55.3 ℃ 30 s,72 ℃ 90 s),72 ℃延伸10 min,4 ℃保存。PCR产物经1.5%琼脂糖凝胶进行电泳检测后直接进行Sanger测序(成都擎科生物有限公司)。为保证序列测定的准确性,采用正反双向测序。
1.2.2 ATP6和Cytb基因序列突变位点鉴定 原始基因序列结果由DNAstar Software(DNAstar Inc. Madison,WI,USA)编辑和比对。借鉴相关研究分组方法[29],从GenBank下载了104条来自欧亚大陆的低海拔山羊群体Cytb和ATP6序列(平均海拔 < 1 000 m,设为LG,表 2)。使用MEGA 7.0[30]鉴定序列变异位点,计算核苷酸组成。以山羊mtDNA基因组(登录号:KR059146)为参考序列。将所有序列进行多重比对后,使用DnaSP v5软件定义单倍型种类[31]。为了更直观的观察单倍型间多态位点的连接关系,使用NETWORK v4.611[32]进行单倍型网络图绘制(分析单倍型使用的参考序列号:KR059210)。
|
|
表 2 低海拔山羊线粒体基因组序列信息 Table 2 The information of mitogenome sequences for the low-altitude goats |
1.2.3 ATP6和Cytb基因突变的功能分析 通过KEGG通路分析(http://www.genome.jp/kegg-bin/show_pathway)ATP6和Cytb氧化磷酸化途径功能关系。SMART程序(http://smart.embl-heidelberg.de/)用于模拟ATP6和Cytb的蛋白结构域二级结构及突变位置。
1.2.4 数据统计分析 使用Mito工具(http://www.mitotool.org/index.html)进行Fisher检验并采用Bonferroni方法进行校正,同时得到95%置信区间(95%CI)的比值比(OR)。所有参数值的显著水平采用P < 0.05表示,极显著水平采用P < 0.01表示。
2 结果 2.1 ATP6和Cytb序列变异和组成及山羊组间多样性经过比对及人工校准,山羊ATP6和Cytb序列全长分别为681和1 140 bp。两个基因的(A+T)%组成均超过50%,TG和LG两组之间碱基组成差异不明显。在ATP6中鉴定出33个单核苷酸多态性位点(SNPs),在Cytb中鉴定出67个SNPs,两个基因中均未检测到碱基的插入或缺失。在ATP6和Cytb两个基因中,LG单倍型种类均高于TG,可能是由于低海拔山羊品种较多导致。ATP6中,LG和TG平均核苷酸差异数(K)组间差异较大,相反Cytb中,LG和TG平均核苷酸差异数(K)组间差异较小。单倍型多样性(Hd)和核苷酸多样性(π)结果表明,ATP6和Cytb总体遗传多样性接近,但低频多态性过多(表 3)。为了检测ATP6和Cytb自然选择印记,本研究对所有藏山羊及下载序列中性检验的参数进行计算(Tajima’s D、Fu and Li’s D*、Fu and Li’s F*、Fu’s Fs)。极显著的负值Tajima’s D(ATP6,-2.472 3; Cytb,-2.515 2)和Fu’s Fs(ATP6,-51.736; Cytb,-92.427)指数表明,在山羊的驯养历史中偏离中性假说,受到了强烈的自然选择(表 4)。
|
|
表 3 藏山羊和低海拔山羊ATP6和Cytb序列变异及多样性指数 Table 3 Sequence variations and the diversity indexes of the ATP6 and Cytb genes of Tibetan goats and low-altitude goats |
|
|
表 4 山羊261条ATP6和Cytb序列的中性检验参数值 Table 4 Neutrality test parameters of 261 sequences of goats ATP6 and Cytb genes |
为了准确筛选出非同义突变,本研究在忽略样本数小于2的错义突变条件下,在ATP6和Cytb中均分别鉴定出6个非同义突变(表 5)。其中,ATP6基因的m.7974G>A只在TG出现,m.8102A>G和m.8348G>A只在LG出现,m.8055G>A,m.8113A>G和m.8476G>A同时在两组中出现;Cytb中5个非同义突变(m.14474A>C,m.14864T> G,m.14867T>A,m.14887A>C和m.14968T>G)为TG特有,m.14794A>G为LG特有。进一步分析发现,ATP6和Cytb基因各有一个错义突变位点,即ATP6 m.8102A>G(P=0.000 6)和Cytb m.14794A>G(P=0.007 7),在TG和LG两组间差异极显著。
|
|
表 5 藏山羊和低海拔山羊ATP6和Cytb序列错义突变位点 Table 5 Missense mutation sites in ATP6 and Cytb genes of Tibetan goats and low-altitude goats |
2.3.1 单倍型种类 在共计261条ATP6序列(157只藏山羊和104只低海拔山羊)中,定义了32种单倍型(H1~H32)。LG包括单倍型H17~H32,TG包括单倍型H2~H5、H8~H9和H11~H16。H1作为最大的共享单倍型,由119只藏山羊和73只低海拔山羊序列构成。同样,261条Cytb序列定义62种单倍型(Ha1~Ha62),LG包括37种单倍型(Ha1~Ha19、Ha21~Ha22、Ha24~Ha36和Ha38~Ha40),TG包括22种特有单倍型(Ha41~Ha62),其余为TG和LG共享单倍型。
2.3.2 单倍型网络图 在ATP6单倍型网络图中(图 1),主单倍型H1为中心点,呈星形向外连接到其他单倍型,然后通过参考序列(序列号:KR059210)与单倍型H10和H27连接。在Cytb单倍型中位连接图中(图 2),主单倍型Ha2为中心点,呈星型向外连接。在两个基因单倍型网络图中都发现一些中间单倍型的缺失。这些缺失的单倍型可能是现存的但没有被抽样,或已灭绝。单倍型网络图结果显示,绝大部分Cytb和ATP6序列在LG和TG群体中为独享单倍型,表明通过极端海拔高度(>3 500 m)分组能较好的分开这两组群体。值得注意的是,Ha2和Ha36之间存在5个SNPs(14460、14794、15219、15003和14745),结合上述错义突变差异显著性分析认为,Cytb基因的多态位点m.14794可能是形成与高原适应性负相关单倍型Ha36的关键位点。
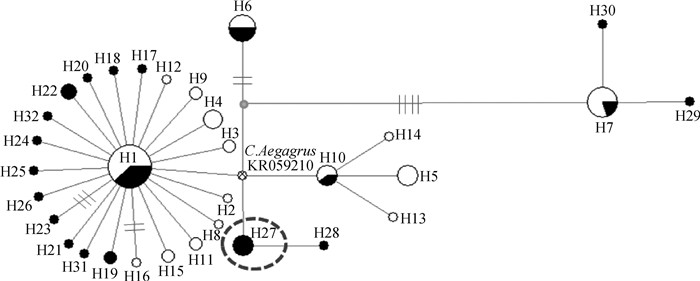
|
藏山羊和低海拔山羊分别用白色和黑色表示,网格圆圈表示野羊参考序列。下同 Tibetan goats and low-altitude goats are represented with white and black circles, respectively. Mesh circular indicates that the reference sequence of C.Aegagrus. The same as below 图 1 ATP6单倍型网络图 Fig. 1 Median-joining network of ATP6 haplotypes |

|
图 2 Cytb单倍体网络图 Fig. 2 Median-joining network of Cytb haplotypes |
2.3.3 单倍型种类与高原适应性关联分析 根据分析,LG特有的ATP6 m.8102A>G(P=0.000 6)和Cytb m.14794A>G(P=0.007 7)组间差异显著。m.8102A>G位点是形成ATP6单倍型H27的关键位点。通过Bonferroni校正检查显著性水平后,ATP6单倍型H27与高适应性进化呈显著负相关(P=0.007 0,表 6)。同样,Cytb单倍型Ha2和Ha37与高海拔适应性呈显著正相关(Ha2,P=0.018 0;Ha37,P=0.001 7;表 6),Ha36与高海拔适应性(P=0.012 3)和错义突变呈负相关,而m.14794A>G是形成单倍型Ha36的关键位点。
|
|
表 6 ATP6和Cytb单倍型分布和Fisher精确检验 Table 6 Haplotype distribution and the Fisher's exact test for ATP6 and Cytb genes |
mtDNA基因ATP6和Cytb参与OXPHOS反应。由ATP6编码的ATP合成酶在跨膜的质子梯度存在下由ADP产生ATP,质子梯度是由呼吸链的电子转运复合物产生的。Cytb参与呼吸链,产生与ATP合成偶联的电化学势。因此,这两个基因的功能改变可能会影响个体的呼吸效率。如图 3A所示,ATP6有3个错义突变(m.7974、m.8348和m.8476)位于跨膜区域,m.8102A>G(Ile→Met,P=0.000 6)位于线粒体膜外部。Cytb的4个错义突变(m.14474、m.14864、m.14867和m.14887)位于跨膜区域(图 3B),m.14794A>G,(Thr→Ala,P=0.007 7)位于线粒体膜内部。

|
箭头表示错义突变在跨膜区域的位置 The arrows indicate the locations of missense mutation distributed in the transmembrane regions 图 3 使用SMART程序预测的ATP6(A)和Cytb(B)蛋白跨膜结构图 Fig. 3 Diagram of transmembrane structure of the ATP6 (A) and Cytb (B) proteins predicted using the SMART program |
藏山羊是适应高海拔环境的重要家畜之一,不仅为青藏高原的原住民和游牧民族提供食物,还起到运输的作用[33]。相反,低地山羊处在相同的高海拔环境时,会产生严重的肺动脉高压反应。通过藏山羊和中国低海拔山羊群体间的外显子组测序比较发现,藏山羊群体与心血管发育相关的基因有明显富集[34]。低氧是高海拔地区的主要特征之一[35],动物体内进行能量代谢主要是由线粒体利用氧气,进行三磷酸腺苷几乎所有的化合反应。研究发现,线粒体基因与动物的高原适应性之间密切相关[11-16]。
本研究发现,TG和LG两组间ATP6和Cytb序列的碱基组成无明显差异,与其它物种线粒体基因序列组成相似[15, 35]。ATP6和Cytb序列在TG和LG两组间的核苷酸多样性和单倍型多样性差异不显著,遗传多样性均低于藏山羊mtDNA D-loop遗传多样性[33],可能原因是mtDNA D-loop突变率更高[36]。中性检验参数结果表明,ATP6和Cytb序列在山羊驯化过程中偏离了中性假说,经历了纯化选择,进而更好的适应了外界环境。本研究在261条 ATP6中发现了33个SNPs,在Cytb中发现了67个SNPs。在ATP6中检测到6个错义突变,其中m.7974G>A只在TG出现,m.8102A>G和m.8348G>A只在LG出现。在Cytb中检测到6个错义突变,m.14794A>G为LG特有,其余5个均为TG特有。部分特有突变尽管没有显示出统计学差异,但仍然值得关注,它们可能对山羊群体到达高原后的适应性进化产生了一定影响。通过ATP6和Cytb单倍型种类和网络图分析发现,TG和LG通过主单倍型呈星状向外发散,共享单倍型极少,有许多中间单倍型未出现在采样或下载序列中,可能是由于采样不完整或该单倍型已经消失,此前研究表明,藏山羊群体间的遗传结构差异正在缩小[37],启发研究者们应加强对藏山羊种质资源的保护,关注山羊保种工作。
在高海拔适应性相关研究中发现,处于不同海拔高度群体的单倍型存在明显差异[38-39],因此本研究中,在高海拔群体中发现的特有单倍型可能是与高原适应性相关的山羊类型。通过对ATP6和Cytb二级结构域模拟,本研究找到了ATP6和Cytb错义突变对应的跨膜区域,并推测这些位点在蛋白结构和功能产生的影响。TG中特有的5个错义突变(m.7974、m.14474、m.14864、m.14867和m.14887)位于跨膜区,可能改变了Cytb和ATP6合成的蛋白质,改变了合成氧的能力,进而在高海拔缺氧环境中起到了关键作用。王召锋[40]利用牦牛及其近缘北美野牛的线粒体蛋白编码序列对其适应性进化分析后发现,牦牛线粒体蛋白跨膜螺旋区域中苏氨酸残基明显增加,其有利于提高氧气结合效率以及有氧呼吸效率,从而更好的适应青藏高原的高海拔环境。
值得注意的是,LG组特异性突变ATP6 m.8102A>G(Ile→Met)和Cytb m.14794A>G(Thr→ Ala)是形成单倍型H27和Ha36的关键位点。在中新世晚期到晚中新世早期,青藏高原发生了快速的隆升,海拔接近于现代的高度,其生态环境也急速向高海拔的极端生态环境发展[41]。此时,早期的山羊在高海拔环境中,具有m.8102A>G和m.14794A>G等位基因A比具有等位基因G的个体具有更好的适应能力,这可能是高原型藏山羊进化的重要支点,为适应寒冷和干旱的瘠薄生存环境提供了遗传基础,并在高原上快速发展、演化。基于以上结论,认为这些错义突变会改变ATP6和Cytb的蛋白结构域,并影响山羊体内H+和e-的转运[42],进而导致不同的呼吸效率,逐渐形成现代具有不同高原适应能力的山羊品种。通过对高原土著动物适应性进化的系统研究,可以为培育耐低温、低氧动物新品种提供理论基础,也为临床预防和治疗高原性疾病提供一些思路,更为藏区家畜生产性能提供技术支撑[7],本研究初步推测了藏山羊高海拔低氧适应性机制,为高原适应性相关研究提供了一定的基础。
4 结论本研究从基因序列变异到功能分析,初步揭示mtDNA基因ATP6和Cytb的功能性突变可能是藏山羊高海拔低氧适应的机制之一,后续应该进一步提供相关的试验结果以揭示这些突变的重要功能。
| [1] | CHANG K C, XU P F, ALLAN S, et al. The formation of Chinese civilization:an archaeological perspective[M]. New Haven, CT: Yale University Press, 2005. |
| [2] | WANG Y, WANG J, ZI X D, et al. Genetic diversity of Tibetan goats of Plateau type using microsatellite markers[J]. Arch Tierzucht, 2011, 54(2): 188–197. |
| [3] | PEACOCK A J. ABC of oxygen:oxygen at high altitude[J]. BMJ, 1998, 317(7165): 1063–1066. DOI: 10.1136/bmj.317.7165.1063 |
| [4] |
江家椿, 何玛利, 嘎玛仁增, 等. 不同海拔高度西藏高原山羊若干血液生理特性的对比分析[J]. 西南农业学报, 1992(1): 79–83.
JIANG J C, HE M L, GAMA R Z, et al. Comparison on several hematologic value of goat in Tibet Plateau at different altitude[J]. Southwest China Journal of Agricultural Sciences, 1992(1): 79–83. (in Chinese) |
| [5] |
欧阳熙, 王杰, 王永, 等. 藏山羊血液生理、生化指标的季节性变化[J]. 西南民族学院学报:自然科学版, 1992, 18(3): 284–288.
OUYANG X, WANG J, WANG Y, et al. Seasonal variety of blood physiological and biochemical targetsof Tibetan goat[J]. Journal of Southwest Nationalities College:Natural Science, 1992, 18(3): 284–288. (in Chinese) |
| [6] |
王杰, 王永, 欧阳熙, 等. 藏山羊种质特性研究[J]. 西南民族学院学报:自然科学版, 1995, 21(3): 272–282.
WANG J, WANG Y, OUYANG X, et al. A study on breeding characters of Tibetan coats[J]. Journal of Southwest Nationalities College:Natural Science Edition, 1995, 21(3): 272–282. (in Chinese) |
| [7] |
张剑搏, 丁学智, AHMADAli Ahmad, 等. 高原土著动物适应性进化的研究进展[J]. 畜牧兽医学报, 2019, 50(9): 1723–1736.
ZHANG J B, DING X Z, AHMAD A A, et al. Advances in research on adaptive evolution of native animals of Tibetan Plateau[J]. Acta Veterinaria et Zootechnica Sinica, 2019, 50(9): 1723–1736. (in Chinese) |
| [8] | KHARRATI-KOOPAEE H, EBRAHIMIE E, DADPASAND M, et al. Genomic analysis reveals variant association with high altitude adaptation in native chickens[J]. Sci Rep, 2019, 9: 9224. DOI: 10.1038/s41598-019-45661-7 |
| [9] | SHEN X J, PU Z Q, CHEN X, et al. Convergent evolution of mitochondrial genes in deep-sea fishes[J]. Front Genet, 2019, 10: 925. DOI: 10.3389/fgene.2019.00925 |
| [10] | KANG L L, ZHENG H X, ZHANG M H, et al. MtDNA analysis reveals enriched pathogenic mutations in Tibetan highlanders[J]. Sci Rep, 2016, 6(1): 31083. DOI: 10.1038/srep31083 |
| [11] | YUAN M L, ZHANG Q L, ZHANG L, et al. Mitochondrial phylogeny, divergence history andhigh-altitude adaptation of grassland caterpillars (Lepidoptera:Lymantriinae:Gynaephora) inhabiting the Tibetan Plateau[J]. Mol Phylogenet Evol, 2018, 122: 116–124. DOI: 10.1016/j.ympev.2018.01.016 |
| [12] | LI M Z, JIN L, MA J D, et al. Detecting mitochondrial signatures of selection in wild Tibetan pigs and domesticated pigs[J]. Mitochondrial DNA A DNA Mapp Seq Anal, 2016, 27(1): 747–752. DOI: 10.3109/19401736.2014.913169 |
| [13] | MOHANDESAN E, FITAK R R, CORANDER J, et al. Mitogenome sequencing in the genus Camelus reveals evidence for purifying selection and long-term divergence between wild and domestic Bactrian camels[J]. Sci Rep, 2017, 7(1): 9970. DOI: 10.1038/s41598-017-08995-8 |
| [14] | GAO Y H, GAUTIER M, DING X D, et al. Species composition and environmental adaptation of indigenous Chinese cattle[J]. Sci Rep, 2017, 7(1): 16196. |
| [15] | SHI Y, HU Y S, WANG J, et al. Genetic diversities of MT-ND1 and MT-ND2 genes are associated with high-altitude adaptation in yak[J]. Mitochondrial DNA A, 2018, 29(3): 485–494. DOI: 10.1080/24701394.2017.1307976 |
| [16] | NIU L L, CHEN X Y, XIAO P, et al. Detecting signatures of selection within the Tibetan sheep mitochondrial genome[J]. Mitochondrial DNA A, 2017, 28(6): 801–809. DOI: 10.1080/24701394.2016.1192614 |
| [17] | GUO J Z, ZHONG J, LI L, et al. Comparative genome analyses reveal the unique genetic composition andselection signals underlying the phenotypic characteristics of three Chinese domestic goat breeds[J]. Genet Sel Evol, 2019, 51: 70. DOI: 10.1186/s12711-019-0512-4 |
| [18] | WU Y P, GUAN W J, ZHAO Q J, et al. A fine map for maternal lineage analysis by mitochondrial hypervariable region in 12 Chinese goat breeds[J]. Anim Sci J, 2009, 80(4): 372–380. DOI: 10.1111/j.1740-0929.2009.00659.x |
| [19] | ZHAO Y J, ZHAO R Z, ZHAO Z Q, et al. Genetic diversity and molecular phylogeography of Chinese domestic goats by large-scale mitochondrial DNA analysis[J]. Mol Biol Rep, 2014, 41(6): 3695–3704. DOI: 10.1007/s11033-014-3234-2 |
| [20] | DI R, FARHAD V S M, MA Y H, et al. Microsatellite analysis revealed genetic diversity and population structure among Chinese cashmere goats[J]. Anim Genet, 2011, 42(4): 428–431. |
| [21] | SCHEFFLER I E. Mitochondria make a come back[J]. Adv Drug Deliv Rev, 2001, 49(1-2): 3–26. DOI: 10.1016/S0169-409X(01)00123-5 |
| [22] | ANDREU A L, BRUNO C, HADJIGEORGIOU G M, et al. Polymorphic variants in the human mitochondrial cytochromebgene[J]. Mol Genet Metab, 1999, 67(1): 49–52. DOI: 10.1006/mgme.1999.2843 |
| [23] | DA FONSECA R R, JOHNSON W E, O'BRIEN S J, et al. The adaptive evolution of the mammalian mitochondrial genome[J]. BMC Genomics, 2008, 9(1): 119. DOI: 10.1186/1471-2164-9-119 |
| [24] | SHEN Y Y, LIANG L, ZHU Z H, et al. Adaptive evolution of energy metabolism genes and the origin of flight in bats[J]. Proc Natl Acad Sci USA, 2010, 107(19): 8666–8671. DOI: 10.1073/pnas.0912613107 |
| [25] | CHRISTIE J R, BEEKMAN M. Uniparental inheritance promotes adaptive evolution in cytoplasmic genomes[J]. Mol Biol Evol, 2017, 34(3): 677–691. |
| [26] | MISHMAR D, RUIZ-PESINI E, GOLIK P, et al. Natural selection shaped regional mtDNA variation in humans[J]. Proc Natl Acad Sci USA, 2003, 100(1): 171–176. DOI: 10.1073/pnas.0136972100 |
| [27] | GOLDING G B, DEAN A M. The structural basis of molecular adaptation[J]. Mol Biol Evol, 1998, 15(4): 355–369. DOI: 10.1093/oxfordjournals.molbev.a025932 |
| [28] | WANG Y, SHEN Y J, FENG C G, et al. Mitogenomic perspectives on the origin of Tibetan loaches and their adaptation to high altitude[J]. Sci Rep, 2016, 6: 29690. DOI: 10.1038/srep29690 |
| [29] | KUMAR C, SONG S, JIANG L, et al. Sequence characterization of DSG3 gene to know its role in high-altitude hypoxia adaptation in the Chinese cashmere goat[J]. Front Genet, 2018, 9: 553. DOI: 10.3389/fgene.2018.00553 |
| [30] | KUMAR S, STECHER G, TAMURA K. MEGA7:molecular evolutionary genetics analysis version 7.0 for bigger datasets[J]. Mol Biol Evol, 2016, 33(7): 1870–1874. DOI: 10.1093/molbev/msw054 |
| [31] | ROZAS J, SÁNCHEZ-DELBARRIO J C, MESSEGUER X, et al. DnaSP, DNA polymorphism analyses by the coalescent and other methods[J]. Bioinformatics, 2003, 19(18): 2496–2497. DOI: 10.1093/bioinformatics/btg359 |
| [32] | BANDELT H J, FORSTER P, ROHL A. Median-joining networks for inferring intraspecific phylogenies[J]. Mol Biol Evol, 1999, 16(1): 37–48. DOI: 10.1093/oxfordjournals.molbev.a026036 |
| [33] | DENG J, FENG J, LI L, et al. Polymorphisms, differentiation, and phylogeny of 10 Tibetan goat populations inferred from mitochondrial D-loop sequences[J]. Mitochondrial DNA A, 2018, 29(3): 439–445. DOI: 10.1080/24701394.2017.1303491 |
| [34] | SONG S, YAO N, YANG M, et al. Exome sequencing reveals genetic differentiation due to high-altitude adaptation in the Tibetan cashmere goat (Capra hircus)[J]. BMC Genomics, 2016, 17: 122. DOI: 10.1186/s12864-016-2449-0 |
| [35] |
王昕, 马月辉, 陈宏. 绵羊Cytb基因序列多态性及系统进化研究[J]. 遗传学报, 2006, 33(12): 1081–1086.
WANG X, MA Y H, CHEN H. Analysis of the genetic diversity and the phylogenetic evolution of Chinese sheep based on Cytb gene sequences[J]. Acta Genetica Sinica, 2006, 33(12): 1081–1086. (in Chinese) |
| [36] | LIU J B, DING X Z, ZENG Y F, et al. Genetic diversity and phylogenetic evolution of Tibetan sheep based on mtDNA D-loop sequences[J]. PLoS One, 2016, 11(7): e0159308. DOI: 10.1371/journal.pone.0159308 |
| [37] |
邓娟, 张红平, 巴贵, 等. 基于Cytb基因多态性研究西藏地区8个藏山羊群体遗传结构及母系起源[J]. 畜牧兽医学报, 2018, 49(1): 65–74.
DENG J, ZHANG H P, BA G, et al. Genetic structure and maternal origins of 8 Tibetan goat populations in Tibet inferred from Cytb gene diversity[J]. Acta Veterinaria et Zootechnica Sinica, 2018, 49(1): 65–74. (in Chinese) |
| [38] | HASSANIN A, ROPIQUET A, COULOUX A, et al. Evolution of the mitochondrial genome in mammals living at high altitude:new insights from a study of the tribe Caprini (Bovidae, Antilopinae)[J]. J Mol Evol, 2009, 68(4): 293–310. DOI: 10.1007/s00239-009-9208-7 |
| [39] | LUO Y J, GAO W X, GAO Y Q, et al. Mitochondrial genome analysis of Ochotona curzoniae and implication of cytochrome c oxidase in hypoxic adaptation[J]. Mitochondrion, 2008, 8(5-6): 352–357. DOI: 10.1016/j.mito.2008.07.005 |
| [40] |
王召锋.牦牛驯化和适应: 线粒体基因组证据[D].兰州: 兰州大学, 2011.
WANG Z F.Yak domestication and adaptation: evidence from mitochondrial genome[D].Lanzhou: Lanzhou University, 2011.(in Chinese) http://cdmd.cnki.com.cn/article/cdmd-10730-1011132955.htm |
| [41] |
贺赤诚, 张岳桥, 李建, 等. 青藏高原东北隅马衔山断裂带及周缘白垩纪——新生代沉积和构造变形历史[J]. 地球学报, 2019, 40(4): 563–587.
HE C C, ZHANG Y Q, LI J, et al. Kinematics of the Maxian Mountain Fault, Northeastern Tibetan Plateau:the history of Cretaceous-Cenozoic sedimentary andtectonic deformation[J]. Acta Geoscientica Sinica, 2019, 40(4): 563–587. (in Chinese) |
| [42] | PAMENTER M E, LAU G Y, RICHARDS J G, et al. Naked mole rat brain mitochondria electron transport system flux and H+ leak are reduced during acute hypoxia[J]. J Exp Biol, 2018, 221: jeb171397. DOI: 10.1242/jeb.171397 |




