多重耐药细菌的增多一直是全球关注的问题,其原因归结为耐药菌的迅速传播以及正在开发的新抗生素数量的急剧下降[1-2]。目前最受关注的是多重耐药革兰阴性菌带来的威胁,如耐碳青霉烯类肠杆菌,正在积极开发的新型抗革兰阴性菌感染的抗菌药物,如CARB-X[3-4],成为主要举措。目前看来,革兰阳性菌抗菌药物起到了不错的效果,特别是第二代糖肽类抗生素(GPAs)—奥利万星(Oritavancin)[3]和达巴万星(Dalbavancin)[4]的开发,但面对不可避免的糖肽类抗生素的耐药性问题,开发新的GPAs药物仍是必要的。在2019年底临床开发的187个传统抗菌剂项目中,有小部分研究(n=35,19%)为已知抗生素类(旧靶标)的改进衍生物,包括β-内酰胺类-青霉素结合蛋白的其他抑制剂、氟喹诺酮类-新型细菌拓扑异构酶抑制剂、氨基糖苷类、多粘菌素和大环内酯类;大部分项目(n=135,72%)专注于新的靶标研究,包括合成和天然抗菌肽(AMPs),天然产物和LpxC抑制剂;而在此基础上,几乎一半的项目主要针对革兰阴性菌,只有约10%的项目针对革兰阳性菌(主要是葡萄球菌)[5]。目前治疗革兰阳性菌感染的药物包括糖肽类[通过与脂质Ⅱ结合抑制肽聚糖合成:万古霉素(Vancomycin)、替考拉宁(Teicoplanin),以及最近的特拉万星(Telavancin)、奥利万星(Oritavancin)和达巴万星(Dalbavancin)]、脂肽类[破坏细胞膜:达托霉素(Daptomycin)]、噁唑烷酮类[抑制蛋白质合成:利奈唑胺(Linezolid)以及最近的特地唑胺(Tedizolid)]、头孢菌素[通过与青霉素结合蛋白结合抑制肽聚糖合成:第五代头孢洛林(Ceftaroline)和头孢吡普(Ceftobiprole)]和甘氨酰四环素[抑制蛋白质合成:替加环素(Tigecycline)]。
在抗生素的临床使用上,万古霉素(图 1)曾一度被作为抗革兰阳性菌感染的最后一道防线[6],几十年来一直是治疗MRSA感染的主要非肠道疗法。与其他抗生素相比,细菌对万古霉素产生抗药性需要更久的时间[7-9],但越来越多的最小抑菌浓度(MIC)证据表明,它可能正在失去对严重MRSA感染的临床疗效[10-11]。此外,万古霉素只能缓慢杀菌,且药物性质不佳,如药代动力学(需要每天两次给药)[9]和复杂可变的组织渗透性[10]。万古霉素已经成功使用了几十年,但危重患者的最佳万古霉素剂量仍然是一个有争议的问题[11],突显了改善GPAs的必要性。
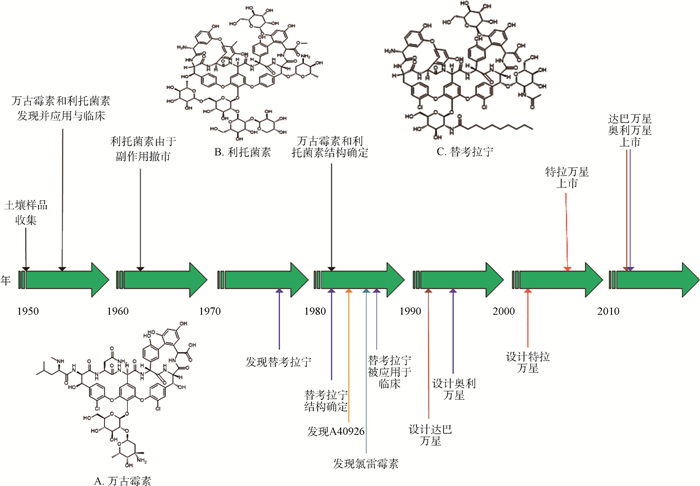
|
图 1 临床使用的糖肽类抗生素类的发现时间线 Fig. 1 Timeline of discovery for the clinically used glycopeptide antibiotics |
GPAs是由几种放线菌属或链霉菌属生物合成的复杂天然产物,其结构为线性多肽,由通过芳香族部分交联高度修饰的七肽骨架组成。另外,它们常通过氯化、糖基化、甲基化、酰化和/或硫酸化进一步修饰。它们通过干扰细胞壁生物合成来抑制细菌生长,作用靶点为细菌胞壁成分D-Ala-D-Ala,从而防止了肽聚糖层的交联。依据所含氨基酸的不同可分为四个族:vancomycin族、ristocetin族、avoparcin族和synmonicin族。由于作用方式独特,GPAs表现出优秀的抗革兰阳性菌的活性,临床应用于治疗MRSA和易感性肠球菌等革兰阳性菌感染引发的各类疾病,各GPAs的抗菌谱详见表 1。目前为止,GPAs发展到了第三代,第一代为两种糖肽类原型肽支架药物(万古霉素和替考拉宁);第二代主要为第一代药物的衍生物,经过了针对性的设计,在某些情况下通过辅助作用机制提高了抗菌活性(特拉万星,达巴万星和奥利万星)。GPAs的发现时间线如图 1所示。
2 GPAs的作用及耐药机制GPAs通过与肽聚糖的膜结合脂质Ⅱ前体结合而起作用(图 2),阻止其与细胞壁成分结合。万古霉素和其他糖肽类与脂质Ⅱ的结合通过协同的“背对背”二聚化方式得到增强,从而提高了它们与脂质Ⅱ的结合亲和力[12]。

|
图 2 万古霉素主链与脂质Ⅱ的D-Ala-D-Ala组分的氢键相互作用 Fig. 2 Hydrogen bond interactions between vancomycin backbone and D-Ala-D-Ala component of lipid Ⅱ |
常见的糖肽耐药性突变(特别是在肠球菌中)是从D-Ala-D-Ala末端多肽到D-Ala-D-Lac(VanA,VanB,VanD)或不太常见的D-Ala-D-Ser(VanC,VanE,VanG)表型的脂质Ⅱ生物合成的分化。由D-Lac取代D-Ala移除了与万古霉素中心形成的五个氢键中的一个,更重要的是,它引入的两个氧原子之间的排斥“孤对-孤对”相互作用,被认为是万古霉素对脂质Ⅱ亲和力降低1 000倍的主要原因[13]。在万古霉素等耐药性金黄色葡萄球菌(VISA)中,抗性是由于多肽聚糖的积累导致细胞壁增厚而产生的,而耐万古霉素的金黄色葡萄球菌(VRSA)则是在耐万古霉素肠球菌(VRE)脂质Ⅱ的VanA表型修饰之后产生的[14]。
3 批准上市的第二代GPAs 3.1 特拉万星特拉万星(Telavancin)(图 3C)是2009年推出的第二代GPAs,通过对万古霉素进行化学修饰得到,包括在万古胺糖上连接一个(癸氨乙基)亲脂性尾部和在芳香氨基酸7的4′-C位上连接一个亲水性的[(膦甲基)氨甲基]基团,前一种修饰提高了对革兰阳性病原体的抗菌效力,抗菌活性谱与万古霉素相似。特拉万星具有快速的杀菌作用,这被认为是由于与新生肽聚糖中的酰基D-Ala-D-Ala亚基结合而产生的协同效应,伴随着亲脂性尾部的膜插入,最终通过膜去极化和改变膜通透性而不是细胞溶解来破坏细菌膜的功能完整性[16]。此外,特拉万星遵循与万古霉素相同的耐药机制,即将D-Ala-D-Ala末端的VanA修饰为D-Ala-D-Lac,以及VISA和异质VISA(hVISA)表型的细胞壁增厚特性[17]。特拉万星可以诱导VRE表达VanA操纵子,但不诱导含有VanB的VRE表达VanA操纵子[18]。在体外耐药选择研究中没有观察到MRSA、MSSA和VRE对特拉万星的高水平耐药[19-21],持续监测表明,特拉万星在体外对一系列革兰阳性病原菌仍然有效[22-23]。
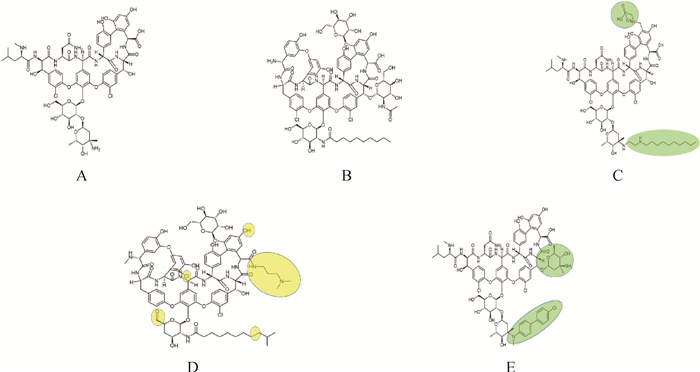
|
A.万古霉素;B.替考拉宁;C.特拉万星;D.达巴万星;E.奥利万星。绿色代表与替考拉宁结构的区别;黄色代表与万古霉素结构的区别 A. Vancomycin; B. Teicoplanin; C. Telavancin; D. Dalbavancin E. Oritavancin. Green represents the difference between the structure of teicoplanin. Yellow represents the difference between the structure of vancomycin 图 3 临床批准的第二代GPAs的化学结构 Fig. 3 Chemical structure of clinically approved second generation GPAs |
达巴万星(Dalbavancin),又称道古霉素(图 3D),是2014年批准临床使用的第二代替考拉宁型GPAs。它是由天然产物糖肽A40926[20]通过氨基酸7的肽羧基与3-(二甲氨基)-1-丙胺酰胺化而得[21],这种取代基的引入增加了对葡萄球菌,特别是凝固酶阴性葡萄球菌(CoNS)的效力[22]。达巴万星在临床上被批准用于治疗革兰阳性相关的急性细菌性皮肤和皮肤结构感染(ABSSSI)[23],以及正在进行的对成人骨髓炎的有效性和安全性评估[24]。和其它脂糖肽一样,达巴万星以肽聚糖前体的C末端的acyl-D-Ala-D-Ala亚单位为靶点,对多种革兰阳性菌表现出体外活性[25]。没有公开数据表明脂质侧链在破环细胞膜稳定性中的作用,相反,它与血清蛋白的相互作用可能是延长其半衰期的原因之一[26]。此外,带正电荷的C端二甲氨基丙基可能与细菌膜的负磷脂头基团相互作用[26]。万古霉素类糖肽的二聚化与配体结合是协同的,而达巴万星的二聚化是反协同的[27]。达巴万星对VanA肠球菌无活性,但对VanB表型仍有活性[28]。由于其较长的半衰期(t1/2>300 h)[29],在治疗过程中患者长期暴露于亚治疗水平,产生了耐药性[30]。
3.3 奥利万星奥利万星(Oritavancin)(图 3E)是礼来公司开发的第二代半合成脂糖肽。它来源于对天然产物氯雷霉素(Chloroeremomycin)的化学修饰,包括将N-烷基-对氯苯基苄取代基拼合到环4氨基酸上双糖所连的表万古胺亚基上。氯雷霉素与万古霉素唯一的不同之处在于万古霉素中的万古胺糖基部分,在环4-和6-氨基酸上有两个L-4表万古胺亚基,而万古霉素中4号位上的是单个L-万古胺亚基。氯雷霉素中增加的L-4-表万古胺亚基具有额外的收益,与万古霉素相比,氯雷霉素对万古霉素敏感细菌具有更强的抗菌活性[31]。在奥利万星的研发过程中,礼来公司发现万古霉素衍生物在万古胺糖上带有烷基(而不是酰基)侧链,对万古霉素耐药细菌表现出更优秀的活性[31]。奥利万星临床被批准用于治疗成人革兰阳性相关ABSSSI[32]。虽然没有被批准用于其他适应症,但它在治疗由VRE引起的人工关节感染[3]和人工瓣膜心内膜炎[33]方面表现出了可能。与特拉万星和达巴万星不同的是,奥利万星是唯一能够通过多种作用模式同时保持对VRSA和VanA型VRE的有效活性的脂糖肽[34]。除了通过与acyl-D-Ala-D-Ala末端结合来抑制转糖基化外,它还可以通过与脂质Ⅱ的五甘醇桥联片段结合来干扰转肽基,从而保持与万古霉素耐药生物修饰的肽聚糖末端的亲和力[35-36]。其次,通过疏水的N-烷基-对氯苯苄基将奥利万星锚定到细胞膜上,与脂质Ⅱ的相互作用加强,导致了靶标亲和力增加,促进奥利万星自结合成二聚体[37]。奥利万星以这种方式与膜相互作用还导致其第三种作用模式,即破坏细菌膜的完整性,致去极化和通透性增加[34]。
4 GPAs的开发策略 4.1 天然产物GPAs分离万古霉素和替考拉宁在临床上的成功,表现在与其他类抗菌药无交叉耐药性,以及发现和出现耐药性之间的显著滞后时间,引发了人们对发现新的天然产物GPAs的关注。光谱学的进步使得能够快速鉴定物质结构,新糖肽的数量在1982年至1996年期间激增[38]。然而,自20世纪90年代中期以来,新糖肽的发现已经减少,需要创新的方法来鉴定和纯化相对不常见的放线菌的菌株[39]。尽管有这些改进,但自20世纪90年代以来,很少有新的糖肽被鉴定为天然产物。
4.2 糖肽衍生物合成糖肽支架含有几个易于修饰的取代基,特别是游离的C末端羧基、万古胺糖伯胺和N末端的伯胺或仲胺。近年来,主要采用几种策略,侧重于合理设计以提高GPAs的效力并克服耐药性,包括膜靶向法、骨架修饰和生物合成。
4.2.1 膜靶向法 传统上药物开发专注于药效学方面优化目标亲和力,选择性和蛋白质结合,但很少评估膜结合特性。相反,自然界利用生物膜成分的细微变化来控制信号转导、囊泡运输和与膜相关蛋白如Ras[40]、Src[41]、Rab[42]、GAG[43]和MARCKS[44]的受体再循环。在这些蛋白质中,N末端和/或C末端亲脂基团(肉豆蔻酰基,棕榈酰基)和碱性氨基酸簇作为经由丝氨酸和酪氨酸残基的磷酸化选择性的静电“开关”到细胞质和膜表面之间的控制易位[41, 45]。将此类基序附加到作用于与膜相关的靶标的药物上可以增强膜结合,并同时增加靶位点上的药物浓度[46]。在Blaskovich等[47]的研究中,通过将膜靶向基序附加到万古霉素的C-末端上,设计并合成了一系列万古霉素衍生物,称为Vancapticins。为了连接含有亲脂膜插入元素(MIE)的基本静电效应肽片段,使用与未保护功能兼容的化学试剂,Blaskovich等[47]将万古霉素与吡啶基二硫代乙胺酰胺化,然后在温和条件下通过二硫化物选择性连接未保护的含有C端Cys残基的基本肽交换(图 4),初始膜靶向糖肽中采用的亲油酰化16-mer的静电效应的肽序列(EEPS)(GSSKSPSKKKKKKPGD)。所得缀合物的抗MRSA、VISA和多重耐药(MDR)肺炎链球菌的活性比万古霉素和达托霉素(Daptomycin)高4~10倍,而抗耐万古霉素肠球菌(VRE)的活性比万古霉素高100倍以上。Vancapticins表现出抗革兰阳性菌活性,包括对耐万古霉素菌株的活性。且Vancapticins在小鼠大腿MRSA感染、肺部肺炎链球菌感染和腹膜内MSSA感染等多种感染模型中非常有效,诱导耐药性实验表明Vancapticins保留了较低的耐药倾向,并且药代动力学(PK)特征更优。
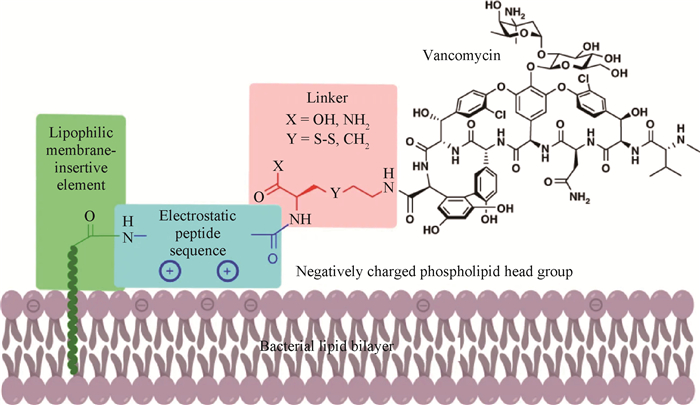
|
MIE.膜插入元件; EEPS.静电效应子肽序列 MIE.Membrane-insertive elementL; EEPS. Electrostatic effector peptide sequence 图 4 赖氨酸连接和半胱氨酸连接的万古霉素-肽缀合物的结构[47] Fig. 4 Structure of lysine-linked and cysteine-linked vancomycin-peptide conjugates[47] |
4.2.2 糖肽核心修饰 Crowley和Boger[48]描述了一种克服万古霉素耐药性的新方法,方法是替换脂质Ⅱ Lys-D-Ala-D-Ala三肽中参与末端酰胺NH结合的关键万古霉素羰基。如前所述,当该三肽被修饰为Lys-D-Ala-D-Lac时,诱导高水平的抗性,其中乳酸的酯氧不能与万古霉素羰基形成氢键,从而大大减少了结合。同时,他们还报道了万古霉素苷元类似物的全合成,其中万古霉素苷元中残基4的羰基被亚甲基取代[48]。与天然苷元的结合相比,Ac-Lys(Ac)-D-Ala-D-Ala三肽配体(天然脂质Ⅱ)的亲和力降低了35倍,而相应的乳酸配体的亲和力增加了40倍。之后的一次新的全合成研究产生了苷元,其中羰基被硫代酰胺取代,硫代酰胺作为一种中间体存在,后续被精制成脒。当硫代酰胺失去所有结合活性时,脒仅对三肽配体失去2倍的活性,并对乳酸盐衍生物保持类似的活性,即与万古霉素苷元的结合增加了600倍。更重要的是,这种配体结合活性转化为对VRE的有效抑制活性,其对粪肠球菌的MIC值为0.31 μg·mL-1[49-51]。
4.2.3 芳环功能化 万古霉素和替考拉宁以往就有氯代物的存在,Nakama等[52]直接对万古霉素氯原子进行Suzuki-Miyaura交叉偶联,以获得带有芳基或烯基的单或双取代万古霉素衍生物(图 5)。大多数二烯基GPAs对敏感菌株没有活性,而在D-O-E环上单取代的一些化合物扩大了抗菌谱,包括对典型抗GPAs菌株的活性。在此,具有最高活性的化合物包含一个双苯基部分,与奥利万星中发现的相似,这清楚地表明了这种修饰对GPAs活性的重要性,而D-O的单取代芳基硼酸的-E环导致对敏感菌株的活性略有损失。此外,使用选择性钯(0)万古霉素糖苷配基催化的硼基化证实了D-上单取代O-E环和硼酸。这可以通过破坏GPAs二聚化来解释,因为已知存在于D-O-E环上的氯化物与该过程有关[53]。接着,两个步骤的脱氯/交叉耦合协议所允许的GPAs C-上发现的氯原子的选择性取代O-D环[54]。这些结果加强了在GPAs中发现的氯原子的重要性,特别是在C-O上-D环,氯化物(以及随后的修饰)面向脂质Ⅱ-结合位点,可能破坏脂质Ⅱ与这些修饰的GPAs的结合。

|
A.用万古霉素探索化学方法;B.用巴利霉素系统测试的体内方法;NBP.N-溴邻苯二甲酰亚胺 A.Chemical approaches explored with vancomycin; B.In vivo approaches tested with the balhimycin system; NBP.N-bromophthalimide 图 5 GPAs的卤素取代修饰模式[55] Fig. 5 Modifying the halogen pattern of GPAs[55] |
4.2.4 GPAs的C和N末端修饰 GPAs的结构刚性和高度交联的性质使糖苷配基核心复合物得到修饰,因为在Ⅰ型和Ⅱ型GPAs中7个氨基酸中有5个是交联的,而在Ⅲ型和Ⅳ型GPAs中,由于额外的F-O-G环,所有7个氨基酸都是交联的。因此,GPAs修饰通常以肽末端为目标。C末端修饰可通过各种氨基酸功能与GPAs的预活化羧酸部分,以生成酰胺键偶联,这一过程是容易实现的。GPAs的C末端修饰的一个共同主题是引入带正电荷的氨基酸(如赖氨酸和精氨酸)以增加对膜的亲和力。例如,用聚D-精氨酸肽对万古霉素进行修饰导致发现了一种能够破坏生物膜的化合物,这是产毒细菌常表现出的机制[56]。额外发现的是,添加单个精氨酸残基也表现出了对革兰阴性细菌的活性[57]。重要的是,GPAs的渗透性破坏机制通常可以通过向GPAs添加脂肪族链进行膜裂解来补充[47],而这种方法最成功的结果之一就是合成了达巴万星[21]。
GPAs N末端修饰重点关注的是去除N-甲基-亮氨酸残基,以便用其他氨基酸取代,并在该位置上具有完整的构效关系。从万古霉素的结构得知,亮氨酸残基的脂肪族链有助于稳定GPAs的三级结构。Zmijewski等[58]将万古霉素添加到含有柠檬马杜拉放线菌(Actinomadura citrea)NRRI 18382的生物转化发酵液中,其被证明负责N-甲基-亮氨酸残基的酶促去除,产生去亮氨酰万古霉素。选择柠檬马杜拉放线菌进行这项工作是因为它是一种稀有的土壤微生物,能够通过降解来灭活万古霉素,形成去亮氨酰万古霉素。用异硫氰酸苯酯处理进行Edman降解后,得到了该化合物及其苷元当量化合物[59]。此外,Brieke等[60]使用了来自Pekiskomycin生物合成的I型GPAs的天然甲基转移酶测试了化学酶法中的N末端修饰,表明这些酶可以有效地对各种GPAs衍生物和前体的N末端进行单或二甲基化。此外,辅因子S-腺苷甲硫氨酸可以用其他合成的辅因子代替,这些辅因子可用于扩展GPA的N末端结构[60]。
4.2.5 生物合成 鉴于几乎所有GPAs都是从天然生物合成途径中直接或半合成修饰后获得的,开发新糖肽的另一种方法是人工操纵生物合成。补全他汀(Complestatin)是一种V型糖肽苷元,具有双环结构,色氨酸残基代替了苯丙氨酸。生物合成基因簇已经被克隆并在变铅青链霉菌(Streptomyces lividans)中表达,以操纵这些基因来产生新的类似物。CYP450单加氧酶基因缺失产生单环和线性衍生物,虽然活性降低了,但该方法可应用于其他糖肽,并用于化学上难以实现的改变[61]。Yim等[62]在天蓝色链霉菌(Streptomyces Coelicolor)中表达了一个最简的替考拉宁骨架(无糖基化或酰基化),并测试了来自不同生产菌的7个GPAs生物合成基因簇的13种骨架修饰酶,然后将一个或两个基因整合到表达平台中组合,用于识别哪些修饰是可能且可行的。
4.3 以系统发育进化为导向筛选GPAsGPAs是由放线菌通过生物合成的基因簇产生的,这些基因簇包括支持糖肽抗生素调节、合成、输出和抗药性的基因。糖肽的化学和生物合成多样性是复杂进化历史的产物。系统发育分析显示,糖肽生物合成的前体比其他组分要古老得多,这意味着这些基因簇来自于先前存在的基因库[63]。Waglechner等[63]研究发现,在整个进化过程中,生物合成基因簇(BGC)至少部分是通过对生成的代谢物生物活性的选择性压力来塑造的。因此,具有进化差异的生物合成基因的BGC也可能产生新的生物活性。这种以系统发育为导向的发现,以前已经被应用于通过从选择的生物合成基因3~5个碱基的短序列标签生成系统发育树来识别天然产品家族的不同成员。Thaker等[64]通过将抗生素耐药性筛选与串联的生物合成基因片段的系统发育分析相结合的策略,识别出肽支架不同的糖肽类化合物(如Pekiskomycin),这些新成员保留了相同的作用机制。与此同时,Guitor等[65]设计了一套探针以准确、快速地鉴定复杂宏基因组样本中的稀有和常见耐药基因,达到靶向捕获耐药的目的。这些方法对于优先处理产生未发现的且具有生物活性的化合物的BGC很有用,但它们不提供任何关于作用机制的信息。抗生素BGC不仅编码生物合成机制,还编码抵抗基因以防止自身被攻击。在BGC中寻找耐药基因的存在已经指导了具有已知或预测机制的抗生素的基因组挖掘[66-67]。Culp等[68]通过鉴定缺乏已知自抗性基因的系统发育上不同的BGC,筛选出了一类具有新的作用机制的抗生素的两个成员,包括现已知的GPAs补全他汀和一种新发现的化合物Corbomycin。它们具有全新的作用模式,通过与肽聚糖结合,阻断了自溶素(生长过程中重塑细胞壁所必需的肽聚糖水解酶)的作用,抑制肽聚糖重塑[68]。同时重新定义了补全他汀的作用机制,此前,研究表明,补全他汀靶向作用于脂肪酸合成[61]。在皮肤MRSA感染的小鼠模型中,Corbomycin和补全他汀的耐药倾向性低,可以有效地降低细菌负担[68]。
5 结 论过去的几十年内,GPAs克服了许多耐药菌带来的困扰,虽然改良抗菌剂暂时摆脱了耐药性的困境,但细菌耐药性的演变仍是一个长期存在的问题。GPAs的多样化为选择性抗菌剂的产生提供了可能的途径。减缓新抗药性机制发展的最佳机会之一依旧是优先开发新的抗生素。除此之外,认真管理与制订抗生素的合理使用也同样重要。
| [1] | FERNANDES P, MARTENS E. Antibiotics in late clinical development[J]. Biochem Pharmacol, 2017, 133: 152–163. DOI: 10.1016/j.bcp.2016.09.025 |
| [2] | OUTTERSON K, REX J H, JINKS T, et al. Accelerating global innovation to address antibacterial resistance:introducing CARB-X[J]. Nat Rev Drug Discov, 2016, 15(9): 589–590. DOI: 10.1038/nrd.2016.155 |
| [3] | YAN Q, KARAU M J, RAVAL Y S, et al. In vitro activity of oritavancin in combination with rifampin or gentamicin against prosthetic joint infection-associated methicillin-resistant Staphylococcus epidermidis biofilms[J]. Int J Antimicrob Agents, 2018, 52(5): 608–615. DOI: 10.1016/j.ijantimicag.2018.07.012 |
| [4] | BOUZA E, VALERIO M, SORIANO A, et al. Dalbavancin in the treatment of different gram-positive infections:a real-life experience[J]. Int J Antimicrob Agents, 2018, 51(4): 571–577. DOI: 10.1016/j.ijantimicag.2017.11.008 |
| [5] | THEURETZBACHER U, OUTTERSON K, ENGEL A, et al. The global preclinical antibacterial pipeline[J]. Nat Rev Microbiol, 2020, 18(5): 275–285. DOI: 10.1038/s41579-019-0288-0 |
| [6] | MERKEL A B, TEMPLE G K, BURKART M D, et al. Purification, crystallization and preliminary structural studies of DTDP-4-Keto-6-Deoxy-Glucose-5-Epimerase (EvaD) from Amycolatopsis orientalis, the fourth enzyme in the DTDP-L-Epivancosamine biosynthetic pathway[J]. Acta Crystallogr D Biol Crystallogr, 2002, 58(7): 1226–1228. DOI: 10.1107/S0907444902007382 |
| [7] | AHMED M O, BAPTISTE K E. Vancomycin-resistant enterococci:a review of antimicrobial resistance mechanisms and perspectives of human and animal health[J]. Microb Drug Resist, 2018, 24(5): 590–606. DOI: 10.1089/mdr.2017.0147 |
| [8] | DIAZ R, AFREIXO V, RAMALHEIRA E, et al. Evaluation of vancomycin MIC creep in methicillin-resistant Staphylococcus aureus infections-a systematic review and meta-analysis[J]. Clin Microbiol Infec, 2018, 24(2): 97–104. DOI: 10.1016/j.cmi.2017.06.017 |
| [9] | RYBAK M J. The pharmacokinetic and pharmacodynamic properties of vancomycin[J]. Clin Infect Dis, 2006, 42(S1): S35–S39. |
| [10] | BEACH J E, PERROTT J, TURGEON R D, et al. Penetration of vancomycin into the cerebrospinal fluid:a systematic review[J]. Clin Pharmacokinet, 2017, 56(12): 1479–1490. DOI: 10.1007/s40262-017-0548-y |
| [11] | ÁLVAREZ R, CORTÉS L E L, MOLINA J, et al. Optimizing the clinical use of vancomycin[J]. Antimicrob Agents Chemother, 2016, 60(5): 2601–2609. DOI: 10.1128/AAC.03147-14 |
| [12] | ZENG D N, DEBABOV D, HARTSELL T L, et al. Approved glycopeptide antibacterial drugs:mechanism of action and resistance[J]. Cold Spring Harb Perspect Med, 2016, 6(12): a026989. DOI: 10.1101/cshperspect.a026989 |
| [13] | STEGMANN E, FRASCH H J, KILIAN R, et al. Self-resistance mechanisms of Actinomycetes producing lipid II-targeting antibiotics[J]. Int J Med Microbiol, 2015, 305(2): 190–195. DOI: 10.1016/j.ijmm.2014.12.015 |
| [14] | GARDETE S, TOMASZ A. Mechanisms of vancomycin resistance in Staphylococcus aureus[J]. J Clin Invest, 2014, 124(7): 2836–2840. DOI: 10.1172/JCI68834 |
| [15] | COURVALIN P. Vancomycin resistance in gram-positive cocci[J]. Clin Infect Dis, 2006, 42(S1): S25–S34. |
| [16] | LUNDE C S, HARTOUNI S R, JANC J W, et al. Telavancin disrupts the functional integrity of the bacterial membrane through targeted interaction with the cell wall precursor lipid Ⅱ[J]. Antimicrob Agents Chemother, 2009, 53(8): 3375–3383. DOI: 10.1128/AAC.01710-08 |
| [17] | KARLOWSKY J A, NICHOL K, ZHANEL G G. Telavancin:mechanisms of action, in vitro activity, and mechanisms of resistance[J]. Clin Infect Dis, 2015, 61(S2): S58–S68. |
| [18] | HILL C M, KRAUSE K M, LEWIS S R, et al. Specificity of induction of the VanA and VanB operons in vancomycin-resistant enterococci by telavancin[J]. Antimicrob Agents Chemother, 2010, 54(7): 2814–2818. DOI: 10.1128/AAC.01737-09 |
| [19] | JONES R N, FLAMM R K, CASTANHEIRA M, et al. Activity of Telavancin against Gram-positive pathogens isolated from bone and joint infections in North American, Latin American, European and Asia-Pacific nations[J]. Diagn Microbiol Infect Dis, 2017, 88(2): 184–187. DOI: 10.1016/j.diagmicrobio.2017.03.003 |
| [20] | GOLDSTEIN B P, SELVA E, GASTALDO L, et al. A40926, a new glycopeptide antibiotic with anti-Neisseria activity[J]. Antimicrob Agents Chemother, 1987, 31(12): 1961–1966. DOI: 10.1128/AAC.31.12.1961 |
| [21] | CANDIANI G, ABBONDI M, BORGONOVI M, et al. In-vitro and in-vivo antibacterial activity of BI 397, a new semi-synthetic glycopeptide antibiotic[J]. J Antimicrob Chemother, 1999, 44(2): 179–192. DOI: 10.1093/jac/44.2.179 |
| [22] | MALABARBA A, GOLDSTEIN B P. Origin, structure, and activity in vitro and in vivo of dalbavancin[J]. J Antimicrob Chemother, 2005, 55(S2): ii15–ii20. DOI: 10.1093/jac/dki005 |
| [23] | GARNOCK-JONES K P. Single-dose dalbavancin:a review in acute bacterial skin and skin structure infections[J]. Drugs, 2017, 77(1): 75–83. DOI: 10.1007/s40265-016-0666-0 |
| [24] | RAPPO U, PUTTAGUNTA S, SHEVCHENKO V, et al. Dalbavancin for the treatment of osteomyelitis in adult patients:a randomized clinical trial of efficacy and safety[J]. Open Forum Infect Dis, 2019, 6(1): ofy331. DOI: 10.1093/ofid/ofy331 |
| [25] | CERCENADO E. Antimicrobial spectrum of dalbavancin.Mechanism of action and in vitro activity against Gram-positive microorganisms[J]. Enferm Infecc Microbiol Clin, 2017, 35(Sl): 9–14. DOI: 10.1016/S0213-005X(17)30029-0 |
| [26] | ECONOMOU N J, NAHOUM V, WEEKS S D, et al. A carrier protein strategy yields the structure of dalbavancin[J]. J Am Chem Soc, 2012, 134(10): 4637–4645. DOI: 10.1021/ja208755j |
| [27] | CHENG M, ZIORA Z M, HANSFORD K A, et al. Anti-cooperative ligand binding and dimerisation in the glycopeptide antibiotic dalbavancin[J]. Org Biomol Chem, 2014, 12(16): 2568–2575. DOI: 10.1039/C3OB42428F |
| [28] | ARHIN F F, SEGUIN D L, BELLEY A, et al. In vitro stepwise selection of reduced susceptibility to lipoglycopeptides in enterococci[J]. Diagn Microbiol Infect Dis, 2017, 89(2): 168–171. DOI: 10.1016/j.diagmicrobio.2017.06.023 |
| [29] | AZANZA J R, SÁDABA B, REIS J. Dalbavancina:pharmacokinetic and pharmacodynamic parameters[J]. Enferm Infecc Microbiol ClÍn, 2017, 35(Sl): 22–27. DOI: 10.1016/S0213-005X(17)30031-9 |
| [30] | WERTH B J, JAIN R, HAHN A, et al. Emergence of dalbavancin non-susceptible, vancomycin-intermediate Staphylococcus aureus (VISA) after treatment of MRSA central line-associated bloodstream infection with a dalbavancin-and vancomycin-containing regimen[J]. Clin Microbiol Infect, 2018, 24(4): 429.e1–429.e5. DOI: 10.1016/j.cmi.2017.07.028 |
| [31] | ALLEN N E. From vancomycin to oritavancin:the discovery and development of a novel lipoglycopeptide antibiotic[J]. Anti Infect Agents Med Chem, 2010, 9(1): 23–47. DOI: 10.2174/187152110790886745 |
| [32] | COREY G R, LOUTIT J, MOECK G, et al. Single intravenous dose of oritavancin for treatment of acute skin and skin structure infections caused by gram-positive bacteria:summary of safety analysis from the phase 3 SOLO studies[J]. Antimicrob Agents Chemother, 2018, 62(4): e01919-17. DOI: 10.1128/AAC.01919-17 |
| [33] | JOHNSON J A, FEENEY E R, KUBIAK D W, et al. Prolonged use of oritavancin for vancomycin-resistant Enterococcus faecium prosthetic valve endocarditis[J]. Open Forum Infect Dis, 2015, 2(4): ofv156. |
| [34] | ZHANEL G G, SCHWEIZER F, KARLOWSKY J A. Oritavancin:mechanism of action[J]. Clin Infect Dis, 2012, 54(S3): S214–S219. |
| [35] | KIM S J, CEGELSKI L, STUEBER D, et al. Oritavancin exhibits dual mode of action to inhibit cell-wall biosynthesis in Staphylococcus aureus[J]. J Mol Biol, 2008, 377(1): 281–293. |
| [36] | COOPER M A, WILLIAMS D H. Binding of glycopeptide antibiotics to a model of a vancomycin-resistant bacterium[J]. Chem Biol, 1999, 6(12): 891–899. DOI: 10.1016/S1074-5521(00)80008-3 |
| [37] | YARLAGADDA V, SAMADDAR S, PARAMANANDHAM K, et al. Membrane disruption and enhanced inhibition of cell-wall biosynthesis:a synergistic approach to tackle vancomycin-resistant bacteria[J]. Angew Chem Int Ed, 2015, 54(46): 13644–13649. DOI: 10.1002/anie.201507567 |
| [38] | NICOLAOU K C, BODDY C N C, BRÄSE S, et al. Chemistry, biology, and medicine of the glycopeptide antibiotics[J]. Angew Chem Int Ed, 1999, 38(15): 2096–2152. DOI: 10.1002/(SICI)1521-3773(19990802)38:15<2096::AID-ANIE2096>3.0.CO;2-F |
| [39] | YAO R C, CRANDALL L W.Glycopeptides: classification, occurrence, and discovery[M]//NAGARAJAN R.Glycopeptide Antibiotics.New York: Marcel Dekker, 1994: 1. |
| [40] | WRIGHT L P, PHILIPS M R. Thematic review series:lipid posttranslational modifications CAAX modification and membrane targeting of Ras[J]. J Lipid Res, 2006, 47(5): 883–891. DOI: 10.1194/jlr.R600004-JLR200 |
| [41] | MURRAY D, HERMIDA-MATSUMOTO L, BUSER C A, et al. Electrostatics and the membrane association of Src:theory and experiment[J]. Biochemistry, 1998, 37(8): 2145–2159. DOI: 10.1021/bi972012b |
| [42] | PYLYPENKO O, HAMMICH H, YU I M, et al. Rab GTPases and their interacting protein partners:structural insights into Rab functional diversity[J]. Small GTPases, 2018, 9(1-2): 22–48. DOI: 10.1080/21541248.2017.1336191 |
| [43] | ZHOU W, PARENT L J, WILLS J W, et al. Identification of a membrane-binding domain within the amino-terminal region of human immunodeficiency virus type 1 gag protein which interacts with acidic phospholipids[J]. J Virol, 1994, 68(4): 2556–2569. |
| [44] | SEYKORA J T, MYAT M M, ALLEN L A H, et al. Molecular determinants of the myristoyl-electrostatic switch of MARCKS[J]. J Biol Chem, 1996, 271(31): 18797–18802. DOI: 10.1074/jbc.271.31.18797 |
| [45] | MURRAY D, ARBUZOVA A, HONIG B, et al. The role of electrostatic and nonpolar interactions in the association of peripheral proteins with membranes[J]. Curr Top Membr, 2002, 52: 277–307. DOI: 10.1016/S1063-5823(02)52012-3 |
| [46] | MCLAUGHLIN S, ADEREM A. The myristoyl-electrostatic switch:a modulator of reversible protein-membrane interactions[J]. Trends Biochem Sci, 1995, 20(7): 272–276. DOI: 10.1016/S0968-0004(00)89042-8 |
| [47] | BLASKOVICH M A T, HANSFORD K A, GONG Y J, et al. Protein-inspired antibiotics active against vancomycin-and daptomycin-resistant bacteria[J]. Nat Commun, 2018, 9(1): 22. DOI: 10.1038/s41467-017-02123-w |
| [48] | CROWLEY B M, BOGER D L. Total synthesis and evaluation of[J]. J Am Chem Soc, 2006, 128(9): 2885–2892. DOI: 10.1021/ja0572912 |
| [49] | XIE J, PIERCE J G, JAMES R C, et al. A redesigned vancomycin engineered for dual D-Ala-D-Ala And D-Ala-D-Lac binding exhibits potent antimicrobial activity against vancomycin-resistant bacteria[J]. J Am Chem Soc, 2011, 133(35): 13946–13949. DOI: 10.1021/ja207142h |
| [50] | XIE J, OKANO A, PIERCE J G, et al. Total synthesis of[J]. J Am Chem Soc, 2012, 134(2): 1284–1297. DOI: 10.1021/ja209937s |
| [51] | OKANO A, JAMES R C, PIERCE J G, et al. Silver(I)-promoted conversion of thioamides to amidines:divergent synthesis of a key series of vancomycin aglycon residue 4 amidines that clarify binding behavior to model ligands[J]. J Am Chem Soc, 2012, 134(21): 8790–8793. DOI: 10.1021/ja302808p |
| [52] | NAKAMA Y, YOSHIDA O, YODA M, et al. Discovery of a novel series of semisynthetic vancomycin derivatives effective against vancomycin-resistant bacteria[J]. J Med Chem, 2010, 53(6): 2528–2533. DOI: 10.1021/jm9017543 |
| [53] | GERHARD U, MACKAY J P, MAPLESTONE R A, et al. The role of the sugar and chlorine substituents in the dimerization of vancomycin antibiotics[J]. J Am Chem Soc, 1993, 115(1): 232–237. DOI: 10.1021/ja00054a033 |
| [54] | WADZINSKI T J, GEA K D, MILLER S J. A stepwise dechlorination/cross-coupling strategy to diversify the vancomycin 'in-chloride'[J]. Bioorg Med Chem Lett, 2016, 26(3): 1025–1028. DOI: 10.1016/j.bmcl.2015.12.027 |
| [55] | MARSCHALL E, CRYLE M J, TAILHADES J. Biological, chemical, and biochemical strategies for modifying glycopeptide antibiotics[J]. J Biol Chem, 2019, 294(49): 18769–18783. DOI: 10.1074/jbc.REV119.006349 |
| [56] | ANTONOPLIS A, ZANG X Y, HUTTNER M A, et al. A dual-function antibiotic-transporter conjugate exhibits superior activity in sterilizing MRSA biofilms and killing persister cells[J]. J Am Chem Soc, 2018, 140(47): 16140–16151. DOI: 10.1021/jacs.8b08711 |
| [57] | ANTONOPLIS A, ZANG X Y, WEGNER T, et al. Vancomycin-arginine conjugate inhibits growth of carbapenem-resistant E. coli and targets cell-wall synthesis[J]. ACS Chem Biol, 2019, 14(9): 2065–2070. DOI: 10.1021/acschembio.9b00565 |
| [58] | ZMIJEWSKI M J, LOGAN R M, MARCONI G, et al. Biotransformation of vancomycin B to vancomycin hexapeptide by a soil microorganism[J]. J Nat Prod, 1989, 52(1): 203–206. DOI: 10.1021/np50061a033 |
| [59] | BOOTH P M, STONE D J M, WILLIAMS D H. The edman degradation of vancomycin:preparation of vancomycin hexapeptide[J]. J Chem Soc Chem Commun, 1987(22): 1694–1695. DOI: 10.1039/c39870001694 |
| [60] | BRIEKE C, YIM G, PESCHKE M, et al. Catalytic promiscuity of glycopeptide N-methyltransferases enables bio-orthogonal labelling of biosynthetic intermediates[J]. Chem Commun, 2016, 52(94): 13679–13682. DOI: 10.1039/C6CC06975D |
| [61] | PARK O K, CHOI H Y, KIM G W, et al. Generation of new complestatin analogues by heterologous expression of the complestatin biosynthetic gene cluster from Streptomyces chartreusis AN1542[J]. ChemBioChem, 2016, 17(18): 1725–1731. DOI: 10.1002/cbic.201600241 |
| [62] | YIM G, WANG W L, THAKER M N, et al. How to make a glycopeptide:a synthetic biology approach to expand antibiotic chemical diversity[J]. ACS Infect Dis, 2016, 2(9): 642–650. DOI: 10.1021/acsinfecdis.6b00105 |
| [63] | WAGLECHNER N, MCARTHUR A G, WRIGHT G D. Phylogenetic reconciliation reveals the natural history of glycopeptide antibiotic biosynthesis and resistance[J]. Nat Microbiol, 2019, 4(11): 1862–1871. DOI: 10.1038/s41564-019-0531-5 |
| [64] | THAKER M N, WANG W L, SPANOGIANNOPOULOS P, et al. Identifying producers of antibacterial compounds by screening for antibiotic resistance[J]. Nat Biotechnol, 2013, 31(10): 922–927. DOI: 10.1038/nbt.2685 |
| [65] | GUITOR A K, RAPHENYA A R, KLUNK J, et al. Capturing the resistome:a targeted capture method to reveal antibiotic resistance determinants in metagenomes[J]. Antimicrob Agents Chemother, 2019, 64(1): e01324-19. DOI: 10.1128/AAC.01324-19 |
| [66] | YAN Y, LIU Q K, ZANG X, et al. Resistance-gene-directed discovery of a natural-product herbicide with a new mode of action[J]. Nature, 2018, 559(7714): 415–418. |
| [67] | TANG X Y, LI J, MILLÁN-AGUIÑAGA N, et al. Identification of thiotetronic acid antibiotic biosynthetic pathways by target-directed genome mining[J]. ACS Chem Biol, 2015, 10(12): 2841–2849. DOI: 10.1021/acschembio.5b00658 |
| [68] | CULP E J, WAGLECHNER N, WANG W L, et al. Evolution-guided discovery of antibiotics that inhibit peptidoglycan remodelling[J]. Nature, 2020, 578(7796): 582–587. DOI: 10.1038/s41586-020-1990-9 |



