小眼畸形相关转录因子(microphthalmia-associated transcription factor, MITF)是黑色素细胞生长必需且最重要的调节器,其在多种细胞中均表达,例如黑色素细胞、破骨细胞、视网膜色素上皮细胞和肥大细胞等。随着研究的深入,MITF对黑色素沉积的影响受到广泛关注,尤其是MITF-M[1-2]。有研究发现,MITF基因表达可能依赖环-磷酸腺苷通路,这一通路中,MITF可诱导一些基因转录,例如酪氨酸酶(tyrosinase, TYR)、酪氨酸酶相关蛋白1(tyrosinase related-protein 1, TYRP1)、酪氨酸酶相关蛋白2(tyrosinase related-protein 2, TYRP2)等酪氨酸酶家族基因(tyrosinase related-protein, TRP)[2-4]。酪氨酸酶家族即TYR、TYRP1、TYRP2这3个蛋白酶是黑色素合成的重要蛋白酶,其家族基因的启动子有M-box、TDE和E-box 3种类型的结构,并且它们共享保守的“CATGTG”、“M-box”碱基序列结构,MITF能够识别并结合该结构,调节控制酪氨酸酶家族基因的正常转录[5],达到影响黑色素形成的目的。
miRNA主要在细胞增殖、分化、个体发育、抗应激等方面发挥作用[6-7]。自从发现microRNA lin-4和let7后[4-6],microRNA已成为研究者热衷的对象,随着对于microRNA研究的深入,人们意识到miRNA在皮肤疾病的发展中也发挥了重要的作用[8-10]。例如较早发现的Lpa-miR-nov-66,它能通过结合靶基因抑制其表达而控制细胞内色素的合成[11]。miRNA可以参与细胞通路中的核心基因来实现对色素沉积的调节控制[12]。
本研究通过生物信息学软件预测发现,miR-96-5p对MITF具有结合作用,本试验在羊驼黑色素细胞中通过过表达和抑制表达miR-96-5p检测MITF及其下游基因的表达情况,且对其进行验证。
1 材料与方法 1.1 靶向MITF的miRNA生物信息学预测利用RAID(http://www.rna-society.org/raid/)、PicTar(https://pictar.mdc-berlin.de/)、TargetScan(http://www.targetscan.org/vert_72/)和miRanda(http://miranda.org.uk/)共4个在线软件预测与MITF基因有一定靶向关系的miRNAs。在RAID结构中选择与MITF靶向评分为0.9~1.0,筛选标准为“Strong Evidence”的靶向miRNAs,在PicTar结果中筛选与MITF评分大于9的靶向miRNAs。用Venny2.0.2对4个在线软件预测得出的miRNAs结果做韦恩图,进一步筛选调节MITF的miRNA。
1.2 羊驼黑色素细胞培养试验中所使用的羊驼黑色素细胞由山西农业大学羊驼生物工程实验室构建。本试验使用的是第6代羊驼黑色素细胞。
黑色素细胞从-80 ℃取出于37 ℃水浴复苏后转移入生物安全柜进行操作,迅速向冻存管中加入等量的含有10%胎牛血清的DMEM培养基(dulbecco’s minimum essential medium, Gibco, New York, USA),以1 000 r·min-1离心10 min,弃去上清液,将细胞沉淀悬浮在黑色素细胞培养基(MelM, ScienCell, Carlsbad, CA, USA)中。将细胞接种到6孔板中,在37 ℃、CO2浓度为5%的培养箱中培养。细胞复苏后,每隔24 h,用显微镜进行细胞形态变化以及生长状态的观察。状态良好时进行转染试验。
1.3 引物设计利用Primer 3.0在线设计软件,根据GenBank上小鼠的MITF、TYR、TYRP1、TYRP2序列设计引物,并且通过NCBI初步检测引物的特异性。以18S rRNA为内参基因。miR-96-5p的引物购自天根生化科技有限公司,以U6为内参基因,引物序列:5′-ATGGACTATCATATGCTTACCGTA-3′。基因的引物序列由擎科生物有限公司合成。引物序列见表 1。
|
|
表 1 荧光定量PCR引物 Table 1 Fluorescence quantitative PCR primers |
取出培养的黑色素细胞,弃去6孔板中的细胞培养液,用37 ℃预热的PBS缓冲液(索莱宝,中国)轻轻洗涤细胞3次,弃掉PBS。迅速在每孔滴加1 mL Trizol裂解液(Invitrogen, Carlsbad, CA, USA)。用微量移液器多次吹打细胞,力度要求稍大,尽可能地使细胞破碎,暴露出其中的核酸片段。收集6孔板中的Trizol裂解液到无RNase的1.5 mL无菌EP离心管中,根据Trizol Reagent试剂盒使用说明从黑色素细胞中提取总RNA,用1%琼脂糖凝胶电泳检测总RNA质量,并用NanoDrop 2000c分光光度计(NanoDrop Technologies,Wilmington,DE,USA)检测总RNA浓度及纯度。
1.5 miR-96-5p与MITF及其下游基因mRNA的表达量检测利用RT-PCR方法分别测定在羊驼黑色素细胞中过表达或抑制表达miR-96-5p及不转染miR-96-5p这3组中的MITF及其下游基因表达的情况。
1.5.1 miR-96-5p表达量的检测 使用miRcute miRNA cDNA合成试剂盒(天根,中国)对不同处理的羊驼黑色素细胞所提取到的总RNA样本分别进行逆转录。对Poly (A)修饰的miRNA进行反转录,体系:0.5 μL Specific primer (10 μmol·L-1),0.5 μL Reverse primer(10 μmol·L-1),2 μL 5× PrimeScript Buffer(for real-time),RNA≤500 ng·L-1,计算上样量并加样,0.5 μL PrimerScript RT Enzyme Mix I,加水至总体积为10 μL。反应程序:37 ℃ 15 min,85 ℃ 5 s,4 ℃保存。
将反转录产物稀释至50 ng·μL-1,作为荧光定量PCR的模板,荧光定量PCR的总体系定为10 μL:5 μL SYBR Premix Ex TaqTMⅡ(2×),0.8 μL miRNAs Specific primer(10 μmol·L-1),1 μL Template DNA,加水至10 μL。反应程序:95 ℃预变性30 s;PCR反应阶段95 ℃ 15 s,60 ℃ 34 s,共进行45个循环;熔解曲线分析阶段95 ℃ 15 s,60 ℃ 1 min,95 ℃ 15 s。
1.5.2 MITF、TYR、TYRP1、TYRP2基因mRNA表达量的检测 根据反转录试剂盒PrimerScript®RT reagent kit(Perfect real-time, 大连TaKaRa公司)的说明书,对各个总RNA样本进行反转录。
体系1:1.0 μL 5×gDNA Eraser Buffer,0.5 μL gDNA Eraser,Total RNA≤1 ng,计算上样量,加水至5 μL。PCR反应程序:42 ℃ 5 min,4 ℃保存。
体系2:体系1反应结束液中加入0.5 μL Prime Script RT Enzyme Mix I,2 μL 5×Prime Script Buffer 2(for Real time),0.5 μL RT Primer Mix,加水至10 μL。反应程序:37 ℃ 13 min,85 ℃ 5 s,4 ℃保存。
将反转录产物稀释至50 ng·μL-1,作为荧光定量PCR的cDNA模板,荧光定量PCR的体系为10 μL:5 μL SYBR Premix Ex TaqTMⅡ(2×),0.8 μL mRNA Specific primer (10 μmol·L-1),Template DNA 1 μL,加水至10 μL。反应程序:95 ℃预变性30 s;PCR反应阶段95 ℃ 15 s,60 ℃ 34 s,共进行45个循环;熔解曲线分析阶段95 ℃ 15 s,60 ℃ 1 min,95 ℃ 15 s。
1.6 免疫细胞化学技术检测MITF、TYR、TYRP1和TYRP2蛋白水平分别取正常组、过表达miR-96-5p组和抑制表达miR-96-5p组细胞样品使其贴壁于破碎的盖玻片上,对细胞进行4%多聚甲醛固定、3% H2O2室温孵育细胞15 min,PBS冲洗3次,每次3 min;滴加封闭液BSA封片,37 ℃,30 min;弃去封闭液后,用PBS稀释一抗:小鼠MITF单克隆抗体(Thermo Fisher Scientific),兔TYR、TYRP1和TYRP2单克隆抗体(Abcam, Cambridge, UK),小鼠β-actin单克隆抗体(TransGen Biotech, Beijing, China),稀释比例为1:100,4 ℃过夜孵育;16 h后取出玻片,置于室温30 min,用1×PBS清洗3次,每次5 min。用PBS将羊抗兔IgG抗体(Cwbio, Beijing, China)作为二抗稀释,稀释比例为1:100,加入适量稀释二抗,37 ℃恒温放置30 min;1×PBS清洗3次,每次5 min;取DAB染液中的A、B、C试剂各50 μL(ABC显色剂最好等量)于1 mL蒸馏水中充分混匀作为显色剂。向玻片上滴加稀释好的显色剂,迅速放置于倒置显微镜下观察,当玻片背景色变为黄色时,立刻用水冲洗;冲洗3次后,苏木精复染2 min;自来水冲洗2次,每次2 min;酒精梯度脱水;滴加100 μL的中性树脂,盖上载玻片进行封片在倒置显微镜下观察片子,用DP软件采集视野,每张片子采集3个视野。
1.7 数据统计分析全部数据采用SPSS 16.0进行统计分析,荧光实时定量PCR结果均用“平均值±标准差(Mean±SD)”表示。各目的基因相对表达量采用△△Ct法计算:目的基因的相对表达水平=2-△△Ct。用Image pro plus分析免疫组化的光密度,用统计学方法分析各试验组平均光密度之间的差异显著性。
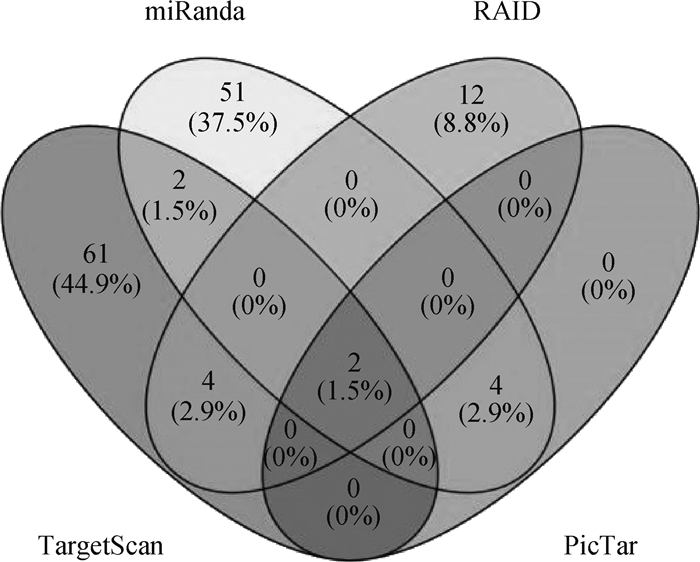
2 结果 2.1 与MITF具有靶向关系miRNAs的预测用RAID、TargetScan、miRanda和PicTar分别预测出与MITF具有靶向关系的miRNAs,用Venny2.0.2在线网站对4个靶基因预测得出的结果进行交互绘制,如图 1所示。4个软件中RAID预测到18个miRNAs,其中12个miRNAs单独在RAID软件中被预测到,4个与TargetScan共同预测到。TargetScan软件预测到69个与MITF具有靶向关系的miRNAs,其中61个miRNAs单独在TargetScan软件中被预测到,2个miRNAs在TargetScan和miRanda软件中被共同预测到,2个miRNAs在TargetScan、PicTar以及miRanda中被共同预测到。miRanda共预测到59个miRNAs,其中有51个miRNAs单独被该软件预测到,4个miRNAs在miRanda和PicTar软件中被共同预测到。2个miRNAs在4个预测软件中被共同预测到:miR-96-5p和miR-137。从各个软件的评分来看,miR-96-5p评分高于miR-137。本试验选择理论上更具有靶向关系的miR-96-5p进行验证。
|
图 1 预测的与MITF基因具有靶向关系的miRNAs Fig. 1 Predicted miRNAs targeting MITF |
体外培养的羊驼黑色素细胞在光学显微镜下观察到的状态见图 2,细胞两端有树状凸起,形状似梭形,细胞状态良好。
|
图 2 羊驼黑色素细胞(200×) Fig. 2 Alpaca melanocyte (200×) |
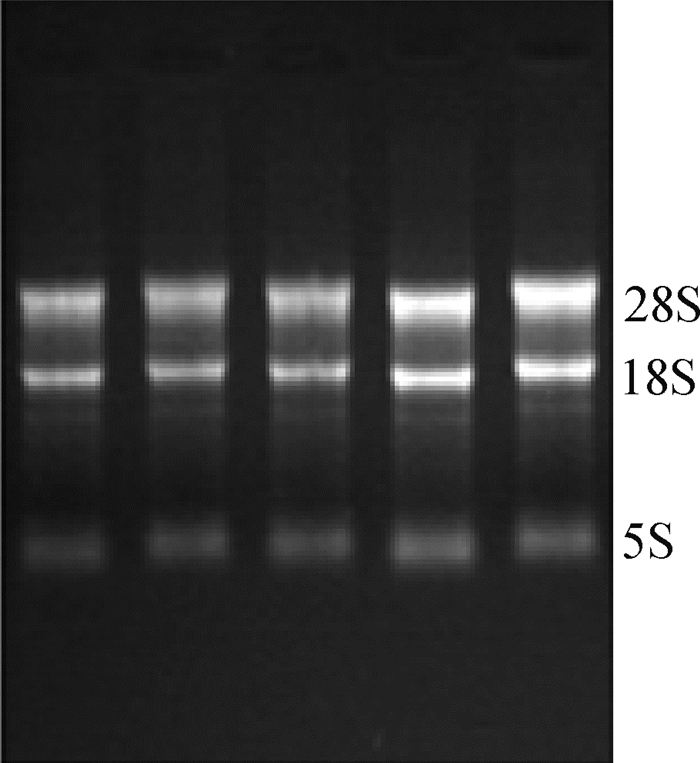
转染miR-96-5p系列物后所提取总RNA,所有样品28S、18S、5S 3条带完整,所有总RNA样品浓度为800~1 000 ng·μL-1,A260nm/A280nm在1.8~2.0之间,样品无DNA和蛋白污染(图 3),可用于后续试验。
|
图 3 RNA电泳图 Fig. 3 Electrophoresis of RNA |
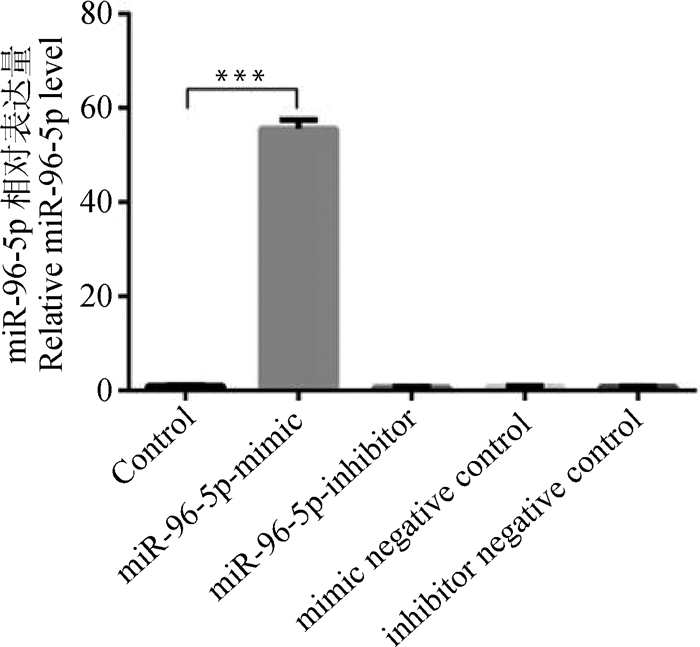
2.4.1 miR-96-5p表达量检测 转染miR-96-5p mimic组的miR-96-5p表达量是空白组的55.65倍,差异极显著(P < 0.001);其余几组miR-96-5p的表达水平与空白对照组相比基本接近,差异不显著(P>0.05,图 4)。
|
Control.空白组;miR-96-5p mimic.转染miR-96-5p mimic组;miR-96-5p inhibitor.转染miR-96-5p抑制物组;mimic negative control. mimic的阴性对照组;inhibitor negative control. inhibitor的阴性对照抑制组。*. P < 0.05;**. P < 0.01;***. P < 0.001,下同 Control. Blank group; miR-96-5p mimic. Transfecting miR-96-5p mimic mature group; miR-96-5p inhibitor. Transfecting miR-96-5p inhibitor group; mimic negative control. Mimic negative control group; inhibitor negative control. Inhibitor negative control. *. P < 0.05; **. P < 0.01; ***. P < 0.001, the same as below 图 4 miR-96-5p表达量检测 Fig. 4 Detection of miR-96-5p expression |
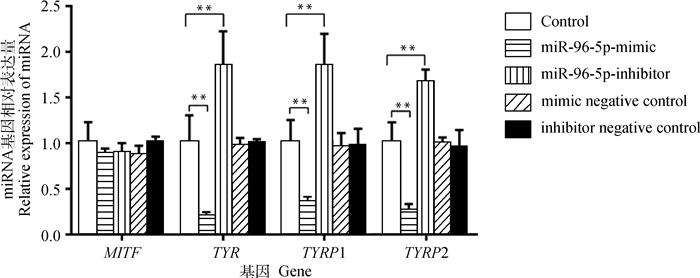
2.4.2 MITF、TYR、TYRP1、TYRP2 mRNA表达量检测 转染miR-96-5p mimic、inhibitor、mimic negative control和inhibitor negative control结果显示,转染组与空白组MITF的相对表达量差异不显著(P>0.05),表明过表达miR-96-5p并未影响MITF mRNA的表达量;转染miR-96-5p inhibitor抑制内源性miR-96-5p表达,也未影响MITF mRNA的表达水平,但是转染miR-96-5p mimic组TYR、TYRP1和TYRP2的相对表达量与空白组相比均极显著降低(P < 0.01);转染miR-96-5p inhibitor组TYR、TYRP1、TYRP2的相对表达量与空白组相比均极显著升高(P < 0.01);转染mimic negative control和inhibitor negative control组TYR、TYRP1和TYRP2的相对表达量与空白组差异不显著(P>0.05,图 5)。
|
图 5 毛色相关基因mRNA的相对表达 Fig. 5 Relative mRNA expression of coat color related genes |
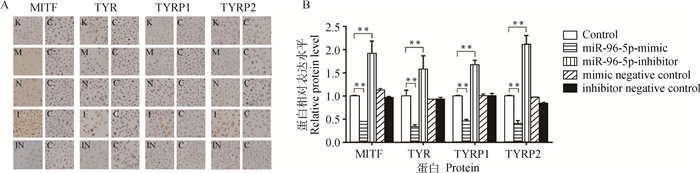
2.4.3 免疫细胞化学检测MITF、TYR、TYRP1和TYRP2蛋白的水平 免疫细胞化学检测结果显示,转染miR-96-5p mimic组MITF、TYR、TYRP1和TYRP2蛋白水平极显著低于空白组(P < 0.01);miR-96-5p inhibitor组MITF、TYR、TYRP1和TYRP2蛋白水平极显著高于空白组(P < 0.01);转染mimic negative control和inhibitor negative control组MITF、TYR、TYRP1和TYRP2的蛋白水平与空白组相比差异不显著(P>0.05,图 6)。
|
A.转染miR-96-5p系列物后各蛋白的表达(100×);B.转染miR-96-5p系列物后对各蛋白的光密度分析结果。K.空白组;M.转染miR-96-5p成熟体组;N. mimic的阴性对照组;I.转染miR-96-5p抑制剂组;IN. inhibitor的阴性对照抑制组;C.阴性对照组,用PBS代替一抗 A.The relative expression of each protein after transfecting the miR-96-5p series (100×); B. The results of optical density analysis of each protein after transfecting the miR-96-5p series. K. Blank group; M. Transfecting miR-96-5p mature group; N. Mimic negative control group; I. Transfecting miR-96-5p inhibitor group; IN. Inhibitor negative control group; C. Negative control group, PBS replaces the primary antibody 图 6 免疫细胞化学分析结果 Fig. 6 Analysis results of immunocytochemistry |
近几年研究发现,在哺乳动物中,黑色素的合成以及被毛颜色的差异与一些miRNA的作用有关。至今,miRNA在参与毛色调控方面的具体作用机制尚不清楚。miRNA在皮肤发育中有调控基因的作用,在皮肤上首次发现的miRNAs是miR-203,随后人们在鼠上发现了miR-203,它在表皮分化方面具有重要的调节作用[13-14]。本研究利用生物信息学预测了miR-96-5p的靶基因,发现MITF理论上可作为它的靶基因。miR-96是miR-182/183/96簇的成员之一,它对哺乳动物耳蜗毛细胞的分化、凋亡等生理功能均有影响[15]。人类DFN50家系遗传性耳聋和miR-96种子区突变存在共分离现象突变;miR-96杂合子小鼠在听力方面表现出渐进性下降,纯合子的小鼠却会发生耳蜗毛细胞退化[16]。MITF突变与听觉损伤有关,MITF等位基因突变会导致内耳缺陷[17]。研究发现,miR-96能够靶向MITF mRNA,过表达miR-96后,发现它可作为一种肿瘤抑制剂,主要通过下调MITF而抑制肿瘤生长,miR-96能够影响卵巢癌细胞的增殖和肿瘤发生[18]。可见,miR-96与MITF对机体的正常运行至关重要。
羊驼的MITF蛋白由419个氨基酸组成[19]。其基因结构分析显示,MITF蛋白存在多种亚型,分别命名为MITF-A、MITF-B、MITF-C、MITF-D、MITF-H、MITF-J、MITF-Mc、MITF-CM和MITF-M等[20-28]。这些氨基酸具有独特的氨基末端结构域M,在黑色素细胞、黑色素瘤细胞中大量表达;最长的一个亚型是由520个氨基酸组成的MITF-A,和MITF-M共享羧基端,主要表达于视网膜色素上皮细胞(retinal pigment epithelium,RPE);MITF-C由具有氨基末端半域C和B1b的519个氨基酸组成,它在许多类型的细胞中均表达;MITF-H由具有氨基酸末端半域H和半域B1b的519个氨基酸组成,与MITF-A在多种细胞中共同表达[29]。每一种亚型由不同的启动子启动,在不同的组织中表现出这些亚型的功能差异性。M启动子是黑色素细胞系中唯一一个功能性启动子,并且能通过LEF-1结合位点被Wnt信号通路上调。除此之外,M启动子还受到了一些转录因子的调控,例如SOX10、CREB和PAX3等。随着研究的深入,MITF对黑色素沉积的影响受到广泛关注[30-31]。
通过在小鼠黑色素细胞中转染miR-27a-3p mimic和miR-27a-3p inhibitor,发现miR-27a-3p对Wnt基因表达无显著作用,但对Wnt蛋白的表达具有抑制作用,并且抑制了黑色素合成[32],在绵羊黑色素细胞中转染pri-miR-27a,黑色素含量显著降低,二者说明miR-27-2a确实抑制黑色素生成[33]。在羊驼黑色素细胞中过表达miR-508-3p后,MITF在基因水平和蛋白水平上均下调,黑色素形成关键基因TYR和TYRP2表达也被下调[34]。在体外培养的羊驼黑色素细胞的试验中得到结果:miR-324-3p和miR-338-3p均可靶向结合MC1R基因从而抑制其基因及其下游黑色素合成相关基因(如MITF、TYR和TYRP2)的表达,最终达到抑制黑色素合成的目的,并且该研究发现, 棕色羊驼黑色素细胞中miR-324-3p的表达量高于白色羊驼黑色素细胞。该研究的结果进一步说明了miRNA与毛色形成有关[35-40]。本研究运用生物信息网站预测到miR-96-5p与MITF基因之间具有靶向位点,通过检测过表达和抑制表达miR-96-5p后羊驼黑色素细胞内MITF及其下游基因TYR、TYRP1和TYRP2的表达情况,证实了miRNA-96-5p可以靶向结合MITF基因达到抑制黑色素形成的目的。该试验为哺乳动物毛色调控提供了理论基础。
4 结论本研究结果表明,miR-96-5p可以靶向结合MITF基因,在MITF转录后水平发挥调控作用,抑制MITF蛋白的表达,抑制MITF及其下游基因TYR、TYRP1、TYRP2的表达。
| [1] | TACHIBANA M. MITF:a stream flowing for pigment cells[J]. Pigm Cell Res, 2000, 13(4): 230–240. DOI: 10.1034/j.1600-0749.2000.130404.x |
| [2] | HANNON G J, RIVAS F V, MURCHISON E P, et al. The expanding universe of noncoding RNAs[J]. Cold Spring Harb Symp Quant Biol, 2006, 71(12): 551–564. |
| [3] | LIN J Y, FISHER D E. Melanocyte biology and skin pigmentation[J]. Nature, 2007, 445(7130): 843–850. DOI: 10.1038/nature05660 |
| [4] |
杜斌, 许冬梅, 张亚玲, 等. miR-146a抑制酪氨酸酶基因家族在小鼠黑色素细胞中表达[J]. 中国生物化学与分子生物学报, 2017, 33(1): 81–87.
DU B, XU D M, ZHANG Y L, et al. miR-146a inhibits the expression of tyrosinase gene family in mouse melanocytes Cells[J]. Chinese Journal of Biochemistry and Molecular Biology, 2017, 33(1): 81–87. (in Chinese) |
| [5] | REINHART B J, SLACK F J, BASSON M, et al. The 21-nucleotide let-7 RNA regulates developmental timing in Caenorhabditis elegans[J]. Nature, 2000, 403(6772): 901–906. DOI: 10.1038/35002607 |
| [6] | LEE R C, FEINBAUM R L, AMBROS V. The C.elegans heterochronic gene lin-4 encodes small RNAs with antisense complementarity to lin-14[J]. Cell, 1993, 75(5): 843–854. DOI: 10.1016/0092-8674(93)90529-Y |
| [7] | O'DONNELL K A, WENTZEL E A, ZELLER K I, et al. c-Myc-regulated microRNAs modulate E2F1 expression[J]. Nature, 2005, 435(7043): 839–843. DOI: 10.1038/nature03677 |
| [8] | BARTEL D P. MicroRNAs:genomics, biogenesis, mechanism, and function[J]. Cell, 2004, 116(2): 281–297. DOI: 10.1016/S0092-8674(04)00045-5 |
| [9] |
蔡婷, 刘志红, 王志新, 等. miRNA在调控皮肤和毛囊发育中的作用[J]. 遗传, 2013, 35(9): 1087–1094.
CAI T, LIU Z H, WANG Z X, et al. miRNA in regulation of skin and hair follicle development[J]. Hereditas (Beijing), 2013, 35(9): 1087–1094. (in Chinese) |
| [10] | MICHAEL M Z, O'CONNOR S M, VAN HOLST PELLEKAAN N G, et al. Reduced accumulation of specific MicroRNAs in colorectal neoplasia11Note:Susan M.O' Connor and Nicholas G.van Holst Pellekaan contributed equally to this work[J]. Mol Cancer Res, 2003, 1(12): 882–891. |
| [11] |
杨姗姗.Lpa-miR-nov-66在羊驼黑色素细胞中的作用研究[D].晋中: 山西农业大学, 2014.
YANG S S.The function of Ipa-miR-nov-66 on the melanogenesis in melanocytes of alpaca (Lama pacos)[D].Jinzhong: Shanxi Agricultural University, 2014.(in Chinese) http://cdmd.cnki.com.cn/Article/CDMD-10113-1014414579.htm |
| [12] |
许冬梅, 赵宇军, 杜斌, 等. miR-186-5p对绵羊黑色素细胞迁移增殖的影响[J]. 畜牧兽医学报, 2017, 48(11): 2068–2075.
XU D M, ZHAO Y J, DU B, et al. The influences of miR-186-5p on migration and proliferation of sheep melanocytes[J]. Acta Veterinaria et Zootechnica Sinica, 2017, 48(11): 2068–2075. DOI: 10.11843/j.issn.0366-6964.2017.11.007 (in Chinese) |
| [13] | NISSAN X, DENIS J A, SAIDANI M, et al. miR-203 modulates epithelial differentiation of human embryonic stem cells towards epidermal stratification[J]. Dev Biol, 2011, 356(2): 506–515. DOI: 10.1016/j.ydbio.2011.06.004 |
| [14] | GODING C R. Mitf from neural crest to melanoma:signal transduction and transcription in the melanocyte lineage[J]. Genes Dev, 2000, 14(14): 1712–1728. |
| [15] | SACHELI R, NGUYEN L, BORGS L, et al. Expression patterns of miR-96, miR-182 and miR-183 in the development inner ear[J]. Gene Expr Patterns, 2009, 9(5): 364–370. DOI: 10.1016/j.gep.2009.01.003 |
| [16] |
王耀文, 程鹏, 包卫亮, 等. 不同月龄BALB/c小鼠耳蜗miR-96表达与ABR阈值及耳蜗形态的关系[J]. 中国耳鼻咽喉头颈外科, 2017, 24(6): 296–300.
WANG Y W, CHENG P, BAO W L, et al. Relationship between the miR-96 expression in the cochlea and ABR threshold and cochlear morphology of BALB/c mice[J]. Chinese Archives of Otolaryngology- Head and Neck Surgery, 2017, 24(6): 296–300. (in Chinese) |
| [17] |
郭维, 丛涛, 杨仕明.Mitf-m突变对耳蜗血管纹中间细胞发育的影响[C]//中华医学会第十三次全国耳鼻咽喉头颈外科学术会议论文集.长沙: 中华医学会, 2013.
GUO W, CONG T, YANG S M.Effect of Mitf-m mutation on the development of intermediate cells in cochlea striatum[C]//Chinese Medical Association National Academic Conference on Otolaryngology - Head and Neck Surgery. Changsha: Chinese Medical Association, 2013.(in Chinese) |
| [18] | XU X F, DONG Z G, LI Y M, et al. The upregulation of signal transducer and activator of transcription 5-dependent microRNA-182 and microRNA-96 promotes ovarian cancer cell proliferation by targeting forkhead box O3 upon leptin stimulation[J]. Int J Biochem Cell Biol, 2013, 45(3): 536–545. DOI: 10.1016/j.biocel.2012.12.010 |
| [19] | LEVY C, KHALED M, FISHER D E. MITF:master regulator of melanocyte development and melanoma oncogene[J]. Trends Mol Med, 2006, 12(9): 406–414. DOI: 10.1016/j.molmed.2006.07.008 |
| [20] | TACHIBANA M. MITF:a stream flowing for pigment cells[J]. Pigm Cell Res, 2000, 13(4): 230–240. DOI: 10.1034/j.1600-0749.2000.130404.x |
| [21] | IDELSON M, ALPER-PINUS R, OBOLENSKY A, et al.Stem cell-derived retinal pigment epithelial cells: CN CN101688178[P]. 2008-04-27. |
| [22] | PARK H Y, GILCHREST B A. More on MITF[J]. J Invest Dermatol, 2002, 119(6): 1218–1219. DOI: 10.1046/j.1523-1747.2002.19651.x |
| [23] | UITTO J, ATANASOVA V S, JIANG Q J, et al. Precision medicine for heritable skin diseases-the paradigm of Epidermolysis Bullosa[J]. J Invest Dermatol Sympos Proc, 2018, 19(2): S74–S76. DOI: 10.1016/j.jisp.2018.09.004 |
| [24] | TAKEDA K, YASUMOTO K I, KAWAGUCHI N, et al. Mitf-D, a newly identified isoform, expressed in the retinal pigment epithelium and monocyte-lineage cells affected by Mitf mutations[J]. Biochim Biophys Acta Gene Struct Expr, 2002, 1574(1): 15–23. DOI: 10.1016/S0167-4781(01)00339-6 |
| [25] | ASAI K, FUNABA M, MURAKAMI M. Enhancement of RANKL-induced MITF-E expression and osteoclastogenesis by TGF-β[J]. Cell Biochem Funct, 2014, 32(5): 401–409. |
| [26] | SAITO H, TAKEDA K, YASUMOTO K I, et al. Germ cell-specific expression of microphthalmia-associated transcription factor mRNA in mouse testis[J]. J Biochem, 2003, 134(1): 143–150. |
| [27] | HERSHEY C L, FISHER D E. Genomic analysis of the Microphthalmia locus and identification of the MITF-J/Mitf-J isoform[J]. Gene, 2005, 347(1): 73–82. |
| [28] | SHIOHARA M, SHIGEMURA T, SUZUKI T, et al. MITF-CM, a newly identified isoform of microphthalmia-associated transcription factor, is expressed in cultured mast cells[J]. Int J Lab Hematol, 2009, 31(2): 215–226. DOI: 10.1111/j.1751-553X.2008.01028.x |
| [29] | LEE S H, LEE J H, LEE Y M, et al. Involvement of MITF-A, an alternative isoform of mi transcription factor, on the expression of tryptase gene in human mast cells[J]. Exp Mol Med, 2010, 42(5): 366–375. DOI: 10.3858/emm.2010.42.5.038 |
| [30] | DYNEK J N, CHAN S M, LIU J F, et al. Microphthalmia-associated transcription factor is a critical transcriptional regulator of melanoma inhibitor of apoptosis in melanomas[J]. Cancer Res, 2008, 68(9): 3124–3132. DOI: 10.1158/0008-5472.CAN-07-6622 |
| [31] | HARTMAN M L, CZYZ M. MITF in melanoma:mechanisms behind its expression and activity[J]. Cell Mol Life Sci, 2015, 72(7): 1249–1260. DOI: 10.1007/s00018-014-1791-0 |
| [32] | LIANG J T, TANG J M, SHI H J, et al. miR-27a-3p targeting RXRα promotes colorectal cancer progression by activating Wnt/β-catenin pathway[J]. Oncotarget, 2017, 8(47): 82991–83008. DOI: 10.18632/oncotarget.19635 |
| [33] |
赵园园.MiR-27a-3p对黑色素生成的作用及机制研究[D].晋中: 山西农业大学, 2015.
ZHAO Y Y.Study of the function and mechanism of mir-27a-3p on melanogenesis in mouse and sheep[D].Jinzhong: Shanxi Agricultural University, 2015. (in Chinese) http://cdmd.cnki.com.cn/Article/CDMD-10113-1016056726.htm |
| [34] | ZHANG J, LIU Y, ZHU Z, et al. Role of microRNA-508-3p in melanogenesis by targeting microphthalmia transcription factor in melanocytes of alpaca[J]. Animal, 2017, 11(2): 236–243. DOI: 10.1017/S1751731116001294 |
| [35] |
曾庆宝.miR-324-3p对羊驼黑素刺激素受体1基因功能的影响[D].晋中: 山西农业大学, 2016.
ZENG Q B.The effect of function for melanocortin-1 receptor gene regulated by mir-324-3p from Melanocytes of Alpaca[D]. Jinzhong: Shanxi Agricultural University, 2016.(in Chinese) |
| [36] |
武良琦, 范瑞文, 曾庆宝, 等. miRNA-338-3p通过靶向抑制MC1R基因表达抑制羊驼黑色素细胞黑色素的生成[J]. 中国生物化学与分子生物学报, 2017, 33(6): 624–629.
WU L Q, FAN R W, ZENG Q B, et al. miRNA-338-3p inhibits Melanogenesis in Alpaca skin melanocytes by targeting MC1R[J]. Chinese Journal of Biochemistry and Molecular Biology, 2017, 33(6): 624–629. (in Chinese) |
| [37] |
曾庆宝, 范瑞文, 张秋月, 等. miR-324-3p通过调控MC1R基因影响黑色素的形成[J]. 畜牧兽医学报, 2016, 47(7): 1381–1388.
ZENG Q B, FAN R W, ZHANG Q Y, et al. The effect of miR-324-3p on melanin synthesis via MC1R gene[J]. Acta Veterinaria et Zootechnica Sinica, 2016, 47(7): 1381–1388. (in Chinese) |
| [38] | CHEN X Q, FENG D, SCHWARZSCHILD M A, et al. Red hair, MC1R variants, and risk for Parkinson's disease-a meta-analysis[J]. Ann Clin Trans Neurol, 2017, 4(3): 212–216. |
| [39] | YANG C W, RAN J S, YU C L, et al. Polymorphism in MC1R, TYR and ASIP genes in different colored feather chickens[J]. 3 Biotech, 2019, 9(5): 203. DOI: 10.1007/s13205-019-1710-z |
| [40] | DUFFY D L, LEE K J, JAGIRDAR K, et al. High Naevus count and MC1R red hair alleles contribute synergistically to increased melanoma risk[J]. Bri J Dermatol, 2019, 181(5): 1009–1016. DOI: 10.1111/bjd.17833 |



