骨骼肌作为人类和动物躯体的重要组成部分,约占其总体重的40%[1],猪肉作为人类所食用的重要肉类产品来源,能提供给人类优质的蛋白质,并含有丰富的营养物质,又因为其产肉性状是非常重要的经济性状,因此研究意义重大。骨骼肌卫星细胞是骨骼肌形成的基本单位[2],当肌肉组织受损时,其本身是不能形成新的骨骼肌纤维[3],而主要是通过骨骼肌卫星细胞来主导肌肉再生[4]。骨骼肌的生长发育过程由多种生长因子和遗传环境因素共同影响,尽管骨骼肌发育的分子机制还并不完全明了,但骨骼肌发育相关的功能基因一直是研究的重点。
大量的研究数据表明,长链非编码RNA(long non-coding RNA,lncRNA)是骨骼肌生长发育的重要调控因子[5],参与骨骼肌细胞增殖和分化的多个过程[6]。lncRNA-133a通过某种基因的互作机制促进牛骨骼肌卫星细胞的增殖和分化[7],lncRNA Snhg8能够通过调控核糖体上某种蛋白基因的转录来调节骨骼肌卫星细胞的增殖过程[8],lincRNA-MD1被miRNA-206/133b在其位点处编码,主要在分化成肌细胞的细胞质中高表达,通过竞争性结合miR-133和miR-135分别把肌肉特异性转录调控因子MAML1和MEF2C激活,从而促进肌肉细胞的分化[9]。在肌肉发育过程中,有关lncRNAs的调控机制研究已有很多,但这些研究大多集中在小鼠和其它一些模式动物上,在猪骨骼肌发育过程中调控和作用机制的研究还很少。
本研究利用Tang等[10]通过生物信息学在猪骨骼肌组织中鉴定出来的长链非编码RNA TCONS_00791383,通过组织表达谱分析,并以在体外增殖和分化的猪骨骼肌卫星细胞为模型,通过在猪骨骼肌卫星细胞中敲低TCONS_00791383,探索其对猪骨骼肌卫星细胞增殖与分化的影响,为进一步研究该lncRNA在猪骨骼肌发育过程中的作用机理奠定重要基础。
1 材料与方法 1.1 试验材料1.1.1 细胞与试验动物 试验选取3头同一胎次、健康状况良好、出生7 d内的大白仔猪(购自华中农业大学种猪场),采集心、脾、肺、肾、背肌和后腿肌肉6种组织。所有组织样品采集后,经液氮速冻,保存于-80 ℃备用。
本试验所用猪骨骼肌卫星细胞分离自上述大白仔猪的腿肌组织。
1.1.2 主要仪器与试剂 主要仪器:CO2细胞培养箱(美国Thermo Scientific公司);荧光定量PCR仪(美国Bio-Rad公司);NanoDrop 2000分光光度计(美国Thermo Scientific公司);体视荧光显微镜(日本Olympus Corporation公司);CoolCell细胞冻存盒(Biocision公司);凝胶成像系统(美国Bio-Rad公司);DYY-6C型电泳仪(北京市六一仪器厂);台式高速离心机及高速冷冻离心机(Eppendorf公司);HH-6数显恒温水浴锅(常州澳华仪器有限公司)等。
主要试剂:RNA提取试剂盒(Epigentek公司);SMARTerTM PCR cDNA Synthesis Kit(TaKaRa公司);dNTP、LaTaq酶、rTaq酶、GXL酶(TaKaRa公司);Taq Plus MasterMix酶(康为世纪公司);UltraSYBR Mixture(康为世纪公司);DMEM、胰蛋白酶(0.25%)、PBS、RPMI1640、CEE、GlutaMax、NEAA、bFGF(美国GIBCO BRL公司);胎牛血清(FBS,杭州四季青公司);LipofectamineTM2000转染试剂(Invitrogen公司)等。
1.2 试验方法1.2.1 引物设计与合成 TCONS_00791383的序列参考文献[10],MyoD、MyoG、MyHC、Myf5、Pax7和18S rRNA基因的mRNA序列由NCBI数据库获得。本试验采用Primer Premier 5软件设计引物,用于实时荧光定量PCR。引物序列由生工生物工程(上海)股份有限公司合成。引物信息详见表 1。
|
|
表 1 本研究所用的引物序列 Table 1 The primer sequences used in this study |
1.2.2 猪组织表达谱构建 从-80 ℃冰箱中取出采集的大白仔猪6种组织,提取总RNA,用Nano -Drop 2000分光光度计把提取的总RNA进行质量和浓度检测,然后反转录成cDNA,通过qRT-PCR技术检测TCONS_00791383在这6种组织中的表达量。以18S rRNA为内参进行定量PCR,反应体系:SYBR Green 5 μL,H2O 3.6 μL,F-Primer 0.2 μL,R-Primer 0.2 μL,cDNA 1 μL。反应条件:95 ℃ 2 min;(95 ℃ 15 s,60 ℃ 20 s,72 ℃ 15 s)40个循环;95 ℃ 5 s;(58 ℃ 5 s,95 ℃ 50 s)1个循环。
1.2.3 猪骨骼肌卫星细胞的培养 从液氮罐中取出实验室前期冻存的猪原代骨骼肌卫星细胞,放入已经预热的37 ℃水浴锅中不断搅动,使细胞迅速融化。然后1 000 r·min-1离心5 min,用75%的酒精棉擦拭冻存管壁(避免污染),把复苏好的细胞加入1 mL增殖培养基并轻轻吹打混匀,待细胞充分悬浮后,将其转移到10 cm培养皿中,并向培养皿中再加入9 mL增殖培养基。上下左右摇晃培养皿,使细胞充分分散,然后将培养皿放入37 ℃,5% CO2培养箱中培养,24 h换液1次。当细胞长满皿底,更换成分化培养基, 放于37 ℃,5% CO2培养箱培养。
1.2.4 猪骨骼肌卫星细胞增殖分化前后TCONS_00791383的表达量检测 本研究在猪骨骼肌卫星细胞增殖6、12、18、30、36和42 h以及分化6、12、24、30、36和42 h时提取卫星细胞的总RNA,反转录成cDNA,采用qRT-PCR技术检测猪骨骼肌卫星细胞增殖分化前后TCONS_00791383的表达量,反应条件和体系同“1.2.1”。
1.2.5 猪骨骼肌卫星细胞的转染以及干扰效果检测 首先设计合成TCONS_00791383的siRNA,将siRNA与其对照组(NC)严格按照本试验所需要的浓度转染猪骨骼肌卫星细胞。细胞接种在底面直径10 cm的细胞培养皿中,待细胞密度达到60%以上后接种到6孔板中,待细胞长到80%左右时进行转染。转染步骤:转染前1 d,将适当数量的细胞用2 mL生长培养基接种到6孔细胞培养板,以便在转染时达到合适的细胞密度;将200 pmol siRNA加到250 μL Opti中,轻轻吹打混匀,室温孵育5 min;将10 μL LipofectamineTM2000加到Opti中,轻轻吹打混匀;将上述液体混合并吹打均匀,室温孵育20 min;等待过程中,将细胞培养板内原来的培养基去掉,加入1.5 mL Opti培养基;将上述混合溶液加入到相应的细胞培养孔内,轻轻摇晃,使试剂充分混匀;将细胞放入37 ℃ CO2培养箱中培养,转染4~6 h后换为新鲜增殖培养基,24 h后更换为分化培养基。细胞诱导分化后24及48 h时收集细胞提取总RNA,反转录成cDNA,通过qRT-PCR技术检测敲低TCONS_00791383后其在猪骨骼肌卫星细胞分化24和48 h的表达量。通过网站(http://sfold.wadsworth.org/cgi-bin/index.)在线设计的敲低lncRNA TCONS_00791383的ASO片段,由生工生物工程(上海)股份有限公司进行全链硫代修饰。ASO序列为GGATCTGGATTATGAACTTG。
1.2.6 猪TCONS_00791383的核质定位 本试验以诱导分化24和48 h的猪骨骼肌卫星细胞为样本,采用核质分离的技术分离其细胞质和细胞核RNA,把RNA反转录成cDNA,运用qRT-PCR技术检测TCONS_00791383在猪骨骼肌卫星细胞细胞质和细胞核的表达量。
1.2.7 TCONS_00791383靶基因预测和功能富集 该试验通过trans(co-expression)对TCONS_00791383进行靶基因预测,然后通过其与蛋白编码基因的表达量来计算Pearson相关系数,把Pearson相关系数|r|>0.95的蛋白编码基因筛选出来,通过Ensemble把这些基因转换为人类同源基因[11],使用DAVID进行GO富集和KEGG通路分析(P < 0.05)[12]。
1.2.8 统计分析 qRT-PCR结果参照2-△△Ct方法[13]来评估TCONS_00791383相对表达量,以18S rRNA作为内参基因进行待测基因表达差异的检测,*表示P < 0.05,差异显著;**表示P < 0.01和***表示P < 0.001, 差异极显著。
2 结果 2.1 TCONS_00791383在猪不同组织中的表达谱本试验采用qRT-PCR技术对心、脾、肺、肾、背肌和腿肌中TCONS_00791383的表达情况进行了定量分析,结果显示,其在不同组织中表达量的高低很不一致,在心脏中的表达量最高,其次是肌肉,在脾和肾组织中不表达(图 1)。
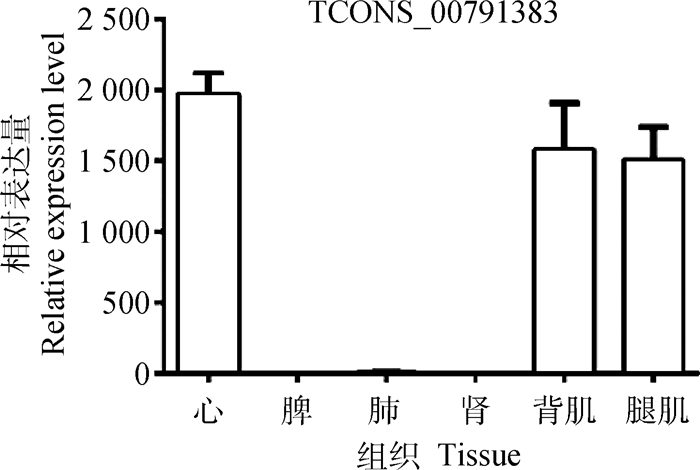
|
图 1 TCONS_00791383在猪各组织中的表达 Fig. 1 TCONS_00791383 expression in various tissues of pigs |
本研究分别提取猪骨骼肌卫星细胞在增殖6、12、18、30、36、42 h以及诱导分化后6、12、24、30、36、42 h猪骨骼肌卫星细胞总RNA,接着反转录成cDNA,通过qRT-PCR检测TCONS_00791383在增殖和分化不同时间点的表达量。结果如图 2所示,与猪骨骼肌卫星细胞增殖期相比,TCONS_00791383在骨骼肌卫星细胞分化期的表达量呈上升趋势,且在分化30 h表达量达到最高,然而在分化36和42 h,表达量又有下降的趋势,提示该lncRNA可能参与猪骨骼肌卫星细胞的增殖和分化过程。
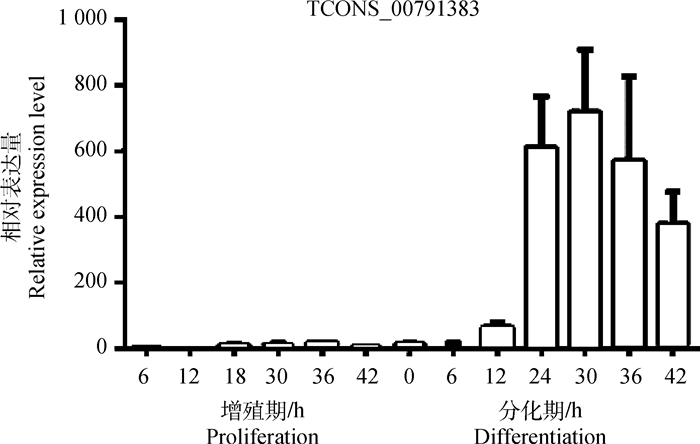
|
图 2 TCONS_00791383在猪骨骼肌卫星细胞增殖分化过程中的表达 Fig. 2 Expression of TCONS_00791383 in the process of proliferation and differentiation of porcine skeletal muscle satellite cells |
利用核质分离技术检测到TCONS_00791383在猪骨骼肌卫星细胞的核质分布情况,结果见图 3。核质分离结果显示,TCONS_00791383在骨骼肌卫星细胞核中的表达量极显著高于细胞质中的表达量(P < 0.01),说明该基因主要分布于猪骨骼肌卫星细胞的细胞核内。已有报道显示[14],lncRNA主要分布在细胞核内,该核质分离结果也与已知的报道相符,验证了核质分离结果的准确性。
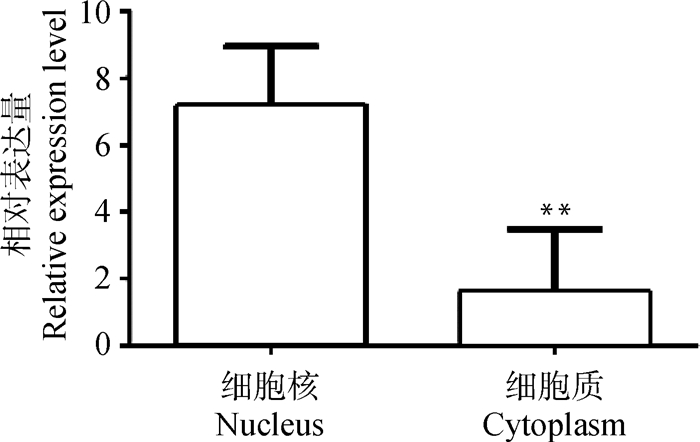
|
**. P < 0.01 图 3 TCONS_00791383的核质分布情况 Fig. 3 Distribution of TCONS_00791383 in the nucleus and cytoplasm |
通过对TCONS_00791383在猪不同组织以及骨骼肌卫星细胞增殖分化中的表达情况分析,发现该lncRNA很有可能在猪骨骼肌卫星细胞的增殖分化阶段发挥重要作用。并且验证了该lncRNA主要分布在猪骨骼肌卫星细胞的细胞核内,因此,本研究设计了特异性ASO片段转染猪骨骼肌卫星细胞,对该lncRNA进行敲低。在诱导分化24和48 h后分别收集细胞,提取细胞总RNA,然后进行反转录,采用qRT-PCR方法检测TCONS_00791383的表达情况(图 4)。结果表明,ASO片段具有敲低TCONS_00791383的效果,且在诱导分化24 h后,极显著降低了TCONS_00791383的表达水平(P < 0.001)。同样在诱导分化48 h后,TCONS_00791383的表达水平也极显著降低(P < 0.01),说明该ASO片段可用于TCONS_00791383的表达调控分析。
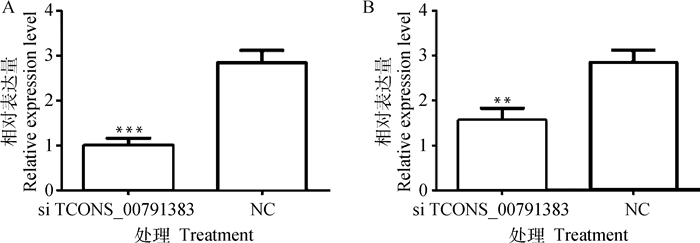
|
**. P < 0.01,***. P < 0.001。下同 **.P < 0.01, ***. P < 0.001. The same as below 图 4 诱导分化24(A)和48 h(B)后TCONS_00791383的敲低效果检测 Fig. 4 Detection of knock-down effect of TCONS_00791383 after induced differentiation for 24 (A) and 48 h (B) |
为了检测TCONS_00791383表达水平的改变对猪骨骼肌卫星细胞增殖分化能力的影响,本研究利用ASO片段转染猪骨骼肌卫星细胞敲低TCONS_00791383表达。分别在诱导分化24和48 h后收集细胞,用qRT-PCR检测骨骼肌卫星细胞增殖标记因子Pax3、Pax7和Myf5以及分化标记因子MyoD、MyoG和MyHC的表达情况(图 5)。结果表明,在分化24 h,增殖标志基因Pax3和Pax7表达量显著或极显著降低(P < 0.05, P < 0.01),分化标志基因MyoG表达量极显著降低(P < 0.01)。在分化48 h,增殖标志基因Pax3表达量极显著降低(P < 0.01),Pax7表达量显著降低(P < 0.05),分化标志基因MyHC表达量显著降低(P < 0.05)。此结果说明TCONS_00791383既可能促进细胞增殖,也可能促进细胞分化。
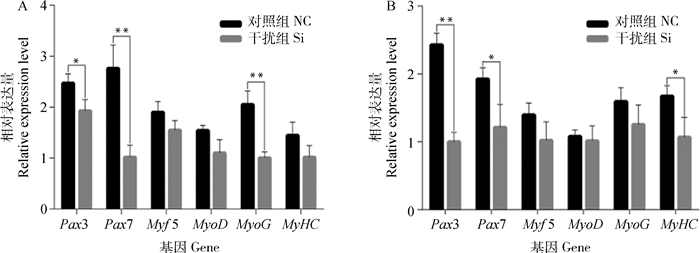
|
图 5 敲低TCONS_00791383后诱导分化24(A)和48 h(B)相关标志基因表达量变化 Fig. 5 The expression of related marker genes after knocking down TCONS_00791383 for 24 (A) and 48 h (B) of induced differentiation |
为了研究该lncRNA功能,通过计算TCONS_00791383与其他蛋白编码基因的表达量相关性,获得TCONS_00791383的多个靶基因。然后,通过DAVID对这些靶基因做GO聚类分析和KEGG通路分析。DAVID结果显示,TCONS_00791383的靶基因参与了27个生物学过程,包括线粒体电子传递、NADH到辅酶Q、转录、DNA模板化和tRNA代谢过程等18个通路,包括AMPK信号通路、类风湿性关节炎和帕金森病等通路(P < 0.05),部分结果见图 6。通过这些数据结果可以推断出TCONS_00791383的靶基因可能调控猪体内的多个生物学过程。
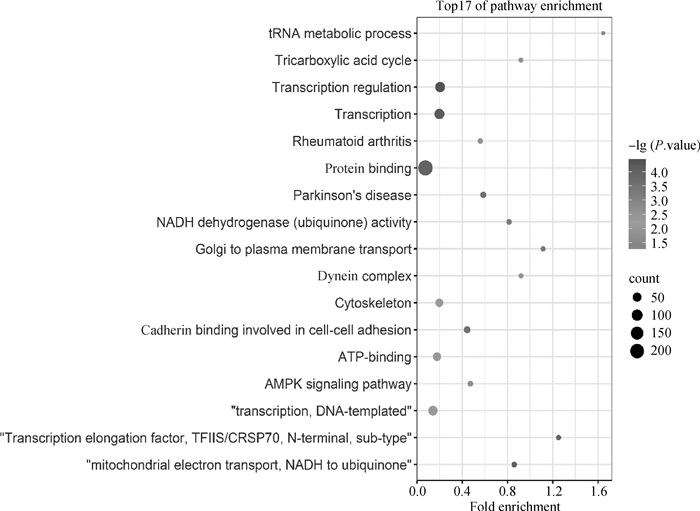
|
图 6 猪TCONS_00791383相关靶基因的GO和通路分析 Fig. 6 GO and pathway analysis for target genes of pig TCONS_00791383 |
lncRNA是指转录本长度超过200 nt,外显子个数大于等于2的一类不具有编码能力的RNA分子,在转录调控、表观遗传调控以及转录后调控等过程中发挥重要作用[15-18]。lncRNA曾经一度被认为是基因转录的“噪音”,不具有生物学意义[19]。Okazaki等[20]在2002年通过小鼠全长cDNA测序技术发现了lncRNA。随着高通量测序技术的快速发展,越来越多lncRNAs被鉴定出来[21-23],lncRNA功能研究也丰富起来。在肌肉发育方面,Wang等[24]发现,lncRNA Dum的表达是呈现动态变化的,它能够对成肌细胞分化进行转录诱导。这个过程是通过其结合MyoD来完成的,并且它能够促进受损伤肌肉组织的再生以及成肌细胞的分化。Zou等[25]利用RNA测序,在猪腿肌的转录组数据中鉴定了323个lncRNAs,并探究了lncRNA甲基化对猪肌肉发育的影响。Luo等[26]分析探究发现,linc-2949和linc-1369由c-Myc直接调节,并且两种lincRNA通过竞争与肌肉分化相关miRNA的结合来参与成肌细胞增殖和分化的调节。Song等[27]研究表明,linc-smad7在成肌细胞分化的早期阶段被上调。体外研究发现,linc-smad7的过表达促进了成肌细胞在G1期的停滞,抑制了DNA复制,并诱导了成肌细胞的分化。已有大量的研究结果说明,在骨骼肌细胞增殖分化过程中lncRNA的确发挥了非常重要的调控功能。
大多数哺乳动物基因组被转录为非编码RNA(ncRNA),例如microRNA、circRNA和lncRNA等。其中,lncRNA是功能最多样化的类别之一,在调节肌肉细胞发育过程中发挥重要作用[28]。利用Tang等[10]在实验室前期通过生物信息学分析鉴定出的一个肌肉增殖分化相关lncRNA TCONS_00791383,本研究通过qRT-PCR技术检测了TCONS_00791383在1周龄健康大白仔猪各个组织中的表达情况,结果显示,TCONS_00791383在各个组织的表达情况差别较大,在心和骨骼肌中显著高表达,在脾和肾组织中不表达,该结果说明,TCONS_00791383具有组织表达特异性,证明其很有可能在肌肉发育过程中发挥作用。猪骨骼肌卫星细胞是研究骨骼肌发育规律的理想材料[29],且已有研究表明,linc-MD1[9]、Malat1[30]和DBE-T[31]等lincRNAs能够促进骨骼肌细胞分化,为了进一步确认这种猜想,本研究分离培养了猪骨骼肌卫星细胞,检测了TCONS_00791383在其增殖及分化不同时间点的表达变化情况。结果表明,TCONS_00791383在卫星细胞从增殖到分化的过程中表达量呈逐渐上升的趋势,且在增殖期表达量很低,在分化30 h达到最高表达量。这些证据表明,TCONS_00791383很有可能在肌肉分化末期发挥调控功能。此外,lncRNA的亚细胞定位决定了其功能和机制[32]。本研究通过核质分离技术得出,TCONS_00791383主要分布在细胞核内,表明TCONS_00791383很可能在转录水平调节肌生成或通过重塑染色质来影响肌细胞生成[33]。
为了研究TCONS_00791383在肌肉发育中的作用,本研究利用特异性ASO片段转染猪骨骼肌卫星细胞,进而对TCONS_00791383进行敲低。在骨骼肌卫星细胞增殖分化的过程中,MyoD、MyoG、MyHC以及Pax3和Pax7这些骨骼肌增殖分化特异性标记因子已经开始表达[34]。众所周知,MyoD能够启动骨骼肌细胞的分化[35],并且MyoD调控MyoG表达的发生。此外,MyoG能够调控肌纤维的形成以及骨骼肌细胞的融合[36],MHC是骨骼肌粗肌丝的主要成分[37]。本研究应用qRT-PCR检测了上述5个增殖分化标记基因的表达情况,来验证骨骼肌细胞增殖分化的进程,结果表明,在敲低TCONS_00791383之后,增殖标志基因Pax3和Pax7以及分化标志基因MyoG和MyHC表达量显著降低,推断TCONS_00791383既能促进细胞增殖也能促进细胞分化。本研究结果可为研究猪骨骼肌发育和肌细胞增殖分化中的lncRNA调控机制提供参考。
反式调控是研究lncRNA功能的重要方法之一,于是本研究通过trans调控作用对TCONS_00791383进行靶基因预测,通过GO和KEGG富集分析发现,相关靶基因例如PIK3CD、RHEB和CRTC2等参与腺苷酸激活蛋白激酶(AMPK)信号通路。AMPK在细胞能量稳态调节中起到关键作用,AMPK对消耗ATP的生物合成过程具有负向调控作用,包括糖异生、脂质和蛋白质合成等过程,进而影响骨骼肌的伸缩和舒张[38]。已有报道显示,CRTC2在通过CREB/CRTC2信号通路的代谢和昼夜节律之间的动态相互作用中起着重要作用,进而间接促进肌肉的生长发育[39]。NDUFS7、VDAC3、NDUFS1和SLC25A4等靶基因富集到了帕金森病(PD)通路。帕金森病又名震颤麻痹,是最常见的神经退行性疾病之一,有肌强直症状,进而引起骨骼肌痉挛[40]。因此,推测TCONS_00791383可能通过调控其潜在的靶基因间接调控骨骼肌的生长和发育过程,但是TCONS_00791383与预测的潜在靶基因的靶向关系需要进一步验证。
4 结 论本研究通过组织表达谱检测发现,TCONS_00791383在心和骨骼肌中表达量最高;然后以猪骨骼肌卫星细胞为研究模型,发现TCONS_00791383在细胞增殖期表达量不断上升,并且在分化30 h表达量达到最高,然而在分化36和42 h表达量又有下降的趋势; 通过核质分离技术进行细胞定位,显示该lncRNA主要在细胞核内表达;使用ASO片段敲低TCONS_00791383表达后,细胞增殖分化标志基因的变化表明,TCONS_00791383既能促进猪骨骼肌卫星细胞增殖,也能促进其分化。本试验对于TCONS_00791383在猪骨骼肌卫星细胞增殖和分化中作用机制的深入研究提供了一定的参考依据。
| [1] |
李伯江, 李平华, 吴望军, 等. 骨骼肌肌纤维形成机制的研究进展[J]. 中国农业科学, 2014, 47(6): 1200–1207.
LI B J, LI P H, WU W J, et al. Progresses in research of the mechanisms of skeletal muscle fiber formation[J]. Scientia Agricultura Sinica, 2014, 47(6): 1200–1207. DOI: 10.3864/j.issn.0578-1752.2014.06.017 (in Chinese) |
| [2] | ANDERSON D M, ANDERSON K M, CHANG C L, et al. A micropeptide encoded by a putative long noncoding RNA regulates muscle performance[J]. Cell, 2015, 160(4): 595–606. DOI: 10.1016/j.cell.2015.01.009 |
| [3] | AKHTAR M M, MICOLUCCI L, ISLAM M S, et al. Bioinformatic tools for microRNA dissection[J]. Nucleic Acids Res, 2016, 44(1): 24–44. DOI: 10.1093/nar/gkv1221 |
| [4] | MEGENEY L A, KABLAR B, GARRETT K, et al. MyoD is required for myogenic stem cell function in adult skeletal muscle[J]. Genes Dev, 1996, 10(10): 1173–1183. DOI: 10.1101/gad.10.10.1173 |
| [5] |
魏彩虹, 吴明明, 刘瑞凿, 等. 肌肉发育相关LncRNA的研究进展[J]. 中国农业科学, 2014, 47(20): 4078–4085.
WEI C H, WU M M, LIU R Z, et al. Research progress in muscular growth and development of long noncoding RNAs[J]. Scientia Agricultura Sinica, 2014, 47(20): 4078–4085. DOI: 10.3864/j.issn.0578-1752.2014.20.016 (in Chinese) |
| [6] | ZHANG H J, YU Y H, CHAI J K. Expression signatures of lncRNAs in skeletal muscles at the early flow phase revealed by microarray in burned rats[J]. Ulus Travma Acil Cerrahi Derg, 2016, 22(3): 224–232. |
| [7] |
李燕, 陈明明, 张俊星, 等. 牛LncRNA-133a对骨骼肌卫星细胞增殖分化的影响[J]. 中国农业科学, 2019, 52(1): 143–153.
LI Y, CHEN M M, ZHANG J X, et al. Effects of bovine LncRNA-133a on the proliferation and differentiation of skeletal muscle satellite cells[J]. Scientia Agricultura Sinica, 2019, 52(1): 143–153. (in Chinese) |
| [8] | CHEN Z, BU N P, QIAO X H, et al. Forkhead box M1 transcriptionally regulates the expression of long noncoding RNAs Snhg8 and Gm26917 to promote proliferation and survival of muscle satellite cells[J]. Stem Cells, 2018, 36(7): 1097–1108. DOI: 10.1002/stem.2824 |
| [9] | CESANA M, CACCHIARELLI D, LEGNINI I, et al. A long noncoding RNA controls muscle differentiation by functioning as a competing endogenous RNA[J]. Cell, 2011, 147(2): 358–369. DOI: 10.1016/j.cell.2011.09.028 |
| [10] | TANG Z L, WU Y, YANG Y L, et al. Comprehensive analysis of long non-coding RNAs highlights their spatio-temporal expression patterns and evolutional conservation in Sus scrofa[J]. Sci Rep, 2017, 7: 43166. DOI: 10.1038/srep43166 |
| [11] | KERSEY P J, ALLEN J E, CHRISTENSEN M, et al. Ensembl genomes 2013:scaling up access to genome-wide data[J]. Nucleic Acids Res, 2014, 42(D1): 546–552. DOI: 10.1093/nar/gkt979 |
| [12] | HUANG D W, SHERMAN B T, ZHENG X, et al. Extracting biological meaning from large gene lists with DAVID[J]. Curr Protoc Bioinformatics, 2009, 27(1): 13.11.1–13.11.13. DOI: 10.1002/0471250953.bi1311s27 |
| [13] |
庾蕾, 刘建平, 庄志雄, 等. 实时RT-PCR基因表达相对定量REST©软件分析与2(-ΔΔCT)法比较[J]. 热带医学杂志, 2007, 7(10): 956–958.
YU L, LIU J P, ZHUANG Z X, et al. Quantitative analysis of real-time PCR expression production by REST© and 2(-ΔΔCT)[J]. Journal of Tropical Medicine, 2007, 7(10): 956–958. DOI: 10.3969/j.issn.1672-3619.2007.10.006 (in Chinese) |
| [14] | GHAFOURI-FARD S, TAHCRI M. Nuclear enriched abundant transcript 1 (NEAT1): A long non-coding RNA with diverse functions in tumorigenesis[J]. Biomed Pharmacother, 2019, 111: 51–59. DOI: 10.1016/j.biopha.2018.12.070 |
| [15] | MERCER T R, DINGER M E, MATTICK J S. Long non-coding RNAs:insights into functions[J]. Nat Rev Genet, 2009, 10(3): 155–159. DOI: 10.1038/nrg2521 |
| [16] | JANDURA A, KRAUSE H M. The new RNA world:growing evidence for long noncoding RNA functionality[J]. Trends Genet, 2017, 33(10): 665–676. DOI: 10.1016/j.tig.2017.08.002 |
| [17] | LI Y Y, CHEN X N, SUN H, et al. Long non-coding RNAs in the regulation of skeletal myogenesis and muscle diseases[J]. Cancer Lett, 2018, 417: 58–64. DOI: 10.1016/j.canlet.2017.12.015 |
| [18] | FLYNN R A, CHANG H Y. Long noncoding RNAs in cell-fate programming and reprogramming[J]. Cell Stem Cell, 2014, 14(6): 752–761. DOI: 10.1016/j.stem.2014.05.014 |
| [19] | LI Y, DUFF M O, GRAVELEY B R, et al. Genomewide characterization of non-polyadenylated RNAs[J]. Genome Biol, 2011, 12(2): R16. |
| [20] | OKAZAKI Y, FURUNO M, KASUKAWA T, et al. Analysis of the mouse transcriptome based on functional annotation of 60, 770 full length cDNAs[J]. Nature, 2002, 420: 563–573. DOI: 10.1038/nature01266 |
| [21] | WILLINGHAM A T, ORTH A P, BATALOV S, et al. A strategy for probing the function of noncoding RNAs finds a repressor of NFAT[J]. Science, 2005, 309(5740): 1570–1573. DOI: 10.1126/science.1115901 |
| [22] | WANG Y Y, XUE S Y, LIU X R, et al. Analyses of long non-coding RNA and mRNA profiling using RNA sequencing during the pre-implantation phases in pig endometrium[J]. Sci Rep, 2016, 6: 20238. DOI: 10.1038/srep20238 |
| [23] | CHEN G, SHI T L, SHI L M. Characterizing and annotating the genome using RNA-seq data[J]. Sci China Life Sci, 2017, 60(2): 116–125. DOI: 10.1007/s11427-015-0349-4 |
| [24] | WANG L J, ZHAO Y, BAO X C, et al. LncRNA Dum interacts with Dnmts to regulate Dppa2 expression during myogenic differentiation and muscle regeneration[J]. Cell Res, 2015, 25(3): 335–350. |
| [25] | ZOU C, LI J X, LUO W Z, et al. Transcriptome analysis reveals long intergenic non-coding RNAs involved in skeletal muscle growth and development in pig[J]. Sci Rep, 2017, 7(1): 8704. DOI: 10.1038/s41598-017-07998-9 |
| [26] | LUO W, CHEN J H, LI L M, et al. c-Myc inhibits myoblast differentiation and promotes myoblast proliferation and muscle fibre hypertrophy by regulating the expression of its target genes, miRNAs and linc-RNAs[J]. Cell Death Differ, 2019, 26(3): 426–442. DOI: 10.1038/s41418-018-0129-0 |
| [27] | SONG C C, WANG J, MA Y L, et al. Linc-smad7 promotes myoblast differentiation and muscle regeneration via sponging miR-125b[J]. Epigenetics, 2018, 13(6): 591–604. DOI: 10.1080/15592294.2018.1481705 |
| [28] | NEGUEMBOR M V, JOTHI M, GABELLINI D. Long noncoding RNAs, emerging players in muscle differentiation and disease[J]. Skelet Muscle, 2014, 4(1): 8. DOI: 10.1186/2044-5040-4-8 |
| [29] | ZAMMIT P S, HESLOP L, HUDON V, et al. Kinetics of myoblast proliferation show that resident satellite cells are competent to fully regenerate skeletal muscle fibers[J]. Exp Cell Res, 2002, 281(1): 39–49. DOI: 10.1006/excr.2002.5653 |
| [30] | HAN X R, YANG F, CAO H Q, et al. Malat1 regulates serum response factor through miR-133 as a competing endogenous RNA in myogenesis[J]. FASEB J, 2015, 29(7): 3054–3064. DOI: 10.1096/fj.14-259952 |
| [31] | CABIANCA D S, CASA V, BODEGA B, et al. A long ncRNA links copy number variation to a polycomb/trithorax epigenetic switch in FSHD muscular dystrophy[J]. Cell, 2012, 149(4): 819–831. DOI: 10.1016/j.cell.2012.03.035 |
| [32] | CHEN L L. Linking long noncoding RNA localization and function[J]. Trends Biochem Sci, 2016, 41(9): 761–772. DOI: 10.1016/j.tibs.2016.07.003 |
| [33] | CHEN L L, CARMICHAEL G G. Decoding the function of nuclear long non-coding RNAs[J]. Curr Opin Cell Biol, 2010, 22(3): 357–364. DOI: 10.1016/j.ceb.2010.03.003 |
| [34] |
王红娜, 孙洪新, 张英杰, 等. 干扰MSTN对绵羊成肌细胞增殖分化及相关基因表达的影响[J]. 畜牧兽医学报, 2018, 49(1): 46–54.
WANG H N, SUN H X, ZHANG Y J, et al. Effects of interfering MSTN on proliferation and differentiation of sheep myoblasts and expression of related genes[J]. Acta Veterinaria et Zootechnica Sinica, 2018, 49(1): 46–54. (in Chinese) |
| [35] | TINTIGNAC L A, LEIBOVITCH M P, KITZMANN M, et al. Cyclin E-cdk2 phosphorylation promotes late G1-phase degradation of MyoD in muscle cells[J]. Exp Cell Res, 2000, 259(1): 300–307. DOI: 10.1006/excr.2000.4973 |
| [36] | BUCKINGHAM M, RIGBY P W J. Gene regulatory networks and transcriptional mechanisms that control myogenesis[J]. Dev Cell, 2014, 28(3): 225–238. DOI: 10.1016/j.devcel.2013.12.020 |
| [37] | TAKAGAKI Y, YAMAGISHI H, MATSUOKA R. Chapter four-Factors involved in signal transduction during vertebrate myogenesis[J]. Int Rev Cell Mol Biol, 2012, 296: 187–272. DOI: 10.1016/B978-0-12-394307-1.00004-7 |
| [38] | CANTÓ C, AUWERX J. AMP-activated protein kinase and its downstream transcriptional pathways[J]. Cell Mol Life Sci, 2010, 67(20): 3407–3423. DOI: 10.1007/s00018-010-0454-z |
| [39] | SUN X J, DANG F B, ZHANG D Y, et al. Glucagon-CREB/CRTC2 signaling cascade regulates hepatic BMAL1 protein[J]. J Biol Chem, 2015, 290(4): 2189–2197. DOI: 10.1074/jbc.M114.612358 |
| [40] | DAUER W, PRZEDBORSKI S. Parkinson's disease:Mechanisms and models[J]. Neuron, 2003, 39(6): 889–909. DOI: 10.1016/S0896-6273(03)00568-3 |



