2. 中国农业科学院北京畜牧兽医研究所, 北京 100193
2. Institute of Animal Science, Chinese Academy of Agricultural Sciences, Beijing 100193, China
随着人们对食品安全的愈发重视和我国相关法律法规的日益完善,人们对牛奶的质量和奶牛的动物福利更加关注。然而,奶牛乳腺炎的发生,不仅严重影响奶牛机体的健康,同时也影响牛奶品质[1],严重损害奶牛产业的经济效益。据统计,无乳链球菌引起的奶牛乳腺炎约占乳腺炎发病的63.02%,占隐性乳腺炎的56.25%[2],由此可见,进一步研究无乳链球菌的致病机制,对临床治疗奶牛乳腺炎的感染具有重要的指导意义。
无乳链球菌(Streptococcus agalactiae,GBS)是引起奶牛乳腺炎的3大病原菌之一,还可以诱发鱼无乳链球菌病及新生的败血症等疾病。GBS为传染性病原体,需通过乳导管侵入乳腺或由体内转移到乳腺组织,透过乳腺上皮细胞,在乳腺组织中存活并增殖。GBS感染过程中的每一步均由特定的毒力因子(virulence factor)调控,主要包括黏附因子、侵袭因子和免疫逃避因子等[3]。研究发现,黏附因子多由细胞黏附素构成,与GBS的黏附、侵袭有关;侵袭因子多为造孔素构成,与侵袭、细胞损伤有关;免疫逃避因子则与GBS的生存相关[3]。这些毒力因子由特定的毒力基因所调控,其中,以CovR/S为首的双组分系统最为重要[4]。下面对上述主要的毒力因子及其研究进展进行综述。
1 黏附因子无乳链球菌侵入乳腺与细胞表面细胞外基质蛋白(ECM)相互作用而黏附到乳腺上皮细胞上,是乳腺炎发生的第1步[5-6]。据报道,无乳链球菌的黏附因子主要包括纤维蛋白原结合蛋白(FbsA/B)和细菌表面黏附素(BsaB或BiBA,又称FbsC)、Srr糖蛋白家族、菌毛(pili)和Alp蛋白家族等(图 1)[5-6]。由于黏附因子的黏附过程对研究乳腺炎的发生发展具有重要的决定性作用,因此,更好地了解黏附因子的作用机制,可为探索治疗乳腺炎的新方法提供理论依据[5-6]。
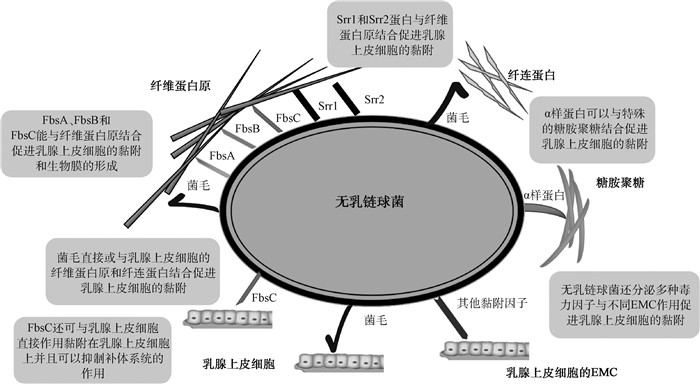
|
图 1 参与无乳链球菌黏附乳腺上皮细胞的毒力因子 Fig. 1 Virulence factors involved in adhesion of Streptococcus agalactiae to mammary gland epithelial cells |
纤维蛋白原结合蛋白(Fbs)及黏附蛋白是一系列免疫原性细菌黏附蛋白,也是GBS表面蛋白的总称。其中,Fbs主要分为两类,即FbsA和FbsB,黏附蛋白被称为FbsC(也称BiBA)。纤维蛋白原结合蛋白(FbsA/B)主要存在于Ia型和Ⅲ型无乳链球菌中,这两种血清也是诱发乳腺炎主要的血清型[7]。据报道,FbsA、FbsB和FbsC均被证明与乳腺上皮细胞的黏附性密切相关,并是链球菌中广泛存在的同源蛋白[8],由双组分调节因子CovS/R(control of virulence sensor and regulator)和RgfA/C[9-10]调节。Safadi等[10]研究表明,在covR基因242位的胞嘧啶被胸腺嘧啶取代后,导致氨基酸从丙氨酸变为缬氨酸,加强了Fbs与纤维蛋白原之间的相互作用,从而增强了无乳链球菌对乳腺上皮细胞的黏附作用并促进生物被膜的形成[11-12]。在杜琳等[13]的研究中,揭示了Fbs的同源蛋白在细菌黏附于乳腺上皮细胞的过程中起着重要的作用,尤其在使用了含有FbsA和FbsB的疫苗后可显著减弱链球菌的致病性。据报道,钙离子可增强纤维结合蛋白的黏附能力,因此,乳汁中富含钙离子更有利于无乳链球菌在乳腺内的定植[7]。此外,FbsC可以促进细菌的传播;当缺失FbsC时,可显著降低GBS在鼠脑内皮细胞中的结合和侵袭能力[3, 14]。FbsC结构为由16个氨基酸以ɑ螺旋和无规则卷曲方式组成的高度重复蛋白,具有相对保守性,因此被用于FbsC亚单位疫苗的研发[7]。柴晨[15]应用FbsA基因制成亚单位疫苗对奶牛进行免疫后,使乳腺中无乳链球菌的感染率显著下降。因此,Fbs蛋白家族的毒力因子可作为未来疫苗和药物研发较好的作用靶点,特别是FbsC具有较大的研究潜力。
1.2 Srr糖蛋白家族Srr糖蛋白家族是无乳链球菌中一组富含的重复丝氨酸的蛋白序列的丝氨酸重复蛋白(serine-rich repeat glycoproteins, Srr),属于革兰阳性细胞壁蛋白家族,主要由Srr1和Srr2两类蛋白组成[16]。Srr蛋白结构上由一个高度重复交替的丝氨酸残基组成[3, 17],具有黏合作用;据报道,Srr蛋白结构通过与乳腺上皮细胞纤维蛋白原的Aa链的结合,使无乳链球菌黏附在细胞上[18]。此外,Pietrocola等[19]证实当Srr蛋白与纤维蛋白原结合后,可引起丝氨酸糖蛋白家族成员Srr1和Srr2发生有序的构象变化,从而进一步增强细菌的黏附能力。
Srr蛋白由纤维蛋白原结合调节因子(RgfA/C)双组分调控系统进行调控[3, 17, 20]。RgfA/C还能够调控黏附相关的纤维蛋白原结合蛋白、荚膜多糖等多种毒力因子,这些毒力因子共同作用增强乳腺上皮细胞的黏附效果[3, 20]。吴金花等[21]利用Srr蛋白的免疫原性所研发的乳腺炎疫苗,证实具有有效的保护作用。Lin[22]研究设计了GBS可以表达任一Srr1或Srr2表面蛋白作为抗原,均得到了较好的免疫效果。那么,将Srr糖蛋白的相对保守性区域作为疫苗研发的可行性位点,进一步研究其在奶牛中的免疫保护效果提供新思路。
1.3 菌毛蛋白菌毛蛋白(pili)由菌毛的骨架蛋白(backbone protein,BP)与辅助蛋白(auxiliiary protein,AP) AP-1、AP-2共同组装而成,均为抗原保守性蛋白,参与GBS对乳腺上皮细胞的黏附、侵袭和致病等过程[23]。菌毛的骨架蛋白作为菌毛的骨干结构,AP1分布于细菌表面,AP2定位于菌毛顶端,包括PI-1、PI-2a和PI-2b等表现型。因此,菌毛在定植过程中通过与宿主细胞分子结合的方式介导乳腺上皮细胞的黏附[24]。白文丽等[25]克隆了奶牛乳腺炎无乳链球菌临床分离株PI-2a菌毛岛屿骨架蛋白BP基因片段,研究证明了该基因的高度保守性和相应蛋白良好的抗原性。因此,菌毛蛋白被认为是最理想的疫苗候选靶标之一。
1.4 Alp蛋白家族Alp蛋白家族由GBS的Alp编码基因产生的,是α相关蛋白(α-like protein)的总称[26]。该蛋白家族成员包括Alpha-C、Rib、Epsilon、Alp2、Alp3、Alp4等蛋白[27]。其中,Alpha-C是Alp蛋白家族在无乳链球菌中首个被鉴别出的抗原表位,也称原型Alp[28],介导GBS的黏附、跨膜通道和易位[28],在侵袭上皮细胞过程中起重要作用。Alpha-C蛋白由bac等位基因编码部分的表面蛋白产生,包含一个糖胺聚糖结合结构域,附着在α1β1整合蛋白或真核细胞相关的糖胺聚糖上,从而介导无乳链球菌侵袭乳腺上皮细胞[29]。此外,bac基因也被用于奶牛乳腺中无乳链球菌毒力的检测[30]。研究发现,无乳链球菌Alp蛋白的侵袭还可由脂筏(lipid rafts)介导[31],通过磷酸肌醇3激酶(phosphoinositide 3-kinase)激活,最终使无乳链球菌黏附在乳腺上皮细胞上而导致炎症的发生。
1.5 其他黏附因子近期研究发现,一些新的黏附因子在无乳链球菌黏附在乳腺上皮细胞的过程中也发挥了作用,丰富了无乳链球菌的黏附因子种类,并逐渐深化了对无乳链球菌的细化研究。Nagarajan等[32]研究发现,无乳链球菌的GAPDH的晶体与细胞外基质分子宿主纤溶酶原(PLG)和纤维蛋白原(Fg)结合并相互作用,可增强毒力并调节宿主免疫系统。Spencer等[33]研究发现CRISPR相关蛋白9(Cas9)可能通过调节其他毒力因子调节GBS基因的表达,有助于无乳链球菌的定植和侵袭。虽然黏附因子是目前无乳链球菌研究得最为广泛的毒力因子,Fbs家族和菌毛等已被研发出可以进行动物试验的疫苗,但在奶牛生产中尚无有效的无乳链球菌型乳腺炎疫苗的临床应用,并且许多新的毒力因子的作用机制和开发利用仍需进一步研究。
2 侵袭因子侵袭因子是指无乳链球菌破坏细胞的免疫屏障侵入机体的物质,大多数由造孔毒素组成[34],这些毒素促进病原体进入宿主细胞,并保证细菌在机体内存活。据报道,无乳链球菌编码多种侵袭因子,主要包括溶血色素、透明质酸酶和细胞外膜囊泡的CAMP因子等(图 2)[34]。
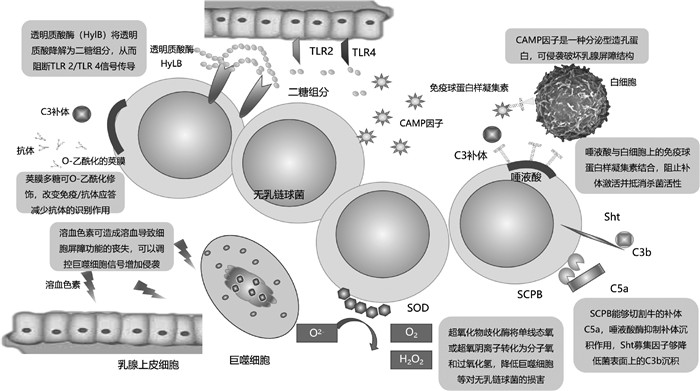
|
图 2 参与无乳链球菌侵袭乳腺上皮细胞和免疫逃避的毒力因子 Fig. 2 Virulence factors involved in invasion of mammary gland epithelial cells and immune evasion by Streptococcus agalactiae |
无乳链球菌是一种β-溶血性细菌,其溶血特性对乳腺炎发生过程中的侵袭乳腺和免疫逃避有重要意义。据报道,溶血色素是由CylE基因产生的一种鸟氨酸鼠李糖多烯色素,是无乳链球菌造成溶血的主要原因,可以促进无乳链球菌对乳腺的侵袭,并导致细胞屏障功能的丧失[34]。Rosa-Fraile等[35]的研究明确了双组分系统CovR/S具有调节溶血色素毒力的作用,并且提出GBS的溶血和细胞溶解活性是由鸟氨酸鼠李糖多烯色素而不是由CylE蛋白引起的。Boldenow等[34]发现,如果溶血色素缺失,可导致细菌传播减少;布日额等[36]认为在奶牛乳腺炎中β-溶血素作为信号分子模式可以影响巨噬细胞信号调控机制,从而调控IL-10的转录与表达,使乳腺炎加重。然而,有研究发现,过高的溶血色素表达,能诱导中性粒细胞胞外陷阱(nets)的形成,反而促进了免疫反应的发生[34]。在乳腺炎方面,因为溶血色素为特异性的溶血素,且具有特异性的鉴别特征,即β溶血,因此,Cyl基因被广泛用于乳腺炎中无乳链球菌的鉴别[37-38]。虽然已经明确该毒力因子在小鼠模型中的溶血有助于对上皮细胞的侵袭作用,但该毒力因子在乳腺炎发生时的作用机制研究较少,仍需进一步研究。
2.2 透明质酸酶透明质酸是一种由二糖单元(N-乙酰D-氨基葡萄糖醛酸)构成的对细胞迁移、信号传递、炎症调节起作用的细胞外基质糖胺多糖[39],无乳链球菌的透明质酸酶(HylB)能够靶向降解宿主透明质酸,从而促进无乳链球菌在乳腺的定植[39-40]。研究发现,HylB可以将宿主的透明质酸降解为具有免疫抑制作用的二糖组分,从而阻断TLR2/TLR4受体结合的信号传导,导致小鼠对全身感染的无乳链球菌清除率增加[39]。而且,HylB广泛存在于奶牛乳腺炎无乳链球菌中,并导致入侵乳腺的毒力增强[38],被认定为乳腺炎中无乳链球菌的优势毒力因子[41]。虽然HylB在分子水平的作用机制得以阐述,证明了其为乳腺炎重要的致病因子,但对其的针对性治疗手段仍需进一步研究。
2.3 CAMP因子CAMP(christine atkins munch-peterson)因子是一种无乳链球菌特有的可作为无乳链球菌鉴定的分泌型造孔蛋白,又称为溶血促进因子(cohemolysin)[3]。它主要分布在由细胞分泌的双层膜结构的囊泡状小体即细胞外微囊泡(MVS)中,与囊泡中的其他毒力因子(HylB、IgA结合蛋白及与ECM结合的结合酶等)共同作用侵袭乳腺屏障结构[42]。Podbielski等[43]发现,CAMP因子能特异性地增加金黄色葡萄球菌对绵羊红细胞的溶解能力,此现象称为CAMP反应,被用于乳腺炎中无乳链球菌的鉴定。此外,产生CAMP因子的cfb基因的PCR方法也被广泛应用于乳腺炎中无乳链球菌的筛查[44-45]。王向柳[46]制造了奶牛乳腺炎牛源无乳链球菌CAMP因子的三联苗,经过田间试验证实产生抗金黄色葡萄球菌、大肠杆菌、无乳链球菌的血清抗体。
3 免疫逃避因子无乳链球菌的免疫逃避因子主要包括唾液酸、抗氧化系统和抗补体系统等(图 2)[47]。目前,对免疫逃避因子的研究主要为免疫逃避因子和机体免疫系统的相互作用机制,这对奶牛乳腺炎的诊断及治疗具有重大意义。
3.1 唾液酸系统唾液酸通常存在于脊椎动物细胞的糖链上,是介导免疫反应的重要物质[48]。Severi等[48]发现,无乳链球菌和其他链球菌可以通过自身合成和从宿主转运利用唾液酸规避免疫系统。姚晶等[49]发现,在乳腺炎发生过程中,无乳链球菌利用唾液酸系统发挥了免疫逃避的作用,因此,对乳腺炎起到了免疫抑制的作用。Tong等[50]认为,荚膜唾液酸的生物学重要性,主要归因于与免疫细胞的相互作用,其中GBS与白细胞上识别唾液酸(siglecs)的免疫球蛋白样凝集素结合,可以阻止补体激活并抵消宿主细胞的杀菌活性。Paoletti等[47]发现,无乳链球菌将唾液酸分泌在囊泡上进行免疫逃避。这可以调节先天性和适应性免疫功能的细胞表面唾液酸结合,凝集素功能受阻从而降低免疫反应[47]。Lewis等[51]认为,无乳链球菌通过在荚膜分泌唾液酸,使机体无法识别无乳链球菌是否为自身物质,从而进行免疫逃避。同时,无乳链球菌能通过唾液酸囊泡或β-蛋白与siglecs受体结合,抑制免疫细胞活化[51]。虽然有间接证据证明其在乳腺炎中发挥的作用,但针对无乳链球菌产生的唾液酸对乳腺炎的作用过程尚不清晰,因此对无乳链球菌唾液酸表达的调控仍需进一步研究。
3.2 超氧化和超氧化物歧化酶无乳链球菌通过产生超氧化物歧化酶(SOD)降低免疫系统对无乳链球菌的氧化作用和对多糖荚膜氧化来逃避乳腺的免疫作用[48]。研究表明,无乳链球菌编码的荚膜多糖可以通过O-乙酰化进行修饰,改变免疫/抗体应答减少抗体对无乳链球菌的识别作用而实现免疫逃避[51-52]。Severi等[48]还发现,O-乙酰化还发生在唾液酸上,这种O-乙酰化修饰加强免疫逃避的作用。负责荚膜多糖的O-乙酰化的酶是由Neua基因编码的,它能定点将天冬氨酸合成丙氨酸(N301A),Neua突变导致无乳链球菌的O-乙酰化增加,使无乳链球菌的免疫逃避能力增强[51, 53]。另一方面,Farhat等[54]研究发现,在乳腺炎发生时通过Toll2受体激活巨噬细胞,来氧化杀伤无乳链球菌。Poyart等[55]证实在乳腺炎中无乳链球菌能编码超氧化物歧化酶SodA,以抵抗氧化应激对无乳链球菌的损害。超氧化物歧化酶将单线态氧或超氧阴离子(O2-)转化为分子氧(O2)和过氧化氢(H2O2),以降低巨噬细胞等免疫细胞对无乳链球菌的损害[52, 56]。另外,SOD能阻碍氧依赖性吞噬细胞来防止菌体被巨噬细胞吞噬建立防御机制[55]。此外超氧化物歧化酶使病原菌能够抵抗感染期间的氧化应激[53]。综上所述,超氧化物歧化酶和无乳链球菌的荚膜多糖的氧化作用在乳腺中达到了一个平衡状态,使无乳链球菌侵袭乳腺并发挥免疫逃避的作用,但这一过程仅仅被阐述,缺乏相应的应对措施和治疗手段,可进一步进行研究。
3.3 抗补体系统补体是抵抗GBS感染的首道防线,在乳腺炎中补体可促进嗜中性粒细胞和巨噬细胞来吞噬细菌,无乳链球菌可通过多种毒力因子抑制补体发挥作用[56]。Zhang等[56]研究表明,无乳链球菌感染引发了复杂的宿主先天免疫反应,涉及补体和凝血级联反应,ECM受体相互作用,局灶性黏附,吞噬体和细菌侵入上皮细胞的途径。Bohnsack等[57]证实,无乳链球菌C5a肽酶(SCPB)是一种由SCPB基因编码的能够切割补体的丝氨酸蛋白酶,它能够定向地切割牛的补体C5a降低补体引起的趋化作用,从而能促进无乳链球菌在乳腺炎中的免疫逃避。且在动物模型中SCPB对无乳链球菌具有明显的保护作用[58]。另外,有报道发现,荚膜的唾液酸酶具有抑制补体沉积的作用[59]。Moulin等[60]也发现Sht家族蛋白的Sht募集因子能够降低无乳链球菌表面上的C3b沉积,从而有助于逃避由膜攻击复合物介导的细菌裂解。虽然无乳链球菌可通过多种途径抑制补体的作用[61],但无乳链球菌对补体的作用缺乏整体和系统的研究,仍处于初级阶段,需要进一步的研究。其对补体的抑制可作为防治无乳链球菌型乳腺炎的重要靶点。
4 小结综上所述,毒力因子对无乳链球菌的生存和感染都有重要的意义,且无乳链球菌的很多毒力因子也是疫苗的靶点,由于无乳链球菌抗原表位的易变性和不同血清型交叉保护性较低等问题,无乳链球菌疫苗距离临床应用还有距离。因此,更好地了解无乳链球菌的毒力因子及其调控机制(图 3),治疗和预防无乳链球菌性乳腺炎,及筛选无乳链球菌的疫苗靶点都是未来可以重点研发的方向和目标。
随着细胞水平及分子水平研究的深入、基因组学和转录组学等方面技术的发展,逐渐加深了对无乳链球菌的毒力因子更全面地认识,使一些新的毒力因子和机制被发现,如Sht因子,双组分调控系统和TSS系统等[62]。而且,一些针对毒力因子的天然活性成分药物不断被探究,例如芦丁、壳聚糖和白藜芦醇等均表现出对毒力因子良好的抑制作用。高新科技的发展及中医药理论的全新解读,对无乳链球菌的毒力因子的致病机制和防控手段的研究将愈加完备,深入了解无乳链球菌毒力因子的调控手段,对无乳链球菌型临床型和亚临床型乳腺炎的预防和治疗具有重要的意义。
| [1] | GUSSMANN M, STEENEVELD W, KIRKEBY C, et al. Economic and epidemiological impact of different intervention strategies for subclinical and clinical mastitis[J]. Prev Vet Med, 2019, 166: 78–85. |
| [2] | LAKEW B T, FAYERA T, ALI Y M. Risk factors for bovine mastitis with the isolation and identification of Streptococcus agalactiae from farms in and around Haramaya district, eastern Ethiopia[J]. Trop Anim Health Prod, 2019, 51(6): 1507–1513. |
| [3] | VORNHAGEN J, WALDORF K M A, RAJAGOPAL L. Perinatal group B streptococcal infections:virulence factors, immunity, and prevention strategies[J]. Trends Microbiol, 2017, 25(11): 919–931. |
| [4] | SIEMENS N, OEHMCKE-HECHT S, HOßMANN J, et al. Prothrombotic and proinflammatory activities of the β-hemolytic group B streptococcal pigment[J]. J Innate Immu, 2020, 12(4): 291–303. |
| [5] | GORI A, HARRISON O B, MLIA E, et al. Pan-GWAS of Streptococcus agalactiae highlights lineage-specific genes associated with virulence and niche adaptation[J]. mBio, 2020, 11(3): e00728–20. |
| [6] | ARMISTEAD B, OLER E, WALDORF K A, et al. The double life of Group B Streptococcus: asymptomatic colonizer and potent pathogen[J]. J Mol Biol, 2019, 431(16): 2914–2931. |
| [7] | CARVALHO-CASTRO G A, SILVA J R, PAIVA L V, et al. Molecular epidemiology of Streptococcus agalactiae isolated from mastitis in Brazilian dairy herds[J]. Braz J Microbiol, 2017, 48(3): 551–559. |
| [8] | PICKERING A C, VITRY P, PRYSTOPIUK V, et al. Host-specialized fibrinogen-binding by a bacterial surface protein promotes biofilm formation and innate immune evasion[J]. PLoS Pathog, 2019, 15(6): e1007816. |
| [9] | THOMAS L, COOK L. Two-component signal transduction systems in the human pathogen, Streptococcus agalactiae[J]. Infect Immun, 2020, 88(7): e00931–19. |
| [10] | SAFADI R A, MEREGHETTI L, SALLOUM M, et al. Two-component system RgfA/C Activates the fbsB gene encoding major fibrinogen-binding protein in highly virulent CC17 clone group B streptococcus[J]. PLoS One, 2011, 6(2): e14658. |
| [11] | RAGUNATHAN P, PONNURAJ K. Expression, purification and structural analysis of a fibrinogen receptor FbsA from Streptococcus agalactiae[J]. Protein J, 2011, 30(3): 159–166. |
| [12] | DEVI A S, PONNURAJ K. Cloning, expression, purification and ligand binding studies of novel fibrinogen-binding protein FbsB of Streptococcus agalactiae[J]. Protein Expr Purif, 2010, 74(2): 148–155. |
| [13] |
杜琳, 吕天星, 赵红梅, 等. 无乳链球菌CP+Sip-FbsA偶联蛋白免疫学特性的研究[J]. 中国预防兽医学报, 2016, 38(12): 976–980.
DU L, LV T X, ZHAO H M, et al. The immune effect of Streptococcus agalactiae recombinant CP+Sip-FbsA fusion protein on S. agalactiae induced mouse mastitis[J]. Chinese Journal of Preventive Veterinary Medicine, 2016, 38(12): 976–980. (in Chinese) |
| [14] | BUSCETTA M, PAPASERGI S, FIRON A, et al. FbsC, a novel fibrinogen-binding protein, promotes Streptococcus agalactiae-host cell interactions[J]. J Biol Chem, 2014, 289(30): 21003–21015. |
| [15] |
柴晨.无乳链球菌α-enolase, PGK和GAPDH基因的克隆、表达及对罗非鱼免疫效果研究[D].广州: 中山大学, 2017.
CHAI C. Studies on cloning, expression and immune effect of α-enolase, PGK and GAPDH genes of Streptococcus lactobacillus on tilapia[D]. Guangzhou: Sun Yat-sen University, 2017. (in Chinese) |
| [16] | SEO H S, MINASOV G, SEEPERSAUD R, et al. Characterization of fibrinogen binding by glycoproteins Srr1 and Srr2 of Streptococcus agalactiae[J]. J Biol Chem, 2013, 288(50): 35982–35996. |
| [17] | MISTOU M Y, DRAMSI S, BREGA S, et al. Molecular dissection of the secA2 locus of group B Streptococcus reveals that glycosylation of the Srr1 LPXTG protein is required for full virulence[J]. J Bacteriol, 2009, 191(13): 4195–4206. |
| [18] | SEIFERT K N, ADDERSON E E, WHITING A A, et al. A unique serine-rich repeat protein (Srr-2) and novel surface antigen (ε) associated with a virulent lineage of serotype Ⅲ Streptococcus agalactiae[J]. Microbiology, 2006, 152(4): 1029–1040. |
| [19] | PIETROCOLA G, ARCIOLA C R, RINDI S, et al. Streptococcus agalactiae Non-Pilus, cell wall-anchored proteins: involvement in colonization and pathogenesis and potential as vaccine candidates[J]. Front Immunol, 2018, 9: 602. |
| [20] | GENDRIN C, LEMBO A, WHIDBEY C, et al. The sensor Histidine kinase RgfC affects group B streptococcal virulence factor expression independent of its response regulator RgfA[J]. Infect Immun, 2015, 83(3): 1078–1088. |
| [21] |
吴金花, 布日额, 王金良, 等. 奶牛乳腺炎无乳链球菌sip、pgk及FbsA基因主要抗原区域的融合表达及抗原性鉴定[J]. 中国兽医学报, 2017, 37(7): 1292–1299.
WU J H, BU R E, WANG J L, et al. Fusion expression of main antigen area of sip, pgk and FbsA subunit genes and the antigenicity identification in Streptococcus agalactiae for dairy cow mastitis[J]. Chinese Journal of Veterinary Science, 2017, 37(7): 1292–1299. (in Chinese) |
| [22] | LIN S. Immunization of 13 amino acid peptide targeting Srr proteins provide a broad spectrum of protections against group B streptococcal infections[M]// 2016 International Meeting of the Microbiological Society of Korea, 2016: 166. |
| [23] | MAIONE D, MARGARIT I, RINAUDO C D, et al. Identification of a universal group B Streptococcus vaccine by multiple genome screen[J]. Science, 2005, 309(5731): 148–150. |
| [24] | DRAMSI S, CALIOT E, BONNE I, et al. Assembly and role of pili in group B streptococci[J]. Mol Microbiol, 2006, 60(6): 1401–1413. |
| [25] |
白文丽, 王金良, 锡林高娃, 等. 奶牛乳腺炎无乳链球菌菌毛岛屿PI-2a骨架蛋白BP基因的克隆及其抗原性预测[J]. 中国病原生物学杂志, 2017, 12(4): 294–297.
BAI W L, WANG J L, XILIN G W, et al. Cloning and prediction of the antigenicity of a gene that codes for a pilus backbone protein (PI-2a) in Streptococcus agalactiae causing bovine mastitis[J]. Journal of Parasitic Biology, 2017, 12(4): 294–297. (in Chinese) |
| [26] | MAELAND J A, AFSET J E, LYNG R V, et al. Survey of immunological features of the alpha-like proteins of Streptococcus agalactiae[J]. Clinical Vaccine Immunol, 2015, 22(2): 153–159. |
| [27] | KONG F R, GOWAN S, MARTIN D, et al. Molecular profiles of group B streptococcal surface protein antigen genes:relationship to molecular serotypes[J]. J Clinical Microbiol, 2002, 40(2): 620–626. |
| [28] | BARON M J, FILMAN D J, PROPHETE G A, et al. Identification of a glycosaminoglycan binding region of the alpha C protein that mediates entry of group B Streptococci into host cells[J]. J Biol Chem, 2007, 282(14): 10526–10536. |
| [29] | CRETI R, FABRETTI F, OREFICI G, et al. Multiplex PCR assay for direct identification of group B streptococcal alpha-protein-like protein genes[J]. J Clinical Microbiol, 2004, 42(3): 1326–1329. |
| [30] |
张保海, 罗梓丹, 芦彪, 等. 四川部分地区奶牛源无乳链球菌的分离鉴定、毒力基因检测及耐药性分析[J]. 西北农业学报, 2020, 29(3): 327–333.
ZHANG B H, LUO Z D, LU B. Isolation, Virulence gene test and drug resistance analysis of Streptococcus agalactiae from dairy cows in some areas of Sichuan[J]. Acta Agriculturae Boreali-occidentalis Sinica, 2020, 29(3):327-333. (in Chinese)[J]. Acta Agriculturae Boreali-occidentalis Sinica, 2020, 29(3): 327–333. (in Chinese) |
| [31] | WÄSTFELT M, STÅLHAMMAR-CARLEMALM M, DELISSE A M, et al. Identification of a family of streptococcal surface proteins with extremely repetitive structure[J]. J Biol Chem, 1996, 271(31): 18892–18897. |
| [32] | NAGARAJAN R, SANKAR S, PONNURAJ K. Crystal structure of GAPDH of Streptococcus agalactiae and characterization of its interaction with extracellular matrix molecules[J]. Microb Pathog, 2019, 127: 359–367. |
| [33] | SPENCER B L, DENG L W, PATRAS K A, et al. Cas9 contributes to group B streptococcal colonization and disease[J]. Front Microbiol, 2019, 10: 1930. |
| [34] | BOLDENOW E, GENDRIN C, NGO L, et al. Group B Streptococcus circumvents neutrophils and neutrophil extracellular traps during amniotic cavity invasion and preterm labor[J]. Sci Immunol, 2016, 1(4): eaah4576. |
| [35] | ROSA-FRAILE M, DRAMSI S, SPELLERBERG B. Group B streptococcal haemolysin and pigment, a tale of twins[J]. FEMS Microbiol Rev, 2014, 38(5): 932–946. |
| [36] |
布日额, 吴金花, 锡林高娃, 等. 牛乳腺炎无乳链球菌β溶血素基因cylE缺失突变株的构建[J]. 中国病原生物学杂志, 2019, 14(7): 773–779.
BU R E, WU J H, XILIN G W, et al. Construction of a β-hemolysin gene deletion mutant strain of bovine mastitis Streptococcus agalactiae[J]. Journal of Pathogen Biology, 2019, 14(7): 773–779. (in Chinese) |
| [37] |
杨学云, 李建军, 王蒴, 等. 改良格拉纳达培养基分离奶牛乳房炎奶样中无乳链球菌的效果评价[J]. 新疆农业科学, 2014, 51(11): 2093–2098.
YANG X Y, LI J J, WANG S, et al. Evaluation of the modified granada medium for detection of Streptococcus agalactiae from mastitis samples[J]. Xinjiang Agricultural Sciences, 2014, 51(11): 2093–2098. (in Chinese) |
| [38] |
阚威, 樊杰, 武小虎, 等. 奶牛乳房炎无乳链球菌的分离及PCR鉴定[J]. 中国兽医学报, 2014, 34(8): 1261–1266.
KAN W, FAN J, WU X H, et al. Isolation and PCR-identification of Streptococcus agalactiaee in milk sampled from mastitic dairy cows[J]. Chinese Journal of Veterinary Science, 2014, 34(8): 1261–1266. (in Chinese) |
| [39] | BAKER J R, PRITCHARD D G. Action pattern and substrate specificity of the hyaluronan lyase from group B streptococci[J]. Biochem J, 2000, 348(2): 465–471. |
| [40] | KOLAR S L, KYME P, TSENG C W, et al. Group B Streptococcus evades host immunity by degrading hyaluronan[J]. Cell Host Microbe, 2015, 18(6): 694–704. |
| [41] |
刘龙海.奶牛乳房炎无乳链球菌血清型分布、耐药性及其相关基因的研究[D].北京: 中国农业科学院, 2017.
LIU L H. Distribution of serotypes, antimicrobial susceptibility and related genes of S. agalactiae isolates from mastitis cases[D]. Beijing: Chinese Academy of Agricultural Sciences, 2017. (in Chinese) |
| [42] | HEATH C. Evolution of Streptococcus iniae after vaccination and molecular underpinnings of capsular antigenicity[D]. Brisbane: The University of Queensland, 2016. |
| [43] | PODBIELSKI A, BLANKENSTEIN O, LVTTICKEN R. Molecular characterization of the cfb gene encoding group B streptococcal CAMP-factor[J]. Me Microbiol Immunol, 1994, 183(5): 239–256. |
| [44] | FOUAD M, ZAKARIA S, METWALLY L, et al. Detection of maternal colonization of group B Streptococcus by PCR targeting cfb and scpb genes[J]. J Microbiol Biotechnol Food Sci, 2016, 6(1): 713–716. |
| [45] | VIEIRA L L, PEREZ A V, MACHADO M M, et al. Group B Streptococcus detection in pregnant women:comparison of qPCR assay, culture, and the Xpert GBS rapid test[J]. BMC Pregnancy Childbirth, 2019, 19(1): 532. |
| [46] |
王向柳.奶牛乳腺炎三联苗田间试验及牛源无乳链球菌CAMP因子的克隆表达[D].乌鲁木齐: 新疆农业大学, 2008.
WANG X L. Field trial of triple inactivated vaccine against mastitis of cows and cloning and expression CAMP gene of Streptococcus agalactiae from mastitis milk[D]. Urumqi: Xinjiang Agricultural University, 2008. (in Chinese) |
| [47] | PAOLETTI L C, KASPER D L. Glycoconjugate vaccines to prevent group B streptococcal infections[J]. Expert Opin Biol Ther, 2003, 3(6): 975–984. |
| [48] | SEVERI E, HOOD D W, THOMAS G H. Sialic acid utilization by bacterial pathogens[J]. Microbiology, 2007, 153(9): 2817–2822. |
| [49] |
姚晶, 任婧, 吴正钧, 等. 唾液酸化路易斯-X合成关键酶基因的克隆表达[J]. 中国生物工程杂志, 2011, 31(12): 51–56.
YAO J, REN J, WU Z J, et al. Cloning and expression of the key enzyme gene in biosynthesis of sialyl lewis X[J]. China Biotechnology, 2011, 31(12): 51–56. (in Chinese) |
| [50] | TONG J, FU Y G, WU N H, et al. Sialic acid-dependent interaction of group B streptococci with influenza virus-infected cells reveals a novel adherence and invasion mechanism[J]. Cell Microbiol, 2018, 20(4): e12818. |
| [51] | LEWIS A L, CAO H Z, PATEL S K, et al. NeuA sialic acid O-acetylesterase activity modulates O-acetylation of capsular polysaccharide in group B Streptococcus[J]. J Biol Chem, 2007, 282(38): 27562–27571. |
| [52] | TAMURA G S, KUYPERS J M, SMITH S, et al. Adherence of group B streptococci to cultured epithelial cells:roles of environmental factors and bacterial surface components[J]. Infect Immun, 1994, 62(6): 2450–2458. |
| [53] | NIZET V, KIM K S, STINS M, et al. Invasion of brain microvascular endothelial cells by group B streptococci[J]. Infect Immun, 1997, 65(12): 5074–5081. |
| [54] | FARHAT K, SAUTER K S, BRCIC M, et al. The response of HEK293 cells transfected with bovine TLR2 to established pathogen-associated molecular patterns and to bacteria causing mastitis in cattle[J]. Vet Immunol Immunopathol, 2008, 125(3-4): 326–336. |
| [55] | POYART C, PELLEGRINI E, GAILLOT O, et al. Contribution of Mn-cofactored superoxide dismutuse (SodA) to the virulence of Streptococcus agalactiae[J]. Infect Immun, 2001, 69(8): 5098–5106. |
| [56] | ZHANG H M, JIANG H R, FAN Y L, et al. Transcriptomics and iTRAQ-Proteomics analyses of bovine mammary tissue with Streptococcus agalactiae-induced mastitis[J]. J Agric Food Chem, 2018, 66(42): 11188–11196. |
| [57] | BOHNSACK J F, CHANG J K, HILL H R. Restricted ability of group B streptococcal C5a-ase to inactivate C5a prepared from different animal species[J]. Infect Immun, 1993, 61(4): 1421–1426. |
| [58] | SANTILLAN D A, ANDRACKI M E, HUNTER S K. Protective immunization in mice against group B streptococci using encapsulated C5a peptidase[J]. Am J Obstet Gynecol, 2008, 198(1): 14. E1–114. E6. |
| [59] | MARQUES M B, KASPER D L, PANGBURN M K, et al. Prevention of C3 deposition by capsular polysaccharide is a virulence mechanism of type Ⅲ group B streptococci[J]. Infect Immun, 1992, 60(10): 3986–3993. |
| [60] | MOULIN P, RONG V, SILVA A R E, et al. Defining the role of the Streptococcus agalactiae Sht-family proteins in zinc acquisition and complement evasion[J]. J Bacteriol, 2019, 201(8): e00757–18. |
| [61] | MAISEY H C, DORAN K S, NIZET V. Recent advances in understanding the molecular basis of group B Streptococcus virulence[J]. Exp Rev Mol Med, 2008, 10: e27. |
| [62] | PETERS J, PRICE J, LLEWELYN M. Staphylococcal and streptococcal infections[J]. Medicine, 2017, 45(12): 727–734. |




