2. 吉林农业大学中药材学院, 长春 130118;
3. 吉林农业大学农业质量标准与检测技术研究中心, 长春 130118;
4. 梅花鹿药用资源利用关键技术研究室, 长春 130118;
5. 动物生产及产品质量安全教育部重点实验室, 长春 130118;
6. 吉林省梅花鹿高效养殖和产品开发技术工程研究中心, 长春 130118
2. College of Chinese Medicine Materials, Jilin Agricultural University, Changchun 130118, China;
3. Research Center of Agriculture Quality Standard and Detecting Technique, Jilin Agricultural University, Changchun 130118, China;
4. Research Laboratory on Key Technology of Development and Resource Utilization Sika Deer, Changchun 130118, China;
5. Key Laboratory of Animal Production and Product Quality Safety of Ministry of Education, Changchun 130118, China;
6. Jilin Province Sika Deer Efficient Breeding and Product Development Technology Engineering Research Center, Changchun 130118, China
结核分枝杆菌(Mycobacterium tuberculosis)是一种可以通过气溶胶传播引起人畜共患病的细菌,2018年全球范围内约有160万人死于这种细菌引起的疾病,是全球十大单一传染因子导致死亡的原因之一[1]。全球约5%的结核感染是由多药耐药(MDR-MTB)和广泛耐药(XDR-MTB)菌株引起的[2],中国是全球耐多药结核病负担第二大的地区[3],因此开发新型抗结核药物和研究结核分枝杆菌耐药机制成为中国防治结核病的重点工作[4]。结核分枝杆菌必需基因的研究一直是个难题,这些必需基因约占结核分枝杆菌基因组的20%,并且编码目前所有抗结核药物靶向的蛋白质[5],同时结核分枝杆菌的基因操作所需的时间和试验操作的复杂性,导致它的基本基因功能分析存在很多困难[6]。新型的CRISPR基因组编辑技术可以有效地对结核分枝杆菌必需基因进行操作,为结核分枝杆菌的功能基因组学、基因调控、基因组编辑、遗传相互作用和药物靶标分析等研究提供了便捷工具,对表征结核分枝杆菌的未知功能的基因和致病机制具有重要意义。
成簇规律间隔短回文重复序列(clustered regularly interspaced short palindromic repeats,CRISPR)于1987年在大肠埃希菌中首次被发现[7],原核生物可以通过CRISPR结构使用核酸内切酶以特定的方式识别和切割外来入侵的病毒或质粒,是广泛存在于细菌和古菌中的获得性免疫机制[8]。2012年6月,Jinek等[9]首次在体外证实了Cas9蛋白可以与靶DNA位点结合并进行剪切,这个发现打开了CRISPR基因基因编辑技术的大门。CRISPR/Cas系统是继锌指核酸酶(zinc finger nuclease,ZFN)[10]、转录激活因子效应物核酸酶(transcription activator-like effector nuclease,TALEN)[11]的第3代基因组编辑技术。ZFNs和TALENs系统因为需要针对不同情况来对酶进行重新设计以适应不同靶点,所以构建复杂、时间长且成本较高,其应用受到很大限制。CRISPR/Cas系统不同于前两种由蛋白质驱动的基因组编辑技术,CRISPR/Cas系统是由RNA进行引导,所以CRISPR相关的(Cas)蛋白保持不变,针对不同的基因靶标,仅需改变指导RNA的短序列就可以重新定位靶点进行特异性切割。因其高效、操作简洁、准确性高和成本低等特点,且具有进行多基因共同编辑的潜力,CRISPR/Cas系统已经成为革命性的基因工程工具[12]。如今,CRISPR系统已经被广泛应用在细菌、真核生物以及古细菌的众多基因组中[13-15]。
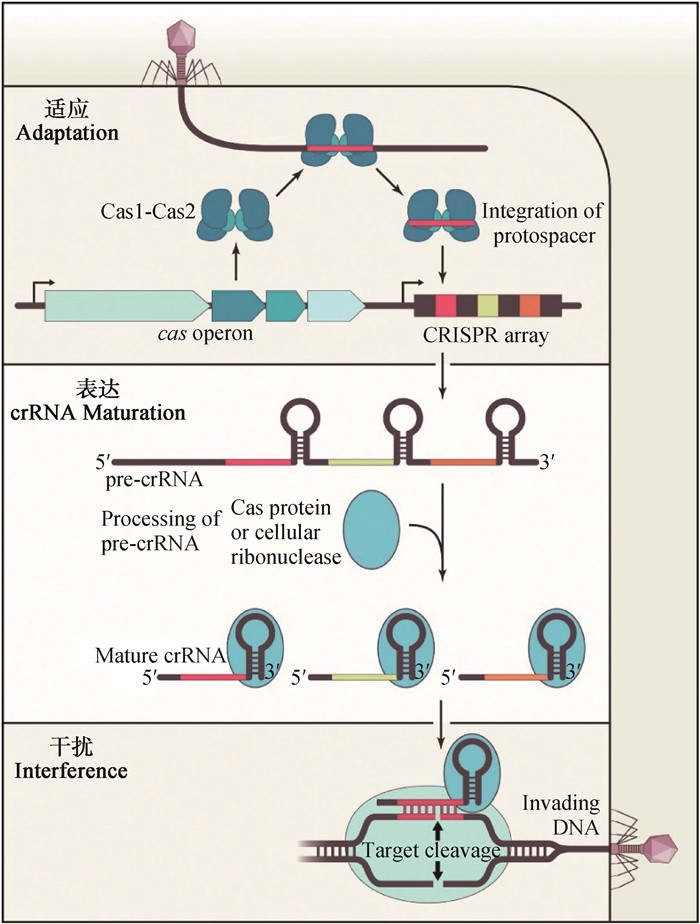
1 CRISPR/Cas系统简介 1.1 CRISPR/Cas系统作用原理CRISPR/Cas系统主要包含CRISPR相关基因(CAS基因)和CRISPR基因座,这些基因座由前导序列、间隔子和直接重复序列组成。CRISPR/Cas系统的适应性免疫分为3个阶段(图 1)[16]:1)适应,当外来的病毒或质粒入侵含有CRISPR系统的细菌时,细菌的多亚基效应复合物会与病毒或者质粒DNA的前间隔序列毗邻基序(protospacer-adjacent motif,PAM)结合,并将与PAM毗邻的一段DNA整合到前导序列与第一个重复序列之间,形成间隔子。2)表达,当外来的病毒和质粒再次入侵细菌时,在前导序列的作用下,CRISPR基因座将转录出前体crRNA(pre-crRNA),然后再加工为成熟crRNA。3)干扰,crRNA与Cas相关蛋白共同作用形成核糖核蛋白复合物(RNP),靶向外来核酸进行切割,使其无法自我复制,进而起到免疫作用[17-18]。
1.2 CRISPR/Cas系统分类CRISPR/Cas系统分布广泛且具有多样性,目前,已经通过干扰蛋白复合物之间的差异,将CRISPR/Cas系统分为两类:第1类是多亚基效应复合物,分为Ⅰ、Ⅲ和Ⅳ型,第2类是单蛋白效应模块,分为Ⅱ、Ⅴ和Ⅵ型[19-20]。
第1类的Ⅰ、Ⅲ和Ⅳ型CRISPR/Cas系统都是采用多亚基效应复合物来防御外来入侵的病毒,其中Ⅰ型系统基因座均包含Cas3基因或者是Cas3的变体[21],Cas3在Ⅰ型CRISPR/Cas系统起重要作用,目前,Ⅰ型被分为IA-IF、IF variant和IU 8个亚型[22],Ⅲ型系统不同于其他CRISPR/Cas系统,除了Ⅲ型系统外,几乎所有的CRISPR/Cas系统都依赖于识别前间隔序列(protospacers)的5′端的短序列(前间隔序列毗邻基序,PAM)来进行剪切干扰外来基因。与Ⅰ型系统相比,Ⅲ型CRISPR/Cas基因座的组成更加多样化,Ⅲ型系统可以同时靶向RNA和DNA,由crRNA编程的效应复合物可以检测到入侵的RNA,从而触发多层防御,其中所有的Ⅲ型系统均包含Cas10基因,其主要通过Cas10的HD核酸酶结构域切割核酸,目前Ⅲ型系统被分为Ⅲ-A、Ⅲ-B、III-B variant、Ⅲ-C、Ⅲ-D 5个亚型[23-24]。Ⅳ型系统的功能尚未得到表征,该系统缺少cas1和cas2基因,并且通常不靠近CRISPR阵列,因其较为罕见,目前, 仅有Ⅳ和Ⅳ variant两种亚型,需要进一步的研究[25]。
第2类的Ⅱ、Ⅴ和Ⅵ型CRISPR/Cas系统仅通过单蛋白效应物发挥作用,其中Ⅴ型的Cas12a(又称Cpf1)和Ⅵ型的Cas13(又称C2c2)都可以通过自身加工crRNA[26-27], Ⅴ型系统被分为Ⅴ-A、Ⅴ-B、Ⅴ-C、Ⅴ-D、Ⅴ-E、Ⅴ-U1、Ⅴ-U2、Ⅴ-U3、Ⅴ-U4、Ⅴ-U5共10种亚型,Ⅵ型系统被分为Ⅵ-A、Ⅵ-B1、Ⅵ-B2、Ⅵ-C共4种亚型。Ⅱ型系统被分为Ⅱ-A、Ⅱ-B、Ⅱ-C、Ⅱ-C variant 4种亚型。Ⅱ型的CRISPR/Cas9系统是目前应用最广泛的基因编辑工具,其准确和高效等特点为快速进行基因工程操作提供了可能。CRISPR/Cas9系统由tracrRNA、crRNA和具有核酸酶活性的Cas9蛋白组成,通过进一步研究发现由tracrRNA和crRNA两部分融合表达的sgRNA也能很好地行使引导功能[28],其在基因组工程应用中显示出了巨大的实用性。
结核分枝杆菌,属于放线菌属的革兰阳性菌,包含1个Ⅲ-A型CRISPR家族[29],该系统由2个分别具有24和18个重复序列的CRISPR阵列组成。2个CRISPR阵列中有36 bp的重复序列高度保守,6个用于靶标干扰的csm基因,1个用于crRNA加工的cas6基因以及1个适应模块[30],最近的研究确定了结核分枝杆菌的一些非典型特征,其成熟crRNA具有均匀的长度(71 nt),其结构与Ⅰ型系统成熟crRNA的结构相似,并且结核分枝杆菌Ⅲ-A型CRISPR/Cas系统中的Cas6还具有独特的金属依赖性[31],使其与其他Ⅲ-A型系统区分开来。
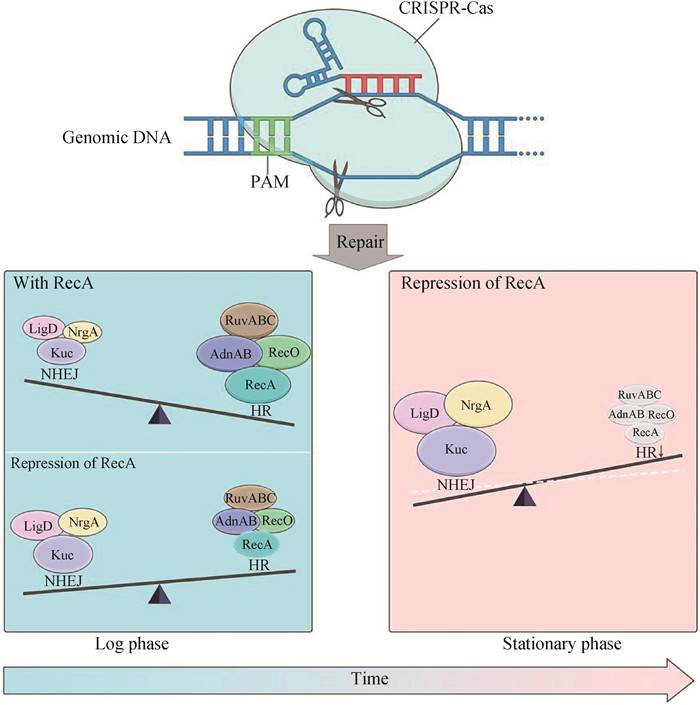
2 CRISPR/Cas系统在结核分枝杆菌研究中的应用 2.1 结核分枝杆菌基因组的编辑结核分枝杆菌因其生长缓慢,细胞壁较厚和高度致病性,导致分枝杆菌的基因工程极具挑战性,在过去的几十年中,已经开发出了多种针对结核分枝杆菌的基因工程方法,目前,主要方法有基于一步法和两步法的等位基因交换的定点突变[32-34],使用分枝杆菌噬菌体的专门传导[35-36]及分枝杆菌定向诱变ORBIT系统[37]。新开发的ORBIT系统结合了两个高效噬菌体重组系统,并且针对不同基因可以使用基本相同的方法生成缺失或者插入,但这些方法通常只能产生少量的突变体,并且需要多个步骤来产生无标记的突变体,如果进行多基因操作可能需要数年时间[2]。严美怡等[38]在2017年首次将CRISPR-Cas12a系统应用到耻垢分枝杆菌中,通过构建含有四环素诱导型启动子(FnCpf1)和gp60、gp61重组蛋白的质粒,和由温度敏感型复制子控制表达crRNAs并含有无标记的dsDNA(或者ssDNA)的质粒,转化到细胞中,形成的crRNA-Cas12a复合物可以结合并切割目标基因,所产生的双链断裂(DSB)通过重组修复,可以导致点突变,缺失或插入。随后Sun(孙冰冰)等[39]在耻垢分枝杆菌中构建了CRISPR-FnCpf1-assisted NHEJ系统,通过筛选14种不同的Cas效应蛋白,最终在使用谷氨酸棒状杆菌(Corynebacterium glutamicum)密码子优化的Cpf1效应蛋白(FnCpf1_cg)构建的CRISPR-FnCpf1_cg系统中检测到非同源末端链接(NHEJ),与之前的CRISPR-Cas12a系统相比,该方法无需添加重组蛋白和同源DNA模板,只需要共表达FnCpf1和靶向目的基因的crRNA即可进行基因编辑。2020年,Yan(严美怡)等[2]通过改善NHEJ修复途径开发了CRISPR-Cas-NHEJ辅助基因组编辑方法(图 2)[2],不需要模板DNA参与就可以进行基因组编辑。因为NHEJ途径需要DNA末端结合蛋白Ku和多官能团连接酶d(LigD),Ku蛋白与DNA末端结合并募集LigD,然后LigD加工并连接DNA末端[40],通过人为增加Ku蛋白和LigD的表达,并抑制可以增加HR途径的RecA活性[41],进而强烈诱导结核分枝杆菌的NHEJ途径。该系统将对分枝杆菌的研究速度、研究范围和药物靶标的鉴定等方面具有重要意义,值得进行更深入的研究。
|
图 2 分枝杆菌中CRISPR-Cas-NHEJ辅助基因组编辑[2] Fig. 2 CRISPR-Cas-NHEJ-assisted genome editing in mycobacteria[2] |
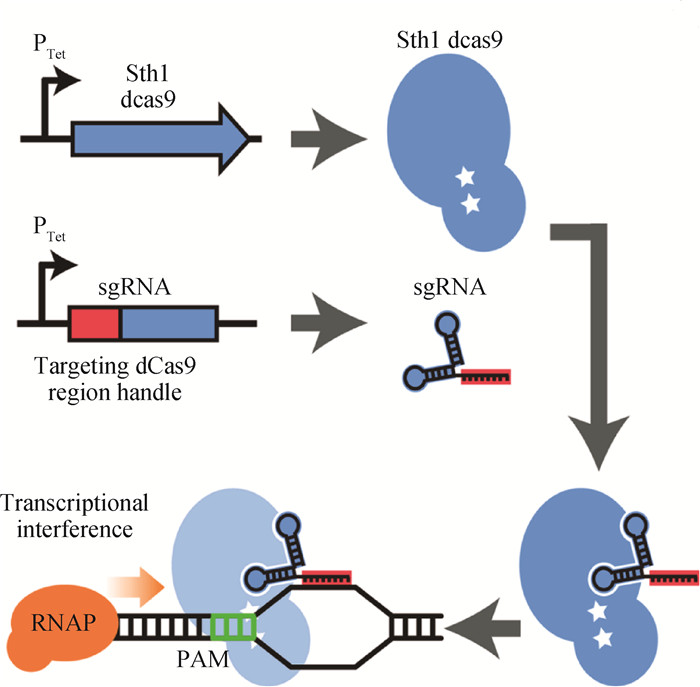
Ⅱ型CRISPR-Cas系统是最常用的基因组编辑工具,Ⅱ型CRISPR-Cas系统的Cas9蛋白可以通过两个核酸裂解域(RuvC和HNH),裂解靶DNA链并产生双链断裂(DSB)[9],从而进行基因编辑。CRISPR干扰(CRISPRi)系统是通过使用核酸内切酶缺陷型Cas9(又称dCas9)开发的一种改良的CRISPR系统,其通过人为在Cas9每个结构域(分别是D10A和H840A)中引入点突变,突变后的Cas9蛋白不具备裂解活性,但不影响其与靶基因的结合,可以通过sgRNA引导至靶基因,达到靶向基因调控的作用,该系统可以将基因表达抑制数千倍以上[42]。该系统与RNAi有显著的不同,RNAi的基因调控需要在翻译之前破坏已转录的信使RNA,而CRISPRi可以利用RNA直接阻断转录。2015年,由Choudhary等[43]首次将CRISPRi应用在结核分枝杆菌中,通过引入化脓性链球菌(革兰阳性菌)的dCas9,并使用ATc诱导型启动子来实现结核分枝杆菌靶基因的受控表达。2016年,Singh等[5]在Choudhary等的研究基础上进一步验证了该系统对结核分枝杆菌必需基因的抑制作用,并证明了CRISPRi对结核分枝杆菌基因组的显著抑制作用。2017年,Rock等[44]筛选了11种不同来源的Cas9蛋白,发现嗜热链球菌(Streptococcus thermophilus)的dCas9 Sth1最为有效,且蛋白毒性最小。加入小分子诱导剂脱水四环素或强力霉素可诱导Sth1 dCas9和sgRNA表达,从而导致靶基因的转录沉默,这种CRISPRi系统为结核基因组(图 3)[6],尤其是针对不能被传统基于重组的基因编辑工具操作的必需基因的研究提供了简单、快速、高效的工具,这也为快速寻找结核药物靶点、开发新型抗结核药物提供了可能[6],并且该系统被证明可以同时抑制多个靶基因,这对于生长缓慢的结核分枝杆菌具有重要意义。目前,这种新型的CRISPRi系统已经在结核分枝杆菌的基因组研究领域得到应用[45-46]。
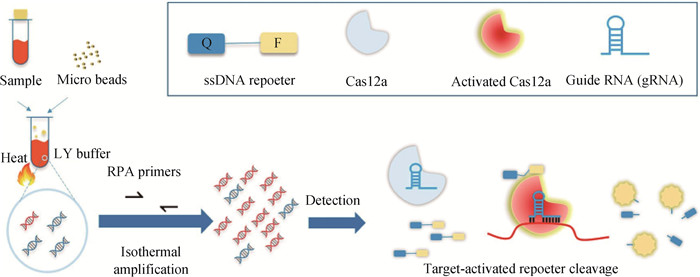
2.3 结核分枝杆菌的核酸检测2018年,Science上发表了Chen等[47]的发现,Cas12a(Cpf1)和相关的Ⅴ型CRISPR干扰蛋白在guide RNA引导下与DNA结合后会释放非特异性的单链(ssDNA)DNase活性,这种强大并且非特异性的ssDNA裂解活性,是由负责特异性双链DNA(dsDNA)切割活性的同一位点催化的,又因为ssDNA的切割需要识别与20个核苷酸的gRNA相匹配的DNA靶序列,所以将依赖性Cas12a ssDNase激活与等温扩增相结合,建立了一种DNA内切酶靶向CRISPR反式报告(DETECTR)的方法,用于快速且特异性的核酸检测。2019年,Ai等[48]在DETECTR基础上开发了一种结核快速检测方法(简称CRISPR-MTB)[48],该方法是将重组酶聚合酶扩增(recombinase polymerase amplification,RPA)和Cas12a检测结合到DETECTR方法中,该方法将样品、正反引物、反应缓冲液、乙酸镁和RPA混合物的反应液在37 ℃下孵育30 min。之后,将所有扩增产物添加到由gRNA、Cas12a和ssDNA报告基因组成的CRISPR反应混合物中,最终反应在37 ℃下温育,然后,通过qPCR仪收集荧光信号(图 4)。作者将CRISPR-MTB和GeneXpert MTB / RIF分析(Xpert)进行比较,发现这种基于CRISPR的活动性结核病诊断方法,具有接近单拷贝的灵敏度,并且该方法还具有同时检测与药物反应相关的多个位点的潜力,这对结核病的检测具有重要意义。
2.4 结核分枝杆菌的基因分型基因分型是表征结核分枝杆菌的细菌进化和系统发育的重要手段[49],可以更好地了解病原体特定的传播和发病机制。CRISPR序列是由一段交替的同向DNA重复序列(DRs)和可变的间隔序列组成。在CRISPR序列中,同向DNA重复序列的大小和顺序几乎没有差别,但间隔序列在不同致病菌、相同致病菌的不同血清型和不同菌株之间差异很大, 所以从数量和性质上分析CRISPR间隔区的结构可以进行有效的基因分型[50]。因为结核分枝杆菌复合体(Mycobacterium tuberculosis complex,MTBC)成员基因组中CRISPR结构的存在,Kamerbeek等[51]开发了间隔区寡核苷酸分型(spacer oligonucletide typing, Spoligotyping),这种方法基于体外扩增的DNA与多个间隔寡核苷酸的应变依赖性杂交模式,对由保守DR隔开的43个寡核苷酸间隔子进行分析,该方法具有高度可重复性,已发展成为大型分子流行病学项目的高通量检测方法,被广泛用于结核分枝杆菌的分型等相关研究中[52-54],同时也已经被应用到其他生物中[55-56]。当前有两种方法用于获取spoligotype。第1种方法(原始方法)基于反向杂交,将PCR产物与含有合成寡核苷酸的膜上杂交,该合成寡核苷酸具有与间隔序列互补的序列,使其中每个间隔子的序列均附着在膜上的特定区域[57],当PCR产物与特定寡核苷酸序列未产生标记,则表明在所检查的菌株中不存在相应的间隔区序列。第2种方法基于Luminex技术(Luminex technology),其中,每个间隔寡核苷酸通过共价连接至微球,使用微球用作捕获探针,同时每个微球上还包含两个共价连接的荧光染料,一束激光将激发荧光染料以识别间隔子,第二激光将激发结合到含有间隔子的PCR产物的报道分子上[55]。
Spoligotype数据库已经被证明可以用于确定和分类世界范围的结核分枝杆菌复合体遗传多样性,第四个国际性基因分型数据库SpolDB4是迄今为止关于结核分枝杆菌复杂遗传多态性的最大的公开数据库之一,有来自122个国家的39 295株菌株的1 939种共享型(STs),使用基于专家和生物信息学的混合方法,暂定将其分为62个进化枝/谱系,它提供了当前MTBC基因组多样性与物种分类和进化史之间的关系[58]。这积极推动了各地区针对不同的结核分枝杆菌基因分型分离株精确制定公共卫生策略和评估防控措施的发展。
3 展望随着生物技术的不断发展,CRISPR/Cas系统已经成为功能多样化的基因编辑工具,被广泛应用在生物医学、遗传育种、药物开发等相关领域。CRISPR/Cas系统极大地提高了结核分枝杆菌的研究效率,在抗结核药物的研发,表征结核分枝杆菌的发病机制和耐药机制等方面具有巨大潜力。然而,CRISPR/Cas系统在结核分枝杆菌中的应用仍然有许多问题需要进一步研究,比如优化相关Cas蛋白在结核菌中的表达和潜在的脱靶问题。其次,需要更加高效安全的递送系统来进行CRISPR-Cas模块的转化。相对于结核分枝杆菌这种古老的病原体,CRISPR/Cas系统在结核菌的应用仍然属于较新的领域,但随着对CRISPR / Cas系统分子机制的深入了解,必将会在不久的将来产生更多新颖的技术,对降低结核分枝杆菌的危害具有重要意义。
| [1] | WHO. Global tuberculosis report 2019[R]. Geneva: World Health Organization, 2019. |
| [2] | YAN M Y, LI S S, DING X Y, et al. A CRISPR-assisted nonhomologous end-joining strategy for efficient genome editing in Mycobacterium tuberculosis[J]. mBio, 2020, 11(1): e02364–19. |
| [3] | YANG C G, LUO T, SHEN X, et al. Transmission of multidrug-resistant Mycobacterium tuberculosis in Shanghai, China:a retrospective observational study using whole-genome sequencing and epidemiological investigation[J]. Lancet Infect Dis, 2017, 17(3): 275–284. DOI: 10.1016/S1473-3099(16)30418-2 |
| [4] | WEI J W, LU N, LI Z Y, et al. The Mycobacterium tuberculosis CRISPR-associated cas1 involves persistence and tolerance to anti-tubercular drugs[J]. Biomed Res Int, 2019, 2019: 7861695. |
| [5] | SINGH A K, CARETTE X, POTLURI L P, et al. Investigating essential gene function in Mycobacterium tuberculosis using an efficient CRISPR interference system[J]. Nucleic Acids Res, 2016, 44(18): e143. DOI: 10.1093/nar/gkw625 |
| [6] | ROCK J. Tuberculosis drug discovery in the CRISPR era[J]. PLoS Pathog, 2019, 15(9): e1007975. DOI: 10.1371/journal.ppat.1007975 |
| [7] | ISHINO Y, SHINAGAWA H, MAKINO K, et al. Nucleotide sequence of the iap gene, responsible for alkaline phosphatase isozyme conversion in Escherichia coli, and identification of the gene product[J]. J Bacteriol, 1987, 169(12): 5429–5433. DOI: 10.1128/JB.169.12.5429-5433.1987 |
| [8] | BARRANGOU R, FREMAUX C, DEVEAU H, et al. CRISPR provides acquired resistance against viruses in prokaryotes[J]. Science, 2007, 315(5819): 1709–1712. DOI: 10.1126/science.1138140 |
| [9] | JINEK M, CHYLINSKI K, FONFARA I, et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity[J]. Science, 2012, 337(6096): 816–821. DOI: 10.1126/science.1225829 |
| [10] | KIM Y G, CHA J, CHANDRASEGARAN S. Hybrid restriction enzymes:zinc finger fusions to Fok I cleavage domain[J]. Proc Natl Acad Sci U S A, 1996, 93(3): 1156–1160. DOI: 10.1073/pnas.93.3.1156 |
| [11] | MOSCOU M J, BOGDANOVE A J. A simple cipher governs DNA recognition by TAL effectors[J]. Science, 2009, 326(5959): 1501. DOI: 10.1126/science.1178817 |
| [12] | COX D B T, PLATT R J, ZHANG F. Therapeutic genome editing:prospects and challenges[J]. Nat Med, 2015, 21(2): 121–131. DOI: 10.1038/nm.3793 |
| [13] | SELLE K, BARRANGOU R. Harnessing CRISPR-Cas systems for bacterial genome editing[J]. Trends Microbiol, 2015, 23(4): 225–232. DOI: 10.1016/j.tim.2015.01.008 |
| [14] | KOMOR A C, BADRAN A H, LIU D R. CRISPR-based technologies for the manipulation of eukaryotic genomes[J]. Cell, 2017, 168(1-2): 20–36. DOI: 10.1016/j.cell.2016.10.044 |
| [15] | NAYAK D D, METCALF W W. Cas9-mediated genome editing in the methanogenic archaeon Methanosarcina acetivorans[J]. Proc Natl Acad Sci U S A, 2017, 114(11): 2976–2981. DOI: 10.1073/pnas.1618596114 |
| [16] | HILLE F, RICHTER H, WONG S P, et al. The biology of CRISPR-cas:backward and forward[J]. Cell, 2018, 172(6): 1239–1259. DOI: 10.1016/j.cell.2017.11.032 |
| [17] | PLAGENS A, RICHTER H, CHARPENTIER E, et al. DNA and RNA interference mechanisms by CRISPR-Cas surveillance complexes[J]. FEMS Microbiol Rev, 2015, 39(3): 442–463. DOI: 10.1093/femsre/fuv019 |
| [18] | BHAYA D, DAVISON M, BARRANGOU R. CRISPR-Cas systems in bacteria and archaea:versatile small RNAs for adaptive defense and regulation[J]. Annu Rev Genet, 2011, 45: 273–297. DOI: 10.1146/annurev-genet-110410-132430 |
| [19] | MAKAROVA K S, WOLF Y I, ALKHNBASHI O S, et al. An updated evolutionary classification of CRISPR-Cas systems[J]. Nat Rev Microbiol, 2015, 13(11): 722–736. DOI: 10.1038/nrmicro3569 |
| [20] | MOHANRAJU P, MAKAROVA K S, ZETSCHE B, et al. Diverse evolutionary roots and mechanistic variations of the CRISPR-Cas systems[J]. Science, 2016, 353(6299): aad5147. DOI: 10.1126/science.aad5147 |
| [21] | SINKUNAS T, GASIUNAS G, FREMAUX C, et al. Cas3 is a single-stranded DNA nuclease and ATP-dependent helicase in the CRISPR/Cas immune system[J]. Embo J, 2011, 30(7): 1335–1342. DOI: 10.1038/emboj.2011.41 |
| [22] | MAKAROVA K S, HAFT D H, BARRANGOU R, et al. Evolution and classification of the CRISPR-Cas systems[J]. Nat Rev Microbiol, 2011, 9(6): 467–477. DOI: 10.1038/nrmicro2577 |
| [23] | SAMAI P, PYENSON N, JIANG W Y, et al. Co-transcriptional DNA and RNA cleavage during type III CRISPR-cas immunity[J]. Cell, 2015, 161(5): 1164–1174. DOI: 10.1016/j.cell.2015.04.027 |
| [24] | TAMULAITIS G, KAZLAUSKIENE M, MANA-KOVA E, et al. Programmable RNA shredding by the type III-A CRISPR-Cas system of Streptococcus thermophilus[J]. Mol Cell, 2014, 56(4): 506–17. DOI: 10.1016/j.molcel.2014.09.027 |
| [25] | ÖZCAN A, PAUSCH P, LINDEN A, et al. Type IV CRISPR RNA processing and effector complex formation in Aromatoleum aromaticum[J]. Nat Microbiol, 2019, 4(1): 89–96. DOI: 10.1038/s41564-018-0274-8 |
| [26] | ABUDAYYEH O O, GOOTENBERG J S, KONERMANN S, et al. C2c2 is a single-component programmable RNA-guided RNA-targeting CRISPR effector[J]. Science, 2016, 353(6299): aaf5573. DOI: 10.1126/science.aaf5573 |
| [27] | DONG D, REN K, QIU X L, et al. The crystal structure of Cpf1 in complex with CRISPR RNA[J]. Nature, 2016, 532(7600): 522–526. DOI: 10.1038/nature17944 |
| [28] | NAJAH S, SAULNIER C, PERNODET J L, et al. Design of a generic CRISPR-Cas9 approach using the same sgRNA to perform gene editing at distinct loci[J]. BMC Biotechnol, 2019, 19(1): 18. DOI: 10.1186/s12896-019-0509-7 |
| [29] | KOONIN E V, MAKAROVA K S, ZHANG F. Diversity, classification and evolution of CRISPR-Cas systems[J]. Curr Opin Microbiol, 2017, 37: 67–78. DOI: 10.1016/j.mib.2017.05.008 |
| [30] | HE L M, FAN X Y, XIE J P. Comparative genomic structures of Mycobacterium CRISPR-Cas[J]. J Cell Biochem, 2012, 113(7): 2464–2473. DOI: 10.1002/jcb.24121 |
| [31] | WEI W J, ZHANG S, FLEMING J, et al. Mycobacterium tuberculosis type Ⅲ-A CRISPR/Cas system crRNA and its maturation have atypical features[J]. FASEB J, 2019, 33(1): 1496–1509. DOI: 10.1096/fj.201800557RR |
| [32] | NORMAN E, DELLAGOSTIN O A, MCFADDEN J, et al. Gene replacement by homologous recombination in Mycobacterium bovis BCG[J]. Mol Microbiol, 1995, 16(4): 755–60. DOI: 10.1111/j.1365-2958.1995.tb02436.x |
| [33] | MALAGA W, PEREZ E, GUILHOT C. Production of unmarked mutations in mycobacteria using site-specific recombination[J]. FEMS Microbiol Lett, 2003, 219(2): 261–268. DOI: 10.1016/S0378-1097(03)00003-X |
| [34] | SONG H H, NIEDERWEIS M. Functional expression of the Flp recombinase in Mycobacterium bovis BCG[J]. Gene, 2007, 399(2): 112–119. DOI: 10.1016/j.gene.2007.05.005 |
| [35] | JACOBS W R, TUCKMAN M, BLOOM B R. Introduction of foreign DNA into mycobacteria using a shuttle phasmid[J]. Nature, 1987, 327(6122): 532–535. DOI: 10.1038/327532a0 |
| [36] | BARDAROV S, KRIAKOV J, CARRIERE C, et al. Conditionally replicating mycobacteriophages:a system for transposon delivery to Mycobacterium tuberculosis[J]. Proc Natl Acad Sci U S A, 1997, 94(20): 10961–10966. DOI: 10.1073/pnas.94.20.10961 |
| [37] | MURPHY K C, NELSON S J, NAMBI S, et al. ORBIT:a new paradigm for genetic engineering of mycobacterial chromosomes[J]. mBio, 2018, 9(6): e01467–18. |
| [38] | YAN M Y, YAN H Q, REN G X, et al. CRISPR-Cas12a-Assisted recombineering in bacteria[J]. Appl Environ Microbiol, 2017, 83(17). DOI: 10.1128/aem.00947-17 |
| [39] | SUN B, YANG J, YANG S, et al. A CRISPR-Cpf1-assisted non-homologous end joining genome editing system of Mycobacterium smegmatis[J]. Biotechnol J, 2018, 13(9): 1700588. DOI: 10.1002/biot.201700588 |
| [40] | SHUMAN S, GLICKMAN M S. Bacterial DNA repair by non-homologous end joining[J]. Nat Rev Microbiol, 2007, 5(11): 852–861. DOI: 10.1038/nrmicro1768 |
| [41] | MOREB E A, HOOVER B, YASEEN A, et al. Managing the SOS response for enhanced CRISPR-Cas-based recombineering in E. coli through transient inhibition of host RecA activity[J]. ACS Synth Biol, 2017, 6(12): 2209–2218. DOI: 10.1021/acssynbio.7b00174 |
| [42] | QI L S, LARSON M H, GILBERT L A, et al. Repurposing CRISPR as an RNA-guided platform for sequence-specific control of gene expression[J]. Cell, 2013, 152(5): 1173–1183. DOI: 10.1016/j.cell.2013.02.022 |
| [43] | CHOUDHARY E, THAKUR P, PAREEK M, et al. Gene silencing by CRISPR interference in mycobacteria[J]. Nat Commun, 2015, 6(1): 6267. DOI: 10.1038/ncomms7267 |
| [44] | ROCK J M, HOPKINS F F, CHAVEZ A, et al. Programmable transcriptional repression in mycobacteria using an orthogonal CRISPR interference platform[J]. Nat Microbiol, 2017, 2(4): 16274. DOI: 10.1038/nmicrobiol.2016.274 |
| [45] | MCNEIL M B, COOK G M. Utilization of CRISPR interference to validate MmpL3 as a drug target in Mycobacterium tuberculosis[J]. Antimicrob Agents Chemother, 2019, 63(8): e00629–19. |
| [46] | CHOUDHARY E, SHARMA R, KUMAR Y, et al. Conditional silencing by CRISPRi reveals the role of DNA Gyrase in formation of drug-tolerant persister population in Mycobacterium tuberculosis[J]. Front Cell Infect Microbiol, 2019, 9: 70. DOI: 10.3389/fcimb.2019.00070 |
| [47] | CHEN J S, MA E B, HARRINGTON L B, et al. CRISPR-Cas12a target binding unleashes indiscriminate single-stranded DNase activity[J]. Science, 2018, 360(6387): 436–439. DOI: 10.1126/science.aar6245 |
| [48] | AI J W, ZHOU X, XU T, et al. CRISPR-based rapid and ultra-sensitive diagnostic test for Mycobacterium tuberculosis[J]. Emerg Microbes Infect, 2019, 8(1): 1361–1369. DOI: 10.1080/22221751.2019.1664939 |
| [49] | COMAS I, GAGNEUX S. The past and future of tuberculosis research[J]. PLoS Pathog, 2009, 5(10): e1000600. DOI: 10.1371/journal.ppat.1000600 |
| [50] | BOTELHO A, CANTO A, LEÃO C, et al. Clustered regularly interspaced short palindromic repeats (CRISPRs) analysis of members of the Mycobacterium tuberculosis complex[M]//CUNHA M V, INÁCIO J. Veterinary infection biology: Molecular diagnostics and high-throughput strategies. New York: Humana Press, 2015: 373-389. |
| [51] | KAMERBEEK J, SCHOULS L, KOLK A, et al. Simultaneous detection and strain differentiation of Mycobacterium tuberculosis for diagnosis and epidemiology[J]. J Clin Microbiol, 1997, 35(4): 907–914. DOI: 10.1128/JCM.35.4.907-914.1997 |
| [52] | SONG E J, JEONG H J, LEE S M, et al. A DNA chip-based spoligotyping method for the strain identification of Mycobacterium tuberculosis isolates[J]. J Microbiol Methods, 2007, 68(2): 430–433. DOI: 10.1016/j.mimet.2006.09.005 |
| [53] | TAFAJ S, ZHANG J, HAUCK Y, et al. First insight into genetic diversity of the Mycobacterium tuberculosis complex in albania obtained by multilocus variable-number tandem-repeat analysis and spoligotyping reveals the presence of beijing multidrug-resistant isolates[J]. J Clin Microbiol, 2009, 47(5): 1581–1584. DOI: 10.1128/JCM.02284-08 |
| [54] | STREICHER E M, VICTOR T C, VAN DER SPUY G, et al. Spoligotype signatures in the Mycobacterium tuberculosis complex[J]. J Clin Microbiol, 2007, 45(1): 237–240. DOI: 10.1128/JCM.01429-06 |
| [55] | COWAN L S, DIEM L, BRAKE M C, et al. Transfer of a Mycobacterium tuberculosis genotyping method, spoligotyping, from a reverse line-blot hybridization, membrane-based assay to the Luminex multianalyte profiling system[J]. J Clin Microbiol, 2004, 42(1): 474–477. DOI: 10.1128/JCM.42.1.474-477.2004 |
| [56] | MOKROUSOV I, LIMESCHENKO E, VYAZOVAYA A, et al. Corynebacterium diphtheriae spoligotyping based on combined use of two CRISPR loci[J]. Biotechnol J, 2007, 2(7): 901–906. DOI: 10.1002/biot.200700035 |
| [57] | DRISCOLL J R. Spoligotyping for molecular epidemiology of the Mycobacterium tuberculosis complex[M]//CAUGANT D A. Molecular Epidemiology of Microorganisms. Totowa: Humana Press, 2009: 117-128. |
| [58] | BRUDEY K, DRISCOLL J R, RIGOUTS L, et al. Mycobacterium tuberculosis complex genetic diversity:mining the fourth international spoligotyping database (SpolDB4) for classification, population genetics and epidemiology[J]. BMC Microbiol, 2006, 6: 23. DOI: 10.1186/1471-2180-6-23 |