塞内卡病毒A(Senecavirus A,SVA),曾被称为塞内卡谷病毒(Seneca valley virus,SVV),属于小RNA病毒科,是无囊膜的单股正链RNA病毒,对其保守多肽区分析发现,SVA的P1、2C、3C和3D多肽与心病毒属的P1、2C、3C和3D多肽的遗传关系最为密切,但经基因组进化分析否定了SVA来源于心病毒属的可能,因此将其独立列为一种新的病毒属,即塞内卡病毒属[1-2]。研究证明该病毒能够特异性靶向神经内分泌细胞,具有良好的溶瘤特性,且对人体无有害影响,为肿瘤的治疗提供了一种可靠的途径,目前SVA肿瘤治疗法已进入不同阶段的临床研究[3-6]。2007—2016年期间,加拿大、美国、巴西、中国以及哥伦比亚等多地相继暴发特发性水疱病(PIVD)[7-16],并从发病猪体内分离到SVA,在排除口蹄疫(FMD)、猪水疱病(SVD)、水泡性口炎(VS)、猪水疱疹(VES)等水疱病毒感染后,相关学者提出了SVA与PIDV的暴发可能存在因果关系。2008—2012年期间,有相关研究报道母猪水疱症状与血清中SVA的RNA量呈正相关[7-8]。但在2016年前,利用已分离的SVA病毒株人工感染猪体,并未观察到预期的水疱症状。直到2016年,Montiel等[17]对9周龄的育肥猪进行鼻腔途径攻毒,最终引起了水疱症状,证明该病毒是引起猪PIDV的一种新病原,而在此之前多次人工感染失败,可能与实验猪的日龄有关。自然感染SVA的发病母猪表现短暂的低热,蹄部叉趾间隙、冠状带周围以及鼻腔黏膜处有充满液体或破裂的囊泡[14, 18],SVA感染可致新生仔猪死亡,在巴西发生的感染仔猪病死率在30%~70%之间[13]。最近中国新分离的SVA毒株多与美国流行毒株同源关系最近[19]。据报道,该病毒感染也与猪流行性短暂新生儿损失(ETNLs)有关,对此目前尚无足够的证据证明两者之间的联系[19-21]。人炭疽毒素受体1(ANTXR1)是一种人类的肿瘤内皮标记物,最近研究揭示了ANTXR1是SVA的细胞受体[22]。
SVA是塞内卡病毒属的唯一成员,基因组全长约7.2 kb,含有编码2 181个氨基酸的开放阅读框(OFR),基因组呈典型的L-4-3-4模式,P1基因区域编码4个结构蛋白(VP1、VP2、VP3和VP4),是病毒衣壳的主要成分,P2和P3基因区域编码7个非结构蛋白(2A、2B、2C、3A、3B、3C和3D),这些蛋白与病毒复制功能相关的[1]。SVV-001的内部核糖体进入位点的元件,与丙型肝炎病毒及其他多种小RNA病毒相似。分析其基因组,发现5′UTR和3D基因区域相对保守,是PCR引物设计的理想基因区域。虽然SVA对猪群致死率低,但由于发病猪表现的水疱症状与口蹄疫难以区分,极易造成错误诊断,尤其是近年来该病毒在我国多个省份扩散迅速,因此迫切需要建立一种简便、快捷、有效的检测方法,以便对疫病进行监测和快速正确诊断,从而采取相应措施控制疾病的扩散。目前国外已建立了多种检测方法,包括间接免疫荧光抗体试验(IFA)、RNA原位杂交技术、竞争性酶联免疫吸附以及间接酶联免疫吸附试验、血清中和试验、荧光定量PCR等[14-15, 23-26],国内樊晓旭等[27-30]也建立了荧光定量PCR、重组酶聚合酶扩增技术(RPA)实时荧光检测方法以及重组酶聚合酶扩增-侧流层析试纸条检测方法。针对病毒核酸的检测,荧光定量PCR方法具有优异的灵敏性和特异性,但容易造成污染,且仪器设备昂贵,对操作人员要求较高,仅适合实验室诊断。而RT-PCR方法操作简便,灵敏度高,特异性强,不但可用于实验室诊断,同样也适用于养殖场SVA的检测,因此本研究在SVA的基因保守区域设计了3对PCR引物,并从中选出最佳引物,对扩增体系和扩增条件优化后,建立了RT-PCR检测方法,以期为SVA疫病的监测、诊断和防控提供一种有效手段。
1 材料与方法 1.1 主要试剂和材料RNA提取试剂盒购自OMEGA生物科技有限公司,Taq DNA聚合酶、Oligo dT18、dNTP、M-MLV反转录酶购自TaKaRa生物科技有限公司。
SVA CH-SD毒株、EMCV NJ08株、PRV ZJ01株、PRRSV经典毒株S1、CSFV、PEDV由本实验室分离并保存,FMDV为购买的商品化疫苗毒株。
100份猪临床病料(水疱液、心、扁桃体)分别采集于山东省的20个养殖场,按1:2体积比加入含250 IU·mL-1青链霉素的0.1 mol·L-1 PBS,匀浆后冻存于-80 ℃备用。
1.2 SVA RNA提取与反转录将SVA接种于BHK-21细胞,待80%细胞产生CPE时,反复冻融3次,10 000 r·min-1离心10 min后,取上清备用。根据RNA提取试剂盒说明书,提取病毒总RNA,加入到10 μL反转录体系中:5×buffer 2 μL、dNTP mix 0.5 μL、Oligo(dT)18 Primer 0.5 μL、病毒RNA模板7 μL,70 ℃水浴5 min,4 ℃冰浴2 min,再加入0.25 μL M-MLV反转录酶,42 ℃水浴1 h后得到病毒cDNA,于-70 ℃保存备用。
1.3 设计引物与特异性检验本研究参照NCBI所报道的40株SVA毒株基因序列,利用Primer5.0软件在保守区域设计了3对引物:P1、P2、P3(表 1),并委托金斯瑞生物技术有限公司合成。按照“1.2”步骤获得SVA的cDNA,同时以实验室保存的EMCV NJ08株、FMDV、PRV ZJ01株、PRRSV经典毒株S1、CSFV、PEDV等病毒cDNA或DNA为模板,分别用3对引物同等条件下进行PCR扩增,从中选出扩增效率最高、特异性最强的引物用于SVA RT-PCR检测方法的建立。
|
|
表 1 扩增SVA保守基因序列的引物设计 Table 1 The list of primers for amplifying the conservative sequences of SVA gene |
12.5 μL Taq DNA聚合酶混合物、1 μL上游引物、1 μL下游引物、1 μL SVA cDNA,ddH2O补足25 μL PCR扩增体系。保持95 ℃预变性5 min,95 ℃变性30 s不变,依次对上、下游引物终浓度(4、4 mmol·L-1,4、2 mmol·L-1,4、1 mmol·L-1,1、4 mmol·L-1,2、4 mmol·L-1)、退火温度(52、54、56、58、60、62 ℃)30 s、72 ℃延长时间(15、20、25、30、35、40 s)、循环次数(15、20、25、30、35、40次)进行优化。
1.5 重复性检验用该方法对同一SVA cDNA进行6次重复PCR扩增,检验其重复性。
1.6 三种RT-PCR方法比较用本研究建立的方法和国外文献[15, 31]报道的两种PCR方法进行以下两组试验的比较。
1.6.1 敏感性试验将已知TCID50为109·mL-1的SVA细胞毒上清,用0.1 mol·L-1 PBS进行一系列10倍比稀释后,提取病毒RNA,分别用三种RT-PCR方法扩增,进行敏感性比较。
1.6.2 符合率试验从来自山东的100份猪组织样匀浆液中随机选取40份,10 000 r·min-1离心10 min后,取250 μL上清,按照“1.2”所述步骤处理后,用以上三种RT-PCR方法同时进行检测,比较符合率。
1.7 临床检测按照“1.6.2”所述步骤,对山东的100份组织匀浆液处理后,再按照“1.2”所述方法提取病毒RNA,反转录后获得cDNA,采用本研究建立的RT-PCR检测方法进行检测,分析SVA在该地区的流行情况。
2 结果 2.1 特异性扩增引物的选择以SVA CH-SD毒株、EMCV NJ08株、FMDV、PRV ZJ01株、PRRSV经典毒株S1、CSFV、PEDV等病毒cDNA或DNA为模板,分别用P1、P2、P3引物对扩增,扩增大小分别为694、469和333 bp。结果显示(图 1),3对引物中,只有P1引物仅能特异性地以SVA cDNA为模板扩增出694 bp的单一目的条带,而与其他病毒cDNA或DNA均无交叉反应,且扩增条带最亮,表明P1引物扩增效率最高,且特异性最好,因此选择P1引物作为本研究的扩增引物。

|
M. DL2000 DNA相对分子质量标准;1~7.SVA、EMCV、FMDV、PRV、PRRSV、CSFV、PEDV;8.阴性对照 M. DL2000 DNA marker; 1-7. SVA, EMCV, FMDV, PRV, PRRSV, CSFV, PEDV; 8. Negative control 图 1 引物特异性鉴定 Fig. 1 The results of specificity detection of the primers |
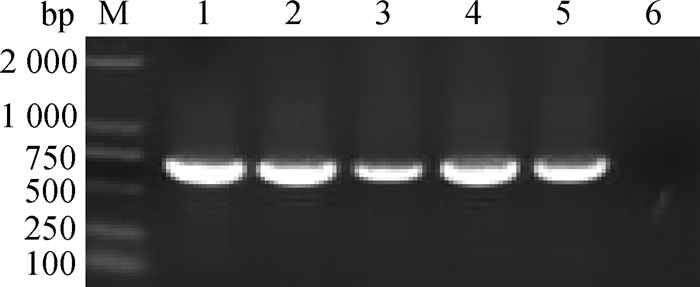
通过对上、下游引物不同浓度的优化,最终确定上、下游引物终浓度均为4 mmol·L-1时,扩增效率最高(图 2)。
|
M. DL2000 DNA相对分子质量标准;1~5.上、下游引物终浓度分别为4、4 mmol·L-1,4、2 mmol·L-1,4、1 mmol·L-1,1、4 mmol·L-1,2、4 mmol·L-1;6.阴性对照 M. DL2000 DNA marker; 1-5. Concentration of primer F and R(4 & 4 mmol·L-1, 4 & 2 mmol·L-1, 4 & 1 mmol·L-1, 1 & 4 mmol·L-1, 2 & 4 mmol·L-1, respectively); 6. Negative control 图 2 最佳引物浓度的确定 Fig. 2 Determination of concentration of the primer |
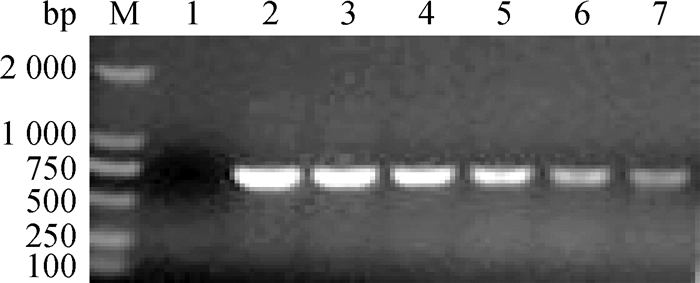
在以上最佳扩增体系条件下,随着退火温度的升高,电泳条带逐渐减弱,在退火温度为54 ℃时,电泳条带最亮,扩增效率最高(图 3)。
|
M. DL2000 DNA相对分子质量标准;1.阴性对照;2~7.退火温度优化(54、56、58、60、62、64 ℃) M. DL2000 DNA marker; 1. Negative control; 2-7. Annealing temperature were 54, 56, 58, 60, 62, 64 ℃, respectively 图 3 最佳退火温度的确定 Fig. 3 Determination of the optimal annealing temperature of PCR |
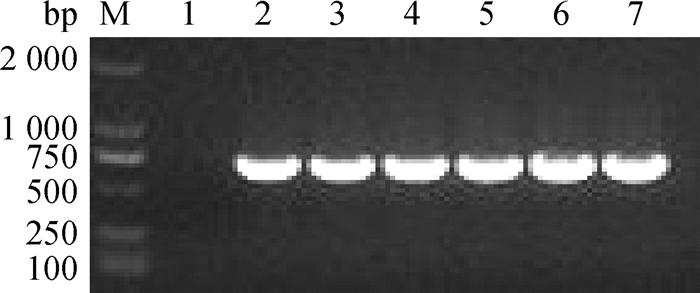
琼脂糖凝胶电泳结果显示(图 4),在不同延长时间条件下,目的条带的亮度无肉眼可辨差异,为缩短扩增时间,确定最佳延长时间为15 s。
|
M. DL2000 DNA相对分子质量标准;1.阴性对照;2~7.延长时间优化(15、20、25、30、35、40 s) M. DL2000 DNA marker; 1. Negative control; 2-7. Extension time were 15, 20, 25, 30, 35, 40 s, respectively 图 4 最佳延长时间的确定 Fig. 4 Determination of the optimal time of extension |
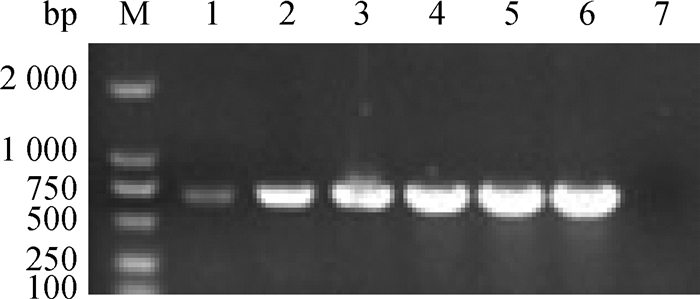
随着PCR循环次数的增加,目的条带亮度逐渐增加(图 5),同时游离碱基被消耗,循环次数35和40的条带几乎无差异,为提高检测效率,并减少非特异扩增,确定最佳循环次数为35。
|
M. DL2000 DNA相对分子质量标准;1~6.循环次数优化(15、20、25、30、35、40);7.阴性对照 M. DL2000 DNA marker; 1-6. 15, 20, 25, 30, 35, 40 cycles, respectively; 7. Negative control 图 5 最佳循环次数的确定 Fig. 5 Determination of the optimal cycles |
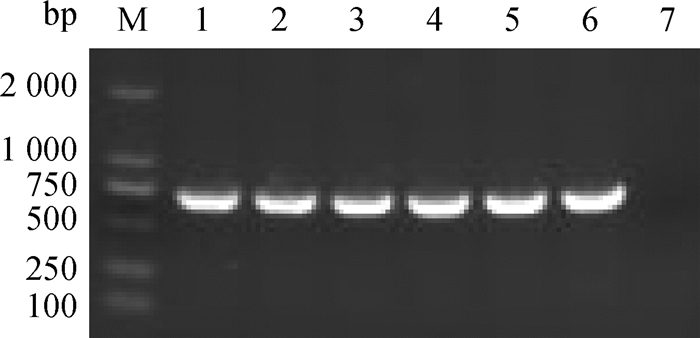
以SVA cDNA为模板,进行6次重复检验,结果显示(图 6),6次扩增产物的电泳条带大小、亮度均一致,表明该方法重复性良好。
|
M. DL2000 DNA相对分子质量标准;1~6. 6次重复PCR检测;7.阴性对照 M. DL2000 DNA marker; 1-6. PCR repeatability; 7. Negative control 图 6 重复性试验 Fig. 6 Repeatability test |
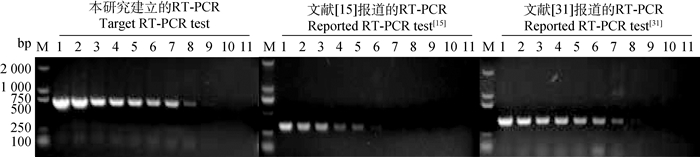
10-1~10-10梯度稀释病毒后,提取核酸进行RT-PCR检测(图 7),该方法的最低检出限为100TCID50,即1 TCID50,而用已报道的PCR方法[15, 31]检测到的最低病毒量分别为103TCID50和101TCID50,本研究建立的方法检测敏感性明显高于其他两种方法,证明该方法敏感性更高。
|
M. DL2000 DNA相对分子质量标准;1~10. 108TCID50、107TCID50、106TCID50、105TCID50、104TCID50、103TCID50、102TCID50、101TCID50、100TCID50、10-1TCID50;11.阴性对照 M. DL2000 DNA marker; 1-10. 108TCID50, 107TCID50, 106TCID50, 105TCID50, 104TCID50, 103TCID50, 102TCID50, 101TCID50, 100TCID50, 10-1TCID50; 11. Negative control 图 7 敏感性试验 Fig. 7 Sensitivity test |
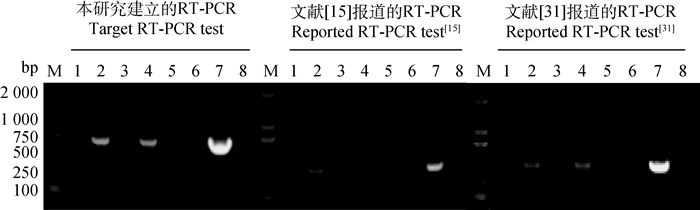
用该方法和文献报道的PCR检测方法[15, 31]检测40份临床样品,部分检测结果见图 8,共检测到阳性2份,阴性38份,三种方法的符合率为100%,表明该方法可用于SVA的检测。
|
M. DL2000 DNA相对分子质量标准;1~6.部分猪组织样品检测检测结果;7.阳性对照;8.阴性对照 M. DL2000 DNA marker; 1-6. The detection results of some samples of pig tissue; 7. Positive control; 8. Negative control 图 8 符合率试验 Fig. 8 Conformity test |
对来自山东的100份组织病料进行检测,结果阳性为2份,阴性为98份,阳性率为2%,表明SVA在该地区尚未流行,但随着病毒基因的进化和毒力的增强,应做好相关疫病的防疫工作。
3 讨论SVA最初作为一种PER.C6细胞培养污染物被发现[1],后被证实是导致巴西、美国、中国广州等多地特发性水疱病的病原,成年猪多呈隐性感染,发病母猪表现水疱症状,新生仔猪发病严重可致死亡,给养猪业造成一定的经济损失。该病毒与口蹄疫病毒、水疱性口炎病毒、猪水疱病病毒、猪传染性水疱病病毒共同组成五大猪水疱病病原,且水疱症状极为相似,只能依赖于实验室诊断。目前各实验室已建立多种SVA检测方法,其中病毒分离鉴定和病毒中和试验需要掌握细胞培养技术,费时、费力;荧光定量RT-PCR方法可对病原进行定量检测,但对试验条件要求比较高。酶联免疫吸附试验主要有竞争ELISA和间接ELISA,前者需要制备单克隆抗体,后者则对抗原的纯度要求较高,且容易产生交叉反应,导致假阳性。RT-PCR特异性好,灵敏度高,且操作简便、快捷,便于养殖场检测人员的掌握。而理想的RT-PCR的建立,关键在于特异性引物的选择,同时要求能尽可能多地检测出不同SVA细胞株,且不与其他病毒基因发生非特异扩增,因此本研究对美国、巴西和中国的流行毒株进行基因比对,从中选择相对保守区域,设计了3对引物P1、P2和P3,P1引物相较于P2和P3引物,以SVA cDNA为模板的PCR电泳目的条带更亮,且与EMCV、FMDV、PRV、PRRSV、CSFV、PEDV等病毒的DNA和cDNA均不反应,是理想的扩增引物。通过对一系列的条件优化,确定最佳引物浓度为4 mmol·L-1,最佳扩增体系:95 ℃预变性5 min,95 ℃变性30 s、54 ℃退火30 s,72 ℃延长15 s,35个循环,72 ℃终延伸5 min。该方法特异性好,不与其他病毒基因交叉反应,SVA的最低检测限可达1 TCID50,比文献[15, 31]的病毒检出限更高,三种RT-PCR的检测符合率为100%。研究表明[8]感染SVA后,病情大多呈一过性,病毒血症随病情发展逐渐消失,因此用该方法可以检出感染前期猪血液中的病毒核酸,而后期可能在血液中无法检出。另外,SVA感染猪的水疱液、心和扁桃体中存在大量的病毒,可作为SVA的最佳检测样品。综上,本研究建立的SVA RT-PCR方法可作为一种可靠的病原检测方法,用于SVA的诊断和分子流行病学的调查和分析。
用该方法对山东地区的100份组织进行检测,阳性率为2%,虽然阳性率低,但是自2015年我国首次报道该病毒感染病例,SVA疫病已迅速在广东、湖北、河南、山东等多省份扩散,未来存在疫病暴发的可能。目前市场上尚无针对SVA的有效疫苗,缺乏相应的应急措施,一旦发生疫病,可参考口蹄疫防控应急预案处理,减少经济损失。
4 结论本研究建立的SVA RT-PCR检测方法特异性好,灵敏度高,适用于SVA的诊断和分子流行病学调查,经初步检测,山东地区SVA感染阳性率处于较低水平,但是随着病毒的基因变异和致病性的增强,疫情防控不容乐观,需政府相关部门和养殖场提高警惕,防患于未然。
| [1] | HALES L M, KNOWLES N J, REDDY P S, et al. Complete genome sequence analysis of Seneca Valley virus-001, a novel oncolytic picornavirus[J]. J Gen Virol, 2008, 89: 1265–1275. DOI: 10.1099/vir.0.83570-0 |
| [2] | ADAMS M J, LEFKOWITZ E J, KING A M Q, et al. Ratification vote on taxonomic proposals to the International Committee on Taxonomy of Viruses (2015)[J]. Arch Virol, 2015, 160(7): 1837–1850. DOI: 10.1007/s00705-015-2425-z |
| [3] | REDDY P S, BURROUGHS K D, HALES L M, et al. Seneca valley virus, a Systemically deliverable oncolytic picornavirus, and the treatment of neuroendocrine cancers[J]. J Natl Cancer Inst, 2007, 99(21): 1623–1633. DOI: 10.1093/jnci/djm198 |
| [4] | KOPPERS-LALIC D, HOEBEN R C. Non-human viruses developed as therapeutic agent for use in humans[J]. Rev Med Virol, 2011, 21(4): 227–239. DOI: 10.1002/rmv.v21.4 |
| [5] | RUDIN C M, SENZER N, STEPHENSON J, et al. Phase I study of intravenous Seneca valley virus (NTX-010), a replication competent oncolytic virus, in patients with neuroendocrine (NE) cancers[J]. J Clin Oncol, 2009, 27(15S): 4629. |
| [6] | BURKE M J. Oncolytic Seneca Valley virus:past perspectives and future directions[J]. Oncolytic Virother, 2016, 5: 81–89. DOI: 10.2147/OV |
| [7] | PASMA T, DAVIDSON S, SHAW S L. Idiopathic vesicular disease in swine in Manitoba[J]. J Can Vet, 2008, 49(1): 84–85. |
| [8] | SINGH K, CORNER S, CLARK S, et al. Seneca Valley virus and vesicular lesions in a pig with idiopathic vesicular disease[J]. J Vet Sci Technol, 2012, 3(6): 123. |
| [9] | GUO B Q, PIÑEYRO P E, RADEMACHER C J, et al. Novel Senecavirus A in swine with vesicular disease, United States, July 2015[J]. Emerg Infect Dis, 2016, 22(7): 1325–1327. DOI: 10.3201/eid2207.151758 |
| [10] | LEME R A, ZOTTI E, ALCÂNTARA B K, et al. Senecavirus A: an emerging vesicular infection in Brazilian pig herds[J]. Transbound Emerg Dis, 2015, 62(6): 603–611. DOI: 10.1111/tbed.2015.62.issue-6 |
| [11] | LEME R A, OLIVEIRA T E S, ALCÂNTARA B K, et al. Clinical manifestations of Senecavirus A infection in neonatal pigs, Brazil, 2015[J]. Emerg Infect Dis, 2016, 22(7): 1238–1241. DOI: 10.3201/eid2207.151583 |
| [12] | LEME R A, ALFIERI A F, ALFIERI A A. Update on Senecavirus infection in pigs[J]. Viruses, 2017, 9(7): 170. DOI: 10.3390/v9070170 |
| [13] | VANNUCCI F A, LINHARES D C L, BARCELLOS D E S N, et al. Identification and complete genome of Seneca valley virus in vesicular fluid and sera of pigs affected with idiopathic vesicular disease, Brazil[J]. Transbound Emerg Dis, 2015, 62(6): 589–593. DOI: 10.1111/tbed.2015.62.issue-6 |
| [14] | WU Q, ZHAO X, BAI Y, et al. The first identification and complete genome of Senecavirus A affecting pig with idiopathic vesicular disease in China[J]. Transbound Emerg Dis, 2017, 64(5): 1633–1640. DOI: 10.1111/tbed.2017.64.issue-5 |
| [15] | QIAN S H, FAN W C, QIAN P, et al. Isolation and full-genome sequencing of Seneca Valley virus in piglets from China, 2016[J]. Virol J, 2016, 13: 173. DOI: 10.1186/s12985-016-0631-2 |
| [16] | SUN D, VANNUCCI F, KNUTSON T P, et al. Emergence and whole-genome sequence of Senecavirus A in Colombia[J]. Transbound Emerg Dis, 2017, 64(5): 1346–1349. DOI: 10.1111/tbed.2017.64.issue-5 |
| [17] | MONTIEL N, BUCKLEY A, GUO B Q, et al. Vesicular disease in 9-week-old pigs experimentally infected with Senecavirus A[J]. Emerg Infect Dis, 2016, 22(7): 1246–1248. DOI: 10.3201/eid2207.151863 |
| [18] | ZHU Z, YANG F, CHEN P, et al. Emergence of novel Seneca Valley virus strains in China, 2017[J]. Transbound Emerg Dis, 2017, 64(4): 1024–1029. DOI: 10.1111/tbed.2017.64.issue-4 |
| [19] | ZHANG X L, ZHU Z X, YANG F, et al. Review of Seneca Valley virus:a call for increased surveillance and research[J]. Front Microbiol, 2018, 9: 940. DOI: 10.3389/fmicb.2018.00940 |
| [20] | CANNING P, CANON A, BATES J L, et al. Vesicular lesions and lameness associated with Senecavirus A in a U. S. sow farm[J]. Transbound Emerg Dis, 2016, 63(4): 373–378. DOI: 10.1111/tbed.2016.63.issue-4 |
| [21] | GIMENEZ-LIROLA L G, RADEMACHER C, LINHARES D, et al. Serological and molecular detection of Senecavirus A associated with an outbreak of swine idiopathic vesicular disease and neonatal mortality[J]. J Clin Microbiol, 2016, 54(8): 2082–2089. DOI: 10.1128/JCM.00710-16 |
| [22] | MILES L A, BURGA L N, GARDNER E E, et al. Anthrax toxin receptor 1 is the cellular receptor for Seneca Valley virus[J]. J Clin Invest, 2017, 127(8): 2957–2967. DOI: 10.1172/JCI93472 |
| [23] | YANG M, VAN BRUGGEN R, XU W H. Generation and diagnostic application of monoclonal antibodies against Seneca Valley virus[J]. J Vet Diagn Invest, 2012, 24(1): 42–50. DOI: 10.1177/1040638711426323 |
| [24] | RESENDE T P, MARTHALER D G, VANNUCCI F A. A novel RNA-based in situ hybridization to detect Seneca Valley virus in neonatal piglets and sows affected with vesicular disease[J]. PLoS One, 2017, 12(4): e0173190. DOI: 10.1371/journal.pone.0173190 |
| [25] | DVORAK C M T, AKKUTAY-YOLDAR Z, STONE S R, et al. An indirect enzyme-linked immunosorbent assay for the identification of antibodies to Senecavirus A in swine[J]. BMC Vet Res, 2016, 13(1): 50. DOI: 10.1186/s12917-017-0967-x |
| [26] | FOWLER V L, RANSBURGH R H, POULSEN E G, et al. Development of a novel real-time RT-PCR assay to detect Seneca Valley virus-1 associated with emerging cases of vesicular disease in pigs[J]. J Virol Methods, 2017, 239: 34–37. DOI: 10.1016/j.jviromet.2016.10.012 |
| [27] | BRACHT A J, O'HEARN E S, FABIAN A W, et al. Real-time reverse transcription PCR assay for detection of Senecavirus A in swine vesicular diagnostic specimens[J]. PLoS One, 2016, 11(1): e0146211. DOI: 10.1371/journal.pone.0146211 |
| [28] |
樊晓旭, 赵永刚, 迟田英, 等. 塞尼卡谷病毒TaqMan荧光定量PCR检测方法的建立[J]. 中国预防兽医学报, 2016, 38(12): 959–962.
FAN X X, ZHAO Y G, CHI T Y, et al. Establishment of a TaqMan real-time PCR assay for detection of Seneca Valley virus[J]. Chinese Journal of Preventive Veterinary Medicine, 2016, 38(12): 959–962. (in Chinese) |
| [29] |
樊晓旭, 哈登楚日亚, 王英丽, 等. 塞尼卡谷病毒重组酶聚合酶扩增技术(RPA)实时荧光检测方法的建立[J]. 中国动物检疫, 2017, 34(8): 81–86.
FAN X X, HADENGCHURIYA, WANG Y L, et al. Establishment of a real-time fluorescent recombinase polymerase amplification (RPA) for the detection of Seneca Valley virus[J]. China Animal Health Inspection, 2017, 34(8): 81–86. (in Chinese) |
| [30] |
樊晓旭, 宋翥远, 赵永刚, 等. 塞尼卡谷病毒重组酶聚合酶扩增-侧流层析试纸条检测方法的建立[J]. 中国预防兽医学报, 2018, 40(5): 406–410.
FAN X X, SONG Z Y, ZHAO Y G, et al. Establishment of recombinase polymerase amplification-lateral flow dipstick for the detection of Seneca valley virus[J]. Chinese Journal of Preventive Veterinary Medicine, 2018, 40(5): 406–410. (in Chinese) |
| [31] | KNOWLES N J, HALES L M, JONES B H, et al. Epidemiology of Seneca Valley virus: identification and characterization of isolates from pigs in the United States[C]//Abstracts of the Northern Lights EUROPIC 2006- XIV Meeting of the European Study Group on the Molecular Biology of Picornaviruses. Saariselka: EUROPIC, 2006. |



