2. 农业部兽用药物与诊断技术北京科学观测实验站, 北京 100193;
3. 北京市畜牧总站, 北京 100107;
4. 中国农业科学院, 北京 100081
2. Scientific Observing and Experiment Station of Veterinary Drugs and Diagnostic Technology of Ministry of Agriculture, Beijing 100193, China;
3. Beijing Animal Husband Service, Beijing 100107, China;
4. Chinese Academy of Agricultural Sciences, Beijing 100081, China
犬细小病毒病是危害我国养犬业的重要疫病之一,其病原是犬细小病毒(canine parvovirus, CPV),该病具有高度的传染性,极易传播,主要发生在幼犬,成年犬也会感染,病犬主要的临床症状表现为带有鱼腥味的出血性粪便和非化脓性心肌炎,死亡率高达80%[1-2]。20世纪70年代,CPV作为一种新的病原首先在澳大利亚和加拿大被发现,接着几个月内就在全球范围内流行起来[3]。在其出现不久后,CPV-2不断突变,连续产生两种新抗原突变体,CPV-2a和CPV-2b,并逐渐取代原来的CPV-2型。2001年,在意大利检测到新的抗原突变体CPV-2c[4-5],并迅速传播到其他国家。近几年,中国在不同省份也有CPV-2c出现的报道[6]。CPV-2c与原始CPV-2相比,抗原性突变体在犬中表现出高的致病性,并且增加宿主范围,能够感染猫并使其致病[7]。流行病学调查表明,最新型的CPV-2c在不同的地理区域中变得普遍,成年犬接种疫苗后也能被感染。然而,通过传统方法检测CPV感染并不敏感,因此,建立新型、灵敏度高、特异性好的犬细小病毒检测方法迫在眉睫。
纳米PCR技术是在PCR反应的buffer中添加一定量的粒径为1~100 nm的纳米金属颗粒,从而形成纳米流体,纳米流体具有极高的导热性,比普通流体要高得多,因此在添加了纳米金属颗粒的PCR热循环中,PCR反应能够更快地达到目标温度,从而缩短了在非目标温度停留的时间,不仅减少了非特异扩增,也提高了反应的特异性[8-11]。
近年来,随着CPV感染数量的逐渐增加,CPV的早期检测越来越受到研究人员的关注和重视。本研究以CPV VP2基因为目标基因,设计特异性引物,拟建立CPV纳米PCR检测方法,为CPV的分子流行病学调查和早期诊断提供试验方法。
1 材料与方法 1.1 病毒犬瘟热病毒(canine distemper virus,CDV)、犬细小病毒(CPV-2a、CPV-2b、CPV-2c型)、狂犬病病毒(rabies virus,RV)和犬冠状病毒(canine coronavirus,CCoV)由中国农业科学院北京畜牧兽医研究所宠物疫病防控科技创新团队保存。
1.2 临床样品2016-2018年在广西省、北京市和吉林省采集的75份疑似感染细小病毒的犬粪便样品,以0.01 mol·L-1 pH7.6 PBS做1:100稀释,反复冻融后涡旋混匀,8 000 r·min-1离心30 min,取上清,-80 ℃保存。应用病毒DNA提取试剂盒提取临床样品的DNA;应用病毒RNA提取试剂盒提取CDV、RV、CCoV的基因组RNA,并以此为模板按照M-MLV反转录试剂盒进行cDNA的合成,-20 ℃保存,待检。
1.3 主要试剂PrimeSTAR® HS DNA高保真酶,DL2000、Pluss Marker,La-Taq,DNA A-Tailing Kit,pET-28a载体,HB101感受态购自TaKaRa公司;质粒提取试剂盒购自OMEGA公司;DNA提取试剂盒购自TIANGEN公司;胶回收试剂盒购自AXYGEN公司;纳米PCR试剂盒购自山东正大医疗器械股份有限公司。
1.4 引物设计参照GenBank中发表的CPV-2(登录号:AB054222)基因组序列的分析,应用Primer 5.0软件设计扩增CPV VP2基因的全长序列引物,通过与已公布的VP2基因全长序列进行比对后筛选保守区域设计扩增574 bp片段的引物(P3/P4)。引物(表 1)由华大基因公司合成。
|
|
表 1 引物信息 Table 1 Primers information |
以CPV-BJ株细胞培养物提取的DNA为模板,以P1/P2为引物,利用PrimeSTAR® HS DNA高保真酶扩增VP2基因全长序列,并将其克隆至pET-28a载体中构建重组质粒pET-28a-VP2,按照小提质粒试剂盒说明书提取质粒,并送至北京华大基因进行测序,测序正确的阳性质粒命名为pET-28a-VP2(+),作为纳米PCR的质粒标准品,于-20 ℃保存备用。
1.6 传统PCR方法的建立传统PCR反应体系预设定:2× LA Taq Buffer Ⅱ (Mg2+ Plus) 10 μL,P3/P4(10 μmol·L-1) 0.6 μL,pET-28a-VP2(+)1 μL,Taq酶(5 U·μL-1)0.5 μL,ddH2O补至20 μL;反应条件预设定:95 ℃ 4 min;95 ℃ 30 s、55 ℃ 30 s、72 ℃ 35 s,30个循环;72 ℃ 10 min。扩增产物经1.0%琼脂糖凝胶电泳检测。
1.7 纳米PCR方法的建立 1.7.1 纳米PCR反应体系及反应条件的优化反应体系:2× nanoPCR buffer 10 μL,P3/P4(10 μmol·L-1) 0.6 μL,pET-28a-VP2(+)1 μL,Taq酶(5 U·μL-1)0.5 μL,ddH2O补至20 μL。反应条件预设定:95 ℃ 4 min;95 ℃ 30 s、退火30 s、72 ℃ 35 s,30个循环;72 ℃ 10 min。优化退火温度和引物浓度。将温度进行梯度设置为45~55 ℃,间隔1 ℃,进行退火温度的优化,反应体系和其他反应条件均相同。引物的使用体积从0.1到1.0 μL进行优化,间隔0.1 μL,按照优化后的退火温度进行。扩增产物经1.0%琼脂糖凝胶电泳检测。
1.7.2 敏感性试验测定阳性模板pET-28a-VP2(+)的质粒浓度,并换算成拷贝数,按照10倍倍比稀释后,分别利用纳米PCR和常规PCR进行扩增,产物经1.0%琼脂糖凝胶电泳检测,比较两者的敏感性;试验重复3次。
1.7.3 特异性试验应用纳米PCR分别对CDV、RV、CCoV的cDNA进行检测,以此验证纳米PCR的特异性;试验重复3次。并对CPV不同抗原突变体CPV-2a、CPV-2b、CPV-2c进行纳米PCR检测。扩增产物经1.0%琼脂糖凝胶电泳检测。
1.8 临床样品的检测对临床样品的检测比较,将75份临床样品提取DNA作为模板,分别应用纳米PCR和常规PCR对提取的DNA进行检测。
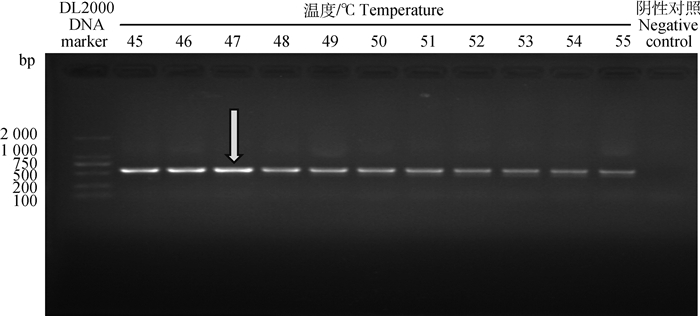
2 结果 2.1 纳米PCR反应条件的优化 2.1.1 CPV纳米PCR检测方法最佳退火温度的确定CPV纳米PCR经退火温度(45~55 ℃)优化,琼脂糖凝胶电泳结果见图 1。由图 1可知,45~55 ℃下,纳米PCR均可扩增得到目的条带,最佳退火温度为47 ℃。最终的反应条件: 95 ℃ 4 min;95 ℃ 30 s、47 ℃ 30 s、72 ℃ 35 s,30个循环;72 ℃延伸10 min。
|
图 1 退火温度的优化 Fig. 1 Optimization of the annealing temperature of the CPV nano PCR |
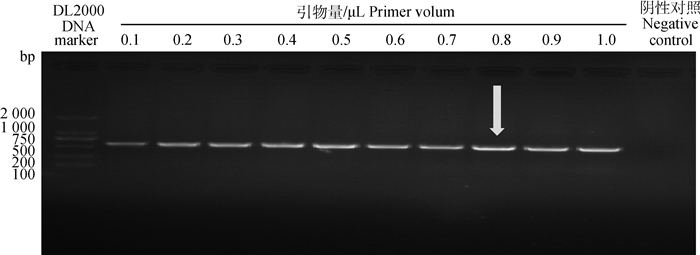
在最佳反应条件下,纳米PCR反应体系为20 μL:2×nano PCR buffer 10 μL,P3/P4 (10 μmol·L-1) 0.1~1.0 μL,pET-28a-VP2(+)1 μL,Taq酶(5 U·μL-1)0.5 μL,ddH2O补至20 μL;纳米PCR反应条件:95 ℃ 4 min;95 ℃ 30 s、47 ℃ 30 s、72 ℃ 35 s,30个循环;72 ℃延伸10 min。引物量分别设置为0.1、0.2、0.3、0.4、0.5、0.6、0.7、0.8、0.9和1.0 μL,进行引物浓度优化。琼脂糖凝胶电泳结果见图 2。结果表明当引物量为0.8 μL时扩增效果最佳。
|
图 2 引物浓度的优化 Fig. 2 Optimization of the primer concentration of the CPV nano PCR |
经反应退火温度及引物浓度的摸索,最终确定CPV纳米PCR反应体系:2×nano PCR buffer 10 μL,P3/P4(10 μmol·L-1)0.8 μL,pET-28a-VP2(+)1 μL,Taq 酶(5 U·μL-1)0.5 μL,ddH2O补至20 μL。最终的反应条件: 95 ℃ 4 min、95 ℃ 30 s、47 ℃ 30 s、72 ℃ 35 s,30个循环;72 ℃终延伸10 min。
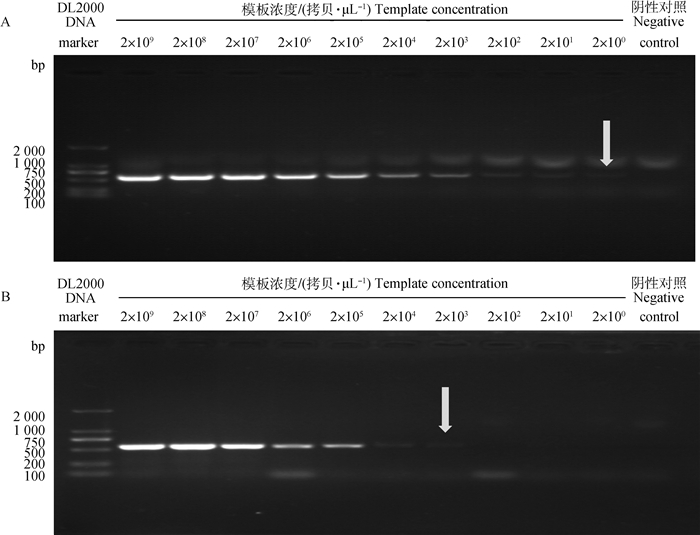
2.2 CPV纳米PCR敏感性试验构建的重组质粒pET-28a-VP2(+)的浓度经测定为174 ng·μL-1,计算出质粒的拷贝数为2×1010拷贝·μL-1。将重组质粒进行10倍倍比稀释,得到浓度标准品为2×109~2×100拷贝·μL-1,取每个稀释度的重组质粒模板分别进行纳米PCR和常规PCR检测。琼脂糖凝胶电泳结果见图 3。由图 3A可知,纳米PCR能检测到2拷贝·μL-1,而常规PCR的最低检测量为2×103拷贝·μL-1(图 3B),敏感性试验结果表明纳米PCR检测方法的灵敏度比普通PCR方法高1 000倍。
|
图 3 CPV纳米PCR(A)和常规PCR(B)敏感性试验 Fig. 3 Sensitivity of CPV nano PCR assay (A) and conventional PCR assay (B) |
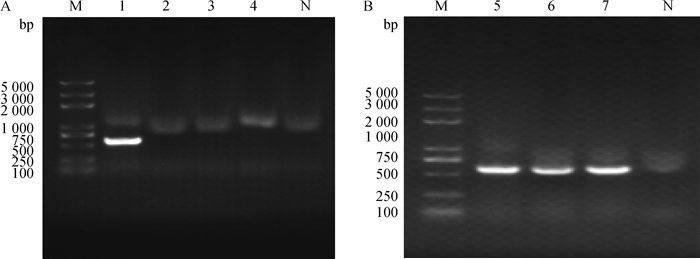
采用P3/P4引物分别对犬瘟热病毒(CDV)、狂犬病毒(RV)、犬冠状病毒(CCoV)进行纳米PCR检测,结果只有CPV能扩增出574 bp的目的片段,其他病毒均未扩增出条带(图 4A),同时对CPV-2a、CPV-2b、CPV-2c检测都能扩增出目的片段(图 4B),试验结果表明CPV纳米PCR检测具有良好的特异性。
|
M. 2K Plus相对分子质量标准;N.阴性对照;1.犬细小病毒;2.犬瘟热病毒;3.狂犬病病毒;4.犬冠状病毒;5. CPV-2a型;6. CPV-2b型;7. CPV-2c型 M. 2K Plus marker; N. Negative control; 1. CPV; 2. CDV; 3. RV; 4. CCoV; 5. CPV-2a; 6. CPV-2b; 7. CPV-2c 图 4 纳米PCR特异性试验结果 Fig. 4 Specificity result of the nano PCR |
提取采集的75份粪便样本的DNA,应用所建立的纳米PCR对CPV进行检测。检测结果显示,纳米PCR和常规PCR检测阳性率分别为89.3%(67/75)和70.7%(53/75)(表 2)。为进一步确定所建立方法检测的准确性,将纳米PCR检出但普通PCR未检出的14份样本的产物送华大基因测序分析。测序结果显示,纳米PCR获得的574 bp特异性片段与CPV相似性均达99%以上。对临床样品的检测结果表明,所建立的纳米PCR比普通PCR更加敏感,适用于低含量CPV临床样品的检测。
|
|
表 2 临床样品的检测结果 Table 2 Test results of clinical samples |
CPV主要感染犬,尤其是幼犬,传染性极强,致死率也很高。一年四季均可发病,以冬、春季节多发。目前,检测CPV感染的技术主要有免疫层析(IC)试验、血凝试验(HA)、病毒分离(Ⅵ)试验、常规PCR[12]、巢式PCR[13]、荧光定量PCR[14]等。与分子生物学技术比较,IC试验的相对灵敏度不超过50%,而特异性为100%;HA试验结果的准确性受红细胞质量、红细胞沉降系数的影响,具有不稳定性;病毒分离(Ⅵ)仅可在实验室细胞上进行,需要一定专业技术和细胞培养能力的实验人员,需要长达数天的时间,并需要进行免疫荧光试验来检测病毒抗原;PCR是比较常见的技术,但其缺陷在于耗时、敏感性不显著且不能定量检测;荧光定量PCR的敏感性和准确性高于普通PCR,但其实施需要昂贵的实时荧光定量PCR仪,在二三线城市的动物诊所或实验室难以普及推广。以上这些缺点限制了上述方法在临床诊断中的应用。
本研究建立了检测犬细小病毒的高效纳米PCR技术,由于纳米流体具有极高导热性,使PCR体系达到温度所需平衡时间短,实现目的条带的快速扩增,提高特异性产物的产量;敏感性试验结果显示纳米PCR检测的敏感性可达到常规PCR技术的1 000倍。目前,纳米PCR已成功应用于水貂肠炎病毒、猪博卡病毒、猪细小病毒等多种动物病原的临床检测中,且其敏感性远远高于普通PCR[15-17]。2018年,Gabriel等[18]将纳米PCR技术成功应用于脑食性变形虫的快速检测中,首次发现了石墨烯氧化物、氧化铜和氧化铝的纳米混合颗粒可以优化棘阿米巴、巴拉木翘和奈格莱里亚的PCR检测效果,表明纳米PCR法在寄生虫感染的临床诊断、流行病学和病理学研究以及其他微生物的环境监测等方面也具有潜在的应用价值。
为证明获得的574 bp是CPV来源的特异性片段,笔者将纳米PCR能检出但普通PCR未检出的14份样本的PCR产物进行了克隆测序,并进行了序列同源性分析,结果显示14个样本的测序结果与CPV VP2基因的相似性均在99%以上,证明纳米PCR获得的574 bp是CPV来源的特异性片段,排除了纳米PCR假阳性的问题。
临床中,在CPV早期感染时病毒量在病犬体内很少,用普通的检测方法很难检测到,但应用本研究建立的纳米PCR技术能够更加高效、快速地检测,且适合基层实验室使用。因此,本方法的建立为CPV的早期检测、流行病学调查、预防和治疗奠定了基础。
4 结论成功建立犬细小病毒的纳米PCR检测方法,该方法适用于CPV低含量临床样品的检测,特异性和灵敏性良好,最低核酸检出量为2拷贝·μL-1,其敏感性比常规PCR高1 000倍,可应用于犬细小病毒感染的早期诊断和流行病学调查,为犬细小病毒感染的鉴定和防控奠定基础,具有重大的临床应用意义和价值。
| [1] | PINTO L D, STRECK A F, GONCALVES K R, et al. Typing of canine parvovirus strains circulating in Brazil between 2008 and 2010[J]. Virus Res, 2012, 165(1): 29–33. DOI: 10.1016/j.virusres.2012.01.001 |
| [2] | SHACKELTON L A, PARRISH C R, TRUYEN U, et al. High rate of viral evolution associated with the emergence of carnivore parvovirus[J]. Proc Natl Acad Sci U S A, 2005, 102(2): 379–384. DOI: 10.1073/pnas.0406765102 |
| [3] | PÉREZ R, BIANCHI P, CALLEROS L, et al. Recent spreading of a divergent canine parvovirus type 2a (CPV-2a) strain in a CPV-2c homogenous population[J]. Vet Microbiol, 2012, 155(2-4): 214–219. DOI: 10.1016/j.vetmic.2011.09.017 |
| [4] | BUONAVOGLIA C, MARTELLA V, PRATELLI A, et al. Evidence for evolution of canine parvovirus type 2 in Italy[J]. J Gen Virol, 2001, 82(12): 3021–3025. DOI: 10.1099/0022-1317-82-12-3021 |
| [5] | DECARO N, BUONAVOGLIA C. Canine parvovirus—A review of epidemiological and diagnostic aspects, with emphasis on type 2c[J]. Vet Microbiol, 2012, 155(1): 1–12. |
| [6] |
张仁舟, 杨松涛, 冯昊, 等. 中国国内首次检测到犬细小病毒CPV-2c[J]. 中国病原生物学杂志, 2010, 5(4): 246–249, 275.
ZHANG R Z, YANG S T, FENG H, et al. The first detection of canine parvovirus type 2c in China[J]. Journal of Pathogen Biology, 2010, 5(4): 246–249, 275. (in Chinese) |
| [7] | PALERMO L M, HAFENSTEIN S L, PARRISH C R. Purified feline and canine transferrin receptors reveal complex interactions with the capsids of canine and feline parvoviruses that correspond to their host ranges[J]. J Virol, 2006, 80(17): 8482–8492. DOI: 10.1128/JVI.00683-06 |
| [8] | LI H K, HUANG J H, LV J H, et al. Nanoparticle PCR:nanogold-assisted PCR with enhanced specificity[J]. Angew Chem Int Ed Engl, 2005, 44(32): 5100–5103. DOI: 10.1002/(ISSN)1521-3773 |
| [9] | SHEN C C, YANG W J, JI Q L, et al. NanoPCR observation:different levels of DNA replication fidelity in nanoparticle-enhanced polymerase chain reactions[J]. Nanotechnology, 2009, 20(45): 455103. DOI: 10.1088/0957-4484/20/45/455103 |
| [10] | LI H X, ROTHBERG L. Colorimetric detection of DNA sequences based on electrostatic interactions with unmodified gold nanoparticles[J]. Proc Natl Acad Sci U S A, 2004, 101(39): 14036–14039. DOI: 10.1073/pnas.0406115101 |
| [11] | MA X J, CUI Y C, QIU Z, et al. A nanoparticle-assisted PCR assay to improve the sensitivity for rapid detection and differentiation of wild-type pseudorabies virus and gene-deleted vaccine strains[J]. J Virol Methods, 2013, 193(2): 374–378. DOI: 10.1016/j.jviromet.2013.07.018 |
| [12] | DECARO N, DESARIO C, BEALL M J, et al. Detection of canine parvovirus type 2c by a commercially available in-house rapid test[J]. Vet J, 2010, 184(3): 373–375. DOI: 10.1016/j.tvjl.2009.04.006 |
| [13] | HIRASAWA T, KANESHIGE T, MIKAZUKI K. Sensitive detection of canine parvovirus DNA by the nested polymerase chain reaction[J]. Vet Microbiol, 1994, 41(1-2): 135–145. DOI: 10.1016/0378-1135(94)90143-0 |
| [14] | DECARO N, ELIA G, MARTELLA V, et al. A real-time PCR assay for rapid detection and quantitation of canine parvovirus type 2 in the feces of dogs[J]. Vet Microbiol, 2005, 105(1): 19–28. |
| [15] | WANG J K, CHENG Y N, ZHANG M, et al. Development of a nanoparticle-assisted PCR (nano PCR) assay for detection of mink enteritis virus (MEV) and genetic characterization of the NS1 gene in four Chinese MEV strains[J]. BMC Vet Res, 2015, 11: 1. DOI: 10.1186/s12917-014-0312-6 |
| [16] | WANG X L, BAI A Q, ZHANG J, et al. A new nano PCR molecular assay for detection of porcine bocavirus[J]. J Virol Methods, 2014, 202: 106–111. DOI: 10.1016/j.jviromet.2014.02.029 |
| [17] | CUI Y, WANG Z, MA X, et al. A sensitive and specific nanoparticle-assisted PCR assay for rapid detection of porcine parvovirus[J]. Lett Appl Microbiol, 2014, 58(2): 163–167. DOI: 10.1111/lam.2014.58.issue-2 |
| [18] | GABRIEL S, RASHEED A K, SIDDIQUI R, et al. Development of nanoparticle-assisted PCR assay in the rapid detection of brain-eating amoebae[J]. Parasitol Res, 2018, 117(6): 1801–1811. DOI: 10.1007/s00436-018-5864-0 |



