2. 山东省农业科学院畜牧兽医研究所, 济南 250100
2. Institute of Animal Science and Veterinary Medicine, Shandong Academy of Agricultural Sciences, Ji'nan 250100, China
猪繁殖与呼吸综合征(porcine reproductive and respiratory syndrome virus, PRRS)是由猪繁殖与呼吸综合征病毒(PRRSV)引起的一种高度接触性传染病,其主要引起母猪流产、死胎、弱胎、木乃伊胎等繁殖障碍及机体免疫力下降[1]。PRRS自20世纪末首次在美国被发现以来,迅速蔓延至各主要养猪国家,并呈现愈演愈烈的趋势[2]。PRRSV为有囊膜的单股正链RNA病毒,其基因组全长约15 kb,包括9个开放阅读框(ORF)[3]。我国于1996年首次分离并报道该病,后在全国各地均分离鉴定到PRRSV。自2006年暴发“高热病”以来,PRRS对养猪业造成了巨大的经济损失,现已成为影响全球养猪业健康发展的重大疫病之一[4-5]。PRRSV基因组中编码RNA复制酶多聚蛋白的ORF1(ORF1a和ORF1b)约占整个基因组全长的80%,该酶可被剪切成13种具有不同生物学功能的非结构蛋白(Nsp1α、Nsp1β、Nsp2~Nsp12),其中以Nsp2的变异性最大[6]。
2006年暴发的HP-PRRSV,以Nsp2第481、533—561位30个不连续氨基酸缺失为特征,且这一特征在高致病性毒株中高度保守[7-8]。在对近几年流行的PRRSV调查研究中发现,其Nsp2出现了新的缺失位点且缺失数量不断变化[9-10]。通过对2017—2018年山东省、江苏省和河南省不同地区PRRSV分离株分析发现,部分HP-PRRSV分离株的Nsp2蛋白增加了新的氨基酸缺失位点,这些新的缺失位点提示,近几年流行的PRRSV可能在致病力方面出现了新的变化。对Nsp2的深入研究可进一步揭示PRRSV流行毒株、经典毒株与高致病性毒株的致病机制与遗传变异特性,为预防和控制该病积累科学数据。相关研究证明[11-12],HP-PRRSV的高复制能力和高致病力与Nsp9基因编码的氨基酸有关,关键氨基酸位点的变异与否直接影响着病毒对宿主的感染能力。通过分析对比Nsp9第561、586和592位关键氨基酸位点,对初步推测PRRSV的复制效率和毒力高低具有参考价值。重组分析一直是了解PRRSV的重要方法,本研究对4株Nsp2缺失了33和73 aa的PRRSV毒株进行重组分析,比较和探索了其变异和重组规律,为PRRS防控提供参考。
1 材料与方法 1.1 样品处理与病毒分离2017年5月—2018年6月从山东、河南和江苏共10个地区的部分规模猪场采集疑似PRRS发病猪的肺、淋巴结、扁桃体等共计70份样品(每场采集6~8份样品)。所采集病料的病猪临床出现不同程度的精神沉郁、食欲减少、发热、呼吸困难;怀孕母猪流产严重,部分猪舍母猪流产率达60%左右,早产、死胎、木乃伊胎现象严重;部分新生仔猪出现呼吸困难、运动失调及偏瘫等症状,且生长速度较其他无症状仔猪慢。剖检病猪可见心、肺部有大小不一的出血点,肾点状出血,肠系膜淋巴结肿大出血,腹股沟淋巴结高度肿大且出血坏死。采集的病料经研磨、离心、过滤等处理后,接种于MARC-145细胞进行病毒分离培养,将出现CPE的细胞传代培养后进行PCR鉴定。
1.2 主要试剂MiniBest Viral Rna/DNA Extraction kit 5.0、MiniBEST Plasmid Purification Kit Ver.4.0、PrimeSTAR Max DNA Polymerase、PrimeScriptTM Ⅱ 1st Strand cDNA Synthesis Kit、DL2000 DNA Marker、E. coli DH5α Competent Cells等购自TaKaRa公司,胶回收试剂盒购自上海生工生物工程技术服务有限公司。
1.3 PRRSV鉴定引物参考GenBank已收录的PRRSV JXA1、VR2332、CH-1a等毒株核苷酸序列,通过DNAstar软件分析对比,采用Primer6.0设计1对可以区分经典毒株与高致病性毒株Nsp2基因的引物R1:5′-CAACACCCAGGCGACTTCA-3′,R2:5′-TGCGTAGCAGGATCACCAAG-3′,经典毒株扩增产物大小930 bp,高致病性毒株扩增产物大小840 bp;扩增Nsp9全基因引物R3: 5′-TTTAAACTGCTAGCCGC-3′,R4:5′-ATTGGACCCTGAGTT-TC-3′。病毒全基因扩增引物序列见表 1,引物由生工生物工程(上海)有限公司合成。
|
|
表 1 扩增PRRSV全基因的引物序列 Table 1 Primers used for the amplification in the full genome of PRRSV |
第2代细胞培养物用于扩增PRRSV全基因组cDNA。20 μL反转录体系中,Random 6 mers 1 μL,RNA 2 μL,dNTP 1 μL,RNase free ddH2O 6 μL置65 ℃保温5 min,于冰上迅速冷却;往上述反应液中加入5×PrimeScript Buffer 4 μL、RNase Inhibitor 0.5 μL、PrimeScript RTase 1 μL、RNase free dH2O 4.5 μL混匀,于30 ℃反应10 min、42 ℃反应50 min、95 ℃孵育5 min,反应完成后冰上放置获得cDNA,保存备用。
1.5 PRRSV Nsp2和Nsp9基因的PCR扩增采用50 μL反应体系,取cDNA 4 μL作为模板,2×Taq Master Mix 24 μL,每个体系分别应用Nsp2基因的9对引物的上、下游各1 μL,补ddH2O至50 μL,Nsp9基因则采用扩增全基因的上、下游引物进行扩增。反应参数为预变性95 ℃ 5 min;95 ℃ 30 s,55 ℃ 30 s,72 ℃ 30 s,共32个循环后再延伸7 min,分别取10 μL PCR产物进行1%琼脂糖凝胶电泳观察,并置4 ℃冰箱保存。
1.6 目的基因的克隆测序胶回收目的片段并连接pMD18-T载体,反应体系:pMD18-T载体1 μL、胶回收DNA 1 μL、缓冲液5 μL、ddH2O 2 μL,16 ℃反应30 min后将其加入100 μL的JM109感受态细胞中冰浴30 min,经42 ℃加热45 s后迅速放入冰中1 min,加入890 μL的SOC培养基并放入恒温摇床37 ℃振荡培养60 min,之后将培养液均匀涂布在含有Amp的琼脂板上培养过夜,挑取白斑接种于LB液体培养基培养,按照质粒小提试剂盒提取质粒并测序(上海生工)。用DNAstar软件对测序结果进行分析,并绘制进化树。
1.7 序列比对分析利用DNA Star-MegAlign软件将分离到的PRRSV全基因组序列与GenBank中已收录的国内外PRRSV毒株和疫苗株进行Blast对比分析,构建基因遗传进化树。
1.8 盲传后病毒Nsp2和Nsp9基因序列测定将盲传第5和10代的分离毒株分别提取病毒RNA,扩增Nsp2和Nsp9基因(见“1.5”)后测序分析。
1.9 基因重组分析根据GenBank中登录的参考序列,利用RDP4软件对分离的4株Nsp2分别缺失33和73 aa的PRRSV进行基因重组分析,筛选出亲本毒株和提供重组片段的病毒株。根据RDP4软件分析结果,利用SimPlot对4株缺失多个氨基酸位点的PRRSV进行全基因序列重组分析。
1.10 部分致病力相关氨基酸位点分析对4株Nsp2分别缺失33和73 aa的PRRSV Nsp9第561、586和592位氨基酸位点进行比较分析,判断其与决定HP-PRRSV复制效率和毒力高低的关键氨基酸是否一致。
1.11 Nsp9蛋白信号肽预测和二级结构分析利用在线软件SignalP 4.1中的神经网络法对分离的4株Nsp2分别缺失33和73 aa的PRRSV Nsp9蛋白进行信号肽预测,并利用在线软件SOPMA对这些毒株Nsp9蛋白的二级结构进行分析。
2 结果 2.1 PRRSV分离株Nsp2基因扩增从70份样品中共分离到11株PRRSV,分离株的名称、分离时间、样品来源等信息见表 2。
|
|
表 2 11株PRRSV分离株采集信息 Table 2 Collecting information about the 11 PRRSV isolates |
11株PRRSV分离株的样品经RT-PCR扩增后,凝胶电泳结果显示在约1 kb处出现大小不一的扩增条带,1、2泳道条带稍小,3、4泳道条带最小,5~11泳道条带大小相似;扩增目的基因的条带提示11株分离株可能存在三种大小不同的Nsp2基因(图 1)。

|
M.DL2000 DNA相对分子质量标准(DNA marker);1. SDLY;2. SDZZ;3. SDRZ01;4. SDDY;5. JSSQ;6. JSHA;7. JSYC;8. SDRZ;9. HNXX;10. HNSQ;11. HNJZ;12.阴性对照(Negative control) 图 1 PRRSV分离株Nsp2部分基因片段RT-PCR扩增 Fig. 1 Amplification of Nsp2 gene of PRRSV isolates by RT-PCR |
扩增11株PRRSV分离株Nsp2基因,将PCR产物连接至pMD-18T载体中,转化后通过PCR和PstⅠ/EcoRⅠ双酶切鉴定,挑选阳性重组质粒进行测序。测序结果分别与GenBank中的PRRSV参考毒株VR2332进行序列比对,结果显示7个PRRSV分离株(表 2, 1~7号)Nsp2基因分别有两处不连续的基因缺失位点,分别为2 608-2 610位和2 762-2 848位,共90个碱基,这与以往分离的HP-PRRSV(如JXA1、HUN4等)缺失的位置相同。2个分离株SDZZ、SDLY发生了3处Nsp2基因位点的缺失,分别位于2 608-2 610位、2 762-2 848位及3 028-3 036位,共99个碱基。2个分离株SDRZ01和SDDY Nsp2基因序列中则存在两处不连续的缺失,但缺失位点分别为2 572-2 703位、2 762-2 848位,共219个碱基(图 2)。
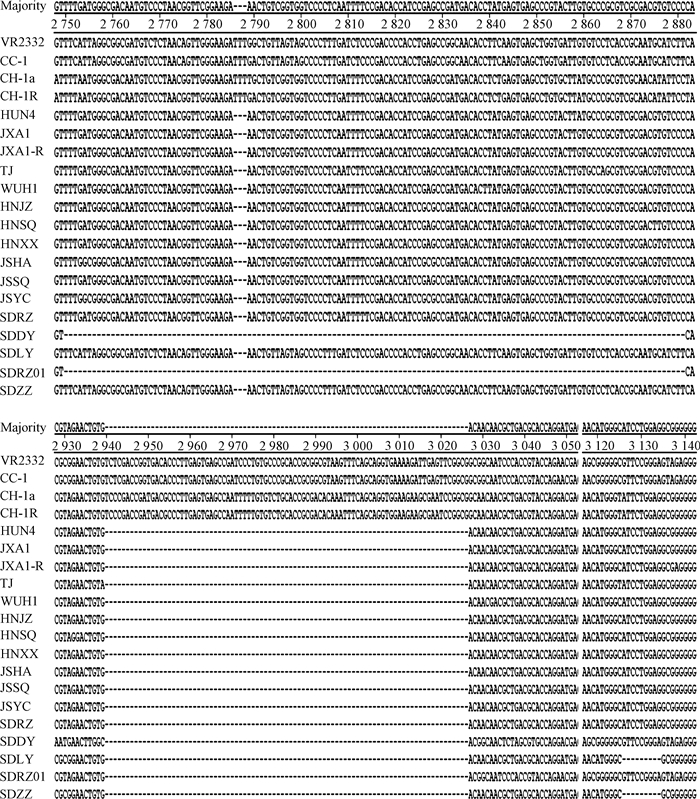
|
图 2 11株PRRSV分离株与参考毒株的部分Nsp2基因序列比对结果 Fig. 2 Sequence alignment of partial Nsp2 gene among 11 PRRSV isolates and reference strains |
将11株PRRSV分离株的Nsp2氨基酸序列与VR2332、HUN4、CC-1等毒株比对,结果显示,7株PRRSV分离株(表 2, 1~7号)的Nsp2蛋白分别在第481及533-561位发生了缺失,共30个氨基酸,与国内流行的高致病性美洲株(如JXA1株等)具有相同的缺失位点;SDZZ和SDLY分离株的Nsp2蛋白分别在第481、533-561及595-597位三个不连续的部位发生了缺失,共33个氨基酸,是一种新的Nsp2氨基酸缺失型毒株;SDRZ01与SDDY分离株的Nsp2蛋白缺失位点则分别位于475-518和533-561位,共73个氨基酸,是一种变异更大的新型Nsp2氨基酸缺失型PRRSV毒株(图 3)。

|
图 3 11株PRRSV分离株与参考毒株的Nsp2氨基酸序列比对结果 Fig. 3 Sequence alignment of Nsp2 aminoacids among 11 PRRSV isolates and reference strains |
利用MegAlign软件将11株PRRSV分离株的全基因组序列与GenBank登录的国内外具有代表性的PRRSV不同毒力参考毒株或疫苗毒株进行系统进化树对比。分析结果显示,本研究分离的11株PRRSV与其他HP-PRRSV(如JXA1、HUN4等)处于同一个大分支中,彼此之间相对有或近或远的亲缘关系,而与经典美洲株(如VR2332等)则亲缘关系较远,与国内广泛使用的疫苗株CH-1R亲缘关系也较远,但与另一疫苗株JXA1-R的亲缘关系相对较近,表明11株分离株的起源较为复杂,呈现一定程度的多样性。4株具有新型氨基酸缺失的分离株(SDDY、SDRZ01、SDZZ和SDLY)与高致病性毒株亲缘关系较远,为高致病性美洲毒株中的一个亚群,但已独立发展为一个较小的分支(图 4),表明具有新缺失位点的4株分离株虽与其他7株分离毒株的分离地区较近(山东及其邻近省份地区),但其进化关系发生了较大变化,具体原因还需进一步深入研究分析;另外7株高致病分离株彼此之间具有不同的亲缘关系,进一步表明其进化的复杂性。同源性分析显示,11株PRRSV分离株之间核苷酸相似性为82.0%~97.2%,氨基酸相似性为80.0%~95.7%;与美洲经典株(如VR2332等)的核苷酸相似性为77.0%~90.7%,氨基酸相似性为75.3%~89.4%;而与国内流行的高致病性美洲株(如JXA1等)的核苷酸相似性为96.8%~99.0%,氨基酸相似性为94.3%~98.1%。
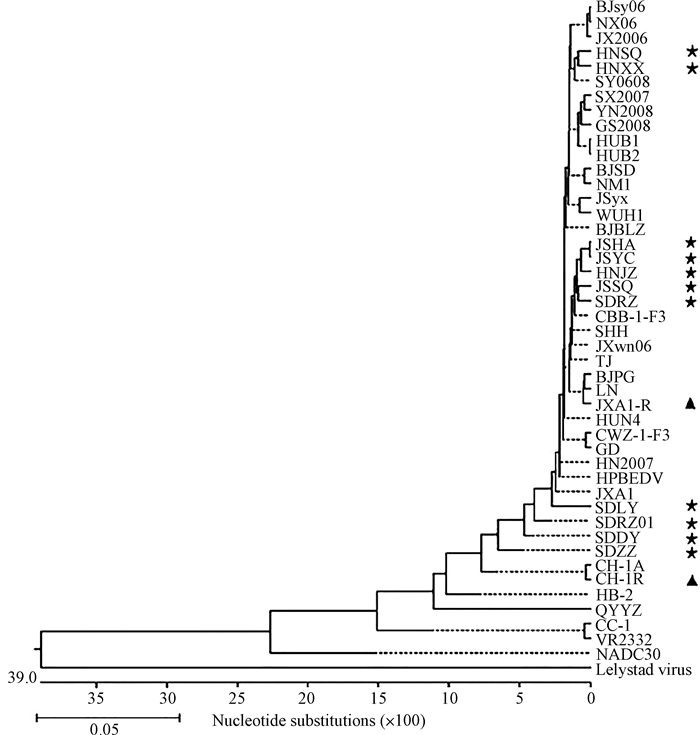
|
★.本研究的分离毒株;▲.疫苗毒株 ★. Isolate strain in this study; ▲. Vaccine strain 图 4 11株PRRSV分离株与参考毒株全基因组序列遗传进化树 Fig. 4 Phylogenetic tree of complete gene nucleotide sequences of 11 PRRSV isolates and reference strains |
相关研究表明,在Nsp2中存在较多数量的脯氨酸,同时包含多个高度免疫原性的B细胞表位,这在病毒的抗原性和感染宿主细胞的过程中发挥重要作用[13]。对PRRSV Nsp2蛋白的B细胞抗原表位进行分析发现,Nsp2蛋白至少存在8个线性化的B淋巴细胞抗原表位,其中包括aa469-aa478(Ⅰ)、aa475-aa490(Ⅱ)、aa496-aa510(Ⅲ)、aa536-aa550(Ⅳ)、aa546-aa560(Ⅴ)、aa576-aa590(Ⅵ)、aa586-aa600(Ⅶ)、aa606-aa620(Ⅷ)。在aa475-aa518与aa533-aa561缺失的SDRZ01与SDDY分离株,其Ⅰ、Ⅱ、Ⅲ和Ⅴ表位的氨基酸发生了较大的抗原性变异;而在aa481、aa533-aa561及aa595-aa597三处缺失的SDZZ和SDLY分离株则发生了Ⅱ、Ⅳ和Ⅶ处表位的抗原性变异;以SDLY为代表的分离株在aa481和aa533-aa561的缺失,可引起表位Ⅱ、Ⅳ和Ⅴ的抗原性变异。4株新型缺失株的Nsp2多个B细胞表位发生变化,其Nsp2蛋白的部分结构也发生了改变,是否引起免疫逃避的变化尚需深入研究[14]。
2.5 Nsp2蛋白抗原性分析将11株PRRSV分离株与GenBank中部分参考毒株(如VR2332、JXA1和HUN4)的Nsp2进行抗原性分析对比,结果显示各分离株普遍在aa475-aa490、aa500-aa510、aa515-aa520、aa560-aa570位点之间与参考毒株存在较大的抗原性差异。其中,SDZZ和SDLY分离株相较于VR2332和JXA1在aa495-aa500、aa505-aa510、aa535-aa540、aa580-aa585、aa590-aa595位点之间表现出较高的抗原性;而SDRZ01和SDDY在aa490-aa510、aa515-aa523、aa557-aa565和aa572-aa575位点之间则表现出较高的抗原性差异。相较于疫苗株CH-1R,SDZZ和SDLY分离株则在aa495-aa500、aa505-aa510、aa535-aa540、aa580-aa585、aa590-aa595位点之间表现出较高的抗原性,另外SDRZ01和SDDY分离株在aa472-aa480、aa480-aa487、aa490-aa500、aa540-aa550相较于疫苗株CH-1R和JXA1-R具有较高的抗原性(图 5)。

|
图 5 PRRSV株Nsp2抗原性预测分析 Fig. 5 Prediction and analysis of Nsp2 antigenicity of PRRSV |
扩增PRRSV分离株盲传第5和10代细胞毒株的Nsp2、Nsp9基因并测序,结果显示其基因序列未发生改变(数据未列出)。
2.7 基因重组分析利用RDP4和SimPlot对缺失33和73 aa的PRRSV分离株和参考株的全基因序列进行重组分析。结果显示,4株缺失多个氨基酸位点的PRRSV分离株均存在重组现象且以JXA1株为亲本毒株,不同毒株的重组位置具有多样性(表 3)。通过SimPlot软件分析进一步证实SDRZ01分离株基因组中的重组交叉区域,其重组部位主要发生在1-1 189 bp(5′UTR+Nsp1)、2 983-4 482 bp (Nsp2-Nsp3)处,分别与VR2332、NADC30-like毒株发生三毒株间重组(图 6);而其他三个分离株则以VR2332、CH-1a或NADC30-like毒株提供重组片段。
|
|
表 3 以JXA1为亲本的4株PRRSV分离株的重组信息分析结果 Table 3 Recombination analysis of the 4 PRRSV isolates based on the parental strain JXA1 |
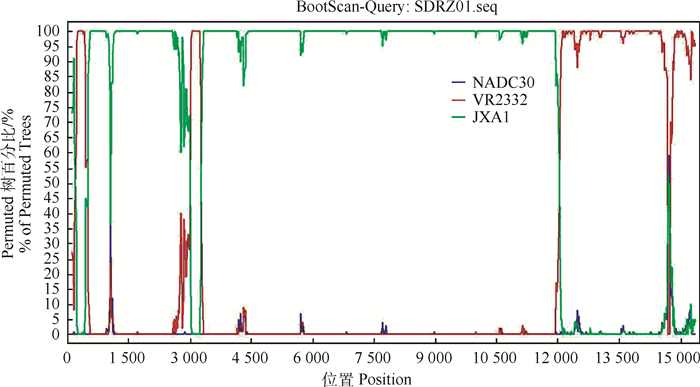
|
Window: 200 bp; Step: 20 bp; GapStrip: On; Reps: 10; Distance model: Kimura (2-parameter); T/t:2.0;Tree model: Neighbor-joining 图 6 PRRSV SDRZ01株的基因重组分析 Fig. 6 Recombination analysis diagram of PRRSV SDRZ01 strain |
相关研究表明[11-12],Nsp9第561、586和592位氨基酸可显著影响PRRSV的复制效率和毒力。经过氨基酸序列比对,分离的SDDY、SDRZ01、SDLY和SDZZ Nsp9的561位氨基酸S和586位氨基酸T分别区别于经典美洲株CC-1、VR2332和CH-1a在该两处位点的氨基酸T和A,而其Nsp9第592位氨基酸S区别于国内最早流行的高致病性毒株HB-1在该位点的氨基酸T(图 7)。上述三处关键氨基酸位点的比较显示该4株PRRSV分离毒株符合HP-PRRSV在以上三处所特有氨基酸的特征,该结果可作为进一步进行动物试验研究其毒力高低的参考。

|
图 7 PRRSV Nsp9第561、586和592位氨基酸比对 Fig. 7 Comparison of the amino acids 561, 586 and 592 in PRRSV Nsp9 |
利用软件SignalP 4.1在线分析功能,采用神经网络法预测分离的4株Nsp2多位点缺失毒株Nsp9蛋白的信号肽,该预测方法主要有三个参数,即S、C和Y值对应每个氨基酸。预测结果中一条起伏较大的曲线表示S值的变化趋势,在信号肽区域内S值较高。C值为剪切位点值,在剪切位点处该值最高。Y值为综合考虑S值和C值之后的参数,要比单独考虑C值更精确。SDRZ01株Nsp9蛋白的信号肽预测结果如图 8所示,1-15 aa区域的S值较高且在11 aa处达到最高0.933,在16 aa处C值和Y值分别达到最高0.215和0.316,由此可知,SDRZ01株Nsp9蛋白存在信号肽,其信号肽区域为1-15 aa,其剪切位点为15-16 aa。
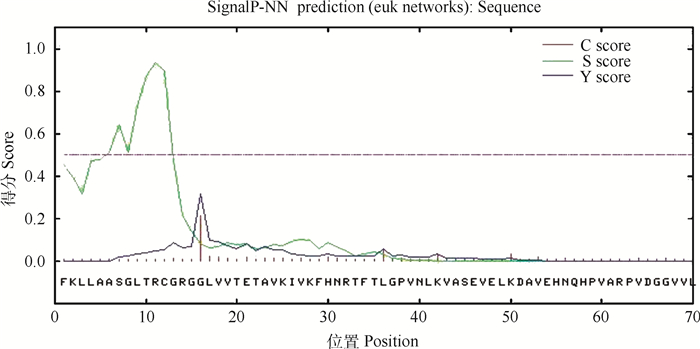
|
图 8 SDRZ01 Nsp9蛋白信号肽预测结果 Fig. 8 Prediction of PRRSV strain SDRZ01 Nsp9 protein signal peptide |
利用SOPMA软件在线分析分离株SDRZ01 Nsp9蛋白二级结构,结果如图 9所示,SDRZ01 Nsp9蛋白的二级结构主要包括α-螺旋(37.07%)、β-折叠(14.64%)、β-转角(5.61%)和无规则卷曲(42.68%)。其中α螺旋和无规则卷曲占较大比例,这对蛋白质的空间结构稳定性有较大帮助。

|
h. α-螺旋; e.β-折叠; t. β-转角; c.无规则卷曲 h. Alpha helix; e. Beta sheet; t. Beta turn; c. Random coil 图 9 SDRZ01 Nsp9蛋白二级结构预测 Fig. 9 Prediction of two-level structure of HP-PRRSV Nsp9 protein |
近年来我国PRRSV流行毒株不断出现新的变异,相继出现美国来源的NADC30-like毒株[15-16]和其他不同的变异毒株[17-18],这些新型毒株的变异主要表现在Nsp2或GP5基因[19]出现了差别巨大的突变或缺失。Nsp2作为PRRSV整个基因组中变异性最大的基因之一,其碱基序列的缺失、突变导致的某些氨基酸位点的变异可以作为监测PRRSV遗传变异的特征性标志[20-21]。Nsp2基因的变异不仅表现在部分碱基位点的突变,更有不同长度的连续性缺失,如HB-2株是我国最早发现的Nsp2存在连续12个氨基酸缺失位点的PRRSV变异株[22],随着2006年高致病性PRRS的暴发,Nsp2的氨基酸序列则表现为不连续的30个氨基酸位点的缺失[23-24]。根据不同报道,新型变异的高致病性PRRSV毒株Nsp2氨基酸缺失位点数量正呈现不断增加的趋势[25-26]。由此可见,PRRSV的Nsp2基因变异程度高、缺失类型多样,而不同毒株之间因遗传特征和抗原性的差异,使得新型突变病毒与以往流行毒株之间的免疫交叉保护力较低,这或许是PRRS难以有效防控的重要原因之一[27]。
分析本研究分离鉴定的PRRSV Nsp2氨基酸序列,发现不同位点存在不同程度的缺失是PRRSV变异的明显特征,11个分离株中出现2种不同的新型缺失,分别为33个不连续氨基酸和73个不连续氨基酸的缺失型,11个分离株均存在aa481和aa533-aa561氨基酸位点的缺失。本试验分离的11株分离株均采集自免疫了不同品系猪繁殖与呼吸综合征疫苗的养殖场,但却存在不同程度的临床及病理症状,母猪流产率和仔猪发病率维持在较高水平。特别是本研究分离鉴定的Nsp2缺失相同73aa的2个病毒株来源于相距几百公里的两个地区,说明PRRSV在不同的地域显示了某种相同的变异趋势。在对比疫苗株如CH-1R和JXA1-R分析发现,11个分离株在多个碱基位点的突变和缺失或是导致机体免疫力低下的根本原因,使原有疫苗诱导的免疫应答显著降低了保护效果,从而导致免疫抑制情况的发生。与以往国内报道的PRRSV毒株不同,本研究中4株PRRSV氨基酸序列均出现不同数量的新缺失位点,表明PRRSV流行株的Nsp2编码的氨基酸位点的数量不断变化,并有进一步扩大的趋势,可能导致PRRSV致病力的进一步变异,因此加强对Nsp2基因变异的监测是十分必要的。Nsp2氨基酸缺失位点大于30 aa的4株PRRSV,其Nsp9的第561、586和592位氨基酸均符合具有高复制能力和毒力的HP-PRRSV的特点,由此可推断Nsp2变异或缺失位点不断扩大与病毒毒力不断变化存在密切关系。
研究表明,PRRSV Nsp2蛋白在影响PRRSV感染宿主细胞和影响病毒毒力方面具有重要作用,主要通过细胞表位来干扰宿主自身的免疫调节功能。本研究中的PRRSV分离株与疫苗株CH-1R和JXA1-R的抗原性对比结果表明,多个部位抗原性的变化可进一步影响B细胞表位的变化,这些变化对PRRSV生物学特性及毒力的影响仍需进一步深入研究。遗传进化分析发现,此次分离的11株PRRSV分离株并不存在于同一分支中,表明PRRSV流行病毒株之间在遗传进化关系上存在交叉现象,一定程度上反映出病原生物的多样性,这可能与近些年来不同省份之间生猪贸易的不断流通有关,从而促进了PRRSV的传播和扩散。基因重组一直是人们关注的焦点之一,揭示PRRSV的基因重组规律对了解PRRSV的流行病学特征有较大的必要性。参照已有的方法,本研究利用RDP4和SimPlot对4株Nsp2氨基酸位点缺失大于30 aa的PRRSV进行重组分析,发现该4株病毒均存在基因重组现象。该4株病毒株均以JXA1为亲本进行重组,或以经典美洲毒株VR2332提供重组片段,又或以国内流行的HP-PRRSV提供重组片段,重组形式和重组位置不尽相同,大多重组在不同的非结构蛋白和次要结构蛋白的区域。
迄今,PRRSV的变异十分活跃且适应性不断增强,在我国境内的流行日趋扩大和复杂化。本研究分离鉴定变异的PRRSV毒株并进行相关分析,为加强对Nsp2基因变异的监测提供了有益的资料与数据,有利于更好地跟踪PRRSV的流行趋势与遗传变异的状况,为有效控制PRRS流行甚至净化提供参考。
4 结论山东及周边部分地区PRRSV检出率为55.7%。对11株PRRSV分离株的序列分析显示,与经典美洲毒株VR2332相比,Nsp2基因已发生明显的新型缺失。7株分离株的Nsp2蛋白分别在第481及533-561位发生了30个氨基酸的不连续缺失;2株PRRSV分离株的Nsp2蛋白在481、533-561位及595-597位发生了三个部位的共33个不连续氨基酸的缺失;2株PRRSV分离株的Nsp2蛋白在475-518及533-561位出现了两个部位的共73个不连续氨基酸的缺失。以上后4株新型缺失株存在较大的基因重组,且重组部位和重组片段数量不相同。另外,Nsp9第561、586和592位氨基酸变异分析显示,该4株病毒在上述三个氨基酸位点与中国HP-PRRSV相符。
| [1] |
杨汉春, 周磊. 猪繁殖与呼吸综合征病毒的遗传变异与演化[J]. 生命科学, 2016, 28(3): 325–336.
YANG H C, ZHOU L. Genetic variation and evolution of porcine reproductive and respiratory syndrome virus[J]. Chinese Bulletin of Life Science, 2016, 28(3): 325–336. (in Chinese) |
| [2] | SONG J, SHEN D, CUI J, et al. Accelerated evolution of PRRSV during recent outbreaks in China[J]. Virus Genes, 2010, 41(2): 241–245. DOI: 10.1007/s11262-010-0507-2 |
| [3] |
刘杏, 王凤雪, 温永俊. 猪繁殖与呼吸综合征病毒感染和复制机制及其毒力研究进展[J]. 病毒学报, 2015, 31(5): 585–592.
LIU X, WANG F X, WEN Y J. Advances in understanding of the infection/replication mechanisms and virulence determinants of the porcine reproductive and respiratory syndrome virus[J]. Chinese Journal of Virology, 2015, 31(5): 585–592. (in Chinese) |
| [4] | TONG G Z, ZHOU Y J, HAO X F, et al. Highly pathogenic porcine reproductive and respiratory syndrome, China[J]. Emerg Infect Dis, 2007, 13(9): 1434–1436. DOI: 10.3201/eid1309.070399 |
| [5] | XIE J X, CUI T T, CUI J, et al. Epidemiological and evolutionary characteristics of the PRRSV in Southern China from 2010 to 2013[J]. Microb Pathog, 2014, 75: 7–15. DOI: 10.1016/j.micpath.2014.08.001 |
| [6] | LU W H, SUN B L, MO J Y, et al. Attenuation and immunogenicity of a live high pathogenic PRRSV vaccine candidate with a 32-amino acid deletion in the nsp2 Protein[J]. J Immunol Res, 2014, 2014: 810523. |
| [7] | ZHOU L, YANG X R, TIAN Y, et al. Genetic diversity analysis of genotype 2 porcine reproductive and respiratory syndrome viruses emerging in recent years in China[J]. BioMed Res Int, 2014, 2014: 748068. |
| [8] | LIU J K, ZHOU X, ZHAI J Q, et al. Genetic diversity and evolutionary characteristics of type 2 porcine reproductive and respiratory syndrome virus in southeastern China from 2009 to 2014[J]. Arch Virol, 2017, 162(9): 2603–2615. DOI: 10.1007/s00705-017-3393-2 |
| [9] |
杨小蓉, 荫硕焱, 潘梦, 等. 一株高致病性猪繁殖与呼吸综合征病毒变异毒株的基因组特征[J]. 畜牧兽医学报, 2012, 43(2): 263–269.
YANG X R, YIN S Y, PAN M, et al. Genomic characterization of a variant of highly pathogenic porcine reproductive and respiratory syndrome virus[J]. Acta Veterinaria et Zootechnica Sinica, 2012, 43(2): 263–269. (in Chinese) |
| [10] | YU L X, WANG X, YU H, et al. The emergence of a highly pathogenic porcine reproductive and respiratory syndrome virus with additional 120aa deletion in Nsp2 region in Jiangxi, China[J]. Transbound Emerg Dis, 2018, 65(6): 1740–1748. DOI: 10.1111/tbed.2018.65.issue-6 |
| [11] | XU L, ZHOU L, SUN W F, et al. Nonstructural protein 9 residues 586 and 592 are critical sites in determining the replication efficiency and fatal virulence of the Chinese highly pathogenic porcine reproductive and respiratory syndrome virus[J]. Virology, 2018, 517: 135–147. DOI: 10.1016/j.virol.2018.01.018 |
| [12] | ZHAO K, GAO J C, XIONG J Y, et al. Two residues in NSP9 contribute to the enhanced replication and pathogenicity of highly pathogenic porcine reproductive and respiratory syndrome virus[J]. J Virol, 2018, 92(7): 9–17. |
| [13] | DE LIMA M, PATTNAIK A K, FLORES E F, et al. Serologic marker candidates identified among B-cell linear epitopes of Nsp2 and structural proteins of a North American strain of porcine reproductive and respiratory syndrome virus[J]. Virology, 2006, 353(2): 410–421. DOI: 10.1016/j.virol.2006.05.036 |
| [14] | YAN Y L, GUO X, GE X N, et al. Monoclonal antibody and porcine antisera recognized B-cell epitopes of Nsp2 protein of a Chinese strain of porcine reproductive and respiratory syndrome virus[J]. Virus Res, 2007, 126(1-2): 207–215. DOI: 10.1016/j.virusres.2007.03.002 |
| [15] | ZHOU L, WANG Z C, DING Y P, et al. NADC30-like strain of porcine reproductive and respiratory syndrome virus, China[J]. Emerg Infect Dis, 2015, 21(12): 2256–2257. DOI: 10.3201/eid2112.150360 |
| [16] | ZHOU L, YANG B N, XU L, et al. Efficacy evaluation of three modified-live virus vaccines against a strain of porcine reproductive and respiratory syndrome virus NADC30-like[J]. Vet Microbiol, 2017, 207: 108–116. DOI: 10.1016/j.vetmic.2017.05.031 |
| [17] | WANG H M, LIU Y G, TANG Y D, et al. A natural recombinant PRRSV between HP-PRRSV JXA1-like and NADC30-like strains[J]. Transbound Emerg Dis, 2018, 65(4): 1078–1086. DOI: 10.1111/tbed.2018.65.issue-4 |
| [18] | SUN Z, WANG J, BAI X F, et al. Pathogenicity comparison between highly pathogenic and NADC30-like porcine reproductive and respiratory syndrome virus[J]. Arch Virol, 2016, 161(8): 2257–2261. DOI: 10.1007/s00705-016-2883-y |
| [19] | GAO G C, XIONG J Y, YE C, et al. Genotypic and geographical distribution of porcine reproductive and respiratory syndrome viruses in mainland China in 1996-2016[J]. Vet Microbiol, 2017, 208: 164–172. DOI: 10.1016/j.vetmic.2017.08.003 |
| [20] | SNIJDER E J, VAN TOL H, ROOS N, et al. Non-structural proteins 2 and 3 interact to modify host cell membranes during the formation of the Arterivirus replication complex[J]. J Gen Virol, 2001, 82: 985–994. DOI: 10.1099/0022-1317-82-5-985 |
| [21] | GRAHAM R L, SIMS A C, BROCKWAY S M, et al. The nsp2 Replicase proteins of murine hepatitis virus and severe acute respiratory syndrome coronavirus are dispensable for viral replication[J]. J Virol, 2005, 79(21): 13399–13411. DOI: 10.1128/JVI.79.21.13399-13411.2005 |
| [22] | GAO Z Q, GUO X, YANG H C. Genomic characterization of two Chinese isolates of Porcine respiratory and reproductive syndrome virus[J]. Arch Virol, 2004, 149(7): 1341–1351. |
| [23] | LENG C L, TIAN Z J, ZHANG W C, et al. Characterization of two newly emerged isolates of porcine reproductive and respiratory syndrome virus from Northeast China in 2013[J]. Vet Microbiol, 2014, 171(1-2): 41–52. DOI: 10.1016/j.vetmic.2014.03.005 |
| [24] | LAGER K M, SCHLINK S N, BROCKMEIER S L, et al. Efficacy of type 2 PRRSV vaccine against Chinese and Vietnamese HP-PRRSV challenge in pigs[J]. Vaccine, 2014, 32(48): 6457–6462. DOI: 10.1016/j.vaccine.2014.09.046 |
| [25] | LI B, XIAO S B, WANG Y W, et al. Immunogenicity of the highly pathogenic porcine reproductive and respiratory syndrome virus GP5 protein encoded by a synthetic ORF5 gene[J]. Vaccine, 2009, 27(13): 1957–1963. DOI: 10.1016/j.vaccine.2009.01.098 |
| [26] |
刘建奎, 魏春华, 杨小燕, 等. 福建地区PRRSV分离株Nsp2基因新变异序列鉴定[J]. 中国预防兽医学报, 2013, 35(4): 271–275.
LIU J K, WEI C H, YANG X Y, et al. Genetic variation of Nsp2 genes of porcine reproductive and respiratory syndrome virus isolated in Fujian province[J]. Chinese Journal of Preventive Veterinary Medicine, 2013, 35(4): 271–275. DOI: 10.3969/j.issn.1008-0589.2013.04.05 (in Chinese) |
| [27] | WANG L J, WAN B, GUO Z H, et al. Genomic analysis of a recombinant NADC30-like porcine reproductive and respiratory syndrome virus in China[J]. Virus Genes, 2018, 54(1): 86–97. |



