天然免疫信号通路作为机体免疫的第一道屏障,通过宿主自身的模式识别受体(pattern recognition receptor, PRR)识别病原体相关分子模式(pathogen associated molecular pattern, PAMP),进而启动下游信号转导,诱导Ⅰ型干扰素等细胞因子表达[1]。TNF受体相关因子(TNF receptor-associated factor, TRAF)家族成员相关的NF-κB活化因子(TANK)结合激酶1 (TANK-binding kinase 1, TBK1)作为干扰素调节因子(interferon regulator factors, IRF)通过活化所必需的磷酸激酶在Ⅰ型干扰素的产生过程中发挥重要作用[2]。对TLR天然免疫信号通路的研究证实宿主细胞感染病毒时,TBK1通过活化IRF3和IRF7来启动IFN的表达[3]。另外,DNA依赖的干扰素调节因子激活剂通过与双链DNA结合促进其与IRF3及TBK1的结合,从而激活纤维母细胞、巨噬细胞以及树突状细胞产生大量的Ⅰ型干扰素,诱导机体免疫应答[4-5]。因此,TBK1在天然免疫和获得性免疫应答过程中都发挥着重要作用。
伪狂犬病病毒(pseudorabies virus, PRV)属于疱疹病毒科A型疱疹病毒亚科猪疱疹病毒1型,可感染多种家畜及野生动物,其传播快、流行广、致死率高,猪作为PRV主要储存宿主和传染源是重点防治对象[6]。有研究证实,野生型PRV感染可以抑制干扰素刺激基因的表达[7]。然而,目前关于PRV宿主逃逸机制研究较少。本试验利用CRISPR/Cas9技术构建猪TBK1基因敲除细胞系,利用PRV-GFP和PRV-QXX毒株检测敲除TBK1基因对PRV复制的影响,以期为PRV免疫逃逸机制研究奠定基础。
1 材料与方法 1.1 试验材料猪肾上皮细胞PK-15、人胚胎肾细胞HEK293T/17和非洲绿猴肾细胞Vero以及猪伪狂犬病病毒PRV-GFP、PRV-QXX以及PRV-gE单克隆抗体均由本实验室保存[8]。LentiCRISPR v2 (#52961)载体购自Addgene。载体质粒pMD2.G和psPAX2购自Sigma。SanPrep柱式DNA胶回收试剂盒(B518131)购自生工生物工程(上海)股份有限公司。CCK-8细胞活力检测试剂盒购自南京建成生物工程研究所。Plasmid Midi kit购自QIAGEN公司。Q5®Hot Start High-Fidelity DNA Polymerase、BsmbⅠ限制性内切酶、T4 DNA连接酶、T7核酸内切酶1购自美国NEB公司。细胞/细菌/酵母基因组DNA提取试剂盒购自上海莱枫生物科技有限公司(TaKaRa中国)。PrimeScriptTM RT reagent Kit、TB GreenTM Premix Ex TaqTM(Tli RNaseH Plus)购自宝日医生物技术(北京)有限公司。β-actin单克隆抗体购自Sigma公司。
1.2 试验方法 1.2.1 引物设计与合成根据National Center for Biotechnology Information (NCBI)网站所公布的猪TBK1基因序列(NM_001105292.1),以第一个外显子为起始,在同源序列区域找到3个合适的靶位点设计sgRNA;其设计规则为以G开头连续20个碱基后为NGG的序列,凡是符合这一规则的序列均可作为Cas9的引导序列;针对3个位点分别设计sgRNA引物,上游引物5′端添加CACC,下游引物5′端添加AAAC (表 1)。参照猪TBK1基因序列(NM_001105292.1),利用Primer 6.0设计TBK1序列扩增引物,用于T7核酸内切酶1(T7E1)检测,引物序列见表 1。
|
|
表 1 引物序列 Table 1 Primers sequence |
根据NCBI网站提供的猪IL-1β、IFN-β、ISG15、β-actin以及PRV-gB、PRV-TK、PRV-gE mRNA序列,利用Primer3Plus在线软件设计Q-PCR引物(表 2)。所有引物序列由生工生物工程(上海)股份有限公司合成。
|
|
表 2 荧光定量PCR引物序列 Table 2 Primers used for quantitative real-time PCR analysis |
将3对sgRNA上、下游引物分别进行退火杂交,退火条件95 ℃ 5 min,关闭PCR仪,使其自然冷却至4 ℃。同时将LentiCRISPR v2质粒用BsmbⅠ37 ℃酶切1 h,然后用SanPrep柱式DNA胶回收试剂盒回收线性片段,退火杂交的引物与酶切后的载体片段用T4 DNA连接酶4 ℃连接过夜。次日,将连接产物转化至E. coli Top10感受态细胞,挑取单菌落送生工生物工程(上海)股份有限公司测序。测序正确菌样扩大培养后用QIAGEN质粒中提试剂盒提取质粒,分别命名为TBK1-sgRNA1、TBK1-sgRNA2、TBK1-sgRNA3。
1.2.3 TBK1敲除细胞系的构建将TBK1-sgRNA重组质粒和pMD2.G、psPAX2包装质粒共转至HEK293T/17细胞收集包装病毒上清液。1 000 r·min-1离心5 min后吸取1 mL病毒液加入提前种好PK-15细胞的6孔板中。48 h后用含8 μg·mL-1 Puromycin的DMEM (含10% FBS)压力筛选细胞,隔天更换压力筛选培养基,连续培养7 d左右,观察到PK-15空白对照组细胞全部死亡,其他三组细胞换用3 μg·mL-1 Puromycin维持培养基继续培养。将筛选获得的细胞接种于60 mm培养皿中,当细胞生长至80%融合度时,取1/3细胞进行基因组提取用于T7E1检测编辑效率,剩余细胞继续传代培养用于单克隆细胞系筛选。T7E1鉴定有编辑的细胞系用有限稀释法筛选单克隆细胞系,单克隆细胞系扩大培养后,提取细胞基因组,用表 1中的P-TBK1引物进行PCR扩增,扩增产物送生工生物工程(上海)股份有限公司测序,挑取有效编辑单克隆细胞系用于后续功能评价。
1.2.4 T7核酸内切酶检测细胞提取基因组后,取200 ng基因组作为模板,用表 1中的P-TBK1引物进行PCR扩增,琼脂糖凝胶电泳后回收814 bp大小的目的条带并检测PCR产物浓度。取200 ng PCR产物进行退火杂交及T7核酸内切酶酶切。体系如下:10×NEB Buffer 21.0 μL,模板200 ng,ddH2O补齐至10 μL。PCR仪中98 ℃ 5 min后,自然冷却至4 ℃,然后加入T7核酸内切酶0.4 μL,37 ℃酶切1 h,用PAGE胶电泳检测T7酶切结果。
1.2.5 CCK-8细胞活力检测参照CCK-8检测试剂盒说明书以每孔1×104个细胞在96孔板中铺种PK-15和PK-15-TBK1-/-细胞。每24 h向指定培养孔中加入10 μL CCK-8溶液37 ℃孵育2 h,然后用酶标仪检测在450 nm处的吸光度。试验重复3次,连续检测5 d。
1.2.6 荧光观察及流式检测以每孔2.5×104个细胞在24孔板中铺种PK-15和PK-15-TBK1-/-细胞。培养细胞生长至50%融合度时,进行感染处理。空白孔细胞计数后按MOI=0.001 PRV-GFP感染两组细胞,37 ℃、5% CO2培养箱吸附1 h后用PBS清洗两次,换含有1%FBS的DMEM培养基37 ℃继续培养6、12、24、36、48 h。使用荧光倒置显微镜观察各组细胞的荧光情况并进行拍照和流式检测。流式操作方法如下:上述培养细胞系用PBS洗涤细胞两次,加入0.25%胰酶消化5~7 min,收集细胞后600 r·min-1离心5 min,离心结束后弃去上清,加入500 μL PBS重悬成单细胞悬液,上机检测,以未感染病毒的PK-15和PK-15-TBK1-/-细胞的荧光数量为空白对照,进行数据收集分析。0 h组用MOI=0.001 PRV-GFP于4 ℃吸附细胞1 h,用预冷的PBS清洗两次后进行后续的荧光观察和流式检测。
1.2.7 Real-time Quantitative PCR (RT-qPCR)按“1.2.5”方法感染MOI=0.1 PRV-QXX后分时间点收取RNA。Trizol裂解法收取各组样品RNA,取1 μg RNA用TaKaRa反转录试剂盒反转录成cDNA,以cDNA为模板,用TB GreenTM Premix Ex TaqTM (Tli RNaseH Plus)进行RT-qPCR检测。β-actin mRNA表达水平为内参值,2-△△Ct法计算PRV-gB、PRV-TK、IFN-β、IL-1β和ISG15 mRNA表达水平,试验重复3次。
1.2.8 Western blot检测PRV-gE蛋白的表达按“1.2.5”方法感染MOI=1 PRV-QXX后分时间点收取蛋白,100 μL RIPA (含protease cocktail)裂解后BCA定量检测样品蛋白含量。取20 μg蛋白样品进行SDS-聚丙烯酰胺凝胶电泳(SDS-PAGE)后转膜,5%脱脂奶室温封闭1 h后,加入PRV-gE单克隆抗体(1:1 000稀释) 4 ℃摇床孵育过夜,1×TBST洗膜3次后,室温摇床孵育HRP标记的山羊抗小鼠IgG (1:1 000稀释) 1 h,1×TBST洗膜3次后进行ECL。以β-actin作为内参,ImageJ软件分析条带灰度值比较TBK1敲除对PRV-gE蛋白表达的影响。
1.2.9 PRV病毒感染力测定(TCID50)以每孔2.0×105个细胞在35 mm dish中铺种PK-15和PK-15-TBK1-/-细胞,当细胞生长至50%融合度时,进行感染处理。按MOI=0.1分别对两组细胞进行感染PRV-QXX,37 ℃、5% CO2培养箱吸附1 h后用PBS清洗两次,换为含有1%FBS的DMEM培养基在37 ℃继续培养36 h,观察细胞病变后收取样品,-80 ℃冰箱中反复冻融两次,用移液枪反复吹打底部细胞,吸出培养基加入离心管中,12 000 r·min-1离心10 min后吸取上清液用Vero细胞进行病毒感染力测定,试验重复3次,用Reed-Muench两氏法计算TCID50·mL-1。
1.2.10 统计学分析试验重复三次,应用GraphPad Prism软件进行统计学分析并作图,P < 0.05具有统计学意义。
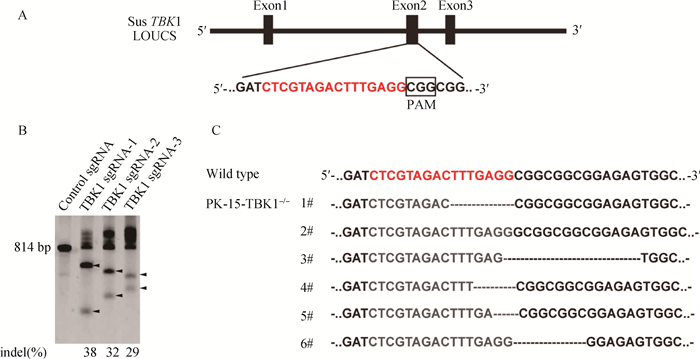
2 结果 2.1 TBK1敲除细胞系的构建以感染慢病毒后的细胞基因组为模板,经T7E1检测结果显示TBK1基因外显子2区的3个sgRNA位点均切出了目的条带(表 1),表明该靶点可能存在基因编辑(图 1A 、B)。利用Image J软件对电泳结果分析计算编辑效率,选取编辑效率最高的TBK1-sgRNA1细胞系进行单克隆化培养。单克隆化培养后获取6株生长状态良好的单克隆细胞系。以细胞基因组为模板,对TBK1基因进行PCR扩增,PCR扩增产物测序,比对分析结果显示与野生型序列相比,1、3、4、5、6号单克隆细胞株打靶区分别缺失了7、14、4、2、7 bp的碱基,2号单克隆细胞株打靶区多了1 bp的碱基(图 1C )。以上结果表明,本试验成功构建稳定敲除TBK1基因的PK-15细胞,随机选取4号细胞株用于后续功能评价,并将该细胞克隆命名为PK-15-TBK1-/-。
|
A.TBK1 sgRNA-1打靶区;B. T7E1酶切鉴定TBK1-sgRNA的活性;C.敲除TBK1基因的单克隆细胞系测序结果 A.Target region of TBK1 sgRNA-1; B. Identification of TBK1-sgRNA activities by T7E1 cutting; C. Sequencing analysis of TBK1 knockout monoclonal cell lines 图 1 敲除TBK1基因的PK-15细胞系构建 Fig. 1 Construction of TBK1 gene knockout PK-15 cell lines |
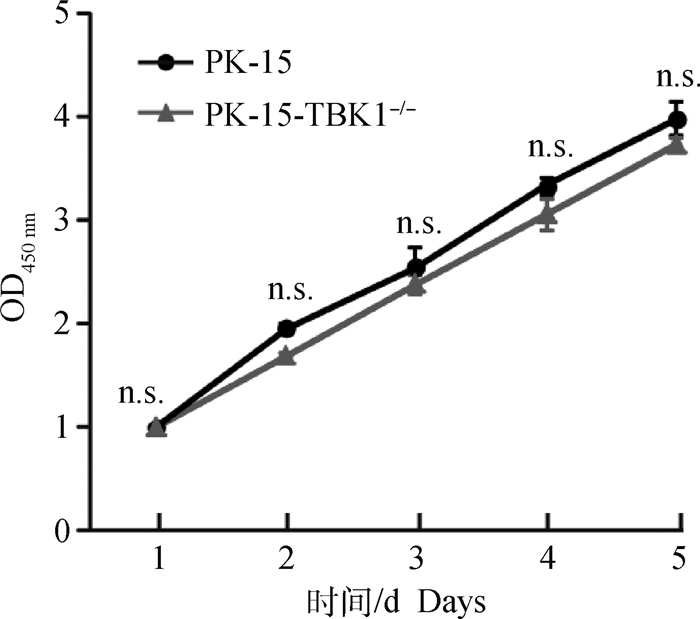
CCK-8检测细胞活力结果显示PK-15-TBK1-/-细胞的存活率与PK-15细胞相比无统计学差异(P>0.05),表明TBK1基因敲除对PK-15细胞活力无影响(图 2)。
|
图 2 敲除TBK1基因不影响PK-15细胞增殖 Fig. 2 TBK1 gene knockout can induce the replication of PRV-GFP |
按MOI=0.001 PRV-GFP感染PK-15和PK-15-TBK1-/-细胞,通过荧光倒置显微镜观察各组细胞的荧光情况并用流式细胞仪检测荧光阳性率。结果显示,TBK1基因敲除显著促进PRV-GFP复制(图 3)。
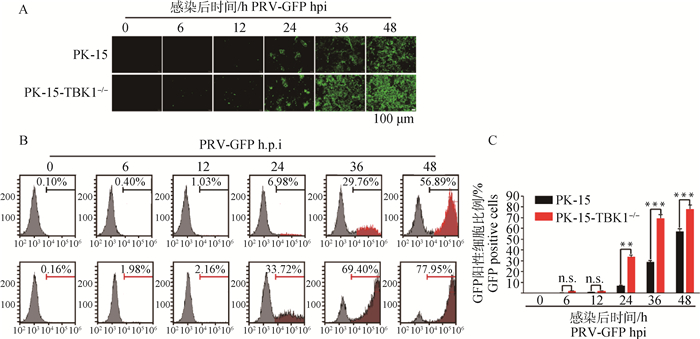
|
A. PRV-GFP荧光显微镜观察结果(4×);B. PRV-GFP流式检测结果;C.流式检测结果的统计分析。标有“*”者,在0.05水平上差异显著,由于0.05已满足统计要求,本试验中不再对其他水平上的检验作出说明,下图同 A. Demonstration of PRV-GFP in cells by fluorescence microscopy (4×); B. Demonstration of PRV-GFP in cells by fluorescence microscopy; C. Statistical analysis of fluorescence microscopy assay. The column marked with * has a significant difference at the 0.05 level. Since 0.05 has met the statistical requirements, we'll no longer explain the significance test at other levels, the same as below 图 3 敲除TBK1基因促进PRV-GFP复制 Fig. 3 TBK1 gene knockout can induce the replication of PRV-GFP |
按MOI=0.1 PRV-GFP感染PK-15和PK-15-TBK1-/-细胞,RT-qPCR检测PRV-gB、PRV-TK mRNA表达水平。如图 4所示,随着PRV复制,PRV-gB、PRV-gE和PRV-TK mRNA表达量递增。敲除TBK1后PRV-gB、PRV-gE和PRV-TK mRNA水平从感染后12 h(12 hpi)就显著高于对照组。结果表明,敲除TBK1促进PRV基因转录。
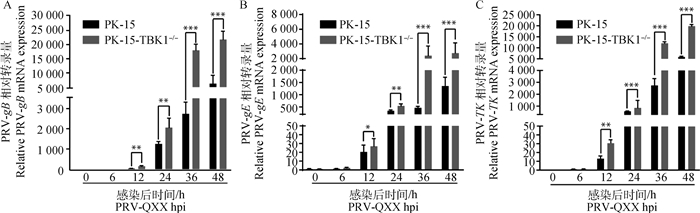
|
A. RT-qPCR检测感染PRV-QXX不同时间PRV-gB基因拷贝数;B. RT-qPCR检测感染PRV-QXX不同时间PRV-gE基因拷贝数;C. RT-qPCR检测感染PRV-QXX不同时间PRV-TK基因拷贝数 A. PRV-gB gene copies were quantified by RT-qPCR; B. PRV-gE gene copies were quantified by RT-qPCR; C. PRV-TK gene copies were quantified by RT-qPCR 图 4 敲除TBK1促进PRV基因转录 Fig. 4 TBK1 gene knockout can induce the transcription of PRV gene |
WB检测结果(图 5)显示PK-15细胞感染PRV 24 h后可以检测到PRV-gE蛋白,48 hpi后表达量达最高。虽然PK-15-TBK1-/-细胞也是在24 hpi可以检测到PRV-gE蛋白,但组间灰度分析结果显示PK-15-TBK1-/-细胞检测到的PRV-gE蛋白显著高于PK-15细胞。结果表明,敲除TBK1促进PRV-gE蛋白表达。
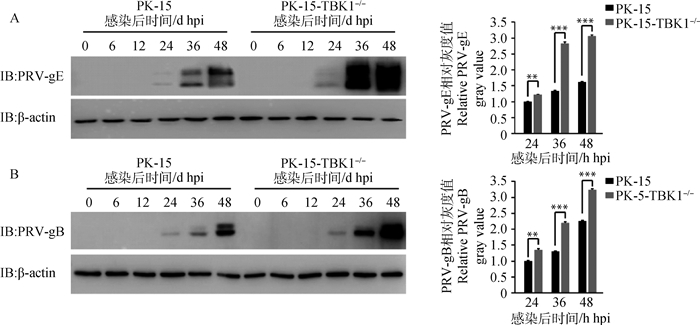
|
A.PRV-gE蛋白Western blot结果(左)及灰度值分析结果(右);B. PRV-gB蛋白Western blot结果(左)及灰度值分析结果(右) A.Western blot results of PRV-gE protein (left) and gray value analysis results (right); B. Western blot results of PRV-gB protein (left) and gray value analysis results (right) 图 5 敲除TBK1促进PRV蛋白表达 Fig. 5 TBK1 gene knockout can induce the expression of PRV protein |
TCID50检测结果显示,PRV-QXX在PK-15细胞复制后TCID50为106.8 TCID50·0.1 mL-1,而在PK-15-TBK1-/-细胞中复制后TCID50为108.5 TCID50·0.1 mL-1。结果表明,敲除TBK1促进PRV-QXX感染性子代病毒的增殖。
2.7 TBK1基因敲除对IL-1β、IFN-β及ISG15转录的影响RT-qPCR检测PK-15细胞及PK-15-TBK1-/-细胞感染PRV后细胞内白细胞介素、干扰素及干扰素刺激基因转录水平。如图 6所示,PK-15细胞内IL-1β、IFN-β和ISG15转录量随着PRV感染时间而上调,48 hpi达到最高值。然而PK-15-TBK1-/-细胞内IL-1β、IFN-β和ISG15转录量并不随着感染时间上调,与PK-15细胞相比组间差异显著。结果表明,敲除TBK1抑制了产生IFN-β和IL-1β信号通路,抑制了干扰素刺激基因的生成进而促进病毒的复制。
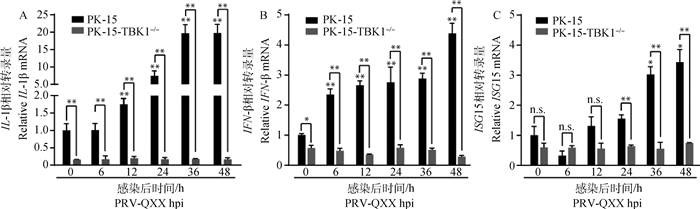
|
A. RT-qPCR检测感染PRV-QXX不同时间IL-1β基因拷贝数;B. RT-qPCR检测感染PRV-QXX不同时间IFN-β基因拷贝数;C. RT-qPCR检测感染PRV-QXX不同时间ISG15基因拷贝数 A.IL-1β gene copies were quantified by RT-qPCR; B. IFN-β gene copies were quantified by RT-qPCR; C. ISG15 gene copies were quantified by RT-qPCR 图 6 敲除TBK1基因抑制PRV诱导的IL-1β、IFN-β及ISG15基因的表达 Fig. 6 TBK1 gene knockout restricts the expression of PRV-induced IL-1β, IFN-β and ISG15 genes |
CRISPR/Cas9基因编辑技术作为第三代基因编辑技术具有设计简单、操作方便、效率高、成本低并且可同时进行多点编辑等优势[9],该技术已经迅速发展为实验室的一种常规试验手段[10]。本试验利用该技术成功构建了稳定敲除TBK1基因的PK-15细胞系,随机选取4号细胞株进行功能评价,并将该细胞克隆命名为PK-15-TBK1-/-。TBK1作为天然免疫中重要的激酶,能够诱导IFN调节因子IRF3的磷酸化以及NF-κB的激活调控Ⅰ型干扰素和干扰素诱导基因的表达[11]。病毒核酸或其中间代谢产物与PRR一旦结合即可募集相应的接头蛋白,形成3条主要的信号通路:RIG1-MAVS、cGAS-STING和TLR3/4-TRIF[2, 12]。接头蛋白MAVS、STING和TRIF均能活化下游的蛋白激酶TBK1,进一步促进IRF3磷酸化以及Ⅰ型干扰素合成[2, 12]。因此,TBK1是不同抗病毒信号通路的汇集点,处于信号网络的中心位置,被认为是抗病毒信号的限速步骤[12]。
虽然TBK1作为关键调节蛋白在抗病毒天然免疫应答中发挥重要作用,但研究显示多种病毒可以通过干预TBK1功能而抑制干扰素表达实现免疫逃逸。研究报道Dengue病毒非结构蛋白NS2A和NS4B以及FMDV 2B蛋白通过阻断TBK1和IRF3的磷酸化抑制宿主细胞Ⅰ型IFN产生[13-14]。PEDV N蛋白以及HCV非结构蛋白NS3通过与TBK1相互作用而抑制IRF3活化及Ⅰ型干扰素的生成[15-16]。Seneca Valley病毒3C蛋白酶和HEV甲基转移酶可以对TBK1去泛素化修饰进而抑制IFN-β表达实现宿主逃逸[17-18]。PRV是一种有囊膜的双链DNA疱疹病毒,该类病毒主要特点是可在神经元细胞中长期潜伏感染[19]。另有报道,PRV感染时可以抑制IFN信号通路以便于病毒在细胞中持续性感染[7, 20]。然而,目前关于PRV免疫逃逸机制研究较少,PRV蛋白与TBK1相互作用研究还未见报道。本试验结果显示敲除TBK1基因可以显著促进PRV-gB[21-22]、PRV-TK[23-25]以及PRV-gE[26] mRNA的转录,PRV-gE和PRV-gB蛋白的翻译以及子代病毒的毒力,表明TBK1在宿主细胞抵抗PRV增殖过程中发挥重要作用。
据报道,抗病毒天然免疫的主要方式是通过释放干扰素和促炎细胞因子直接参与抗病毒天然免疫反应或介导启动适应性免疫应答[27]。另外,Ⅰ型IFN通过与受体结合后启动JAK-STAT信号通路激活IFN刺激基因(ISG)的转录,干扰病毒基因转录或病毒蛋白组分的翻译进而阻止或限制病毒感染[28-29]。本试验结果显示随着PRV感染时间延长,PK-15细胞内IL-1β[30]、IFN-β[31]和ISG15[31]转录水平显著上调。表明PRV感染激活宿主细胞内天然免疫系统。然而,PK-15-TBK1-/-细胞感染PRV后IL-1β、IFN-β和ISG15转录水平组内差异不显著,组间比较显著降低。结果表明TBK1在PRV诱导的干扰素表达发挥重要的调控作用,推测敲除TBK1基因通过抑制干扰素的生成从而促进PRV的复制。本试验所构建的稳定敲除TBK1细胞系可为PRV免疫逃逸机制研究奠定基础。
4 结论首先利用CRISPR/Cas9基因编辑技术构建了TBK1稳定敲除PK-15细胞系,CCK-8检测细胞活力与PK-15细胞无显著差异。其次,通过流式检测显示本试验成功构建的TBK1稳定敲除细胞系可以促进PRV-GFP的复制。另外,进一步研究显示该细胞系感染PRV-QXX时抑制IFN-β、ISG15以及IL-1β的表达,促进病毒基因转录、蛋白表达以及子代病毒的感染力。以上研究结果表明TBK1在宿主细胞抵抗PRV感染发挥重要功能。本试验所构建的稳定敲除TBK1细胞系可为PRV免疫逃逸机制研究奠定基础。
| [1] | TAKEUCHI O, AKIRA S. Pattern recognition receptors and inflammation[J]. Cell, 2010, 140(6): 805–820. DOI: 10.1016/j.cell.2010.01.022 |
| [2] |
王雪, 张毓川, 陈玮. TANK结合激酶1在抗病毒免疫应答中的作用研究进展[J]. 浙江大学学报:医学版, 2016, 45(5): 550–557.
WANG X, ZHANG Y C, CHEN W. Research progress on the role of TANK-binding kinase 1 in anti-virus innate immune response[J]. Journal of Zhejiang University:Medical Sciences, 2016, 45(5): 550–557. (in Chinese) |
| [3] | TENOEVER B R, SHARMA S, ZOU W, et al. Activation of TBK1 and IKKε kinases by vesicular stomatitis virus infection and the role of viral ribonucleoprotein in the development of interferon antiviral immunity[J]. J Virol, 2004, 78(19): 10636–10649. DOI: 10.1128/JVI.78.19.10636-10649.2004 |
| [4] | TAKAOKA A, TANIGUCHI T. Cytosolic DNA recognition for triggering innate immune responses[J]. Adv Drug Deliv Rev, 2008, 60(7): 847–857. DOI: 10.1016/j.addr.2007.12.002 |
| [5] |
韩斌. TBK-1增强HBcAg核酸疫苗特异性免疫应答的实验研究[D].杭州: 浙江大学, 2011.
HAN B. Experimental study on TBK-1 enhancing specific immune responses to HBcAg nucleic acid vaccine[D]. Hangzhou: Zhejiang University, 2011. (in Chinese) http://cdmd.cnki.com.cn/Article/CDMD-10335-1011057877.htm |
| [6] |
刘博涛, 宋昌军, 刘翊中. 伪狂犬病毒致病机理的研究进展[J]. 西北民族大学学报:自然科学版, 2008, 29(4): 65–68.
LIU B T, SONG C J, LIU Y Z. Research progress of pathogenesis of pseudorabies virus[J]. Journal of Northwest University for Nationalities:Natural Science, 2008, 29(4): 65–68. (in Chinese) |
| [7] | BRUKMAN A, ENQUIST L W. Suppression of the interferon-mediated innate immune response by pseudorabies virus[J]. J Virol, 2006, 80(13): 6345–6356. DOI: 10.1128/JVI.00554-06 |
| [8] | WANG J, ZENG L, ZHANG L, et al. Cholesterol 25-hydroxylase acts as a host restriction factor on pseudorabies virus replication[J]. J Gen Virol, 2017, 98(6): 1467–1476. DOI: 10.1099/jgv.0.000797 |
| [9] | AIN Q U, CHUNG J Y, KIM Y H. Current and future delivery systems for engineered nucleases:ZFN, TALEN and RGEN[J]. J Control Release, 2015, 205: 120–127. DOI: 10.1016/j.jconrel.2014.12.036 |
| [10] |
舒磊磊, 甲芝莲, 吴勇浒, 等. CRISPR/Cas9技术的应用性研究[J]. 畜牧兽医学报, 2016, 47(7): 1316–1323.
SHU L L, JIA Z L, WU Y H, et al. The application research of CRISPR/Cas9[J]. Acta Veterinaria et Zootechnica Sinica, 2016, 47(7): 1316–1323. (in Chinese) |
| [11] | FITZGERALD K A, MCWHIRTER S M, FAIA K L, et al. IKKε and TBK1 are essential components of the IRF3 signaling pathway[J]. Nat Immunol, 2003, 4(5): 491–496. DOI: 10.1038/ni921 |
| [12] | LIU S Q, CAI X, WU J X, et al. Phosphorylation of innate immune adaptor proteins MAVS, STING, and TRIF induces IRF3 activation[J]. Science, 2015, 347(6227): aaa2630. DOI: 10.1126/science.aaa2630 |
| [13] | DALRYMPLE N A, CIMICA V, MACKOW E R. Dengue virus NS proteins inhibit RIG-I/MAVS signaling by blocking TBK1/IRF3 phosphorylation:dengue virus serotype 1 NS4A is a unique interferon-regulating virulence determinant[J]. mBio, 2015, 6(3): e00553–15. |
| [14] | LI M, XIN T, GAO X T, et al. Foot-and-mouth disease virus non-structural protein 2B negatively regulates the RLR-mediated IFN-β induction[J]. Biochem Biophys Res Commun, 2018, 504(1): 238–244. DOI: 10.1016/j.bbrc.2018.08.161 |
| [15] | DING Z, FANG L R, JING H Y, et al. Porcine epidemic diarrhea virus nucleocapsid protein antagonizes beta interferon production by sequestering the interaction between IRF3 and TBK1[J]. J Virol, 2014, 88(16): 8936–8945. DOI: 10.1128/JVI.00700-14 |
| [16] | OTSUKA M, KATO N, MORIYAMA M, et al. Interaction between the HCV NS3 protein and the host TBK1 protein leads to inhibition of cellular antiviral responses[J]. Hepatology, 2005, 41(5): 1004–1012. DOI: 10.1002/hep.v41:5 |
| [17] | XUE Q, LIU H S, ZHU Z X, et al. Seneca valley virus 3C protease negatively regulates the type I interferon pathway by acting as a viral deubiquitinase[J]. Antiviral Res, 2018, 160: 183–189. DOI: 10.1016/j.antiviral.2018.10.028 |
| [18] | KANG S M, CHOI C, CHOI I, et al. Hepatitis E virus methyltransferase inhibits type I interferon induction by targeting RIG-I[J]. J Microbiol Biotechnol, 2018, 28(9): 1554–1562. |
| [19] | VAN OPDENBOSCH N, DE REGGE N, VAN POUCKE M, et al. Effects of interferon on immediate-early mRNA and protein levels in sensory neuronal cells infected with herpes simplex virus type 1 or pseudorabies virus[J]. Vet Microbiol, 2011, 152(3-4): 401–406. DOI: 10.1016/j.vetmic.2011.05.010 |
| [20] | SUN Y, LUO Y Z, WANG C H, et al. Control of swine pseudorabies in China:opportunities and limitations[J]. Vet Microbiol, 2016, 183: 119–124. DOI: 10.1016/j.vetmic.2015.12.008 |
| [21] | METTENLEITER T C. Immunobiology of pseudorabies (Aujeszky's disease)[J]. Vet Immunol Immunopathol, 1996, 54(1-4): 221–229. DOI: 10.1016/S0165-2427(96)05695-4 |
| [22] | YIN Y, XU Z W, LIU X W, et al. A live gI/gE-deleted pseudorabies virus (PRV) protects weaned piglets against lethal variant PRV challenge[J]. Virus Genes, 2017, 53(4): 565–572. DOI: 10.1007/s11262-017-1454-y |
| [23] | ENQUIST L W, HUSAK P J, BANFIELD B W, et al. Infection and spread of alphaherpesviruses in the nervous system[J]. Adv Virus Res, 1998, 51: 237–347. DOI: 10.1016/S0065-3527(08)60787-3 |
| [24] | FERRARI M, GUALANDI G L, CORRADI A, et al. Experimental infection of pigs with a thymidine kinase negative strain of pseudorabies virus[J]. Comparat Immunol Microbiol Infect Dis, 1998, 21(4): 291–303. DOI: 10.1016/S0147-9571(98)00012-5 |
| [25] | WANG J C, SONG Z C, GE A M, et al. Safety and immunogenicity of an attenuated Chinese pseudorabies variant by dual deletion of TK & gE genes[J]. BMC Vet Res, 2018, 14(1): 287. DOI: 10.1186/s12917-018-1536-7 |
| [26] | SETAS PONTES M, DEVRIENDT B, FAVOREEL H W. Pseudorabies virus triggers glycoprotein gE-mediated ERK1/2 activation and ERK1/2-dependent migratory behavior in T cells[J]. J Virol, 2015, 89(4): 2149–2156. DOI: 10.1128/JVI.02549-14 |
| [27] |
刘兴楼, 马迪, 舒赛男, 等. 人巨细胞病毒感染THP-1源性巨噬细胞对IL-1β表达的影响[J]. 国际病毒学杂志, 2017, 24(3): 151–155.
LIU X L, MA D, SHU S N, et al. Effects of human cytomegalovirus infection in THP-1 derived macrophages on the expression of IL-1β[J]. International Journal of Virology, 2017, 24(3): 151–155. DOI: 10.3760/cma.j.issn.1673-4092.2017.03.002 (in Chinese) |
| [28] |
甘霖, 赵俊, 陈敬贤, 等. 干扰素抗病毒效力影响因素的研究进展[J]. 微生物与感染, 2018, 13(1): 43–48.
GAN L, ZHAO J, CHEN J X, et al. Research progress on the influencing factors of antiviral efficacy of interferon[J]. Journal of Microbes and Infections, 2018, 13(1): 43–48. DOI: 10.3969/j.issn.1673-6184.2018.01.008 (in Chinese) |
| [29] |
温正旺, 陈洁, 金龙腾, 等. TLR3-TBK1-IRF-3信号通路在EV71手足口病脑损伤中的机制研究[J]. 浙江临床医学, 2016, 18(11): 1995–1997.
WEN Z W, CHEN J, JIN L T, et al. Study on the mechanism of TLR3-TBK1-IRF-3 signaling pathway in brain injury caused by EV71 hand-foot-and-mouth disease[J]. Zhejiang Clinical Medical Journal, 2016, 18(11): 1995–1997. (in Chinese) |
| [30] | LIN H W, CHANG T J, YANG D J, et al. Regulation of virus-induced inflammatory response by β-carotene in RAW264. 7 cells[J]. Food Chem, 2012, 134(4): 2169–2175. DOI: 10.1016/j.foodchem.2012.04.024 |
| [31] | LIU H M, LI S S, YANG X, et al. Porcine ISG15 modulates the antiviral response during pseudorabies virus replication[J]. Gene, 2018, 679: 212–218. DOI: 10.1016/j.gene.2018.09.007 |



