2. 中国牧工商(集团)总公司, 北京 100070
2. China Animal Husbandry Group(CAHG), Beijing 100070, China
玻璃化冷冻是指在高浓度抗冻保护剂的作用下快速降至-196 ℃的过程,同时避免冰晶的形成。1985年Rall和Fahy等[1]首次使用玻璃化冷冻方法成功冷冻了小鼠的胚胎,1998年Mukaida等[2]首次报道使用玻璃化冷冻方法冷冻人胚胎并成功分娩。玻璃化冷冻技术由于成本低、操作简单、效率高等优点,日益被广大科研工作者和临床工作人员采用。然而,该技术仍然面临有许多挑战,如玻璃化冷冻会影响卵母细胞重要发育基因的表达,玻璃化冷冻卵母细胞IVF囊胚发育能力严重降低[3-4]。如朱士恩[5]研究发现,小鼠卵母细胞玻璃化冷冻的体外受精率显著低于新鲜卵母细胞(46.67%和86.06%),在家畜上,牛卵母细胞玻璃化冷冻体外受精后的卵裂率和囊胚发育率仅为62.7%和18.9%[6]。
哺乳动物的DNA甲基化作为重要的表观遗传修饰,在胚胎发育、印记基因、X染色体失活等过程中发挥重要作用[6-7]。Cheng等[8]使用亚硫酸氢盐测序分析研究表明,玻璃化冷冻MⅡ期小鼠卵母细胞IVF囊胚基因组中H19、Peg3、Snrpn的DMR甲基化缺失。刘佳[9]研究发现,玻璃化冷冻小鼠卵母细胞获得的胚胎中DNA甲基化状态出现异常,如囊胚阶段的Dnmt3b表达量显著下降。Chen等[10]使用RT-PCR研究牛玻璃化冷冻卵母细胞IVF囊胚发现Dnmt3A显著下调。也有使用亚硫酸氢钠转化和焦磷酸测序方法进行相关研究的[11]。但目前研究主要集中在少数基因的DNA甲基化模式,且是在不同组织、不同处理方法的甲基化差异分析,迄今为止,关于玻璃化冷冻卵母细胞IVF囊胚在全基因组水平上的研究尚未见报道。
单细胞全基因组甲基化测序技术(single cell-whole genome methylation sequencing, scWGMS)是指利用单细胞全基因组亚硫酸氢盐测序(single cell-whole genome bisulfite sequencing,scWGBS)得到全基因组DNA甲基化图谱的方法[12]。哺乳动物的胞嘧啶甲基化主要发生在CG位点上,scWGMS技术是在单细胞水平获得全基因组范围内所有C位点的甲基化水平,可精确解析单细胞中每个胞嘧啶的甲基化状态。目前,Li等[13]使用scWGBS技术研究了妇女的多囊卵巢综合征(PCOS),同样该技术在构建人胚胎发育过程的动态甲基化图谱[14]、小鼠肝癌细胞甲基化研究[15]等方面都已取得了成功应用。
本试验采用scWGMS技术对新鲜、玻璃化冷冻卵母细胞IVF囊胚全基因组甲基化模式进行初步探索。随后,对测序结果采用生物信息学分析,包括全基因组甲基化水平、差异甲基化区域(differentially methylated region,DMR)的聚类分析、DMR的GO和KEGG分析等。通过分析研究有助于阐明玻璃化冷冻引起卵母细胞表观遗传差异的机制,进而为提高玻璃化冷冻卵母细胞IVF囊胚的质量奠定生物信息学基础。
1 材料与方法 1.1 试剂和仪器除特殊说明,所有试验试剂均购于Sigma公司(美国)。牛的常规精液购于山东奥克斯畜牧种业有限公司。
1.2 牛卵母细胞采集和体外成熟将采集好的牛卵巢在2 h内运回实验室,提前组装好真空泵和加热垫,使用提前预热并加入双抗(青霉素+链霉素)的生理盐水洗涤2~3次,选择卵巢表面直径大小约为2~8 mm的卵泡,并用针头抽取其卵泡液于15 mL离心管中。将15 mL离心管放置于恒温加热台上静置,吸取沉淀于90 mm培养皿中,在显微镜下挑选含3层及以上卵丘细胞的卵丘-卵母细胞复合体(COCs),依次在卵母细胞洗涤液、卵母细胞成熟液中洗2~3次。最后放入含有成熟液的四孔板中(750 μL·孔-1,每孔50枚COCs),在5% CO2、38.5 ℃和100%湿度的恒温培养箱中培养22~24 h。
1.3 牛卵母细胞玻璃化冷冻及解冻玻璃化冷冻及解冻步骤参照Hou等[6]的方法,略作改动。玻璃化冷冻前,将室温调整至(25±1) ℃,在恒温台(38~39 ℃)进行试验。使用OPS管吸取M Ⅱ期卵母细胞,先在预处理液中平衡30 s,再转移至EDFSF40中平衡25 s,之后将MⅡ期卵母细胞吸入OPS管,并直接投放于液氮中。
先将OPS管从液氮中取出,将其浸入到含有浓度为0.25 mol·L-1蔗糖溶液的表面皿中进行解冻,之后使用口吸管将卵母细胞从OPS管中吹出,先将卵母细胞在0.25 mol·L-1蔗糖溶液中平衡约1 min,再转移到0.15 mol·L-1蔗糖溶液中平衡约5 min,之后在成熟液中洗2次。最后挑选出解冻后细胞膜完整性较好、胞质较均匀的卵母细胞放入成熟液中待用。
1.4 解冻常规精液参照Brackett和Oliphant[16]的方法,略作改动。将液氮罐中的冻精取出,并在38 ℃水浴锅中进行解冻,将解冻后的常规精液移入加有7 mL洗精液的15 mL离心管中,低速离心机中离心2次(5 min·次-1,1 500 r·min-1),离心结束后弃上清液,取20 μL精液与80 μL受精液混合,调整精子密度为1×106个·mL-1,成100 μL受精滴,放入恒温培养箱(38.5 ℃、5% CO2、100%湿度)孵育1.5 h。
1.5 牛新鲜、玻璃化冷冻卵母细胞体外受精将新鲜、冷冻卵母细胞移入100 μL受精滴(25枚·滴-1)中进行体外受精,在恒温培养箱中培养6~8 h后,脱去卵丘细胞,移入胚胎前期培养液中培养48 h, 统计并计算卵裂率。挑选卵裂的受精卵移入胚胎后期培养液中,且隔天半量换液,第7天统计并计算囊胚率。
1.6 牛新鲜、玻璃化冷冻卵母细胞体外受精囊胚的收集分别收集新鲜、玻璃化冷冻卵母细胞IVF第7天的囊胚,在体视显微镜下放入含有链霉蛋白酶(5 mg·mL-1)的DPBS溶液中2~3 min,去除透明带,挑选一个完整的去除透明带的囊胚放入DPBS中洗2遍后,转移至裂解缓冲液中,此过程中尽量排除细胞所在原环境中的液体,重复3次,-80 ℃冻存备用。
1.7 新鲜、玻璃化冷冻卵母细胞IVF囊胚单细胞全基因组甲基化测序将收集到的新鲜、玻璃化冷冻卵母细胞IVF囊胚的6个样品(各3个)放于干冰,送北京安诺优达基因科技有限公司进行单细胞全基因组甲基化检测。以每组样品为起始进行建库,使用EZ DNA Methylation-GoldTM Kit DNA甲基化试剂盒(D5006)方法裂解单细胞并进行Bisulfite处理,使用带有生物素biotin标记的oligo1引物进行一链合成,以链霉亲和素磁珠(M-280磁珠)进行钓取,加入带接头序列的oligo1引物进行二链合成,通过PCR扩增技术引入Index序列,得到最终文库。本试验中共建立6个scBS文库,分别为IVF_B_1、IVF_B_2、IVF_B_3(新鲜卵母细胞体外受精囊胚的scBS文库)以及V-IVF_B_1、V-IVF_B_2、V-IVF_B_3(玻璃化冷冻卵母细胞体外受精囊胚的scBS文库)。scBS文库构建完成后,使用Agilent 2100/LabChip GX Touth对文库的片段及长度进行检测,符合预期结果后,在HiSeq测序平台上运行PE150双端测序程序,进行单细胞全基因组甲基化测序。
1.8 新鲜、玻璃化冷冻卵母细胞IVF囊胚单细胞全基因组甲基化测序的生物信息学分析为保证数据质量,先采用Trimmomatic(v0.36)软件[17]对2组样品的原始序列进行过滤,之后采用Bismark(v0.16.3)将过滤后序列与参考基因组序列进行比对[18],先将测序基因组和参考基因组[Bos_ taurus_UMD_3.1.1 genome]分别进行反向互补的转换;随后将转换后的测序基因组同转换后的参考基因组序列底层调用Bowtie2进行两两比对,在4种平行比对结果中选取最好的一个作为最终比对结果,唯一比对位点进行后续甲基化信息的分析。基于比对后的结果,对全基因组C碱基的甲基化情况进行检测,由于甲基化位点主要集中在CpG二核苷酸上,因此后续分析主要集中于全基因组CpG位点,即样品甲基化位点覆盖度统计;对全部甲基化CpG位点不同细胞样品的整体甲基化水平进行计算,即样品的全基因组甲基化水平。
采用DSS软件进行DMR的筛选,最终保留P < 0.05、组间平均甲基化水平差异≥0.2、包含3个及以上CpG位点的区域作为最终的DMR。基因本体(Gene Ontology,GO)是一个在生物信息学领域中广泛使用的本体,可挖掘出所研究的生物学问题相关的生物学过程。GO分为3个Ontology:分子功能(molecular function,MF)、细胞组分(cellular component,CC)和生物过程(biological process,BP)。
KEGG(Kyoto Encyclopedia of Genes and Genomes)是基因组破译方面有关通路的主要公共数据库[19],Pathway显著性富集分析以KEGG Pathway为单位,应用超几何检验,找出与整个基因组背景相比,在DMR相关基因中显著富集(P < 0.05)的通路。
1.9 数据分析IVF囊胚试验重复至少3次。IVF囊胚数据采用SAS软件进行分析,百分数比较时经反正弦转化后采用方差分析,结果用“平均值±标准差”表示,差异显著性判断标准为P < 0.05。
2 结果 2.1 新鲜、玻璃化冷冻牛卵母细胞体外受精囊胚的卵裂率和囊胚率分析如表 1所示,本试验中新鲜卵母细胞IVF(IVF组)的卵裂率((75.49±6.35)%)显著高于玻璃化冷冻卵母细胞IVF(V-IVF组)的卵裂率((50.98±4.82)%,P < 0.05);且IVF组的囊胚率((31.82±2.78)%)显著高于V-IVF组的囊胚率((11.19±1.02)%,P < 0.05)。
|
|
表 1 新鲜、玻璃化冷冻卵母细胞IVF囊胚的分析 Table 1 Analysis on fresh and vitrified oocyte IVF blastocysts |
本试验中,IVF组和V-IVF组的6个样品数据过滤和数据比对情况见表 2。经统计,过滤后IVF、V-IVF组的Q30平均值分别为92.88%、92.26%;过滤后IVF、V-IVF组序列数目占原始序列的平均比例分别为90.97%、90.82%。IVF、V-IVF组过滤后比对到基因组上的数目占过滤后序列数目的平均百分比分别为34.27%、33.56%;IVF、V-IVF组唯一比对到基因组上的数目占过滤后序列的平均百分比分别为28.2%、27.7%。
|
|
表 2 数据过滤和数据比对统计结果 Table 2 Statistical results of clean Reads and mapped Reads |
本试验中IVF组和V-IVF组的6个样品甲基化位点覆盖度的统计情况见表 3。IVF、V-IVF组覆盖深度≥3X的CpGs占全基因组CpGs的平均百分比分别为13.82%、12.98%。
|
|
表 3 样品甲基化位点覆盖度统计结果 Table 3 Statistical results of sample CpG coverage |
IVF和V-IVF组6个样品的全基因组甲基化水平见图 1,横轴表示样品名称,纵轴表示全基因组甲基化水平。其中,IVF 3组的整体甲基化水平分别为9.0%、6.0%、4.2%,平均值为6.4%;而V-IVF 3组的整体甲基化水平分别为5.0%、3.0%、7.0%,平均值为5.0%。经SAS统计学计算发现,IVF组的整体甲基化平均水平(6.4%)显著高于V-IVF组的整体甲基化平均水平(5.0%,P < 0.05)。

|
图 1 IVF、V-IVF组的全基因组甲基化水平 Fig. 1 Genome-wide methylation levels of IVF and V-IVF groups |
DMR是指不同比较组间具有显著甲基化水平修饰差异的基因组区域。采用DSS软件进行DMR的筛选,最终保留P < 0.05、组间平均甲基化水平差异≥0.2、包含3个及以上CpGs位点的区域作为最终的DMR。本试验共找到143个DMR区域,表 4列举了样品中部分DMR中高甲基化区域和DMR中低甲基化区域及其对应的基因名称。
|
|
表 4 DMR区域甲基化水平统计结果 Table 4 DMR methylation level statistics result |
图 2为新鲜、冷冻卵母细胞IVF囊胚DMR聚类图。如图 2所示,IVF组和V-IVF组聚类明显,并且IVF组的甲基化水平明显高于V-IVF组的甲基化水平。

|
行表示DMR,列表示细胞样品。右侧标尺数值为甲基化水平(0~100,%) Lines indicate DMR; Columns indicate sample. The right scale value is the methylation level(0-100, %) 图 2 IVF、V-IVF组DMR聚类图 Fig. 2 DMR clustering diagrams of IVF and V-IVF groups |
如图 3所示,GO富集分析分为3大类,分别是生物过程(biological process,BP)、细胞组分(cellular component,CC)和分子功能(molecular function,MF)。横坐标为GO分析下的每一个大类,左纵坐标为该类基因所占的比例,右纵坐标为富集于该类具体的基因数。不同的颜色分别代表不同的组别。生物过程主要在新陈代谢、生长发育、细胞定位、细胞刺激反应;细胞组分主要在细胞膜、细胞器、高分子配合物、细胞连接等;分子功能主要在核酸结合调节因子、催化剂、转运体等。
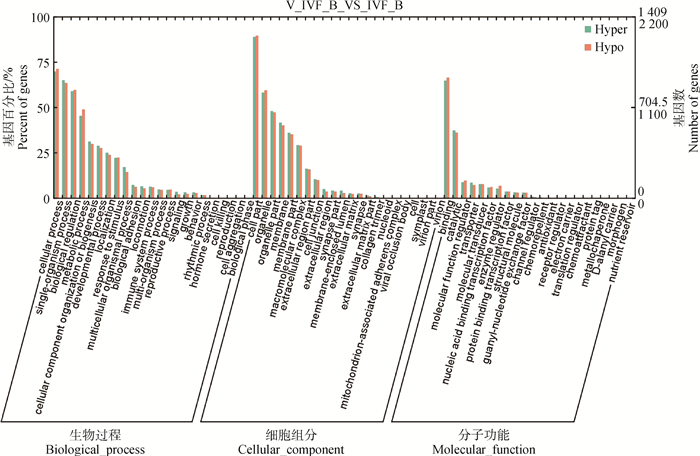
|
横坐标为GO分析下的每一个大类。左纵坐标为富集于该类下基因百分比,右纵坐标为富集于该类具体的基因数。不同的颜色分别代表不同的组别 The horizontal coordinates are each large class under GO analysis, the left vertical coordinates are the percentage of genes under this class, and the right vertical coordinates are the gene numbers under this class. Different colors represent different groups 图 3 差异甲基化区域GO富集结果 Fig. 3 Results of GO enrichment in different methylation regions |
如图 4所示,KEGG富集散点图是KEGG富集分析结果的图形化展示方式,笔者选取富集最显著的20个通路条目进行展示,其中Q值的取值范围为[0, 1],Q值越小,富集越显著。纵轴表示通路名称,横轴表示富集因子(rich ratio),Q值的大小用点的颜色来表示,每个通路下包含的差异基因的多少用点的大小来表示。图中富集通路依次是癌症发生,其次依次是黏着斑、肌动蛋白细胞骨架信号通路、钙离子信号通路、轴突导向、谷氨酸能突触、多巴胺能神经突触、胆碱能突触、血管平滑肌收缩、粘合连接、GnRH信号通路、磷脂酰肌醇信号通路、胰岛素分泌、子宫内膜癌发生、卵巢类固醇激素生成等过程。
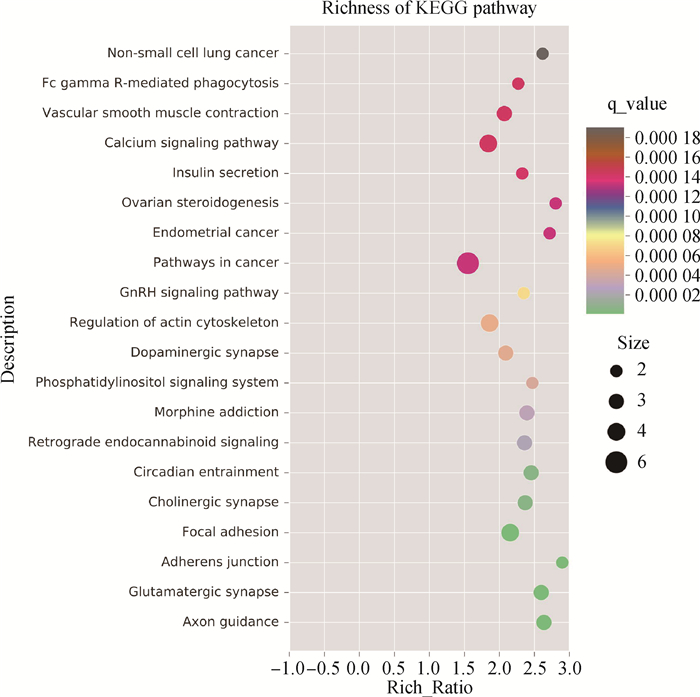
|
纵轴代表通路名称,横轴代表富集因子(rich ratio),点的颜色和大小分别表示富集在某通路差异基因的数量和富集显著性 The vertical axis represents the name of pathway, the horizontal axis represents rich ratio, the color and size of the points respectively indicate the number and significance of differentially expressed genes enriched in a certain pathway 图 4 KEGG富集散点图 Fig. 4 KEGG enrichment scatter plot |
近年来,许多DNA甲基化检测方法不断发展并应用于全基因组DNA甲基化分析,比如,DNA甲基化免疫共沉淀并结合高通量测序(MeDIP-seq)[20]、重亚硫酸氢盐测序(RRBS)[21]以及WGBS。RRBS由于数据分析的复杂性,现有的分析软件不能完全满足要求[22];而WGBS是一种很好的测定DNA甲基化的方法,且具有很高的分辨率[23]。近几年,该技术的应用逐步扩展,如Zhang等[24]使用WGBS技术首次对小鼠脑组织的DNA甲基化特征进行系统研究,以寻找新的甲基化位点对抗疾病,Zhang等[25]使用WGBS技术对多产和低产的湖羊卵巢组织进行研究,并构建了全基因组甲基化图谱,郭添福等[26]使用WGBS技术对不同性别大白猪背最长肌的全基因组DNA甲基化和DMR区域进行研究,并构建了高分辨率单碱基全基因组甲基化图谱。
本试验中,样品过滤后IVF、V-IVF组的Q30平均值分别为92.88%、92.26%;过滤后IVF、V-IVF组序列数目占原始序列的平均比例分别为90.97%、90.32%,说明样品测序质量较好,可以利用过滤后序列数据进行后续分析。样品过滤后IVF、V-IVF组比对到基因组上的数目占过滤后序列数目的平均百分比分别为34.27%、33.56%,比对率>30%,满足后续分析的要求[27]。
Liang等[28]使用免疫荧光染色法发现,玻璃化冷冻小鼠MII卵母细胞会导致整体基因组甲基化水平的降低,这可能是引起小鼠早期胚胎体外发育能力降低的重要因素。Ma等[29]研究发现,玻璃化冷冻降低了牛GV期卵母细胞的成熟率,进而改变了受精相关基因(CD9、CD81)和DNA甲基转移酶在卵母细胞体外成熟过程中的表达。本试验采用单细胞全基因组甲基化测序技术的结果表明,IVF组囊胚整体甲基化平均水平显著高于V-IVF组的整体甲基化平均水平,这也进一步验证了玻璃化冷冻会对卵母细胞造成损伤,影响早期胚胎的表观遗传重编程,进而影响体外受精进程。
DMR在基因表达调控方面发挥着重要作用,分布于启动子、外显子、内含子等多种基因组功能区域,故DMR对基因的调控方式具有多样性。本研究中,IVF和V-IVF组共找到143个DMR区域。玻璃化冷冻过程中所采用的抗冻保护剂、急剧降温和升温过程,都是造成DMR的重要因素。进一步分析表明,它们位于不同染色体上,且各个DMR区域的长度不等,同时存在以下有趣的现象:1)IVF组的部分DMR区域高甲基化,而V-IVF组相对应部分DMR区域甲基化水平为零;2)V-IVF组的部分DMR区域甲基化水平高,而IVF组的相对应部分DMR区域较低。这些生物学信息很明确的表明,卵母细胞玻璃化冷冻会引起其IVF囊胚甲基化水平的显著异常。
对143个DMR区域中的部分DMR高甲基化和低甲基化区域中对应的基因功能进行查阅,FARP2(FERM、RhoGEF和含有pleckstrin结构域的蛋白1)及同源FARP1被鉴定为RhoGTP酶的鸟嘌呤核苷酸交换因子(GEF),其在神经元发育中发挥调节作用[30]。FARP2还与PlexinA家族成员相互作用调节Plexin介导的排斥性轴突导向和树突发育,还发现FARP2参与Plexin介导的骨稳态调节[31]。PI4KA是磷脂酰肌醇4-激酶,可催化磷脂酰肌醇转化为磷脂酰肌醇4-磷酸(PtdIns4P),已知哺乳动物中的4种PI4激酶为PI4KA、PI4KB、PI4K2A和PI4K2B,它们参与细胞内脂质和蛋白质的转运[32]。FAM3D是FAM3基因家族4个成员(FAM3A、FAM3B、FAM3C和FAM3D)之一,它们是重要的糖脂代谢调节因子。FAM3D通过mkp1依赖性的ERK1/2信号通路抑制胰高血糖素的分泌[33]。这些差异基因在新鲜、玻璃化冷冻卵母细胞IVF囊胚中表达差异极显著,为后续的表观遗传修饰提供理论支持。
GO是一个在生物信息学领域中广泛使用的本体,可挖掘出所研究生物学问题相关的生物学过程,即:分子功能、细胞组分和生物过程。对IVF和V-IVF组进行GO富集分析时发现,143个DMR富集的生物学过程主要包括:1)生物过程:新陈代谢、生长发育、细胞定位、细胞刺激反应;2)细胞组分:细胞膜、细胞器、高分子配合物、细胞连接等;3)分子功能:核酸结合调节因子、催化剂、转运体等。同时,与上述功能相关的候选基因主要是生长发育基因:XAB2(XPA binding protein 2)和IFT140(intraflagellar transport 140),核酸结合因子:KLF11(kruppel like factor 11)、ZNF827(zinc finger protein 827)和ZNF618(zinc finger protein 618),组蛋白乙酰化基因:NACC2(NACC family member 2)、NCOR2(nuclear receptor corepressor 2)和WDR5(WD repeat-containing protein 5)。KEGG作为基因组破译方面的数据库,通过Pathway显著性富集能确定DMR相关基因参与的最主要生化代谢途径和信号转导途径。本试验结果表明,IVF和V-IVF组DMR富集的通路主要包括:钙离子信号通路、GnRH信号通路、磷脂酰肌醇信号通路、子宫内膜癌发生、卵巢类固醇激素生成等过程。
4 结论本研究采用scWGBS技术发现,卵母细胞玻璃化冷冻会引起其IVF囊胚全基因甲基化水平显著降低,对143个DMR区域分析发现,生物学功能主要显著富集在新陈代谢、生长发育、细胞定位等过程,通路主要富集在生长发育、核酸结合及组蛋白乙酰化上,并筛选出几个与之相关的候选基因。本试验有助于阐明玻璃化冷冻引起卵母细胞表观遗传差异的机制,进而为提高玻璃化冷冻卵母细胞IVF囊胚的质量奠定生物信息学基础。
| [1] | RALL W F, FAHY G M. Ice-free cryopreservation of mouse embryos at -196℃ by vitrification[J]. Nature, 1985, 313(6003): 573–575. DOI: 10.1038/313573a0 |
| [2] | MUKAIDA T, WADA S, TAKAHASHI K, et al. Vitrification of human embryos based on the assessment of suitable conditions for 8-cell mouse embryos[J]. Hum Reprod, 1998, 13(10): 2874–2879. DOI: 10.1093/humrep/13.10.2874 |
| [3] | CHANG H C, GRIFO J, KREY L C. Vitrification of metaphase Ⅱ oocytes impacts oct-4 expression during subsequent preimplantation development[J]. Fertil Steril, 2005, 84(Suppl 1): S37–S38. |
| [4] | PRENTICE-BIENSCH J R, SINGH J, MAPLETOFT R J, et al. Vitrification of immature bovine cumulus-oocyte complexes:effects of cryoprotectants, the vitrification procedure and warming time on cleavage and embryo development[J]. Reprod Biol Endocrinol, 2012, 10: 73. DOI: 10.1186/1477-7827-10-73 |
| [5] |
朱士恩.卵母细胞与胚胎玻璃化冷冻保存技术研究新进展[C]//中国畜牧兽医学会动物繁殖学分会第十五届学术研讨会论文集(上册).天津: 中国畜牧兽医学会, 2010: 5.
ZHU S E.Recent advances in vitrification of oocytes and embryos[C]//Proceedings of the 15th Symposium of Animal Reproduction Branch of Chinese Animal Husbandry and Veterinary Society.Tianjin: Chinese Association of Animal Science and Veterinary Medicine, 2010: 5.(in Chinese) |
| [6] | HOU Y P, DAI Y P, ZHU S E, et al. Bovine oocytes vitrified by the open pulled straw method and used for somatic cell cloning supported development to term[J]. Theriogenology, 2005, 64(6): 1381–1391. DOI: 10.1016/j.theriogenology.2005.03.012 |
| [7] | FANG X B, ZHAO Z H, YU H B, et al. Comparative genome-wide methylation analysis of longissimus dorsi muscles between Japanese black (Wagyu) and Chinese red steppes cattle[J]. PLoS One, 2017, 12(8): e0182492. DOI: 10.1371/journal.pone.0182492 |
| [8] | CHENG K R, FU X W, ZHANG R N, et al. Effect of oocyte vitrification on deoxyribonucleic acid methylation of H19, Peg3, and Snrpn differentially methylated regions in mouse blastocysts[J]. Fertil Steril, 2014, 102(4): 1183–1190. DOI: 10.1016/j.fertnstert.2014.06.037 |
| [9] |
刘佳.玻璃化冷冻小鼠MⅡ期卵母细胞对附植前胚胎DNA甲基化的影响[D].北京: 中国农业大学, 2015.
LIU J.Effects of vitrification cryopreservation of mouse MⅡ stage oocytes on DNA methylation of preimplantation embryos[D].Beijing: China Agricultural University, 2015.(in Chinese) |
| [10] | CHEN H H, ZHANG L, DENG T F, et al. Effects of oocyte vitrification on epigenetic status in early bovine embryos[J]. Theriogenology, 2016, 86(3): 868–878. DOI: 10.1016/j.theriogenology.2016.03.008 |
| [11] | SONG S, GHOSH J, MAINIGI M, et al. DNA methylation differences between in vitro- and in vivo-conceived children are associated with ART procedures rather than infertility[J]. Clin Epigenetics, 2015, 7(1): 41. |
| [12] | CLARK S J, SMALLWOOD S A, LEE H J, et al. Genome-wide base-resolution mapping of DNA methylation in single cells using single-cell bisulfite sequencing (scBS-seq)[J]. Nat Protoc, 2017, 12(3): 534–547. DOI: 10.1038/nprot.2016.187 |
| [13] | LI Q N, GUO L, HOU Y, et al. The DNA methylation profile of oocytes in mice with hyperinsulinaemia and hyperandrogenism as detected by single-cell level whole genome bisulphite sequencing (SC-WGBS) technology[J]. Reprod Fertil Dev, 2018, 30(12): 1713–1719. DOI: 10.1071/RD18002 |
| [14] | GUO H S, ZHU P, YAN L Y, et al. The DNA methylation landscape of human early embryos[J]. Nature, 2014, 511(7511): 606–610. DOI: 10.1038/nature13544 |
| [15] | GRAVINA S, DONG X, YU B, et al. Single-cell genome-wide bisulfite sequencing uncovers extensive heterogeneity in the mouse liver methylome[J]. Genome Biol, 2016, 17(1): 150. |
| [16] | BRACKETT B G, OLIPHANT G. Capacitation of rabbit spermatozoa in vitro[J]. Biol Reprod, 1975, 12(2): 260–274. |
| [17] | BOLGER A M, LOHSE M, USADEL B. Trimmomatic:a flexible trimmer for Illumina sequence data[J]. Bioinformatics, 2014, 30(15): 2114–2120. DOI: 10.1093/bioinformatics/btu170 |
| [18] | KRUEGER F, ANDREWS S R. Bismark:a flexible aligner and methylation caller for bisulfite-seq applications[J]. Bioinformatics, 2011, 27(11): 1571–1572. DOI: 10.1093/bioinformatics/btr167 |
| [19] | KANEHISA M, ARAKI M, GOTO S, et al. KEGG for linking genomes to life and the environment[J]. Nucleic Acids Res, 2007, 36(S1): D480–D484. |
| [20] | SU J M, WANG Y S, XING X P, et al. Genome-wide analysis of DNA methylation in bovine placentas[J]. BMC Genomics, 2014, 15: 12. DOI: 10.1186/1471-2164-15-12 |
| [21] | CHAN M M, SMITH Z D, EGLI D, et al. Mouse ooplasm confers context-specific reprogramming capacity[J]. Nat Genet, 2012, 44(9): 978–980. DOI: 10.1038/ng.2382 |
| [22] |
祝海军.RRBS数据分析新流程的建立及其在肿瘤细胞中等位基因特异甲基化分析的应用[D].上海: 华东师范大学, 2016.
ZHU H J.Establishment of a new pipeline for RRBS data analysis and application of identification and characterization of allele specific methylations in cancer cells[D].Shanghai: East China Normal University, 2016.(in Chinese) |
| [23] | LAIRD P W. Principles and challenges of genome-wide DNA methylation analysis[J]. Nat Rev Genet, 2010, 11(3): 191–203. DOI: 10.1038/nrg2732 |
| [24] | ZHANG S, QIN C X, CAO G Q, et al. Genome-wide analysis of DNA methylation profiles in a senescence-accelerated mouse prone 8 brain using whole-genome bisulfite sequencing[J]. Bioinformatics, 2017, 33(11): 1591–1595. |
| [25] | ZHANG Y L, LI F Z, FENG X, et al. Genome-wide analysis of DNA methylation profiles on sheep ovaries associated with prolificacy using whole-genome bisulfite sequencing[J]. BMC Genomics, 2017, 18(1): 759. DOI: 10.1186/s12864-017-4068-9 |
| [26] |
郭添福, 张志燕, 陈冬, 等. 不同性别大白猪肌肉全基因组高分辨率单碱基甲基化差异分析[J]. 畜牧兽医学报, 2018, 49(11): 2326–2339.
GUO T F, ZHANG Z Y, CHEN D, et al. High resolution and single base genome-wide methylation variance analysis of muscle of large white pigs with different sexes[J]. Acta Veterinaria et Zootechnica Sinica, 2018, 49(11): 2326–2339. (in Chinese) |
| [27] | GRAVINA S, DONG X, YU B, et al. Single-cell genome-wide bisulfite sequencing uncovers extensive heterogeneity in the mouse liver methylome[J]. Genome Biol, 2016, 17(1): 150. |
| [28] | LIANG Y, FU X W, LI J J, et al. DNA methylation pattern in mouse oocytes and their in vitro fertilized early embryos:effect of oocyte vitrification[J]. Zygote, 2014, 22(2): 138–145. DOI: 10.1017/S0967199412000512 |
| [29] | MA Y S, PAN B, YANG H X, et al. Expression of CD9 and CD81 in bovine germinal vesicle oocytes after vitrification followed by in vitro maturation[J]. Cryobiology, 2018, 81: 206–209. DOI: 10.1016/j.cryobiol.2018.02.011 |
| [30] | KUO Y C, HE X J, COLEMAN A J, et al. Structural analyses of FERM domain-mediated membrane localization of FARP1[J]. Sci Rep, 2018, 8(1): 10477. DOI: 10.1038/s41598-018-28692-4 |
| [31] | HAYASHI M, NAKASHIMA T, TANIGUCHI M, et al. Osteoprotection by semaphorin 3A[J]. Nature, 2012, 485(7396): 69–74. DOI: 10.1038/nature11000 |
| [32] | TAI A W, BOJJIREDDY N, BALLA T. A homogeneous and nonisotopic assay for phosphatidylinositol 4-kinases[J]. Anal Biochem, 2011, 417(1): 97–102. |
| [33] | ZHANG X Y, YANG W L, WANG J P, et al. FAM3 gene family:a promising therapeutical target for NAFLD and type 2 diabetes[J]. Metabolism, 2018, 81: 71–82. DOI: 10.1016/j.metabol.2017.12.001 |



