2. 四川省草原科学研究院, 成都 611731;
3. 西藏自治区农牧科学院 省部共建青稞和牦牛种质资源与遗传改良国家重点实验室, 拉萨 850009
2. Sichuan Academy of Grassland Sciences, Chengdu 611731, China;
3. State Key Laboratory of Hulless Barley and Yak Germplasm Resources and Genetic Improvement, Tibet Academy of Agricultural and Animal Husbandry Sciences, Lhasa 850009, China
microRNA(miRNA)是一类长约20~25个核苷酸的非编码小RNA,以互补配对的方式与靶基因转录出的mRNA结合,引导RNA诱导沉默复合物(RICS)清除mRNA[1]或抑制mRNA的翻译[2],从而影响编码蛋白基因的表达。miRNA最先在秀丽线虫中被发现[3],广泛存在于哺乳动物、果蝇和植物等生物中。miRNA基因尽管不编码蛋白,但在生物整个生命活动中发挥着重要作用,参与细胞的分裂增殖、分化、生物发育及代谢等多个生物学过程[4-6]。miRNA对肌肉发育的调节是肌肉发育调控机制研究的热点之一,越来越多的研究表明,miRNA对哺乳动物骨骼肌发育的多个环节具有调控作用,在肌肉细胞增殖和分化过程中起关键作用[7]。研究发现,miR-192通过靶向RB1 3′-UTR抑制绵羊骨骼肌卫星细胞的成肌分化,促进其增殖[8],而miR-378通过降低GRB2表达促进成肌细胞分化[9]。miRNA的表达具有时序性,在不同发育阶段哺乳动物肌肉组织miRNAs研究中,发现秦川牛出生前和出生后肌肉组织共有36个显著差异miRNAs,过表达miR-10020能够抑制Pax7基因表达,延迟早期肌细胞分化过程或在出生后肌肉组织中抑制肌卫星细胞分化[10];miR-128、miR-133a、miR-489在4个不同发育阶段的猪骨骼肌中都差异表达,miR-1通过靶向Cnn3和Sfrp1基因,调控猪骨骼肌生长发育[11];在冷季与暖季营养胁迫下牦牛背最长肌组织中差异表达microRNAs主要通过MAPK、GnRH、Wnt、PI3K-Akt和胰岛素信号通路等调控肌肉代谢,其中,miR-133b、miR-378、miR-378c、miR-29a、miR-29b以及miR-29c发挥重要的调控作用[12];有研究表明,miR-378靶向POLA2以促进牛骨骼肌衍生卫星细胞[13]。这些研究成果都充分体现了miRNA对肌肉发育有重要的调控作用。我国是牦牛主产国,牦牛因具有生产“绿色肉食品”的独特优势引起国内外的广泛关注。研究牦牛不同年龄肌肉组织microRNA表达谱和特征对于进一步揭示其在牦牛肌肉细胞增殖、分化等生长发育过程中的作用及机理具有重要意义。
本研究拟通过对牦牛幼年期到成年期肌肉组织小RNA进行测序,并进行RT-qPCR验证和生物信息学分析,挖掘调控肌肉发育的特定miRNAs,为进一步研究miRNA在牦牛生长发育中的调控机制奠定基础。
1 材料与方法 1.1 样品采集试验牛为0.5、2.5、4.5和7.5岁各3头健康的类乌齐母牦牛。对其进行屠宰后采集臀大肌样品,经DEPC冲洗后,锡箔纸包装迅速置于液氮保存,带回实验室备用。
1.2 牦牛臀大肌microRNA文库构建及Solexa测序构建12头牦牛的臀大肌测序文库:用TRIzol从样本中提取总RNA,琼脂糖凝胶电泳切胶选择18~30 nt的片段。分别连接3′接头和5′接头,然后对已连接两侧接头的small RNA进行反转录和PCR扩增。最后琼脂糖凝胶电泳回收并纯化约140 bp的条带,由此构建0.5、2.5、4.5和7.5岁牦牛肌肉小RNA文库各3个。构建好的文库使用Agilent 2100以及qPCR进行质量控制,并上机测序。
1.3 牦牛臀大肌microRNA测序数据处理与分析对上述测序获得的原始序列数据进行去接头、去低质量、去污染等过程的处理,然后统计序列总数(total)、种类(unique)和长度分布,最后得到的全部有效序列用于后续分析:1)运用blastn软件将sRNA序列与GenBank和Rfam(11.0)数据库进行比对,尽可能的发现并去除样本中可能存在的rRNA、scRNA、snoRNA、snRNA和tRNA。2)利用Bowtie 1.1.2软件与基因组比对,确定测序得到的sRNA序列位于基因组的位置,根据外显子和内含子在基因组的位置,找出来自mRNA降解片段的sRNA。3)利用RepeatMasker version open-4.0.6软件将sRNA与重复序列进行比对,得到repeat associate sRNA。4)根据miRNA在不同物种中的保守型,利用Bowtie(version 1.1.2)软件与miRBase数据库中所有物种的miRNA序列进行比对分析,鉴定出已知保守的miRNA及其含量,利用MIREAP_v0.2软件预测novel miRNA,并统计这些miRNAs各位点的碱基分布。5)利用edgeR软件对数据进行统计分析,差异miRNA的筛选标准为TPM变化2倍以上并且P < 0.05,获得差异表达miRNA。6)统计不同年龄之间肌肉组织差异表达的保守miRNA,用Venny2.1.0(http://bioinfogp.cnb.csic.es/tools/venny/index.html)软件做韦恩图,分析统计0.5、2.5、4.5岁与7.5岁肌肉组织共同差异表达的miRNAs,并进一步筛选TPM>100的miRNAs。7)用RNAhybrid(v2.1.2)+svm_light(v6.01)、Miranda(v3.3a)和TargetScan 7.0 3种方法对共同差异表达miRNA进行靶基因预测,预测结果取交集作为miRNA靶基因预测的结果,运用R语言中的clusterProfilter软件包和org.Hs.eg.db数据库,根据靶基因注释信息进行GO富集和KEGG通路分析。
1.4 牦牛臀大肌microRNA测序结果的RT-qPCR验证用于测序结果验证的样本为用于小RNA文库构建的12头母牦牛。从与7.5岁共同差异表达的miRNAs中随机选取8个miRNAs,以U6[14]作为内参基因,每个样品设置3个重复。根据这8个miRNAs的成熟序列设计反转录及荧光定量PCR引物。利用PrimeScriptTMRT逆转录试剂盒和SYBR Premix Ex TaqTMⅡ定量试剂盒,对目标miRNAs在牦牛肌肉组织中的表达进行检测。检测严格参照试剂盒说明书进行,相对表达量结果采用2-△△Ct法进行计算[15-16]。miRNA引物及内参基因序列见表 1。
|
|
表 1 荧光定量引物列表 Table 1 The primers of qRT-PCR |
经Solexa测序和生物信息学分析,去除接头序列以及低质量序列,12个文库共获得146 188 329条原始reads,经过滤后,对每个年龄段对应的3个文库求平均值,分别统计获得0.5、2.5、4.5、7.5岁4个文库的12 005 605、10 777 987、10 240 742、11 430 439条纯净序列,结果见表 2。筛选4个年龄段肌肉组织中获得的纯净序列,选取一定长度范围内的sRNA进行长度统计分布,本试验过滤掉18 bp以下及30 bp以上的reads。大多数reads分布在20~24 nt之间(图 1),其中,长度为22 nt的序列频率最高,在0.5、2.5、4.5、7.5岁4个文库中依次占42.52%、40.31%、39.74%、43.05%;其次是21 nt,在4个文库中频率依次为18.20%、19.57%、18.96%和18.15%。
|
|
表 2 数据过滤信息 Table 2 The information of raw data filtering |

|
图 1 reads长度分布情况 Fig. 1 The distribution of read lengths from Solexa sequencing data |
将所有纯净序列与GenBank、Rfam、外显子、内含子和重复序列数据库进行比对,注释情况按照优先级(rRNA等>已知miRNA>重复序列>外显子>新miRNA>内含子)的顺序对所有纯净序列进行分类,没有比对上任何注释信息的用“unann”表示。随后分别统计0.5、2.5、4.5、7.5岁4个文库中小RNA的种类及数量,这4个文库都以miRNA的数量最多,所占的比例依次为93.25%、92.36%、89.88%、93.14%;无注释信息的sRNA依次占2.63%、3.12%、4.43%、2.98%;其他种类的RNAs含量较低(图 2)。

|
图 2 牦牛0.5(A)、2.5(B)、4.5(C)和7.5岁(D)肌肉组织文库中小RNA的分类注释 Fig. 2 Annotation of small RNAs in 0.5(A), 2.5(B), 4.5(C) and 7.5 years(D) muscle tissue of yak |
由于miRBase目前还没有收录牦牛的miRNA,所以有效去除rRNA、tRNA、snoRNA、snRNA和repeat等非编码小RNAs后,将剩余序列比对到miRBase所有物种已知miRNA的成熟序列或前体序列(默认G-U配对,允许1~2个碱基错配),从而得到牦牛基因组中存在的物种间保守的miRNA,表达量前12位的miRNA如表 3所示,其表达量都在20 000以上,其中miR-1表达量最高。分析已知miRNAs各位点的碱基偏向性,0.5、2.5、4.5、7.5岁4个sRNA文库中首碱基都偏向U,频率依次为75.50%、73.11%、74.03%、71.35%;其次为A,频率依次为15.86%、17.25%、15.92%、17.34%;4个文库中的首位和末端位点处富含U碱基,第15位富含G碱基,频率依次为59.03%、56.50%、57.35%、61.28%(图 3)。
|
|
表 3 牦牛肌肉组织中表达量前12的miRNAs Table 3 The most abundantly expressed miRNAs in yak muscle tissues (Top 12) |

|
图 3 牦牛0.5(A)、2.5(B)、4.5(C)和7.5岁(D)肌肉组织文库中miRNAs片段各位点碱基的偏向性 Fig. 3 Nucleotide bias at each position of miRNAs in 0.5(A), 2.5(B), 4.5(C) and 7.5 years(D) muscle tissue of yak |
将0.5、2.5、4.5、7.5岁4个文库中获得的已知miRNA用edgeR软件进行统计分析,根据TPM值,获得4个年龄段之间差异表达miRNA(图 4)。相较于0.5岁肌肉组织,7.5岁肌肉组织中差异表达的miRNA数量最多,共126个,其中30个上调,96个下调;2.5、4.5岁肌肉组织中分别与7.5岁差异表达的miRNAs数量依次减少,分别为99个和93个;2.5与4.5岁肌肉组织中差异表达的miRNA数量最少,共14个差异表达miRNAs。随着牦牛年龄增大,肌纤维直径显著增大[17],即试验样品中,7.5岁牦牛肌纤维直径最大,依次为4.5、2.5、0.5岁,因此,运用Venny2.1.0软件获得0.5、2.5、4.5岁与7.5岁共同差异表达的miRNAs,共鉴定出58个共同差异表达miRNAs,其中5个共同在7.5岁肌肉组织中上调表达,53个下调表达(图 5),并筛选出TPM>100的miRNA,共获得10个,均在7.5岁肌肉组织中下调表达(表 4),可以看出miR-381-3p、miR-154-3p和miR-3958-3p倍数差异最大;miR-199-5p表达量最高,但倍数差异不大。

|
图 4 牦牛肌肉组织4个年龄段之间差异表达的保守miRNAs(上调和下调) Fig. 4 Conserved miRNAs differentially expressed between 4 age stages in yak muscle tissue(UP and DOWN) |

|
图 5 牦牛肌肉组织中0.5(A)、2.5(B)、4.5(C)分别与7.5岁(D)差异表达的保守miRNAs(上调和下调) Fig. 5 Conserved miRNAs differentially expressed in yak muscle tissue at 0.5(A), 2.5(B), 4.5(C) and 7.5 years(D)(UP and DOWN) |
|
|
表 4 牦牛肌肉组织0.5、2.5、4.5岁共同与7.5岁差异表达的保守miRNAs Table 4 Conserved miRNAs differentially expressed in yak muscle tissue at 0.5, 2.5, 4.5 and 7.5 years |
先去除鉴定为已存在的miRNA、已存在miRNA编辑、已知的miRNA、来自mRNA降解片段、重复序列,以及rRNA、scRNA、snoRNA、snRNA、tRNA等其它小RNA的纯净序列;挑选出能够比对上基因组的纯净序列,使用MIREAP_v0.2软件,预测miRNA的特殊二级结构,找出可能存在的新miRNA。下表列出了在肌肉组织中表达的前5个novel miRNA的相关信息(表 5和表 6)和预测的miRNA结构(图 6),结果表明预测的novel miRNA表达量都普遍较低。
|
|
表 5 牦牛肌肉组织中预测的novel miRNAs表达量 Table 5 The most abundantly expressed novel miRNAs in yak muscle tissues(top 5) |
|
|
表 6 牦牛肌肉组织中预测的novel miRNAs前体信息 Table 6 Predicted precursor sequences and genome locations of 5 novel miRNAs |

|
图 6 预测的部分novel miRNAs结构 Fig. 6 Predicted secondary structures of partial novel miRNAs |
目前应用于预测miRNA靶基因的软件很多,为避免单一的软件预测可能造成的高假阳性,本试验用RNAhybrid(v2.1.2)+svm_light(v6.01)、Miranda(v3.3a)和TargetScan(Version: 7.0) 3种方法对58个共同差异表达的miRNAs进行靶基因预测,筛选3种方法预测结果的交集,共预测出9 172个靶基因位点。为了更好地将这些基因进行归类并统计分析,运用R语言中的clusterProfilter软件包和org.Hs.eg.db数据库对预测出的靶基因进行GO富集和KEGG信号通路分析。
GO富集分析结果表明,靶基因与泛素蛋白转移酶活性、蛋白质丝氨酸/苏氨酸激酶活性、细胞粘附分子结合等分子功能相关(图 7);KEGG通路分析共发掘出60个通路,图 8中仅列出前30个可能作用于肌肉组织的通路,根据信号通路中包含的靶基因数量及可能的生物学意义,在这些通路当中,PI3K-Akt、MAPK、Ras信号通路及肌动蛋白细胞骨架的调控通路(regulation of actin cytoskeleton)可能参与调节牦牛肌肉细胞的增殖与分化,4个通路分别包含预测出的197、184、144及129个靶基因。肌动蛋白细胞骨架参与细胞的收缩、黏着斑的形成、细胞外基质的重构以及相关基因转录与翻译的调控,促进成肌纤维细胞的分化及组织的纤维化过程。在regulation of actin cytoskeleton通路中(图 9),发现许多基因和Focal adhesion信号通路、MAPK信号通路相关,Rac(蛋白激酶B,又称PKB或Akt)是调控肌肉发育的关键蛋白,且同时作用于PI3K-Akt、MAPK、Ras信号通路,这3个信号通路共同靶向53个基因,包括成纤维细胞生长因子(FGF)及受体(FGFR)、胰岛素生长因子(IGF)及受体(INSR)、AKT、MAP2K1等重要的靶基因,表明这58个差异miRNAs可能主要通过这3个信号通路调控肌纤维的形成。

|
图 7 牦牛肌肉组织中0.5、2.5、4.5岁与7.5岁共同差异表达miRNA靶基因GO分析 Fig. 7 GO analysis of common differentially expressed miRNA target genes in yak muscle tissue at the age of 0.5, 2.5, 4.5 and 7.5 years |

|
图 8 牦牛肌肉组织中0.5、2.5、4.5与7.5岁共同差异表达miRNA靶基因通路分析 Fig. 8 Pathway analysis of predicted target genes of common differentially expressed miRNA in yak muscle tissue at the age of 0.5, 2.5, 4.5 and 7.5 years |

|
图 9 肌动蛋白细胞骨架调节通路 Fig. 9 Regulation pathway of actin cytoskeleton |
在0.5、2.5、4.5岁共同与7.5岁差异表达的58个miRNAs中随机选择8个miRNAs进行验证,8个miRNAs在牦牛4个年龄段肌肉组织中的相对表达量列于图 10A。这些miRNAs在牦牛肌肉组织中都有表达,除miR-382-5P外,不同年龄间的差异表达趋势与Solexa测序结果(图 10B)一致,测序结果显示,miR-382-5P在2.5岁肌肉组织中表达高于4.5岁,而验证结果与之相反。
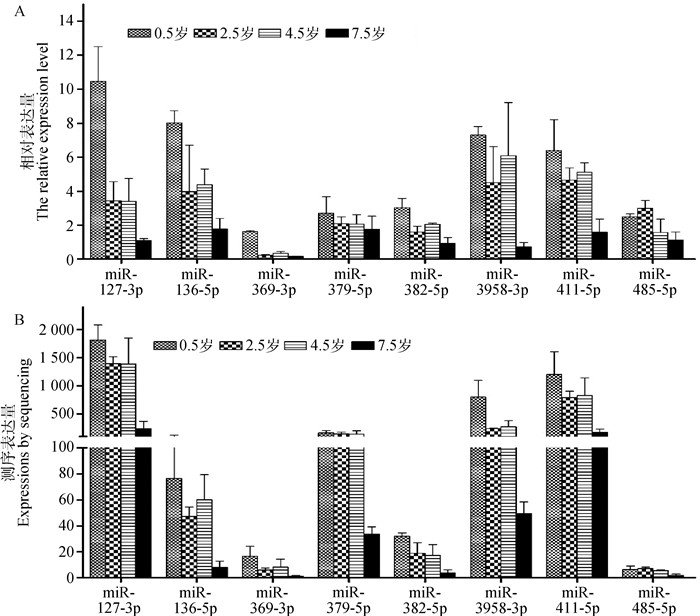
|
图 10 随机选取的8个差异表达miRNAs在牦牛不同年龄肌肉组织中的定量表达量(A)和测序表达量(B) Fig. 10 Expressions by quantification(A) and sequencing(B) of 8 randomly selected differentially expressed miRNAs at different ages of yak muscle tissue |
目前,高通量测序技术已广泛应用于家畜小RNA组学研究[17-19]。近年来通过高通量测序在牛、羊、猪等动物肌肉组织中挖掘到大量的新miRNAs,并鉴定出差异性、特异性表达的miRNAs[20-22],关于其功能及作用机制的研究仍在不断扩展。年龄对牦牛肌纤维直径的影响显著,随着牦牛年龄增大,肌纤维直径显著增大[15]。本研究利用高通量测序技术对0.5、2.5、4.5和7.5岁牦牛肌肉组织中的miRNA进行测序,对8个随机选择的miRNAs进行RT-qPCR验证,结果表明,有7个miRNAs的表达与测序结果一致,这表明测序结果具有较高的准确性和可靠性。miR-382-5p验证结果显示,在4.5岁肌肉组织中的表达量高于2.5岁,与测序结果相反,这种不一致在其他研究中也有报道,可能原因是测定方法不同而产生的差异[23-24]。本研究中,4个sRNA文库中已知miRNA的首位碱基都偏向U,与前人研究中5′端碱基为U有利于miRNA与Agol蛋白结合形成RNA诱导沉默复合体,进而有利于该复合体切割靶标基因的结果一致。miRNA碱基的分布可能影响miRNA的作用机制,对靶基因的调控有关[25]。
从鉴别出的保守miRNAs中获得各年龄段之间差异表达的miRNA,筛选出0.5、2.5、4.5岁共同与7.5岁差异表达的58个miRNAs,其中5个在7.5岁肌肉组织中上调表达,53个下调表达,在0.5岁时表达量最高,其中miR-199-5p表达量最高。53个miRNAs在牦牛幼年期-青年期-成年期肌肉组织中的表达量依次下降,而牦牛幼年期-青年期-成年期肌肉组织肌纤维直径逐渐增大,表明牦牛肌肉组织中53个miRNAs表达量与肌纤维直径呈负相关,其中表达量较高的miR-199-5p和miR-127-3p具有延迟肌肉分化和负调控肌细胞增殖的作用[26-27],推测这53个miRNAs可能对肌纤维增大共同发挥抑制作用,负调控牦牛肌肉发育过程。
Ras/MAPK与PI3K/Akt信号转导通路可以相互影响,在大多数细胞中,PI3K正向调节Ras/MAPK一系列蛋白激酶的级联反应,PI3K可加剧Ras的活化导致MAPK传导信号上调,促进诱导ERK激活,而激活的ERK反过来负向控制PI3K/Akt通路,在细胞的分化、增殖、凋亡等生物学过程中具有重要的调节作用[28-29]。本研究对0.5、2.5、4.5岁共同与7.5岁差异表达的58个miRNAs进行靶基因预测及功能分析,发现肌肉发育与PI3K-Akt、MAPK、Ras信号通路及肌动蛋白细胞骨架调节通路均有关。前人研究发现,Ras单链DNA与Ras蛋白结合可抑制Ras和MEK1/2、ERK1/2磷酸化,中断PI3K/Akt途径的信号转导,从而抑制血管平滑肌细胞的增殖和迁移[30],PI3K-Akt、MAPK、Ras 3个信号通路共同靶向53个靶基因,表明在肌肉组织中这3个信号通路相互关联影响肌细胞增殖。MAPK通路参与调控牛胎儿骨骼肌成肌细胞分化过程,MAPK能够激活肌源性转录因子MyoD和MEF2家族成员的活性,促进成肌细胞分化[31-32],表明MAPK通路在肌肉发育过程中具有重要调控功能,本研究以牦牛幼年期-青年期-成年期肌肉组织为研究对象,发现MAPK通路与PI3K-Akt通路、Ras通路联系密切,Akt同时作用于3个信号通路,有研究报道,急性激活Akt可快速诱导小鼠体内骨骼肌显著肥大,且伴随脂肪组织的显著减少[33],表明Akt在哺乳动物肌肉发育过程中通过3个关联的信号通路发挥重要的调控作用。
关于miRNA与3个信号通路在肌肉组织中的研究发现,miR-432和miR-138分别通过靶向降低PI3K/Akt信号通路中P55PIK和PDK1的表达,从而抑制肌细胞的增殖[34-35];miR-133b通过抑制MAPK信号通路中ERK1/2的磷酸化负面调节肌细胞增殖[36]。结合本研究结果说明,miRNAs介导的Ras/MAPK与PI3K/Akt信号转导通路在牦牛肌肉发育过程中有重要调控作用。已有研究发现,miR-199-5p通过PI3K/Akt途径降低DDR1表达从而抑制癌细胞的迁移和侵袭[37],推测在牦牛不同年龄肌肉组织中,miR-199-5p等58个差异表达miRNAs通过靶向关联Ras/MAPK与PI3K/Akt信号通路的53个信号基因,阻碍3个信号通路之间信号的传导,尤其是Akt蛋白激酶的活化,从而抑制肌细胞增殖和肌肉分化,负调控不同年龄段牦牛肌肉的发育,这仍需要进一步研究。
4 结论本研究通过高通量测序及生物信息学分析获得牦牛不同年龄肌肉组织miRNA表达谱,筛选出肌肉组织中0.5、2.5、4.5岁共同与7.5岁差异表达的58个miRNAs;靶基因预测和功能分析表明,信号通路涉及PI3K/Ak、MAPK、Ras、肌动蛋白细胞骨架调节等调控肌肉发育的多种途径,其中FGF、FGFR、IGF、INSR、AKT、MAP2K1等是重要的靶基因。这为进一步鉴定调控肌肉发育的miRNA,探究其功能及作用机制奠定了理论基础。
| [1] | XIE Z X, KASSCHAU K D, CARRINGTON J C. Negative feedback regulation of Dicer-Like1 in Arabidopsis by microRNA-guided mRNA degradation[J]. Curr Biol, 2003, 13(9): 784–789. DOI: 10.1016/S0960-9822(03)00281-1 |
| [2] | THERMANN R, HENTZE M W. Drosophila miR2 induces pseudo-polysomes and inhibits translation initiation[J]. Nature, 2012, 447(7146): 875–878. |
| [3] | LEE R C, FEINBAUM R L, AMBROS V. The C.elegans heterochronic gene lin-4 encodes small RNAs with antisense complementarity to lin-14[J]. Cell, 1993, 75(5): 843–854. DOI: 10.1016/0092-8674(93)90529-Y |
| [4] |
徐盛玉, 王定越, 吴德. microRNA及其对哺乳动物繁殖的影响[J]. 畜牧兽医学报, 2011, 42(6): 747–753.
XU S Y, WANG D Y, WU D. Effects of microRNA on the mammalian reproduction[J]. Acta Veterinaria et Zootechnica Sinica, 2011, 42(6): 747–753. (in Chinese) |
| [5] |
杨雪梅, 顾以韧, 梁艳, 等. miR-1、miR-27a、miR-369和miR-378在7个地方猪种背最长肌中的表达与肉质性状的关系研究[J]. 中国畜牧杂志, 2018, 54(2): 30–34.
YANG X M, GU Y R, LIANG Y, et al. Relationship between expression of miR-1, miR-27a, miR-369, miR-378 and meat quality traits in the longissimus dorsi muscle of 7 local pig breeds[J]. Chinese Journal of Animal Science, 2018, 54(2): 30–34. (in Chinese) |
| [6] | MA T H, CHENG L, WANG B, et al. miR-378 induces apoptsis of granulosa cells during follicle development in cattle[J]. J Anim Plant Sci, 2017, 27(5): 1738–1742. |
| [7] |
李新云, 付亮亮, 程会军, 等. microRNA调控哺乳动物骨骼肌发育[J]. 遗传, 2017, 39(11): 1046–1053.
LI X Y, FU L L, CHENG H J, et al. Advances on microRNA in regulating mammalian skeletal muscle development[J]. Hereditas, 2017, 39(11): 1046–1053. (in Chinese) |
| [8] |
赵谦.miR-192在绵羊骨骼肌卫星细胞增殖和成肌分化中的功能研究[D].北京: 中国农业科学院, 2016.
ZHAO Q.The role of miR-192 in proliferation and myogenic differentiation of sheep skeletal muscle satellite cells[D].Beijing: Chinese Academy of Agricultural Sciences, 2016.(in Chinese) http://cdmd.cnki.com.cn/Article/CDMD-82101-1016174599.htm |
| [9] |
侯欣华.猪骨骼肌相关miRNA的鉴定及miR-378对肌肉发育调控的研究[D].南京: 南京农业大学, 2011.
HOU X H.Identification of porcine skeletal muscle related miRNA and regulation role of miR-378 in myogenesis[D].Nanjing: Nanjing Agricultural University, 2011.(in Chinese) http://cdmd.cnki.com.cn/Article/CDMD-10307-1016040412.htm |
| [10] |
孙加节.秦川牛肌肉与脂肪组织发育相关miRNA鉴定及miR-10020调控机制解析[D].杨凌: 西北农林科技大学, 2015.
SUN J J.Identification of microRNAs from Chinese Qinchuan bovine muscle and adipose tissues and functional analysis of miR-10020 during myogenesis[D].Yangling: Northwest A&F University, 2015.(in Chinese) |
| [11] | WANG Q, QI R L, WANG J, et al. Differential expression profile of miRNAs in porcine muscle and adipose tissue during development[J]. Gene, 2017, 618: 49–56. DOI: 10.1016/j.gene.2017.04.013 |
| [12] |
褚敏.营养胁迫条件下牦牛皮下脂肪和背肌差异microRNAs的筛选与鉴定[D].兰州: 中国农业科学院, 2015.
CHU M.Identification and analysis of differential expressed microRNAs in subcutaneous and longissimus muscle of yak under nutrition pressure[D].Lanzhou: Chinese Academy of Agricultural Sciences, 2015.(in Chinese) http://cdmd.cnki.com.cn/Article/CDMD-82101-1016174758.htm |
| [13] | TONG H L, JIANG R Y, LIU T T, et al. bta-miR-378 promote the differentiation of bovine skeletal muscle-derived satellite cells[J]. Gene, 2018, 668: 246–251. DOI: 10.1016/j.gene.2018.03.102 |
| [14] |
张阳阳.miR-378在牛前体脂肪细胞分化的作用与机制[D].长春: 吉林大学, 2014.
ZHANG Y Y.Effect and mechanism of bovine miR-378 in preadipocyte differentiation[D].Changchun: Jilin University, 2014.(in Chinese) http://cdmd.cnki.com.cn/Article/CDMD-10183-1014281818.htm |
| [15] | DEO A, CARLSSON J, LINDLÖF A. How to choose a normalization strategy for miRNA quantitative real-time (qPCR) arrays[J]. J Bioinform Comput Biol, 2011, 9(6): 795–812. DOI: 10.1142/S0219720011005793 |
| [16] |
习欠云, 周莲莲, 李虹仪, 等. 不同品种猪肌肉组织miR-1和miR-133基因的表达分析[J]. 畜牧兽医学报, 2012, 43(6): 843–848.
XI Q Y, ZHOU L L, LI H Y, et al. The expression analysis of miR-1 and miR-133 in Longissimus dorsi muscle from different pig breeds[J]. Acta Veterinaria et Zootechnica Sinica, 2012, 43(6): 843–848. (in Chinese) |
| [17] |
徐瑛, 文鹏程, 梁琪, 等. 不同岁龄牦牛肉肉用品质的变化规律[J]. 食品工业科技, 2014, 35(20): 121–124.
XU Y, WEN P C, LIANG Q, et al. Changing cule of meat quality of yak in different ages[J]. Science and Technology of Food Industry, 2014, 35(20): 121–124. (in Chinese) |
| [18] |
熊显荣, 兰道亮, 李键, 等. 牦牛卵巢小RNA高通量测序及生物信息学分析[J]. 畜牧兽医学报, 2016, 47(1): 55–63.
XIONG X R, LAN D L, LI J, et al. Solexa sequencing of small RNAs in yak (Bos grunniens) ovaries and bioinformatics analysis[J]. Acta Veterinaria et Zootechnica Sinica, 2016, 47(1): 55–63. (in Chinese) |
| [19] | ZHOU Y, TANG X, SONG Q, et al. Identification and characterization of pig embryo microRNAs by Solexa sequencing[J]. Reprod Domest Anim, 2013, 48(1): 112–120. DOI: 10.1111/rda.2013.48.issue-1 |
| [20] |
凌英会, 张晓东, 王丽娟, 等. 山羊肌肉组织microRNA Solexa测序与生物信息学分析[J]. 畜牧兽医学报, 2013, 44(3): 481–487.
LING Y H, ZHANG X D, WANG L J, et al. Solexa sequencing and bioinformatics analysis on microRNA from the goat muscle[J]. Acta Veterinaria et Zootechnica Sinica, 2013, 44(3): 481–487. (in Chinese) |
| [21] |
张翔宇, 邝良德, 李丛艳, 等. 家兔肌肉组织miRNA Solexa测序与生物信息学分析[J]. 湖北农业科学, 2018, 57(16): 97–101.
ZHANG X Y, KUANG L D, LI C Y, et al. Solexa sequencing and bioinformatics analysis on miRNA from rabbit muscle[J]. Hubei Agricultural Sciences, 2018, 57(16): 97–101. (in Chinese) |
| [22] | HOU X H, YANG Y L, ZHU S Y, et al. Comparison of skeletal muscle miRNA and mRNA profiles among three pig breeds[J]. Mol Genet Genomics, 2016, 291(2): 559–573. DOI: 10.1007/s00438-015-1126-3 |
| [23] |
陈晨, 柴进, 蒋思文.Solexa测序鉴定大白猪和梅山猪背部脂肪组织中的microRNAs[C]//第十六次全国动物遗传育种学术讨论会暨纪念吴仲贤先生诞辰100周年大会论文集.扬州: 中国畜牧兽医学会, 2011.
CHEN C, CHAI J, JIANG S W.Identification of microRNAs in the adipose tissue of the Large White and Meishan pigs by Solexa sequencing[C]//The 16th National Symposium on Animal Genetics and Breeding and the 100th Anniversary of the Birth of Mr.Wu Zhongxian.Yangzhou: Chinese Association of Animal Science and Veterinary Medicine, 2011.(in Chinese) |
| [24] |
张方, 胡子乔, 景炅婕, 等. 绵羊不同部位脂肪组织microRNA高通量测序及生物信息学分析[J]. 畜牧兽医学报, 2016, 47(6): 1093–1101.
ZHANG F, HU Z Q, JING J J, et al. High-throughput sequencing and bioinformatics analysis on microRNAs expressed in adipose tissues of sheep[J]. Acta Veterinaria et Zootechnica Sinica, 2016, 47(6): 1093–1101. (in Chinese) |
| [25] | MI S J, CAI T, HU Y G, et al. Sorting of small RNAs into Arabidopsis argonaute complexes is directed by the 5' terminal nucleotide[J]. Cell, 2008, 133(1): 116–127. DOI: 10.1016/j.cell.2008.02.034 |
| [26] | CHEN X.Molecular mechanism of muscle growth of broiler chicken: a role of miR-199-5p in regulating differentiation of skeletal myoblasts[D].Nanjing: Nanjing University, 2015. |
| [27] | YUAN R Q, ZHANG X M, FANG Y, et al. mir-127-3p inhibits the proliferation of myocytes by targeting KMT5a[J]. Biochem Biophys Res Commun, 2018, 503(2): 970–976. DOI: 10.1016/j.bbrc.2018.06.104 |
| [28] | AKSAMITIENE E, KIYATKIN A, KHOLODENKO B N. Cross-talk between mitogenic Ras/MAPK and survival PI3K/Akt pathways:a fine balance[J]. Biochem Soc Trans, 2012, 40(1): 139–146. DOI: 10.1042/BST20110609 |
| [29] |
袁向飞, 陆敏. Ras/MAPK与PI3K/Akt信号转导通路及其相互作用[J]. 国际检验医学杂志, 2006, 27(3): 261–263.
YUAN X F, LU M. Ras/MAPK and PI3K/Akt signaling pathway and their interaction[J]. International Journal of Laboratory Medicine, 2006, 27(3): 261–263. DOI: 10.3969/j.issn.1673-4130.2006.03.025 (in Chinese) |
| [30] | HU X P, WANG Z W, WU H B, et al. Ras ssDNA aptamer inhibits vascular smooth muscle cell proliferation and migration through MAPK and PI3K pathways[J]. Int J Mol Med, 2015, 35(5): 1355–1361. |
| [31] |
邢义珅, 胡鑫, 任玲, 等. 牛胎儿骨骼肌来源成肌细胞分化相关microRNA的筛查与鉴定[J]. 畜牧兽医学报, 2018, 49(6): 1134–1144.
XING Y S, HU X, REN L, et al. Identification of micrornas involved in myogenic differentiation of bovine fetal skeletal muscle derived myoblasts[J]. Acta Veterinaria et Zootechnica Sinica, 2018, 49(6): 1134–1144. (in Chinese) |
| [32] | KEREN A, TAMIR Y, BENGAL E. The p38 MAPK signaling pathway:a major regulator of skeletal muscle development[J]. Mol Cell Endocrinol, 2006, 252(1-2): 224–230. DOI: 10.1016/j.mce.2006.03.017 |
| [33] | LAI K M V, GONZALEZ M, POUEYMIROU W T, et al. Conditional activation of akt in adult skeletal muscle induces rapid hypertrophy[J]. Mol Cell Biol, 2004, 24(21): 9295–9304. DOI: 10.1128/MCB.24.21.9295-9304.2004 |
| [34] | MA M L, WANG X M, CHEN X C, et al. microRNA-432 targeting E2F3 and P55PIK inhibits myogenesis through PI3K/AKT/mTOR signaling pathway[J]. RNA Biol, 2017, 14(3): 347–360. DOI: 10.1080/15476286.2017.1279786 |
| [35] | LIU Y, YANG K Z, SUN X Z, et al. miR-138 suppresses airway smooth muscle cell proliferation through the PI3K/AKT signaling pathway by targeting PDK1[J]. Exp Lung Res, 2015, 41(7): 363–369. DOI: 10.3109/01902148.2015.1041581 |
| [36] |
冯阳.MiR-133b, miR-214和miR-495通过MAPK信号通路调节成肌细胞增殖和分化的研究[D].武汉: 华中农业大学, 2011.
FENG Y.Regulation of myoblasts proliferation and differentiation by miR-133b, miR-214 and miR-495 through MAPK signaling[D].Wuhan: Huazhong Agricultural University, 2011.(in Chinese) http://cdmd.cnki.com.cn/Article/CDMD-10504-1011405185.htm |
| [37] | PALLADINO C, MATÀ R, NICOLOSI M L, et al. Abstract 4612:In breast cancer cells IGF-I induces upregulation of DDR1 by suppressing miR-199a-5p via the PI3K/Akt pathway[J]. Cancer Res, 2016, 76(S14): 4612. |



