病毒性腹泻已成为世界各地养猪业的常见疾病之一,尤其在仔猪中具有较高的发病率。冠状病毒(特别是PEDV)、杯状病毒、星状病毒等是仔猪腹泻的常见重要病原体。冠状病毒感染人、鼠、猪、鸟等脊椎类动物,具有胃肠、呼吸道、肝和神经系统的嗜性[1]。近年来,冠状病毒感染人和动物导致疾病流行的事件屡有发生,2002—2003年期间,我国暴发的非典疫情即由SARS-CoV引起[2-4]。2012年9月,沙特阿拉伯王国首先出现由MERS-CoV引起的中东呼吸综合征(MERS)病例,继而在中东其他国家及欧洲蔓延[5]。2015年5月29日,我国出现首例输入性MERS确诊病例[6]。这两种高死亡率的新发传染病的出现,引起了人们对冠状病毒的高度重视[7-12]。
冠状病毒属于尼多病毒目冠状病毒科,病毒粒子呈球形或不规则形,有囊膜,大小为80~120 nm。其基因组为单股正链RNA,大小为27~32 kb,5′端带有帽子结构,其后包含6~10个开放阅读框(open reading frames,ORFs),3′端有polyA尾[1, 13-16]。在所有冠状病毒的全基因组中,编码复制酶以及纤突蛋白(S)、小包膜蛋白(E)、囊膜蛋白(M)和核蛋白(N)4种结构蛋白的基因序列均相同,即5′-ORF1ab-S-E-M-N-3′,编码辅助蛋白的基因散布在结构蛋白基因之间[17]。根据基因组的特点,目前将冠状病毒分为4个属,即α、β、γ和δ[18]。α冠状病毒包括猪流行性腹泻(PEDV)、人冠状病毒NL63(HCoV-NL63)、犬冠状病毒(CCoV)以及传染性肠胃炎病毒(TGEV)等;β冠状病毒包括SARS、MERS、鼠肝炎病毒(MHV)等;γ冠状病毒包括鸡传染性支气管炎(IBV);δ冠状病毒包括鹦鹉冠状病毒(PaCoV)、猪δ冠状病毒(PDCoV)等[18-19]。
近年来基于高通量测序的病毒宏基因组学方法在对自然环境、人和动物的病毒组学分析上已经成为一个高效的工具[20-23]。为了解上海周边地区腹泻仔猪肠道病毒组成情况,特别是从2010年秋季开始,对我国养猪业构成巨大威胁的α冠状病毒中的PEDV[24]的感染情况以及基因型变异情况,本研究对来自上海周边地区6个猪场的90份腹泻仔猪粪便样本进行检测,将为仔猪腹泻的防治提供一定的参考依据[25]。
1 材料与方法 1.1 样品采集从上海周边地区6所猪场采集腹泻仔猪粪便样本,各猪场的样品相关信息见表 1。采集过程均使用一次性用具以避免样本交叉污染。粪便样本用无钙、镁离子的DPBS制备成约20%的悬液,充分混匀后,冻存于-80 ℃。
|
|
表 1 各猪场粪便样本信息 Table 1 Information of stool sampled from different pig farms |
PCR产物纯化试剂盒(Qiaquick PCR purification kit)、病毒核酸提取试剂盒(QIAampMinElute Virus Spin Kit)购自Qiagen公司;逆转录试剂盒(SuperScript Ⅲ reverse transcriptase)购自Life technologie公司;(高通量测序)DNA文库构建试剂盒(Nextera XT DNA Sample Preparation Kit)、DNA文库接头添加试剂盒(Nextera XT Index Kit)购自Illumina公司;核酸纯化试剂盒(AgencourtAMPure XP Kit)购自Beckman公司;核酸消化酶(DNA和RNA消化酶)购自Life Technologie公司;离心管(RNase-free)、一次性手套、移液枪枪头等常用试剂耗材购自上海生工公司以及扬州博瑞公司等。
1.3 样品制备 1.3.1 粪样中病毒基因组提取每份样本加入0.8~1.0 mL无钙、镁离子的DPBS溶液,涡旋振荡,在4 ℃静置5 min;混合液于4 ℃、12 000 r·min-1离心5 min;取500 μL上清液以相同条件再次离心;再取上清液用0.45 μm Spin Column滤器(Mil-lipore)过滤小型颗粒物和固体漂浮物;每166 μL的滤过液添加20 μL 10×Buffer Cocktail、7 μL Cocktail of DNases、5 μL Micrococcal Nuclease以及2 μL RNase A在37 ℃消化60~90 min;随后用QIAamp MinElute Virus Spin Kit提取核酸(包括RNA和DNA在内)。
1.3.2 基因组RNA反转录及双链DNA合成为了保证后续试验的稳定进行,对获取RNA样本进行随机反转录,取获得的RNA 12 μL,dNTP(10 nmol·L-1)1 μL,Random 6I primer(100 μmol·L-1)1 μL,涡旋混匀后离心,再置入PRC仪中65 ℃反应5 min,移至冰上,在冰浴中加入以下试剂:Buffer Cocktail 4 μL,SuperScript Ⅲ RT Enzyme Mix I 1 μL,DTT 1 μL,此时总体系为20 μL,涡旋混匀瞬时离心,反转录条件如下:25 ℃,10 min;50 ℃,60 min;85 ℃,5 min;95 ℃,2 min,转移至冰浴冷却。随后进行Klenow反应合成双链:向反转录产物中加入1 μL Klenow酶,混匀,瞬时离心5 s,进行PCR反应,反应条件如下:37 ℃,60 min,75 ℃,20 min,10 ℃保存。
1.3.3 深度测序文库构建和扩增双链DNA合成后可直接用于深度测序DNA文库的构建,取出制备的双链DNA、ATM试剂、TD试剂、NPM试剂置于冰上融化;取出NT buffer,Illumina Index primer置于室温,融化后涡旋振荡瞬时离心置于冰上待用,取15 μL (每样品用量)的NPM buffer到新试管,取PCR管,加入如下试剂:TD buffer 10 μL,ATM 5 μL,制备好的双链DNA 5 μL,混匀,300 r·min-1离心1 min,再置于PCR仪中,55 ℃反应5 min,温度下降到10 ℃时,迅速加入5 μL NT buffer混匀,300 r·min-1离心1 min,取新的PCR管,每管加入5 μL N primer和5 μL S primer混合成的Illumina index primer,再加入15 μL NPM buffer,摇匀100 r·min-1离心1 min,再向混合液中加入4 μL之前制备好待用的核酸混合液,摇匀,300 r·min-1离心1 min,PCR反应,条件如下:72 ℃,3 min;95 ℃,30 s;扩增反应程序为94 ℃变性10 s,55 ℃退火30 s,72 ℃延伸30 s进行15~16个循环;72 ℃,5 min;4 ℃保存。
1.3.4 Misaeq文库深度测序、质控及组装用Qiaquick PCR purification kit产物纯化试剂盒除去文库中的引物二聚体和用Ampure Beads对产物片段大小进行筛选优化,以保证基因片段满足Miseq测序平台的300~800 bp之间的要求,分别从符合测序要求的文库里取出1 μL DNA核酸混合液,调配成浓度为4 nmol·L-1的DNA溶液,送到上海派森诺公司的Illumina Miseq测序平台进行深度测序;测序要求为Miseq 2×250 bp。输出数据格式设置为FASTAQ。Illumina高通量测序结束后,DNA序列拼接、质控由加州大学旧金山分校协助完成。测序结果表明基因组测序质量较好,具有研究意义,对原始测序数据进行质控,剪切adapter,修剪低质量reads末端和含N比例较高的reads(>10%),去除小片段。
2 结果 2.1 Illumina测序结果概况通过高通量测序,获得了3 402 918条可注释到病毒的序列,共计注释到70个病毒科,其中有257 525条序列被定义为疑似病毒序列但未被分类。在这70个科中,hits数大于20(视为相关病毒呈阳性)的有46个病毒科,笔者挑取这46个病毒科进行比对分析。其中,69.77%(2 194 335/3 145 264)的序列与脊椎动物病毒相关,包括小RNA病毒科(Picornaviridae)、星状病毒科(Astroviridae)、杯状病毒科(Caliciviridae)、细小病毒科(Parvoviridae)、圆环病毒科(Circoviridae)、冠状病毒科(Coronaviridae)以及小双节RNA病毒科(Picobirnaviridae)等17个病毒科;昆虫病毒相关序列占到了0.40%(12 531/3 145 264),包括双顺反子病毒科(Dicistroviridae)、传染性软化症病毒科(Iflaviridae)和肥大唾液腺病毒科(Hytrosaviridae)等6个病毒科;植物病毒序列占0.25%(7 989/3 145 264),包括豇豆花叶病毒科(Tymoviridae)、矮缩病毒科(Nanoviridae)、番茄丛矮病毒科(Tombusviridae)、帚状病毒科(Virgaviridae)和伴生豇豆病毒科(Secoviridae)等10个病毒科;另外,还有29.58%(930 409/3 145 264)的序列与噬菌体相关,包括微小噬菌体科(Microviridae)、轻小噬菌体科(Leviviridae)等13个科(图 1)。
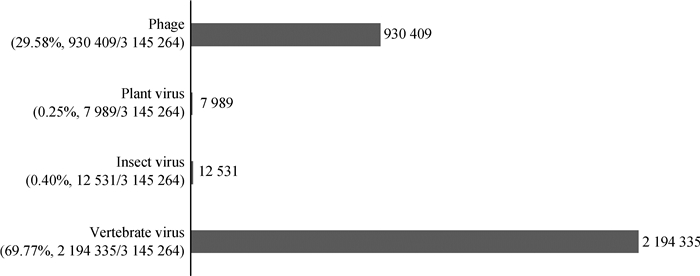
|
图 1 Illumina高通量测序获得的重叠序列注释 Fig. 1 Annotation of Illuminac ontigs |
对数据分析发现,除去噬菌体外,腹泻仔猪中的病毒主要是由小RNA病毒科主导,冠状病毒在总病毒序列中占比0.51%(图 2)。
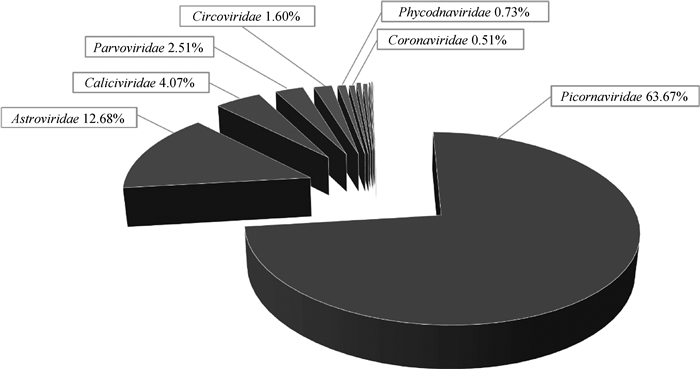
|
图 2 仔猪粪便文库中病毒组成情况 Fig. 2 Composition of virus communities in 90 piglets's feces libraries |
通过分析还发现,6个猪场中,星状病毒(PAstV)样本阳性率为53.33%(48/90),杯状病毒(PECV)的样本阳性率为32.22%(29/90)。所有的样本中,PAstV和PECV同时呈阳性的样本有22个,共感染率为24.44%(22/90)。在PAstV阳性样本发现,主要以PAstV-2和PAstV-4混合感染为主。
2.2 冠状病毒阳性样本数据分析6个猪场中,冠状病毒阳性样本率为15.56%(14/90)。14份冠状病毒阳性样本中,α冠状病毒阳性率为92.86%(13/14),且均为PEDV感染;余下的一份为Bat_Hp-betacoronavirus,属于β冠状病毒(表 2)。
|
|
表 2 不同仔猪的冠状病毒感染情况 Table 2 The infection of Coronaviridae in different piglets |
对这14个冠状病毒感染呈阳性的样本进行整合分析,发现它们的主要病毒群落组成如下:小RNA病毒科61.58%;星状病毒科11.57%;藻类去氧核糖核酸病毒科8.54%;冠状病毒科6.36%;双顺反子病毒科3.58%等(图 3)。
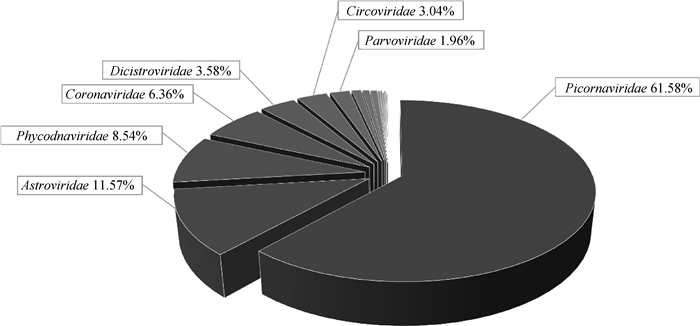
|
图 3 14个冠状病毒阳性文库中病毒群落组成 Fig. 3 Composition of virus community in 14 Coronaviridae positive feces libraries |
综合分析表 1各猪场粪便信息和表 2检出结果,13份PEDV感染阳性的腹泻仔猪均来源于C猪场,而β冠状病毒阳性的样本来源于F猪场。其中,因检测到的β冠状病毒为蝙蝠冠状病毒,推测仔猪仅为其中间宿主,并不具有持续致病性,因此暂不关注。而C猪场中,PEDV感染阳性率为43.33%(13/30)。
2.3 PEDV ORF3基因遗传进化分析利用Geneious软件对阳性样品的测序数据进行拼接,获得了全长为675 bp的ORF3全基因序列。根据测序结果,总共获得4个完整ORF3序列读长,来自于4个不同文库的PEDV ORF3基因序列根据分析结果显示,76和77号文库的毒株聚类为同一株,而来源于73号和91号的序列为分别为不同的毒株,三株毒株之间的ORF3的核苷酸序列相似性为97.04%~98.22%,氨基酸相似性为99.11%~99.55%。
参照16个GenBank中登陆的国内外PEDV流行株和经典株的ORF3基因,分析本研究获得的2个ORF3基因的同源性和遗传进化地位。结果显示,4个ORF3基因序列与G1型经典毒株和疫苗株的ORF3基因核苷酸相似性为95.11%~99.41%,与G2型当代流行毒株的ORF3基因核苷酸相似性为96.44%~99.70%(图 4)。本研究获得的三株流行株中,76和77号流行株属于G2亚型,73和91号两个流行株位于G1型。本研究的15株参考株序列分别为10株中国野毒株、2株韩国流行株,2株美国流行株和1株法国流行株。

|
图 4 3株PEDV分离株的ORF3基因与参考株ORF3基因进化关系分析 Fig. 4 The phylogenetic tree based on ORF3 of 3 PEDV isolates sequences with the reference strains |
西北农林科技大学几位学者的研究证实,PEDV ORF3基因序列在病毒进化史上相对而言比较保守[25]。PEDV根据基因型共分为三种毒株,分别为经典毒株、弱毒毒株以及流行毒株,且每种毒株均有各自典型的分子生物学特性[26]。本研究通过PEDV ORF3基因检测分析表明,在PEDV阳性的C猪场中,G1基因型和G2基因型均能检测到阳性结果。
2.4 ORF3基因分子特性G1基因型包含4株,分别是1株韩国经典株(SM98-5P)和1株韩国弱毒株(DR13),1株欧洲经典株(Br1/87),1株中国经典株(LZC)。本研究测序获得的两个ORF3基因序列,与韩国经典株分支DR13(序列号EU054929)距离较近。G2基因型包含10株中国流行株,2株美国流行株。10株中国流行株里主要流行区域位于中国南部地区,其中各有2株位于上海市内(SH1和PF5),2株位于江苏省(PEDV-LYG和JS-HZ2012),1株位于浙江省(CH/ZJCX-1/2012),1株位于广东省(CHYJ130330),1株位于福建省(CH/FJND-2/2011),1株位于河北省(HeBXTE),1株位于河南省(CH/ZMDZY/11),1株位于山东省(SD/QD/2015)。本研究测序获得的piglet76和piglet77号ORF3基因序列就属于山东株SD/QD/2015(序列号KU641638)分支。在C猪场分离出的3株PEDV ORF3基因均由编码224个氨基酸的675个核苷酸组成。在基因编码过程中,特定位点的核苷酸变化会导致特定的氨基酸变化。
选取本研究测序获得3个ORF3基因序列与传统CV777株ORF3进行序列比对(表 3)。发现与传统CV777株相比,除F2S以外,3株试验所得株存在一致性突变,分别是V21A、I70M、V79I、F80V、L85I、L92F,这一特性,有可能是PEDV传统株与流行株基因型的鉴别依据。这些位点的突变是否影响了ORF3蛋白的结构,改变了其生物学功能还不得而知,需要将来进一步探究。
|
|
表 3 ORF3基因特定位点的核苷酸突变引起对应位点的氨基酸变化 Table 3 Specific nucleotide changes resulted amino acid changes in ORF3 gene |
病毒宏基因组学旨在挖掘特定环境中的病毒群落,已经在污水处理、海洋、葡萄种植、人类粪便、人肠道、蝙蝠粪便和动物组织等特定的环境中得到应用[27-33]。该方法应用随机引物合成双链cDNA和PCR扩增病毒遗传物质,避免了传统方法的限制[34]。在养猪业中,病毒性腹泻是引起家养猪腹泻甚至死亡的重要病因之一,现有的检测手段,并不能快速高效地明确腹泻猪病因。本试验通过病毒宏基因组学手段,分析了上海周边猪场腹泻仔猪粪便中的病毒群落,结果显示腹泻仔猪粪便中病毒群落主要由小RNA病毒科(Picornaviridae)、星状病毒科(Astroviridae)、杯状病毒科(Caliciviridae)、细小病毒科(Parvoviridae)、圆环病毒科(Circoviridae)、藻类去氧核糖核酸病毒科(Phycodnaviridae)、冠状病毒科(Coronaviridae)等组成,不同文库的组成情况有所差别,但大多由小RNA病毒科主导。而关于健康猪粪便中病毒的组成情况,在笔者实验室的另一篇论文中,已经获得了较为全面的结果[35],可以看出健康仔猪粪便中的病毒主要集中在Kobuvirus、Sapovirus、Teschovirus和Sapelovirus上,且病毒滴度较低,在文中就不做过多讨论。
上海周边地区冠状病毒的整体阳性率为15.56%,其中:α-CoV属中的PEDV阳性率占92.86%,β-CoV属中的Bat_Hp-betacoronavirus阳性率占7.14%。对于在F猪场检测出的一株β-CoV属中的Bat_Hp-betacoronavirus阳性推测为经蝙蝠粪便排出,以仔猪作为中间宿主暂时寄存性感染,本研究不多作讨论。而对于PEDV阳性样本,阳性者全部来自于C猪场,在C猪场中, PEDV阳性率高达43.33%,则C猪场中PEDV是致仔猪腹泻的重要病原之一。对获得的PEDV基因序列进行遗传进化分析,结果显示,在本研究获得3个PEDV ORF3基因序列中,有1个属于G2基因型,与其他参考PEDV病毒株同源性较高,相对保守;另外2个ORF3基因序列属于G1基因型,同源性相对较低。与PEDV传统株CV777蛋白序列比较分析发现,除piglet91株产生F2S突变以外,三株PEDV研究所得株存在一致性突变,分别是V21A、I70M、V79I、F80V、L85I、L92F,这一特性,有可能是PEDV传统株与流行株基因型的鉴别依据。
本研究报道了上海周边地区CoV的感染情况,并对α-CoV属中PEDV的基因序列进行了遗传进化分析,从碱基突变基础上分析了PEDV突变株与传统株之间潜在的差异性。
综上所述,上海虽然不与韩国以及山东地区临近,但作为农畜产品交易大省,畜类交易密集,畜载量过大,而山东属于农畜产品主要生产地,其与上海和韩国农畜产品交易活动频繁,这就为疫病的传播提供了机会。因此,交通运输环节在防止疫病传播方面应该重点考虑,做好交通运输环节的防护和消毒,是控制新病毒的流入和传播的关键[35]。另外,C猪场同时出现G1和G2基因型的PEDV,提示了从事动物疫控的机构和相关研究人员应注意PEDV的跟踪研究,在发展养猪业时一定要考虑到如何加强包括PED在内的传染病的有效控制,在出现新问题时不要违背传染病防治“早快严小”的原则,应及时发现,及时研究,集中全行业的力量攻坚克难,共同解决。
4 结论上海周边地区腹泻仔猪肠道病毒组成丰富,在不同猪群中,病原感染情况有所差异,除PEDV外,星状病毒和杯状病毒也是仔猪腹泻的主要病原。PEDV的ORF3基因与传统分离株相比具有独特的分子特征,新检测到的氨基酸位点与病毒毒力的关系有待于进一步试验验证。研究结果有助于了解腹泻仔猪肠道的病毒谱,并为仔猪病毒病防控提供一定的基础数据。
| [1] | ZHANG J Q, GUY J S, SNIJDER E J, et al. Genomic characterization of equine coronavirus[J]. Virology, 2007, 369(1): 92–104. DOI: 10.1016/j.virol.2007.06.035 |
| [2] | JUNG K, HU H, EYERLY B, et al. Pathogenicity of 2 porcine deltacoronavirus strains in gnotobiotic pigs[J]. Emerg Infect Dis, 2015, 21(4): 650–654. DOI: 10.3201/eid2104.141859 |
| [3] | CHENG V C C, LAU S K P, WOO P C Y, et al. Severe acute respiratory syndrome coronavirus as an agent of emerging and reemerging infection[J]. Clin Microbiol Rev, 2007, 20(4): 660–694. DOI: 10.1128/CMR.00023-07 |
| [4] | PEIRIS J S, LAI S T, POON L L M, et al. Coronavirus as a possible cause of severe acute respiratory syndrome[J]. Lancet, 2003, 361(9366): 1319–1325. DOI: 10.1016/S0140-6736(03)13077-2 |
| [5] | LIM P L, LEE T H, ROWE E K. Middle east respiratory syndrome coronavirus (MERS CoV):update 2013[J]. Curr Infect Dis Rep, 2013, 15(4): 295–298. DOI: 10.1007/s11908-013-0344-2 |
| [6] |
凌云, 瞿嵘, 罗裕锋. 中国首例输入性中东呼吸综合征患者临床救治分析[J]. 中华危重病急救医学, 2015, 27(8): 630–634.
LING Y, QU R, LUO Y F. Clinical analysis of the first patient with imported Middle East respiratory syndrome in China[J]. Chinese Critical Care Medicine, 2015, 27(8): 630–634. DOI: 10.3760/cma.j.issn.2095-4352.2015.08.002 (in Chinese) |
| [7] | SUN R Q, CAI R J, CHEN Y Q, et al. Outbreak of porcine epidemic diarrhea in suckling piglets, China[J]. Emerg Infect Dis, 2012, 18(1): 161–163. DOI: 10.3201/eid1801.111259 |
| [8] | STEVENSON G W, HOANG H, SCHWARTS K J, et al. Emergence of Porcine epidemic diarrhea virus in the United States:clinical signs, lesions, and viral genomic sequences[J]. J Vet Diagn Invest, 2013, 25(5): 649–654. DOI: 10.1177/1040638713501675 |
| [9] | HUANG Y W, DICKERMAN A W, PIÑEYRO P, et al. Origin, evolution, and genotyping of emergent porcine epidemic diarrhea virus strains in the united states[J]. mBio, 2013, 4(5): 00737–13. |
| [10] | KSIAZEK T G, ERDMAN D, GOLDSMITH C S, et al. A novel coronavirus associated with severe acute respiratory syndrome[J]. N Engl J Med, 2003, 348(20): 1953–1966. DOI: 10.1056/NEJMoa030781 |
| [11] | PEIRISM. Severe acute respiratory syndrome[M]. Malden: Blackwell Pub., 2005: 88-97. |
| [12] | ZAKI A M, VAN BOHEEMEN S, BESTEBROER T M, et al. Isolation of a novel coronavirus from a man with pneumonia in Saudi Arabia[J]. N Engl J Med, 2012, 367(19): 1814–1820. DOI: 10.1056/NEJMoa1211721 |
| [13] | BELOUZARD S, MILLET J K, LICITRA B N, et al. Mechanisms of coronavirus cell entry mediated by the viral spike protein[J]. Viruses, 2012, 4(6): 1011–1033. DOI: 10.3390/v4061011 |
| [14] | MILLET J K, WHITTAKER G R. Host cell proteases:Critical determinants of coronavirus tropism and pathogenesis[J]. Virus Res, 2015, 202: 120–134. DOI: 10.1016/j.virusres.2014.11.021 |
| [15] | DEHAAN C A M, STADLER K, GODEKE G J, et al. Cleavage inhibition of the murine coronavirus spike protein by a furin-like enzyme affects cell-cell but not virus-cell fusion[J]. J Virol, 2004, 78(11): 6048–6054. DOI: 10.1128/JVI.78.11.6048-6054.2004 |
| [16] | KAWASE M, SHIRATO K, MATSUYAMA S, et al. Protease-mediated entry via the endosome of human coronavirus 229E[J]. J Virol, 2009, 83(2): 712–721. DOI: 10.1128/JVI.01933-08 |
| [17] | SIMMONS G, GOSALIA D N, RENNEKAMP A J, et al. Inhibitors of cathepsin L prevent severe acute respiratory syndrome coronavirus entry[J]. Proc Natl Acad Sci U S A, 2005, 102(33): 11876–11881. DOI: 10.1073/pnas.0505577102 |
| [18] | QIU Z Z, HINGLEY S T, SIMMONS G, et al. Endosomal proteolysis by cathepsins is necessary for murine coronavirus mouse hepatitis virus type 2 spike-mediated entry[J]. J Virol, 2006, 80(12): 5768–5776. DOI: 10.1128/JVI.00442-06 |
| [19] | WICHT O, LI W T, WILLEMS L, et al. Proteolytic activation of the porcine epidemic diarrhea coronavirus spike fusion protein by trypsin in cell culture[J]. J Virol, 2014, 88(14): 7952–7961. DOI: 10.1128/JVI.00297-14 |
| [20] |
方肇寅, 谢华萍, 吕红霞, 等. 1999~2005年我国婴幼儿人杯状病毒腹泻研究[J]. 病毒学报, 2007, 23(1): 9–15.
FANG Z Y, XIE H P, LV H X, et al. Investigation of human calicivirus (HuCV) diarrhea among infantile and young children in China, 1999-2005[J]. Chinese Journal of Virology, 2007, 23(1): 9–15. DOI: 10.3321/j.issn:1000-8721.2007.01.002 (in Chinese) |
| [21] |
王新珍.东北稻田水体蓝藻病毒基因多样性与水体病毒宏基因组学研究[D].长春: 中国科学院大学(中国科学院东北地理与农业生态研究所), 2017.
WANG X Z. Analysis of cyanophage genetic diversity and viral metagenomics in paddy waters in Northeast China[D]. Changchun: Chinese Academy of Sciences (Northeast Institute of Geography and Agroecology), 2017. (in Chinese) http://cdmd.cnki.com.cn/Article/CDMD-80062-1017930196.htm |
| [22] |
刘文彬, 张翠媛, 虞结梅, 等. 中国广西龙虎山野生猕猴粪便的病毒宏基因组学分析[J]. 病毒学报, 2016, 32(3): 273–282.
LIU W B, ZHANG C Y, YU J M, et al. Metagenomic analysis of wild rhesus monkey virome in Longhu Mountain in Guangxi Area, China[J]. Chinese Journal of Virology, 2016, 32(3): 273–282. (in Chinese) |
| [23] |
舒鹏.宏基因组测序技术在肠道菌群研究和临床样本检测中的应用[D].合肥: 安徽医科大学, 2016.
SHU P. The application of metagenomics sequencing technology in the research of intestinal floraand the detection of clinical samples[D]. Hefei: Anhui Medical University, 2016. (in Chinese) http://cdmd.cnki.com.cn/Article/CDMD-10366-1016144002.htm |
| [24] |
张若曦, 张志, 顾文源, 等. 河北省猪腹泻相关病毒的检测及猪流行性腹泻病毒S基因遗传变异分析[J]. 畜牧兽医学报, 2018, 49(3): 597–605.
ZHANG R X, ZHANG Z, GU W Y, et al. Detection of porcine diarrhea associated virus and genetic variation analysis of S gene of porcine epidemic diarrhea virus in Hebei province[J]. Acta Veterinaria et Zootechnica Sinica, 2018, 49(3): 597–605. (in Chinese) |
| [25] |
施标, 董世娟, 朱于敏, 等. 中国猪流行性腹泻病毒分子流行病学研究进展[J]. 中国农业科学, 2013, 46(20): 4362–4369.
SHI B, DONG S J, ZHU Y M, et al. Advances in study of molecular epidemiology of porcine epidemic diarrhea virus in China[J]. Scientia Agricultura Sinica, 2013, 46(20): 4362–4369. DOI: 10.3864/j.issn.0578-1752.2013.20.020 (in Chinese) |
| [26] | SUN R Q, LENG Z M, ZHAI S L, et al. Genetic variability and phylogeny of current Chinese porcine epidemic diarrhea virus strains based on Spike, ORF3, and Membrane genes[J]. Sci World J, 2014, 2014: 208439. |
| [27] | GE F F, YANG D Q, JU H B, et al. Epidemiological survey of porcine epidemic diarrhea virus in swine farms in Shanghai, China[J]. Arch Virol, 2013, 158(11): 2227–2231. DOI: 10.1007/s00705-013-1722-7 |
| [28] |
韩文, 罗玉子, 赵碧波, 等. 基于宏基因组学的猪群样本病毒探测方法的建立[J]. 微生物学报, 2013, 53(2): 197–203.
HAN W, LUO Y Z, ZHAO B B, et al. Metagenomics-based detection of swine viruses[J]. Acta Microbiologica Sinica, 2013, 53(2): 197–203. (in Chinese) |
| [29] | BREITBART M, HEWSON I, FELTS B, et al. Metagenomic analyses of an uncultured viral community from human feces[J]. J Bacteriol, 2003, 185(20): 6220–6223. DOI: 10.1128/JB.185.20.6220-6223.2003 |
| [30] | LI L L, VICTORIA J G, WANG C L, et al. Bat guano virome:predominance of dietary viruses from insects and plants plus novel mammalian viruses[J]. J Virol, 2010, 84(14): 6955–6965. DOI: 10.1128/JVI.00501-10 |
| [31] | WILLNER D, FURLAN M, HAYNES M, et al. Metagenomic analysis of respiratory tract DNA viral communities in cystic fibrosis and non-cystic fibrosis individuals[J]. PLoS One, 2009, 4(10): e7370. DOI: 10.1371/journal.pone.0007370 |
| [32] | ALLANDER T, EMERSON S U, ENGLE R E, et al. A virus discovery method incorporating DNase treatment and its application to the identification of two bovine parvovirus species[J]. Proc Natl Acad Sci U S A, 2001, 98(20): 11609–11614. DOI: 10.1073/pnas.211424698 |
| [33] | RONDON M R, AUGUST P R, BETTERMANN A D, et al. Cloning the soil metagenome:a strategy for accessing the genetic and functional diversity of uncultured microorganisms[J]. Appl Environ Microbiol, 2000, 66(6): 2541–2547. DOI: 10.1128/AEM.66.6.2541-2547.2000 |
| [34] | HENNE A, SCHMITZ R A, BOMEKE M, et al. Screening of environmental DNA libraries for the presence of genes conferring lipolytic activity on Escherichia coli[J]. Appl Environ Microbiol, 2000, 66(7): 3113–3116. DOI: 10.1128/AEM.66.7.3113-3116.2000 |
| [35] |
单同领.病毒宏基因组学分析儿童和猪肠道病毒群落及23株病毒的初步研究[D].上海: 上海交通大学, 2011.
SHAN T L. Viral metegenomic analysis of viromes of stool samples in children and swine and preliminary study of 23 strains of viruses[D]. Shanghai: Shanghai Jiaotong University, 2011. (in Chinese) http://med.wanfangdata.com.cn/Paper/Detail/DegreePaper_D633632 |



