2. 河北科技师范学院, 秦皇岛 066004;
3. 河北省牛羊胚胎工程技术研究中心, 保定 071000
2. Hebei Normal University of Science & Technology, Qinhuangdao 066004, China;
3. Research Center of Cattle and Sheep Embryo Engineering Technique of Hebei, Baoding 071000, China
哺乳动物毛色是由色素决定的,决定毛色的物质基础主要是酪氨酸源性色素,即黑色素及其衍生物[1-3]。多巴色素异构酶(dopachrometautomerase,DCT),也叫作酪氨酸酶相关蛋白2(TYRP2),是定位于黑素体膜上的跨膜蛋白,参与黑色素的生物合成过程。DCT主要控制5,6-二羟基吲哚羧酸与5,6-二羟基吲哚的比例,从而影响黑色素生成,因此是影响动物毛色的重要蛋白[4-5]。Costin等[6]发现,DCT属于Ⅰ型膜蛋白,并通过化学分析显示,DCT突变体的黑素细胞中褐黑素产量增加,真黑素产量降低,因此在色素生物合成中DCT的酶活性决定了选择合成褐黑素途径还是真黑素途径。Guyonneau等[7]通过敲除小鼠DCT基因外显子1序列,使小鼠DCT基因的mRNA和蛋白表达缺失,但小鼠能够正常存活且除黑色素含量减少,使得小鼠出现了毛色变浅的表型外,在DCT基因正常表达部位(如皮肤、视网膜色素上皮细胞和大脑)未发现任何畸形或异常情况。Okumura等[8]利用辐射杂交和SNP连锁分析将猪DCT基因定位于11号染色体上,并发现了与猪毛色表型相关的SNP位点。
DNA甲基化主要位于富含CpG岛的启动子区或第一外显子区,一般发生在二核苷酸胞嘧啶的第五位碳原子上[9]。启动子区与转录因子结合的DNA序列发生甲基化形成“转录抑制复合物”,可引起转录抑制[10-15]。启动子单核苷酸多态性可能会引起基因上某些特异转录因子结合位点的改变,从而影响基因的表达[16]。鉴于DCT基因表达在动物毛色中的重要作用,其表达水平受启动子和转录因子调控,而启动子活性和转录因子结合与基因甲基化和SNP突变密切相关,因此,研究DCT基因核心启动子区甲基化水平和单核苷酸多态性与毛色特征和启动子活性变化的关系对揭示DCT基因在动物毛色形成中的作用具有重要意义。
本试验通过CpG岛预测、甲基化测序、多态性位点筛选、测序结果比对、转录因子在线预测及启动子活性检测,以期为探究DCT基因启动子区甲基化水平、SNP突变对山羊毛色的影响提供理论依据。
1 材料与方法 1.1 材料和试剂用于多态性分析的DNA由唐山奶山羊、济宁青山羊、南江黄羊和南江黄羊黑色系血液样本中提取;甲基化检测所用DNA由唐山奶山羊和南江黄羊黑色系血液样本中提取。
EasyPure Genomic DNA Kit、TransStart Taq DNA Polymerase、EasyPure PCR Purification Kit购自北京全式金公司,限制性内切酶Sac Ⅰ和Xho Ⅰ、质粒载体T-Vector PMD19、DNA Ligat1 on Kit购自大连宝生物工程有限公司,San-Prep柱式质粒DNA小量提取抽提试剂盒和San-Prep柱式DNA胶回收试剂盒购自生工公司,无内毒素质粒DNA大量提取试剂盒购自天根公司,双荧光素酶报告基因检测试剂盒购自Promega公司,T4连接酶和Lipofectamine2000 Transfection Reagent购自Thermo Fisher Science公司,EZ DNA Methylation-GoldTMKit试剂盒购自ZYMO公司,A375细胞、质粒载体pGL3-basic和pRL-TK均为本实验室保存。
1.2 引物设计根据NCBI(National Center for Biotechnology)中GenBank数据库公布的山羊DCT基因序列(NC030819.1),结合真核生物启动子数据库(Eukaryotic Promoter Database,EPD)公布的小鼠和人的DCT启动子信息,截取位于山羊DCT基因g.-2 000~+1 000区域(外显子1第1个碱基定义为+1位)。利用在线软件MethPrimer(http://www.urogene.org/cgi-bin/methprimer/methprimer.cgi)预测CpG岛,参数设置为Island size ≥ 100,Obs/Exp ≥ 0.6,GC Percent ≥ 50.0。利用在线程序NEBuilder(http://nebuilder.neb.com/)对预测的CpG岛序列设计甲基化测序引物(甲1和甲2),核心启动子区引物(核1)参考刘春杨等[17]的研究,核1引物上下游序列5′端分别引入SacⅠ和XhoⅠ限制性酶切位点及保护碱基,引物序列及扩增条件见表 1。
|
|
表 1 核心启动子区扩增和甲基化水平检测引物 Table 1 Primers for amplifying core promoter region and detecting methylation level |
按照EZ DNA Methylation-GoldTMKit使用说明处理唐山奶山羊和南江黄羊黑色系各3只的基因组,以亚硫氢酸盐修饰后的基因组为模板进行扩增,甲1和甲2引物的扩增条件完全一致。PCR扩增体系为50 μL:0.5 μL TransStart Taq DNA Polymerase,36 μL ddH2O,5 μL 10× Trans Start Taq Buffer,4 μL 2.5 mmol·L-1 dNTPs,2.5 μL修饰的DNA模板,上下游引物各1 μL。PCR反应条件:94 ℃预变性5 min;94 ℃变性30 s,60 ℃退火30 s,72 ℃延伸45 s,35个循环;72 ℃最终延伸10 min,4 ℃保存。纯化回收PCR产物,连接到pMD19-Simple载体,转化DH5α感受态细胞,菌液PCR检测重组质粒,每个样本测序10个单克隆。利用甲基化水平分析软件BISMA(http//services.ibc.uni-stuttgart.de/BDPC/BISMA)统计甲基化位点,分析不同毛色山羊DCT基因甲基化水平差异。
1.4 山羊DCT基因核心启动子区SNP筛选和转录因子结合位点分析对24只唐山奶山羊、27只南江黄羊黑色系、28只济宁青山羊和20只南江黄羊快长系,利用核1引物进行核心启动子区克隆。PCR反应体系:10×PCR Buffer 5 μL,dNTP(2.5 mmol·L-1)4 μL,上下游引物(10 μmol·L-1)各1 μL,基因组DNA 2.5 μL,TransStart Taq酶(2.5 U·μL-1)0.5 μL,最后ddH2O补足至50 μL。PCR反应条件:95 ℃预变性5 min;95 ℃变性30 s,65 ℃退火30 s,72 ℃延伸0.5 min,35个循环;最终72 ℃延伸10 min;4 ℃终止反应。纯化回收的PCR产物送华大基因公司进行测序,比对所有个体的DCT基因核心启动子区序列,筛选SNP变异位点。
将包含SNP位点的不同单倍型启动子序列利用JASPAR(http://jaspar.genereg.net/cgi-bin/jaspar_db.pl)和Nsite(http://www.softberry.com/berry.phtml?topic=nsite&group=programs&subgroup=promoter)预测转录因子结合位点,分析SNP是否影响转录因子结合。
1.5 山羊DCT基因g.-804 T > G突变片段启动子活性分析依照1.4的PCR反应体系和条件扩增DCT基因g.-804位点T→G突变的两种单倍型基因序列,产物测序确定单倍型后纯化回收,用限制性内切酶SacⅠ和XhoⅠ双酶切回收产物和表达载体pGL3-basic。取线性化载体50 ng,纯化回收PCR目的片段250 ng,5 μL 2 × Lightening Cloning Master Mix,ddH2O补足至10 μL,50 ℃水浴条件下反应1 h,连接目的片段与pGL3-Basic,获取重组质粒。鉴定阳性的重组质粒瞬时转染A375细胞,48 h后收获并裂解细胞,检测萤火虫荧光素酶荧光值(F)和海肾荧光素酶荧光值(R),计算相对荧光素酶活性(F/R),并与阴性对照(Basic)的相对荧光素酶表达量做比,以消除不同转染批次的系统误差。比较鉴定核心启动子区g.-804 T > G突变前后的DCT基因启动子活性。
1.6 数据分析利用SPSS24.0中描述统计的列联表进行χ2检验,分析白色唐山奶山羊与其他毛色山羊SNP位点基因型频率的差异性和黑白两种毛色山羊DCT基因碱基位点甲基化率的差异;利用单因素方差分析对不同片段的启动子活性进行分析,Duncan’s多重比较组间差异;利用独立样本t检验分析黑白两种山羊DCT基因CpG岛区域平均甲基化水平差异。P < 0.01为统计学差异极显著,P < 0.05为统计学差异显著。
2 结果 2.1 山羊DCT基因CpG岛预测预测得到DCT基因g.-2 000~+1 000区域2处CpG岛序列,分别位于DCT基因g.-258~ -158和g.+144~ +438区域。如图 1所示,特异性甲1和甲2引物扩增片段分别为g.-348 ~ -150和g.+222~+502。
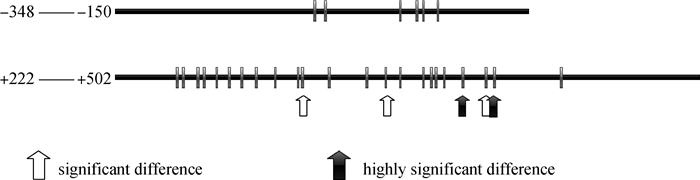
|
图 1 预测的甲基化位点 Figure 1 Methylation sites predicted |
检测黑色和白色两种毛色山羊各3个个体的10个单克隆序列,发现DCT基因g.-348~-150区域包含6个甲基化位点,g.+222~+502区域包含23个甲基化位点(图 1)。g.-348~-150区域两种毛色各位点甲基化水平统计结果见表 2,6个位点的甲基化水平在白色和黑色山羊间均无显著差异(P > 0.05),白色山羊平均甲基化水平((90.3±2.9) %)高于黑色山羊((89.4±2.5) %),但差异不显著(P > 0.05)。
|
|
表 2 两种毛色山羊DCT基因g.-348~-150区域甲基化水平差异 Table 2 Methylation differences of two goat populations with different coat colors in g. -348--150 region of DCT gene |
两种毛色山羊DCT基因g.+222~+502区域各位点甲基化水平统计结果见表 3。白色山羊平均甲基化水平((90.6±5.3) %)极显著高于黑色山羊((83.7±8.5) %)(P < 0.01),其中,g.+312、g.+352和g.+400位点白色山羊甲基化水平显著高于黑色山羊(P < 0.05),g.+389和g.+404位点白色山羊甲基化水平极显著高于黑色山羊(P < 0.01)。其他位点甲基化水平白色和黑色山羊间差异不显著(P > 0.05)。
|
|
表 3 两种毛色山羊DCT基因g.+222~+502区域甲基化水平差异 Table 3 Methylation differences of two goat populations with different coat colors in g.+222-+502 region of DCT gene |
DCT基因核心启动子区共发现4个SNPs位点,依次为g.-804位点的T>G、g.-714位点的T>C、g.-705位点的C>T和g.-679位点的G>A突变,分别定义为SNP1、SNP2、SNP3和SNP4(图 2)。

|
图 2 DCT基因启动子区SNPs位点示意图 Figure 2 Schematic diagram of SNPs sites in core promoter region of DCT gene |
Hardy-Weinberg平衡分析发现,除济宁青山羊在SNP2位点偏离了平衡状态外,其它群体和其它位点都处于平衡状态,SNP的群体分布结果见表 4。白色的唐山奶山羊SNP1突变与其它3个有色群体存在显著差异,其中济宁青山羊与南江黄羊和南江黄羊黑色系的T等位基因频率分别显著和极显著高于唐山奶山羊(P < 0.05,P < 0.01),唐山奶山羊的TT基因型频率低于其它群体,而GG基因型频率高于其它群体。唐山奶山羊SNP2的T等位基因频率显著高于济宁青山羊(P < 0.05),但与其它山羊间没有显著差异(P > 0.05),这可能与济宁青山羊该位点的不平衡状态有关。唐山奶山羊SNP3的基因频率与其它3个群体间均存在显著差异,C等位基因频率极显著高于济宁青山羊和南江黄羊(P < 0.01),显著高于南江黄羊黑色群体(P < 0.05),CC基因型频率高于3个有色群体。SNP4位点A等位基因频率白色唐山奶山羊极显著高于3个有色群体(P < 0.01),而GG基因型频率低于有色群体。综上,4个SNPs位点中的g.-804 T > G、g.-705 C > T和g. -679 G > A变异可能与山羊的白色和有色被毛特征形成相关。
|
|
表 4 4个SNPs位点在4个山羊群体的等位基因频率和基因型频率 Table 4 Allele frequency and genotype frequency of the 4 SNPs in 4 goat populations |
通过JASPAR和Nsite的转录因子预测分析发现,SNP1(g.-804 T > G)位点的突变型G序列较野生型的T序列缺失了SOX10的结合位点,如图 3A。将野生型与突变型核心启动子序列构建双荧光素酶报告基因,并测试表达活性,结果如图 3B所示。突变型的等位基因G序列启动子活性(2.08)比野生型T序列(5.84)显著降低(P < 0.05),下降2.8倍;虽然突变型活性是阴性对照(pGL3-Basic)的2.08倍,但与阴性对照没有显著差异(P>0.05)。

|
图 3 DCT基因核心启动子区g.-804 T > G突变前后转录因子和启动子活性变化 Figure 3 Changes of transcription factors and promoter activity caused by g.-804 T > G mutation in core promoter region of DCT gene |
DNA的甲基化会影响基因的转录表达[9],本试验检测了黑色和白色两种毛色山羊DCT基因两处CpG岛的DNA甲基化水平,结果显示,g.+222~+502区域中有5个甲基化位点在白色山羊中的甲基化水平显著或极显著高于黑色山羊。但处于第一外显子上游启动子区的CpG区域的甲基化水平在两种毛色山羊中没有差异,说明启动子区的甲基化水平可能不参与调控不同毛色的形成,而处于内含子区的g.+222~+502区域在两种毛色中的甲基化差异对基因表达存在调控作用。由于DNA的甲基化修饰会抑制基因的转录表达[10-15],因此可以推测,g.+222~+502区域的高甲基化水平抑制了山羊DCT基因的表达,使得山羊毛色变浅。而之前的研究证明,DCT基因在浅毛色动物体内的表达量低于深毛色动物[18],进一步说明了g.+222~+502区域的甲基化与山羊不同毛色的形成有关。
基因启动子的活性调控机制已有较多研究[19-22],但单核苷酸变异引起的序列多态性对启动子活性影响的研究尚不多见,其可能会影响基因的转录表达,其中发生在相关转录因子结合位点的序列变异会阻碍转录因子与DNA序列的结合,从而抑制基因的表达[23]。部分位点的变异会导致毛色的改变[6-8],Guyonneau等[7]的研究即是由于某一位点的突变,导致产生了鼠毛色变浅的表型。本试验所检测的变异位点g.-804、g.-714和g.-679在唐山奶山羊中的等位基因频率与其它3种毛色山羊群体存在显著差异,而唐山奶山羊被毛颜色为白色,其它3个群体皆为有色群体,揭示g.-804、g.-714和g.-679 3个位点的突变可能与山羊白色毛色形成有关。
已有研究表明,SOX10能够激活DCT基因的表达[24],课题组的前期研究发现,点突变引起SOX10转录因子结合位点的3处突变缺失,均造成DCT基因启动活性的显著降低[17]。本研究中,白色唐山奶山羊SNP1的G等位基因频率显著高于3个有色山羊群体,同时发现,该突变引起SOX10转录因子结合位点的缺失,通过双荧光素酶活性检测发现,缺失SOX10的突变G型序列启动子活性降低到阴性对照水平。另外,课题组对皮肤转录组数据分析发现,唐山奶山羊DCT基因表达显著低于南江黄羊黑色系(未发表)。这一结果充分证明了SOX10对DCT基因具有正向调控作用,同时说明DCT基因启动子区的g.-804T>G突变引发SOX10转录因子结合位点的缺失,继而引起DCT基因转录启动活性降低,mRNA表达水平下降,这与唐山奶山羊的白色被毛特征有关。
本试验在山羊DCT基因核心启动子区发现了4个SNPs,只有SNP1位点变化引起SOX10转录因子结合位点缺失,并未发现其他SNP引起转录因子结合变化,也可能这些突变位点改变了其它未知的参与DCT基因转录调控的转录因子的结合,从而在不同毛色个体中起到调控作用。
4 结论白色山羊DCT基因g.+222~+502区域的高甲基化水平,g.-804、g.-714和g.-679 3个位点的突变,尤其是g.-804 T > G造成SOX10转录因子结合位点的缺失,突变的G型序列显著降低DCT基因的启动子活性。因此DCT基因启动子区SNP突变和高甲基化水平可能抑制了基因的表达从而形成山羊白色被毛。
| [1] |
吴宇婷. 哺乳动物毛色形成机制与影响因素[J]. 四川动物, 2011, 30(6): 1003–1007.
WU Y T. The formation mechanism of mammal coat color and its possible influencing factors[J]. Sichuan Journal of Zoology, 2011, 30(6): 1003–1007. DOI: 10.3969/j.issn.1000-7083.2011.06.033 (in Chinese) |
| [2] |
王乐, 吕雪峰, 郑文新, 等. 动物毛色形成的原因和机制[J]. 草食家畜, 2009(1): 20–21.
WANG L, LV X F, ZHENG W X, et al. The cause and mechanism of animal coat color formed[J]. Grass-Feeding Livestock, 2009(1): 20–21. DOI: 10.3969/j.issn.1003-6377.2009.01.007 (in Chinese) |
| [3] |
张俊珍, 董常生, 范瑞文, 等. 哺乳动物毛色形成研究进展[J]. 动物医学进展, 2006, 27(S1): 65–68.
ZHANG J Z, DONG C S, FAN R W, et al. Advance in pigment melanin of mammalian[J]. Progress in Veterinary Medicine, 2006, 27(S1): 65–68. (in Chinese) |
| [4] |
舒文, 毛华明. 黑色素的研究进展[J]. 国外畜牧学-猪与禽, 2003, 23(2): 31–34.
SHU W, MAO H M. Research progress of melanin[J]. Foreign Animal Husbandry-Pigs and Poultry, 2003, 23(2): 31–34. DOI: 10.3969/j.issn.1001-0769.2003.02.012 (in Chinese) |
| [5] |
刘伟兰, 李祥龙, 周荣艳, 等. 不同物种TYRP2基因完整编码区生物信息学分析[J]. 河南农业科学, 2011, 40(10): 144–148.
LIU W L, LI X L, ZHOU R Y, et al. Bioinformatics analysis of complete coding regions of TYRP2 gene among 21 species[J]. Journal of Henan Agricultural Sciences, 2011, 40(10): 144–148. DOI: 10.3969/j.issn.1004-3268.2011.10.041 (in Chinese) |
| [6] | COSTIN G E, VALENCIAJ C, WAKAMATSU K, et al. Mutations in dopachrome tautomerase (DCT) affect eumelanin/pheomelanin synthesis, but do not affect intracellular trafficking of the mutant protein[J]. Biochem J, 2005, 391(2): 249–259. DOI: 10.1042/BJ20042070 |
| [7] | GUYONNEAU L, MURISIER F, ROSSIER A, et al. Melanocytes and pigmentation are affected in Dopachrome Tautomerase knockout mice[J]. Mol Cell Biol, 2004, 24(8): 3396–3403. DOI: 10.1128/MCB.24.8.3396-3403.2004 |
| [8] | OKUMURA N, HAYASHI T, SEKIKAWA H, et al. Sequences and mapping of genes encoding porcine tyrosinase (TYR) and tyrosinase-related proteins (TYRP1 and TYRP2)[J]. Anim Genet, 2005, 36(6): 513–516. DOI: 10.1111/j.1365-2052.2005.01353.x |
| [9] | FENG J, FAN G P. The role of DNA methylation in the central nervous system and neuropsychiatric disorders[J]. Int Rev Neurobiol, 2009, 89: 67–84. DOI: 10.1016/S0074-7742(09)89004-1 |
| [10] | JONES P A, TAKAI D. The role of DNA methylation in mammalian epigenetics[J]. Science, 2001, 293(5532): 1068–1070. DOI: 10.1126/science.1063852 |
| [11] | SINGAL R, GINDER G D. DNA methylation[J]. Blood, 1999, 93(12): 4059–4070. |
| [12] | MEEHAN R R, LEWIS J D, MCKAY S, et al. Identification of a mammalian protein that binds specifically to DNA containing methylated CpGs[J]. Cell, 1989, 58(3): 499–507. DOI: 10.1016/0092-8674(89)90430-3 |
| [13] | LEWIS J D, MEEHAN R R, HENZEL W J, et al. Purification, sequence, and cellular localization of a novel chromosomal protein that binds to methylated DNA[J]. Cell, 1992, 69(6): 905–914. DOI: 10.1016/0092-8674(92)90610-O |
| [14] | ZHANG Y, NG H H, ERDJUMENT-BROMAGE H, et al. Analysis of the NuRD subunits reveals a histone deacetylase core complex and a connection with DNA methylation[J]. Genes Dev, 1999, 13(15): 1924–1935. DOI: 10.1101/gad.13.15.1924 |
| [15] | YU F, THIESEN J, STR TLING W H. Histone deacetylase-independent transcriptional repression by methyl-CpG-binding protein 2[J]. Nucleic Acids Res, 2000, 28(10): 2201–2206. DOI: 10.1093/nar/28.10.2201 |
| [16] |
李圣彦, 郎志宏, 黄大昉. 真核生物启动子研究概述[J]. 生物技术进展, 2014, 4(3): 158–164.
LI S Y, LANG Z H, HUANG D F. Research progress on eukaryotic promoter[J]. Current Biotechnology, 2014, 4(3): 158–164. DOI: 10.3969/j.issn.2095-2341.2014.03.02 (in Chinese) |
| [17] |
刘春杨, 张乐超, 王麒, 等. 山羊DCT基因启动子活性区及其转录因子调控探究[J]. 畜牧兽医学报, 2018, 49(1): 55–64.
LIU C Y, ZHANG L C, WANG Q, et al. The exploration of the promoter activity area and regulation by transcription factors of goat DCT gene[J]. Acta Veterinaria et Zootechnica Sinica, 2018, 49(1): 55–64. (in Chinese) |
| [18] |
高莉, 董常生, 赫晓燕, 等. 羊驼酪氨酸酶基因家族在不同毛色个体中的基因表达水平[J]. 畜牧兽医学报, 2008, 39(7): 895–899.
GAO L, DONG C S, HE X Y, et al. Gene expression levels of alpaca tyrosinase gene family in individuals of different colors[J]. Acta Veterinaria et Zootechnica Sinica, 2008, 39(7): 895–899. DOI: 10.3321/j.issn:0366-6964.2008.07.009 (in Chinese) |
| [19] |
周纬男, 史铭欣, 乔书培, 等. 鸡Perilipin1基因启动子的克隆及分析[J]. 畜牧兽医学报, 2016, 47(2): 249–259.
ZHOU W N, SHI M X, QIAO S P, et al. Promoter cloning and analysis of chicken Perilipin1 gene[J]. Acta Veterinaria et Zootechnica Sinica, 2016, 47(2): 249–259. (in Chinese) |
| [20] |
刘心, 刘浩, 王义涛, 等. 人LOXL2启动子的鉴定与初步分析[J]. 中国生物化学与分子生物学报, 2018, 34(5): 540–547.
LIU X, LIU H, WANGY T, et al. Structural and functional analysis of Human LOXL2 promoter[J]. Chinese Journal of Biochemistry and Molecular Biology, 2018, 34(5): 540–547. (in Chinese) |
| [21] |
刘小辉, 彭永东, 周荣艳, 等. 坝上长尾鸡MITF基因核心启动子鉴定与分析[J]. 畜牧兽医学报, 2018, 49(6): 1116–1123.
LIU X H, PENG Y D, ZHOU R Y, et al. Identification and analysis of the core promoter of MITF gene in Bashang long-tail chicken[J]. Acta Veterinaria et Zootechnica Sinica, 2018, 49(6): 1116–1123. (in Chinese) |
| [22] |
王涵, 陈烨, 周荣艳, 等. 蛋鸡胱硫醚β合酶基因启动子鉴定与分析[J]. 畜牧兽医学报, 2018, 49(1): 211–217.
WANG H, CHEN Y, ZHOU R Y, et al. Identification and analysis of promoter of Cystathionine beta synthase in layer[J]. Acta Veterinaria et Zootechnica Sinica, 2018, 49(1): 211–217. (in Chinese) |
| [23] |
郭晓敏, 刘向萍, 常国斌, 等. 鸡NLRC5基因启动子多态性分析[J]. 中国兽医学报, 2015, 35(11): 1845–1849.
GUO X M, LIU X P, CHANG G B, et al. Polymorphism analysis of NLRC5 gene promoter of chickens[J]. Chinese Journal of Veterinary Science, 2015, 35(11): 1845–1849. (in Chinese) |
| [24] | POTTERF S B, MOLLAAGHABABA R, HOU L, et al. Analysis of SOX10 function in neural crest-derived melanocyte development:SOX10-dependent transcriptional control of Dopachrome Tautomerase[J]. Dev Biol, 2001, 237(2): 245–257. DOI: 10.1006/dbio.2001.0372 |



