2. 扬州大学兽医学院, 扬州 225009;
3. 山东省动物生物工程与疾病防治重点实验室, 泰安 271000;
4. 山东省畜禽疫病防制工程技术研究中心, 泰安 271000
2. College of Veterinary Medicine, Yangzhou University, Yangzhou 225009, China;
3. Shandong Provincial Key Laboratory of Animal Biotechnology and Disease Control and Prevention, Tai'an 271000, China;
4. Shandong Provincial Engineering Technology Research Center of Animal Disease Control and Prevention, Tai'an 271000, China
20世纪50年代以来我国的规模化毛皮动物饲养方式开始兴起,近年来发展迅速,已经成为一项重要产业。随着毛皮动物饲养规模不断扩大,制约产业发展的问题日趋凸显,其中包括饲养管理的水平、传染性疾病的危害、多重耐药细菌的污染等;而细菌性疫病目前已经成为阻碍该产业向前发展不可忽视的问题之一[1-4]。大肠埃希菌(Escherichia coli)广泛存在于人和动物的肠道中,一直以来认为是正常菌群。直到20世纪中叶,人们发现有些大肠杆菌具有条件致病性,可引起食物中毒,致使正常机体发生腹泻,甚至出现败血症。引起致病性的大肠杆菌血清型众多,近年来的流行病学调查表明,不同地区的优势血清型存在差异,且同一区域不同疫群的优势血清型也可能表现不同[5],这给人们对畜禽进行大肠杆菌病的防控带来巨大难题。水貂大肠杆菌病多数是由致病性血清型大肠杆菌所引起,主要侵害幼貂和妊娠母貂,可产生腹泻、败血症及母貂流产等一系列危害[6],威胁着水貂养殖业的稳定发展。临床上经常使用抗生素治疗水貂的大肠杆菌病,但随着抗生素的滥用导致多重耐药大肠杆菌的检出率不断上升,因此在对水貂大肠杆菌病进行治疗时需要选择合适的抗菌药物[7-8]。
大肠杆菌是具有克隆结构的种群,由不同的克隆发育群组成[9]。国内有关水貂源大肠杆菌的研究相对较少,本研究从诸城地区的水貂养殖场采集水貂粪便分离大肠杆菌进行致病性、耐药性以及克隆关系等方面的研究;将水貂源大肠杆菌和人源大肠杆菌在MLST基因分型上进行对比分析,借以探究不同克隆发育群的大肠杆菌与宿主特异性之间的关系;以及利用菌株的7对看家基因序列构建系统发育树来探究相同克隆菌株耐药谱和耐药基因的携带特点[10]。目的是了解和掌握大肠杆菌在该地区的流行情况以及探究相同克隆群菌株的遗传相似性,继而阐明水貂养殖场粪便中大肠杆菌对公共卫生的影响。
1 材料与方法 1.1 样品采集2017年7月从山东省诸城地区大型水貂养殖场(水貂养殖量超过10 000条)用无菌棉拭子和灭菌的2 mL离心管采集新鲜粪便样品(采样过程中不会伤害水貂,故本次研究不需要动物伦理许可)。所有样本均放入4 ℃冰盒保存,在6 h内转移到实验室进行后续的实验分析。
1.2 分离鉴定和菌株保存把采集的粪便拭子样品移入含有10 mL Luria-Bertani(LB)肉汤的无菌摇管中,放于37 ℃恒温摇床(220 r·min-1)中进行增菌培养8~12 h。将增菌液使用三线法接种于伊红美蓝(EMB)琼脂培养基上,37 ℃培养18 h。在EMB平板上选择3~5株呈金属光泽的单菌落用16S rRNA PCR进行菌种鉴定(重复检测两次,并取部分PCR阳性结果进行测序比对)。为避免相同来源的菌株,从EMB平板上的多株阳性单菌落中挑取一株作为编号菌株置于5 mL LB液体培养基中进行增菌培养8 h,将增菌液与无菌甘油按照6:4的比例混合,然后使用玻璃纸密封离心管,把处理好的菌液先置于4 ℃冰箱30 min,然后保存于-20 ℃冰箱中备用。试验所用的培养基均选自青岛高科园海博生物技术有限公司,2×PCR Master Mix购于南京诺唯赞生物科技有限公司。
1.3 血清型鉴定采用玻片凝集试验并按照所诊断血清试剂盒说明书的标注对大肠杆菌分离株进行血清型鉴定。诊断血清试剂盒由肠致病性大肠杆菌(EPEC)诊断血清、肠侵袭性大肠杆菌(EIEC)诊断血清、肠产毒性大肠杆菌(ETEC)诊断血清、肠出血性大肠杆菌(EHEC)诊断血清组成。血清型诊断试剂盒均购自宁波天润生物药业有限公司。
1.4 耐药表型的检测耐药表型采用美国临床实验室标准化委员会(CLSI)所推荐的Kirby-Bauer法(K-B法)来测定,检测分离株对环丙沙星(CIP)、氯霉素(CHL)、萘啶酸(NAL)、头孢曲松(CRO)、头孢他啶(CTA)、头孢西丁(COX)、庆大霉素(GEN)、复方新诺明(SXT)、四环素(TET)、氨苄西林(AMP)、氟苯尼考(FFN)、妥布霉素(TOB)、丁胺卡那霉素(AMI)、奥格门丁(AUG)共14种抗菌药的敏感性。大肠杆菌ATCC 25922作为质控菌株,试验过程设一个平行重复,药敏试验结果所表现出的敏感(sensitive,S)、耐药(resistance,R)、中介(intermediary,I)参照CLSI 2017判定标准确定。对3类及3类以上药物产生耐药的菌株定义为多重耐药菌株(MDR)。试验所用药敏纸片选自杭州天和微生物试剂有限公司,大肠杆菌ATCC 25922质控菌株来自山东农业大学家禽疫病防控实验室。
1.5 耐药基因和Ⅰ类整合子基因盒的检测使用细菌DNA试剂盒提取分离菌株的DNA,根据GenBank已知序列和参考文献[11]设计了相关的特异性引物。通过PCR反应对分离菌中可能存在的Ⅰ类整合子可变区域基因盒和四环素类、磺胺类、氯霉素类、喹诺酮类、氨基糖苷类、β-内酰胺类等的24种耐药基因进行扩增,PCR产物用含有Dured(核酸染料)的1%琼脂糖凝胶进行电泳分析。选取部分耐药基因和所有基因盒的阳性扩增产物送上海生物工程有限公司进行测序,将所得的DNA序列使用BLAST工具与GenBank中的相关序列进行比较,进而确定Ⅰ类整合子可变区域基因盒的结构和耐药基因的种类。试验所用的DNA试剂盒选购于天根生化科技(北京)有限公司,PCR所用引物由生工生物工程(上海)有限公司合成。
1.6 多位点序列分型和系统发育分析通过参考Wirth等[12]对大肠杆菌7个看家基因(adK、fumC、gyrB、icD、mdH、purA、recA)的描述设计合成7对特异性引物,对分离菌株DNA进行特异性扩增,PCR产物进行测序。将每个菌株的7个基因序列通过BioEdit(https://www.bioedit.com)软件进行拼接、分析,获得基因序号;将7个看家基因序号输入到巴斯德在线数据库(http://enterobase.warwick.ac.uk/species/ecoli)进行在线比较,从而得出该分离株的ST型。利用BioNumerics 7.6软件(Applied Maths, Kortrijk, Belgium)对MLST分型结果构建最小生成树,探讨分离菌的亲缘关系[10]。
使用eBURST v3 (http://eburst.mlst.net/)将分离株的ST型与中国目前人源大肠杆菌的340个ST型进行对比并制成eBURST图。其中人源大肠杆菌的STs作对照,人源大肠杆菌STs数据来自EnteroBase (http://enterobase.warwick.ac.uk/species/ecoli/search_strains? query=st_search)。
根据Ooka等[13]的描述,把7个看家基因的串联序列导入MEGA 7.0软件中(http://www.mega software.net/),通过分析处理,把串联序列中所有的排列间隙进行删除,最终将串联序列中的3 423个位置作为系统发育计算的数据集。使用最大似然法(MCL)估计进化距离,采取1 000次重复的自展检验(bootstrap)验证拓补结构[14-16],最后在MEGA 7.0中构建系统发育树,为方便研究分支编号记为菌株序号-STs-整合子结构。本研究以大肠杆菌O157:H7 Sakai(GenBank:BA000007,ST11)为参考菌株[17]。为了探究相同克隆群菌株的耐药谱和耐药基因分布规律,将系统发生树导入Evolview(http://www.evolgenius.info)软件中进行修饰。
1.7 数据处理使用SAS V8软件(SAS Inc., Raleigh, N Car, USA)采用X2检验分析大肠杆菌分离株的耐药表型与对应耐药基因的相关性,P < 0.01时差异极显著,P < 0.05时差异显著。
2 结果 2.1 大肠杆菌分离鉴定为保证所采样品能够在一定程度上代表该水貂养殖场,根据养殖场分笼饲养的特点,每个笼子采集1~3份新鲜粪便,共采集82份样品。通过对每份样品进行增菌培养、选择性培养基鉴定、16S rRNA PCR检测、部分样品测序、Blast对比,82份水貂粪便样品中分离到62株大肠杆菌,分离率为75.61%(62/82)。
2.2 耐药表型分析62株大肠杆菌对14种抗菌药物的耐药率在1.61%~91.94%。氨苄西林抗生素的耐药率最高,为91.94%(57/62);耐药率超过50%的如下:四环素90.32%(56/62)、萘啶酸66.13%(41/62)、复方新诺明62.90%(39/62);对头孢西丁、丁胺卡那霉素这两种抗生素的抗性最低,耐药率分别为1.61%(1/62)、9.68%(6/62),详见图 1。分离菌中存在42种耐药谱,其中最常见的是AMP-SXT-TET(n=4,6.25%)。有2株分离菌对所检的14种抗菌药都表现为敏感,1株菌对除头孢西丁外的其他13种抗菌药都具有抗性(图 2)。多重耐药菌株(MDR)所占比率为85.48%(53/62), 在MDR中对5种抗菌药物产生抗性的比例最高,占多重耐药菌株的18.87%(10/53)。

|
AMP.氨苄西林;TET.四环素;NAL.萘啶酸;SXT.复方新诺明;CHL.氯霉素;CIP.环丙沙星;AUG.奥格门丁;TOB.妥布霉素;FFN.氟苯尼考;CTA.头孢他啶;GEN.庆大霉素;CRO.头孢曲松;AMI.丁胺卡那霉素;COX.头孢西丁 AMP. Ampicillin; TET. Tetracycline; NAL. Nalidixic acid; SXT. Compound sulfamethoxazole; CHL. Chloramphenicol; CIP. Ciprofloxacin; AUG. Ogmentin; TOB. Tobramycin; FFN. Florfenicol; CTA. Ceftazidime; GEN. Gentamicin; CRO. Ceftriaxone; AMI. Amikacin; COX. Cefoxitin 图 1 62株大肠杆菌的耐药表型 Fig. 1 Antimicrobial resistance phenotypes of 62 E. coli isolates |
|
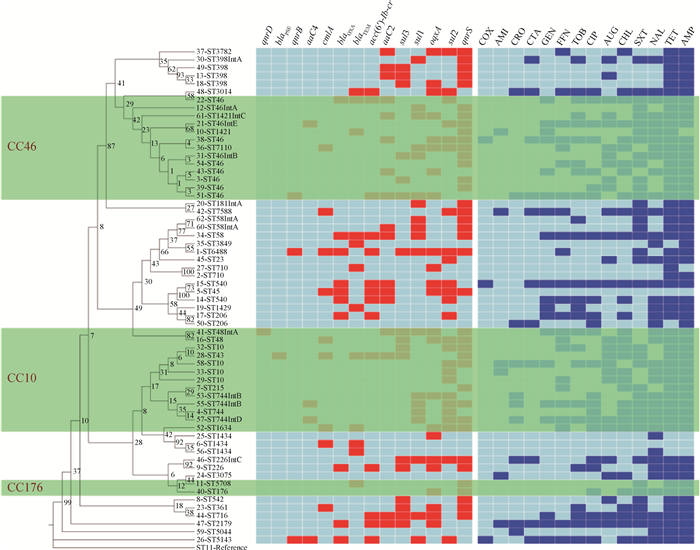
ST11为参照序列;IntA表示dfrA27-aadA2-qnrA,IntB表示dfrA17-aadA5,IntC表示dfrA1-aadA1,IntD表示dfr17-aadA5,IntE表示dfrA12-aadA2;耐药基因种类见左侧矩阵上方,下方的红色方格表示该菌株携带该抗性基因,浅蓝色方块表示不携带;抗生素类型见右侧矩阵上方,下面的蓝色方格红色方块表示菌株对抗生素有抗性,浅蓝色方块表示中介或者敏感 ST11 is the reference sequence; IntA represents dfrA27-aadA2-qnrA, IntB represents dfrA17-aadA5, IntC represents dfrA1-aadA1, IntD represents dfr17-aadA5, and IntE represents dfrA12-aadA2; the types of drug resistance genes are shown at the top of the left matrix. The red square at the bottom indicates that the strain carries the resistance gene, while the light blue square indicates that it does not. Antibiotic types are shown at the top of the matrix on the right. The red square on the blue square below indicates that the strain is resistant to antibiotics, and the light blue square indicates intermediate or sensitive 图 2 采用最大似然法对水貂源的62株大肠杆菌构建的系统发育树 Fig. 2 Phylogenetic trees of 62 E. coli strains from mink sources by maximum likelihood method |
耐药基因的检测结果显示,分离株中共检测到14种不同的耐药基因,对应耐受的药物包括磺胺类、氯霉素类、喹诺酮类、氨基糖苷类、β-内酰胺类等,其中qnrS检出率最高,为61.29%(38/62),其次为sul2(40.32%)、oqxA(37.10%)、sul1(30.64%),见图 2所示。没有检测到blaSHV、blaCMY-2、aaC1、aaC3、ant(2')、qnrA、qnrC、tetA、tetB、stCM。统计分析结果表明,分离株对庆大霉素、妥布霉素、复方新诺明、环丙沙星的耐药表型与其携带对应种类的部分耐药基因成正相关(P < 0.01)。携带耐药基因的菌株对抗菌药的耐药率和不携带耐药基因的菌株对抗菌药的耐药率相比差异显著,如表 1所示。
|
|
表 1 相关耐药基因对分离株耐药率的影响 Table 1 Effect of related drug-resistant genes on drug resistance rate of isolated strains |
水貂源所分离的62株大肠杆菌中,Ⅰ类整合子基因盒的检出率为20.97%(13/62),目的条带大小为1 369 bp。经测序、Blast对比,结果显示Ⅰ类整合子可变区域内有5种不同的基因盒结构,分别为dfrA27-aadA2-qnrA(9.68%)、dfrA17-aadA5(4.84%)、dfrA1-aadA1(3.23%)、dfr17-aadA5(1.61%)、dfrA12-aadA2(1.61%),见表 2。携带整合子基因盒的菌株都是多重耐药菌且在这些菌株中喹诺酮类和磺胺类耐药基因的携带率为100%(13/13)。
|
|
表 2 大肠杆菌MLST、血清型和Ⅰ类整合子结构的多样性分布 Table 2 Diversity profiles of E. coli isolates based on MLST, serovar and class Ⅰ Integron structure |
结果显示,所分离的62株大肠杆菌中有7株是致病性大肠杆菌,分为4种血清型(表 2)。检出率最高的是肠出血性大肠杆菌(EHEC)的O104:H4(3/62,4.84%),其次是产肠毒性大肠杆菌(ETEC)的O20:K17(2/62,3.23%)和O9:K9(1/62,1.61%),肠侵袭性大肠杆菌(EPEC)的O152:K?(1/62,1.61%),未发现肠致病性大肠杆菌(EPEC)。
2.6 MLST序列类型和克隆群分析MLST分析结果表明,62株水貂源大肠杆菌包含33个不同的等位基因谱(见表 2)。在所有的STs中,ST46为主要分型占16.13%(10/62),ST398、ST744、ST10各占6.45%(4/62);ST58、ST540、ST1434各占4.84%(3/62);ST48、ST206、ST226、ST710、ST1421各占3.23%(2/62);其余的21个STs均占1株,没有发现新的ST型,故未将测序结果上传至GenBank。使用eBurst算法的原理将18个STs分配给10个ST Cplx,15个STs没有ST Cplx,分离菌株中ST10 Cplx的占比最高为27.42%(17/62)。最小生成树结果显示,62株大肠杆菌中存在3个克隆群分别为CC10、CC46、CC176,CC10由ST10、ST43、ST48、ST215、ST744、ST1634组成,CC46由ST46、ST1421、ST7110组成,CC176由ST176和ST5708组成,见图 3。

|
每个圆圈代表一个ST型,圆圈的面积对应ST型的数量,圆圈的颜色表示STs所属的ST Cplx,灰色区域代表一个克隆群(clonal complexes);粗实线表示两个ST型相差1个等位基因,细实线表示两个ST型差两个等位基因;虚线则代表两个ST型之间相差三个或以上的等位基因 Each circle represents a ST type, and the area of the circle corresponds to the number of ST types. The color of the circle represents the ST Cplx to which STs belongs, and the gray area represents a clonal complex. The thick solid line represents one allele difference between the two ST types, and the thin solid line represents two alleles difference between the two ST types. The dotted line represents alleles with three or more differences between the two ST types 图 3 62株水貂源大肠杆菌构建的最小生成树 Fig. 3 Minimum spanning tree of 62 mink-derived Escherichia coli strains |
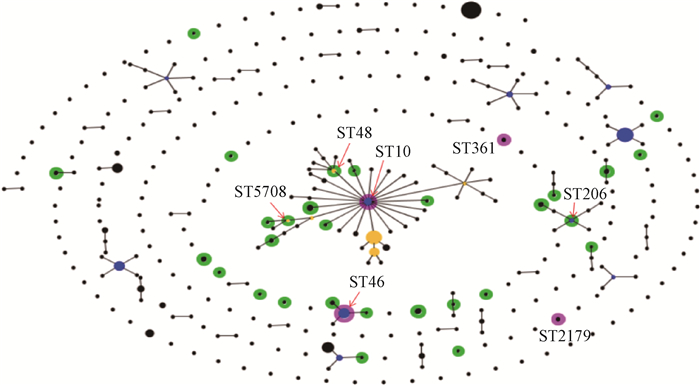
水貂源大肠杆菌分离株的33种STs与中国目前人源大肠杆菌的340种STs的比较结果显示(图 4),在MLST基因分型水平上,分离株的21个STs与中国的人源STs存在关联,其中19个STs与中国的人源STs形成9个新的克隆群(clonal complexes);水貂源STs中作为新克隆群的核心STs(the primary founder)的是ST10、ST46、ST206,成为新克隆群的亚核心STs(subgroup founder)的是ST48和ST5708,与人源STs相同的是ST10、ST46、ST361、ST2179。从系统发生树中可以看出,不同ST型的菌株,亲缘关系存在差异,同一克隆群的菌株遗传关系密切,如CC46和CC176的菌株集中分布于系统发育树中。CC10克隆群的菌株则分布在系统发育树的两个区域,ST48与克隆群里的其他菌株亲缘关系相对较远(图 2),由等位基因的差异可知,ST48和ST10之间adk基因的差异所导致的。从系统发育树中可以看出相同STs菌株或同一克隆群菌株的耐药谱组成与耐药基因携带状况存在显著差异,如39-ST46和51-ST46,38-ST46和36-ST7110,携带不同基因盒或血清型的菌株与菌株STs之间没有明显相关性。
|
不同颜色的点表示两个数据集中STs的不同关系,粉红色表示该ST型在两个数据集中都存在,绿色表示ST仅在水貂源STs中存在,蓝色表示克隆群的主要ST核心序列型(the primary founder),黄色表示克隆群的亚核心序列型(subgroup founder),黑色表示参考数据集中其他的STs The different colored dots indicate the different relationship between STs in both datasets. Pink indicates that the ST type exists in both datasets, green indicates that ST only exists in the mink source STs, blue indicates the primary founder of the clone group, yellow indicates the subgroup founder of the clone group, and black indicates other STs in the reference data set 图 4 以我国人源大肠杆菌STs数据集为对照构建的分离菌STs的eBURST图 Fig. 4 eBURST map of isolated bacteria STs contrasted with STs data set of human E. coli in China |
大肠杆菌分布广泛,在一定条件下可引起动物及人类的腹泻、尿路感染和败血症等疾病[18],特别是一些致病性的大肠杆菌不仅对畜牧业的发展造成危害, 而且会威胁到人类健康。国内对水貂源大肠杆菌的相关研究还比较少。在本试验中,82份水貂源粪便样品检出62株大肠杆菌,分离率为75.61%,高于Oliveira等[19]在巴西圣保罗州Alto Paranapanema河流域水貂粪便中大肠杆菌30%的分离率。近年来的流行病学调查显示,各地区流行的优势血清型差异较大,血清型的多样性给该病的防治带来极大困难[5]。有关报道显示,近年来山东地区E. coli的主要血清型有O78、O35、O18[20-21],本研究中水貂源致病性E. coli的检出率为11.29%(7/62),其中出血性大肠杆菌O104:H4为优势致病性血清型,值得关注的是该血清型具有多种毒力因子,包括产志贺毒素大肠杆菌(STEC)和肠聚集性大肠杆菌(EAEC)[22],曾引起德国地区暴发大范围的传染病,它能够在易感人群引发高频率溶血性尿毒症综合征(HUS),因而成为公众健康的重大威胁[23]。结合药敏试验结果发现,本研究中分离到的致病性血清型菌株都具有多重耐药性,不同菌株之间的耐药谱差异明显,这说明水貂养殖场粪便中不仅携带潜在的致病因子,而且存在着威胁人类健康的致病菌,因此需要对养殖场的粪便进行及时处理,防止病原菌扩散传播,同时大力加强养殖场病原菌的监测和防控。
近几十年以来,随着抗生素的滥用,导致高抗药或多重耐药的致病性大肠杆菌不断被检出。2010年英国医学杂志《柳叶刀》发表有关产NDM-1细菌的相关研究报道,引起人们的高度关注,该细菌可编码Ⅰ型金属β-内酰胺酶并同时含有多种耐药基因,从而导致对几乎所有抗生素都具有耐药性,感染者死亡率高,故称作超级细菌[24]。细菌的耐药性已经对公共卫生安全造成严重威胁,本研究药敏试验结果显示,85.48%分离菌株为多重耐药菌株(MDR),高于家兔的50.9%[11]和丹麦水貂的60%[25],说明该水貂养殖场的大肠杆菌耐药现象严重。通过与Radhouani等[26]的研究相对比,发现养殖场毛皮动物所分离的大肠杆菌对相关抗生素的耐药表现远远高于野生毛皮动物的耐药表现,说明水貂养殖场大肠杆菌的流行情况更应引起关注。在我国养殖业中头孢类抗生素没有被广泛的应用,但本试验检测出耐第二代、三代头孢类的菌株,也有在畜牧养殖业中发现具有耐头孢类抗生素菌株的报道[27],值得引起重视。
造成细菌耐药的机制有很多,其中包括耐药基因和整合子介导的耐药机制[11]。分离菌中耐药基因的检测结果表明携带编码超广谱β-内酰胺酶(ESBLs)相关联的基因包括blaTEM(19.35%)、blaOXA(16.13%)、blaPSE(1.61%),这些β-内酰胺类耐药基因被认为是引起菌株对氨苄西林和头孢菌素类抗生素产生耐药的常见机制[11],但本次研究中携带β-内酰胺类耐药基因的菌株与氨苄西林和头孢菌素类耐药无关联(P>0.05),这可能与基因的表达状态以及在抗生素的选择压力下细菌产生抗性机制的多样性等多种因素有关。据报道,四环素类耐药基因tetA和tetB与四环素的抗性有关[28],本试验四环素的耐药率为90.32%,虽然没检测到tetA和tetB,但是四环素类耐药基因有多种tet(A~M)很有可能是由四环素类其他耐药基因所引起。统计分析结果显示,本试验菌株携带氨基糖苷类的aaC2和aaC4耐药基因,磺胺类的sul1基因以及喹诺酮类的aac(6')-Ib-cr基因与对应种类的耐药表型之间呈正相关(P < 0.01),但其他耐药基因表现为没有关联,这说明,分离株对抗生素的耐药机制复杂多样,在研究细菌耐药性应综合考量多方面因素。整合子可引起肠杆菌科细菌产生多重耐药性,它能同时捕获多个外源耐药基因并整合进质粒或转座子基因组中使之转变为功能性基因的表达单位;携带耐药基因的整合子可随着质粒或者转座子在肠杆菌科细菌间水平传播[29],根据整合酶的不同将整合子分为三种,其中Ⅰ类整合子最为常见。Ⅰ类整合子检测结果显示,在62个分离株中检测到13个携带基因盒的菌株,在Ⅰ类整合子可变区域发现5种不同的基因盒结构,存在5个可转移的DHFR基因盒,分别是dfrA1、dfrA12、dfr17、dfrA17和dfrA27,这些基因盒编码甲氧苄啶抗性基因;3个可转移的编码氨基糖苷类抗性基因的AAD基因盒,分别是AADA1、AADA2和AADA5;1个qnra基因盒可编码为喹诺酮类耐药基因,值得注意的是,Ⅰ类整合子通常与MDR大肠杆菌分离株相关,这与本研究的结果一致。分离菌中耐药基因和Ⅰ类整合子基因盒的携带特点可能与饲养环境,物种差异以及地区差异有关。
在群体遗传学研究中,越来越多地使用基因分型的方法来描述细菌病原体的特征[30]。MLST作为一种有效的细菌基因分型方法,可以反映细菌种群内的进化关系,由于实验具有高度重复性和可比性,与PFGE相比更易于对菌株的亲缘关系和微生物遗传学进行分析,可用于大范围的流行病学调查[31]。如大肠杆菌ST131分离株通常被报道可产生超广谱β-内酰胺酶,比如CTX-M-15,几乎所有对氟喹诺酮类药物有抵抗力,由于其引起的感染谱和含有大量毒力相关基因,也被认为是致病菌[30]。本试验MLST基因分型结果显示,62株分离菌分为33个ST型,优势ST型为ST46,存在3个克隆群,分别为CC10、CC46、CC176,形成克隆群的菌株占比分别为45.16%(28/62);其中ST10菌株与动物肠道的定殖有关,一般只与低毒力和抗生素敏感性存在相关性,很少有ST10的大肠杆菌菌株与动物和人类疾病存在关联[32]。通过对系统发育树结果的研究,笔者发现在系统发育树的同一区域分布着相同的ST菌株,它们之间的关系最为密切,如ST10、ST46和ST1434;同一个CC的大多数菌株集中在系统发生树的同一区域,但CC10中的ST48与CC10中的其他菌株相差甚远,我们推测可能是由ST48和ST10之间adk基因的差异所导致的;因此可以说明与聚类分析图相比系统发育树更能准确地展示菌株间的亲缘关系。通过对系统发育树上不同遗传关系菌株的耐药谱和耐药基因谱进行观察,可以看出不同血清型和携带整合子菌株在系统发生树中的分布没有明显的规律性。从系统发生树的整体来看,相同克隆群的菌株其耐药谱的组成与耐药基因的携带类型存在明显差异。Clermont等[9]利用MLST的数据分析表明,动物和人类的许多致病菌株密切相关,具有共同的遗传背景。通过与来自中国的340株人源大肠杆菌STs相比,我们可以看出水貂中4个STs与人源STs相同,17个STs与人源STs亲缘关系密切,感兴趣的是水貂源中部分STs(如ST206、ST48)与人源STs形成新的克隆群并成为核心序列类型的有力候选。结果表明,部分水貂源大肠杆菌与中国的人源大肠杆菌密切相关,这些STs与腹泻、败血症等疾病存在一定的关联[33]。因此,为了防止对公众健康的威胁,需要对水貂养殖场粪便中的大肠杆菌进行持续监测。
4 结论从诸城地区分离的水貂源大肠杆菌对氨苄西林和四环素的耐药情况最为严重,耐药基因和Ⅰ类整合子分布广泛。分离菌中ST型复杂多样,其中ST46为主要基因型,与致病性相关菌株的ST型与人源大肠杆菌具有共同的遗传背景。养殖场的水貂受到致病性和多重耐药性大肠杆菌的污染,相同克隆群菌株的耐药基因分布具有多态性以及部分表观特征差异明显。
| [1] |
陈晨, 李朋, 韩铠怿, 等. 貂源绿脓杆菌的分离鉴定及其致病性[J]. 兽类学报, 2015, 35(2): 196–202.
CHEN C, LI P, HAN K Z, et al. Identification and Pathogenicity test of Pseudomonas aeruginosa from mink[J]. Acta Theriologica Sinica, 2015, 35(2): 196–202. (in Chinese) |
| [2] |
钟世勋, 迟珊珊, 王振, 等. 山东规模化养殖场毛皮动物多重感染病原学分析[J]. 中国兽医学报, 2014, 34(11): 1770–1777.
ZHONG S X, CHI S S, WANG Z, et al. The etiology analysis of multiple infections in Shandong scale fur farms[J]. Chinese Journal of Veterinary Science, 2014, 34(11): 1770–1777. (in Chinese) |
| [3] |
芮萍, 刘曜综, 马增军, 等. 貂源伪狂犬病病毒的分离鉴定及gE基因分子特征[J]. 畜牧兽医学报, 2015, 46(7): 1268–1272.
RUI P, LIU Y Z, MA Z J, et al. Identification and glycoprotein e characteristic of pseudorabies virus from mink[J]. Acta Veterinaria et Zootechnica Sinica, 2015, 46(7): 1268–1272. (in Chinese) |
| [4] |
王春梅, 何启盖, 操继跃. 细菌多重耐药泵的研究进展[J]. 畜牧兽医学报, 2011, 42(4): 455–467.
WANG C M, HE Q G, CAO J Y. Multidrug efflux systems involved in resistance to antibacterial drugs[J]. Acta Veterinaria et Zootechnica Sinica, 2011, 42(4): 455–467. (in Chinese) |
| [5] |
冯敏燕, 林树乾, 房玉波, 等. 山东省鸡源致病性大肠杆菌O-抗原混合血清型分析[J]. 中国兽医学报, 2017, 37(9): 1676–1679.
FENG M Y, LIN S Q, FANG Y B, et al. Analysis of mixed O-antigen serotypes of E. coli isolated from chickens in Shandong province[J]. Chinese Journal of Veterinary Science, 2017, 37(9): 1676–1679. (in Chinese) |
| [6] |
金美伶, 白雪, 王峰, 等. 水貂大肠杆菌性腹泻的诊断与治疗[J]. 中国兽医杂志, 2015, 51(10): 45–46.
JIN M L, BAI X, WANG F, et al. Diagnosis and treatment of diarrhea caused by Escherichia coli in mink[J]. Chinese Journal of Veterinary Medicine, 2015, 51(10): 45–46. DOI: 10.3969/j.issn.0529-6005.2015.10.014 (in Chinese) |
| [7] |
田甜甜.环境多重耐药细菌中分子遗传特征及耐药传播机制研究[D].新乡: 河南师范大学, 2017.
TIAN T T. Molecular characterization and transmission of antibiotic resistance in environmental multidrug resistant bacteria[D]. Xinxiang: Henan Normal University, 2017. (in Chinese) http://cdmd.cnki.com.cn/Article/CDMD-10476-1017120734.htm |
| [8] | KOCZURA R, MOKRACKA J, BARCZAK A, et al. Association between the presence of class 1 integrons, virulence genes, and phylogenetic groups of Escherichia coli isolates from river water[J]. Microb Ecol, 2013, 65(1): 84–90. |
| [9] | CLERMONT O, OLIER M, HOEDE C, et al. Animal and human pathogenic Escherichia coli strains share common genetic backgrounds[J]. Infect, Genet Evol, 2011, 11(3): 654–662. DOI: 10.1016/j.meegid.2011.02.005 |
| [10] |
王龙光, 姜雯, 逄春华, 等. 利用MLST技术探讨不同地区致病性耐药鸡源大肠杆菌的遗传进化关系[J]. 中国兽医学报, 2017, 37(9): 1680–1686.
WANG L G, JIANG W, PANG C H, et al. Genetic evolutionary relationship of drug-resistant and pathogenic chicken originated Escherichia coli strains from different regions with MLST[J]. Chinese Journal of Veterinary Science, 2017, 37(9): 1680–1686. (in Chinese) |
| [11] | ZHAO X N, YANG J, JU Z J, et al. Molecular characterization of antimicrobial resistance in Escherichia coli from rabbit farms in Tai'an, China[J]. BioMed Res Int, 2018, 2018: 8607647. |
| [12] | WIRTH T, FALUSH D, LAN R, et al. Sex and virulence in Escherichia coli:an evolutionary perspective[J]. Mol Microbiol, 2006, 60(5): 1136–1151. DOI: 10.1111/j.1365-2958.2006.05172.x |
| [13] | OOKA T, SETO K, KAWANO K, et al. Clinical significance of Escherichia albertii[J]. Emerg Infect Dis, 2012, 18(3): 488–492. DOI: 10.3201/eid1803.111401 |
| [14] | EVANGELISTA D, THOUZÉ F, KOHLI M K, et al. Topological support and data quality can only be assessed through multiple tests in reviewing Blattodea phylogeny[J]. Mol Phylogenet Evol, 2018, 128: 112–122. DOI: 10.1016/j.ympev.2018.05.007 |
| [15] | TELLES G P, ARAÚJO G S, WALTER M E M T, et al. Live neighbor-joining[J]. BMC Bioinformatics, 2018, 19: 172. DOI: 10.1186/s12859-018-2162-x |
| [16] | TRUCCO M I, BURATTI C C. Taxonomic review of Argentine mackerel Scomber japonicus (Houttuyn, 1782) by phylogenetic analysis[J]. Mol Biol Res Commun, 2017, 6(3): 141–152. |
| [17] | JI X W, LIAO Y L, ZHU Y F, et al. Multilocus sequence typing and virulence factors analysis of Escherichia coli O157 strains in China[J]. J Microbiol, 2010, 48(6): 849–855. DOI: 10.1007/s12275-010-0132-8 |
| [18] | LEWIS Ⅱ J S, HERRERA M, WICKES B, et al. First report of the emergence of CTX-M-type extended-spectrum β-lactamases (ESBLs) as the predominant ESBL isolated in a U. S. health care system[J]. Antimicrob Agents Chemother, 2007, 51(11): 4015–4021. DOI: 10.1128/AAC.00576-07 |
| [19] | OLIVEIRA M, FREIRE D, PEDROSO N M. Escherichia coli is not a suitable fecal indicator to assess water fecal contamination by otters[J]. Braz J Biol, 2017, 78(1): 155–159. DOI: 10.1590/1519-6984.167279 |
| [20] |
李莎莎.山东省禽源大肠杆菌血清型鉴定及喹诺酮类药物耐药性研究[D].泰安: 山东农业大学, 2010.
LI S S. Fluoroquinolones resistance and serogroups of Escherichia coli from Shandong province of China[D]. Tai'an: Shandong Agricultural University, 2010. (in Chinese) http://cdmd.cnki.com.cn/Article/CDMD-10434-1011019430.htm |
| [21] |
潘鑫.山东省高密地区鸭源大肠杆菌的分离鉴定及耐药基因检测[D].泰安: 山东农业大学, 2012.
PAN X. Isolation, identification and detection of drug resistance genes of duck E. coli isolates from Gaomi in Shandong province[D]. Tai'an: Shandong Agricultural University, 2012. (in Chinese) |
| [22] | SAFADI R A, ABU-ALI G S, SLOUP R E, et al. Correlation between in vivo biofilm formation and virulence gene expression in Escherichia coli O104:H4[J]. PLoS One, 2012, 7(7): e41628. DOI: 10.1371/journal.pone.0041628 |
| [23] | DEVAUX I, VARELA-SANTOS C, PAYNE-HALLSTRÖM L, et al. Investigation of travel-related cases in a multinational outbreak:example of the Shiga-toxin producing E. coli outbreak in Germany, May-June 2011[J]. Epidemiol Infect, 2015, 143(16): 3468–3474. DOI: 10.1017/S0950268814003823 |
| [24] | KUMARASAMY K K, TOLEMAN M A, WALSH T R, et al. Emergence of a new antibiotic resistance mechanism in India, Pakistan, and the UK:a molecular, biological, and epidemiological study[J]. Lancet Infect Dis, 2010, 10(9): 597–602. DOI: 10.1016/S1473-3099(10)70143-2 |
| [25] | PEDERSEN K, HAMMER A S, SØRENSEN C M, et al. Usage of antimicrobials and occurrence of antimicrobial resistance among bacteria from mink[J]. Vet Microbiol, 2009, 133(1-2): 115–122. DOI: 10.1016/j.vetmic.2008.06.005 |
| [26] | RADHOUANI H, IGREJAS G, GONÇALVES A, et al. Antimicrobial resistance and virulence genes in Escherichia coli and enterococci from red foxes (Vulpes vulpes)[J]. Anaerobe, 2013, 23: 82–86. DOI: 10.1016/j.anaerobe.2013.06.013 |
| [27] | ROTIMI V O, JAMAL W, PAL T, et al. Emergence of CTX-M-15 type extended-spectrum β-lactamase-producing Salmonella spp. in Kuwait and the United Arab Emirates[J]. J Med Microbiol, 2008, 57(7): 881–886. DOI: 10.1099/jmm.0.47509-0 |
| [28] | BRYAN A, SHAPIR N, SADOWSKY M J. Frequency and distribution of tetracycline resistance genes in genetically diverse, nonselected, and nonclinical Escherichia coli Strains isolated from diverse human and animal sources[J]. Appl Environ Microbiol, 2004, 70(4): 2503–2507. DOI: 10.1128/AEM.70.4.2503-2507.2004 |
| [29] | GILLINGS M R. Class 1 integrons as invasive species[J]. Curr Opin Microbiol, 2017, 38: 10–15. DOI: 10.1016/j.mib.2017.03.002 |
| [30] | NICOLAS-CHANOINE M H, BERTRAND X, MADEC J Y. Escherichia coli ST131, an intriguing clonal group[J]. Clin Microbiol Rev, 2014, 27(3): 543–574. DOI: 10.1128/CMR.00125-13 |
| [31] | LARSEN M V, COSENTINO S, RASMUSSEN S, et al. Multilocus sequence typing of total-genome-sequenced bacteria[J]. J Clin Microbiol, 2012, 50(4): 1355–1361. DOI: 10.1128/JCM.06094-11 |
| [32] | MANGES A R, JOHNSON J R. Food-borne origins of Escherichia coli causing extraintestinal infections[J]. Clin Infect Dis, 2012, 55(5): 712–719. DOI: 10.1093/cid/cis502 |
| [33] | CHEN Y, CHEN X, ZHENG S, et al. Serotypes, genotypes and antimicrobial resistance patterns of human diarrhoeagenic Escherichia coli isolates circulating in southeastern China[J]. Clin Microbiol Infect, 2014, 20(1): 52–58. DOI: 10.1111/1469-0691.12188 |



