2. 国家民族事务委员会青藏高原动物疫病防控创新团队, 成都 610041
2. Animal Disease Prevention and Control Innovation Team in the Qinghai-Tibet Plateau of State Ethnic Affairs Commission, Chengdu 610041, China
藏猪是青藏高原特有的高原型地方猪种,生活在青藏高原海拔2 500~4 300 m的农区和半农半牧区,属放牧饲养较原始的瘦肉型小型猪种,对高海拔恶劣气候有极强的适应性,抗病耐粗[1]。藏猪常年野外放牧,与牛羊混群饲养过程中,各畜种之间相互接触,一定程度上为病毒的交叉感染提供了契机[2]。同时,在日常放牧过程中,与人群的密切接触,也有可能会造成一些人畜共患传染病在人畜之间循环传播。
腹泻在藏猪群体中普遍存在,造成了一定的经济损失。能引起藏猪腹泻的病毒,目前已报道有流行性腹泻病毒(porcine epidemic diarrhea virus,PEDV)[3]和猪丁型冠状病毒(porcine deltacoronavirus,PDCoV)[4],是否感染其他病毒尚不清楚。猪腹泻可大幅度增加仔猪的死亡率和发病率,特别是在集约化猪场,造成全世界养猪业的重大经济损失。多种病毒可以引起猪腹泻,包括PEDV、猪轮状病毒(porcine rotavirus,PoRV)、猪传染性胃肠炎病毒(transmissible gastroenteritis virus of swine,TGEV)等。近些年来,仔猪养殖场暴发了与腹泻有关的新病毒病[5],相关病毒如博卡病毒(Bocavirus)[6]、库布病毒(Kubovirus)[7]、杯状病毒(calicivirus)[8]、萨佩罗病毒(Sapelovirus)[9]和星形病毒(astrovirus,AstV)[10]等,是否存在新病毒引起猪腹泻还有待进一步研究,新病毒是否在藏猪中流行也值得重点关注。
病毒宏基因组学是一种新兴的病毒组学研究技术,从样本中直接获得所有病毒的核酸,通过建库和高通量测序,与病毒数据库比对,从而无偏差地鉴定出所有的病毒种类。该技术突破了传统技术的局限,具有从混合感染的样本中鉴定出全部病毒的种类,发现新病原,监控病毒遗传变异等优势[11]。近年来基于高通量测序对自然环境、人和动物的病毒组学分析已经成为一个高效的工具[12-15]。最近,有学者运用病毒宏基因组学方法对我国猪腹泻粪便病毒种群进行了分析[16-18]。为了解藏猪腹泻粪便病毒组成,本研究从青藏高原地区的16个藏猪场采集藏猪腹泻粪便样本运用病毒宏基因组学技术进行分析,为藏猪腹泻的防治提供一定的参考依据。
1 材料与方法 1.1 样本采集从青藏高原地区的16个猪场采集藏猪腹泻粪便样本146份。采集过程中均使用一次性用具以避免样本交叉污染,冻存于-80 ℃。
1.2 试剂与材料Quick Taq HS DyeMix(含Taq酶、dNTP、Mg2+)购自东洋纺(上海)生物科技有限公司;反转录试剂盒、核酸消化酶(DNA和RNA消化酶)和Marker DL2000均购自大连宝生物工程有限公司;QIAamp Viral RNA Mini Kit购自QIAGEN公司;高速冷冻离心机Sorvall ST 8R购自ThermoFisher Scientific公司;普通PCR仪、核酸电泳仪和凝胶成像系统均购自Bio-Rad公司。
1.3 样本的处理及总核酸的提取根据已报道的方法[16]对临床样本进行处理。简述如下:每份粪便样本各取50 μL上清液组装成一个pool样。用一次性0.22 μm过滤器过滤pool样,目的是除去完整的细菌和大的细胞碎片。将滤液用10 U的DNase酶和1.5 μg的RNase酶在37 ℃下单独处理90 min以除去游离的核酸。使用QIAamp Viral RNA Mini Kit提取合并后粪便样本的总RNA并逆转录得到cDNA,送华大基因科技有限公司建库测序。
1.4 建库和TruSeq Illumina测序通过Qubit分光光度计初步测定pool样中cDNA的质量,利用TruSeq RNA样品制备试剂盒将cDNA制备物用于单独构建文库。简述如下;利用超声法将大片段cDNA随机打断以产生长度小于500 bp的片段,使用T4多核苷酸激酶、T4 DNA Polymerase和Klenow DNA Polymerase对打断后形成的cDNA片段进行末端修复,通过3'端增加的碱基“A”作为衔接子,使DNA片段能与带“T”碱基的特殊接头连接,再用琼脂糖凝胶电泳回收目的片段连接产物,然后对两端带有接头的DNA片段进行PCR扩增,最后将得到的片段加载HiSeq 4000上进行测序。
1.5 数据分析、序列组装及系统发育分析对HiSeq 4000生成的原始序列读长(reads)进行筛选,将Raw data使用fastp软件进行质控和过滤低质量的数据。针对clean reads中非病毒的部分,删除用NCBI refseq数据库中比对得到的宿主、细菌或真菌等序列。通过数据处理程序留下有用的序列,这些序列分别使用BLASTn、BLASTx、NCBI核酸序列和氨基酸序列数据库中的序列进行比对,选择具有最佳BLAST值的序列,并用于进一步分组分析。利用SOAP比对软件对病毒的含量进行扫描。同时,使用SOAP组装软件对所有不低于序列相似性90%标准的重叠群序列(contigs)从头组装成更大的重叠群或基因组序列。使用BLASTN和BLASTP算法将组装好的病毒核苷酸和氨基酸序列与NCBI数据库进行比对,以计算它们的百分比恒等式。利用MUSCLE v3.7构建完整或近似全长的病毒基因组序列的多核酸序列比对。最后利用MEGA 7.0软件,采用最大似然法建立系统发生树,并基于1 000个自举复制进行树拓扑评价。
2 结果 2.1 Illumina测序结果概述通过高通量测序,从藏猪腹泻粪便的pool样本中获得884 471 reads(GenBank: SRR9276460),与哺乳动物相关病毒reads占比为7.2%,经NCBI比对,总共获得12 291 reads(可以在NCBI比对到哺乳动物相关病毒序列)。用BLASTX软件比对,结果发现,藏猪腹泻粪便病毒种群包括11个病毒科(图 1A),按病毒序列占所有reads百分比排列顺序如下:小环状DNA病毒科(Smacoviridae)31.09%,细小病毒科(Parvoviridae)21.50%,Genomoviridae 11.77%,戊型肝炎病毒科(Hepeviridae)10.58%,腺病毒科(Adenoviridae)10.20%,小RNA病毒科(Picornaviridae)8.30%,环状病毒科(Circoviridae)2.82%,星状病毒科(Astroviridae)2.33%,冠状病毒科(Coronaviridae)0.81%,黄病毒科(Flaviviridae)0.33%,杯状病毒科(Caliciviridae)0.27%;对应19种病毒(图 1B),包括猪粪相关环状病毒7型(porcine stool-associated circular virus type 7)31.09%,猪博卡病毒(porcine bocavirus,PBoV)16.80%,双环病毒(gemycircularvirus)11.77%,戊型肝炎病毒(hepatitis E virus,HEV)10.58%,猪腺病毒(porcine adenovirus)10.20%,肠道病毒(enterovirus)4.46%,Sapelovirus 3.04%,猪圆环病毒2型(porcine circovirus type 2,PCV-2)2.82%,猪细小病毒6型(porcine parvovirus type 6,PPV-6)2.51%,AstV 2.33%,PPV-2 1.33%,porcine bufavirus 0.86%、PEDV 0.81%,波萨病毒(Posavirus)0.41%,牛病毒性腹泻病毒1型(bovine viral diarrhea virus type 1,BVDV-1)0.33%,猪库布病毒(porcine Kobuvirus,PKV)0.29%,萨波病毒(Sapovirus)0.27%,rabovirus 0.08%,pasivirus 0.02%。
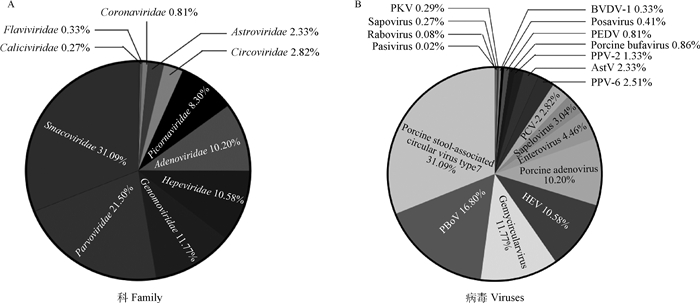
|
图 1 腹泻藏猪粪便样本中检测到的病毒的序列分类 Fig. 1 Sequence classification of viruses detected in fecal samples of diarrheal Tibetan pig |
藏猪腹泻粪便病毒种群以DNA病毒为主,其中DNA病毒占77.38 %,包括porcine stool-associated circular virus type 7、PBoV、gemycircularvirus、porcine adenovirus、PCV-2、PPV-6、PPV-2和porcine bufavirus 8种,分属于Smacoviridae、Parvoviridae、Genomoviridae、Adenoviridae和Circoviridae 5个病毒科;RNA病毒占22.62%,包括HEV、enterovirus、Sapelovirus、AstV、PEDV、Posavirus、BVDV-1、PKV、Sapovirus、rabovirus和pasivirus 11种,分属于Hepeviridae、Picornaviridae、Astroviridae、Coronaviridae、Flaviviridae和Caliciviridae 6个病毒科。
2.2 4种病毒基因序列组装及系统发育分析使用SOAP组装软件对19种病毒的contigs进行组装,从中使用相应的病毒序列contigs组装出3个完整或接近全长的基因组序列,包括PPV-6、PCV-2、PBoV-2,组装出HEV完整的ORF2基因序列,对PPV-6、PCV-2、PBoV-2和HEV ORF2进行遗传进化分析。
2.2.1 PPV-6在粪便样本中检测到总共309条具有对应于PPV-6的序列同一性的序列reads,并组装接近完整的基因组序列,长度为5 982 bp,覆盖PPV-6参考毒株全基因组序列96.5%,GC含量为47.17%。与23株PPV-6 GenBank参考病毒序列相比,PPV-6的核苷酸相似性为97.1%~100%。系统发育分析表明(图 2A),本研究中的PPV-6与Br/Brazil/2016株聚为一簇。
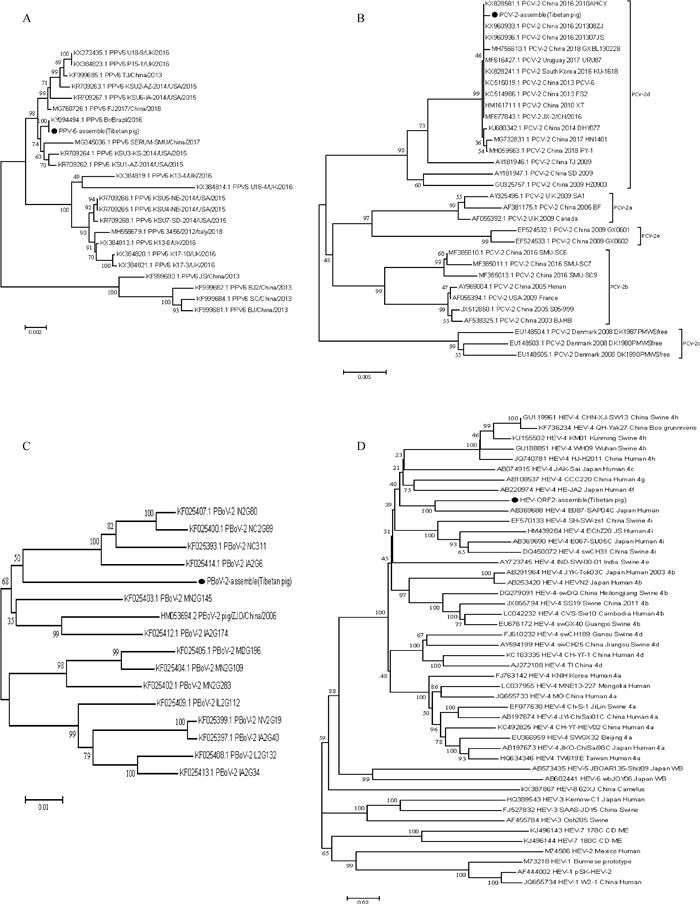
|
图 2 拼接序列系统发育分析(本研究组装的病毒序列用黑点标记) Fig. 2 Phylogenetic analysis of assembled sequences (The assembled viral sequences in this study are marked by black spot) |
在粪便样本中检测到总共346条具有对应于PCV-2的序列同一性的序列reads,并组装了完整的基因组序列,长度为1 767 bp, 覆盖PCV-2参考毒株全基因组序列100%,GC含量为48.27%。与28株PCV-2 GenBank参考病毒序列相比,PCV-2的核苷酸相似性为95.4%~100%。系统发育分析表明(图 2B),本研究中的PCV-2与2010AHCY株聚为一簇,位于PCV-2d亚群中。
2.2.3 PBoV-2在粪便样本中检测到总共2 065条具有对应于PBoV-2的序列同一性的序列reads,并组装了近似完整的PBoV-2基因组序列,长度为5 091 bp,覆盖PBoV-2参考毒株全基因组序列98.60%,GC含量为53.98%。与15株PBoV-2 GenBank参考病毒序列相比,PBoV-2的核苷酸相似性为89.5%~92.4%。系统发育分析表明(图 2C),本研究中的PBoV-2单独成一支,可能为新的基因型。
2.2.4 HEV在粪便样本中检测到总共1 300条具有对应于HEV的序列同一性的序列reads,并组装完整的HEV ORF2基因序列,长度为2 025 bp,覆盖HEV参考毒株ORF2基因序列100%,GC含量为53.28%。与45株HEV GenBank参考病毒序列相比,与HEV-4 ORF2基因的核苷酸相似性为85.9%~91.3%,与其他HEV ORF2基因的核苷酸相似性为77.4%~80.1%。系统发育分析表明(图 2D),本研究中的HEV与HEV-4 E087-SAP04C Japan Human聚为一支,提示HEV有可能在藏猪与人之间循环。
2.3 样本中PCV-2检测及全基因分析本研究首次在藏猪中发现PCV-2的存在,说明藏猪也可感染PCV-2。为进一步探明PCV-2在藏猪中检出率,根据本实验室报道的方法[19],对146份藏猪腹泻样本进行PCV-2检测,检出率为10.96%(16/146,95% CI: 6.4%~17.2%)。为进一步了解其分子特征,参考已发表文献[19]全基因扩增引物,对16份PCV-2阳性藏猪腹泻样本进行了全基因序列扩增,获得的三株病毒全基因组命名为SMU-TP-L1、SMU-TP-J1和SMU-TP-J6,全长均为1 767 bp,GenBank登录号为MK990814~MK990816。进一步进行序列相似性分析和遗传进化分析(图 3)。
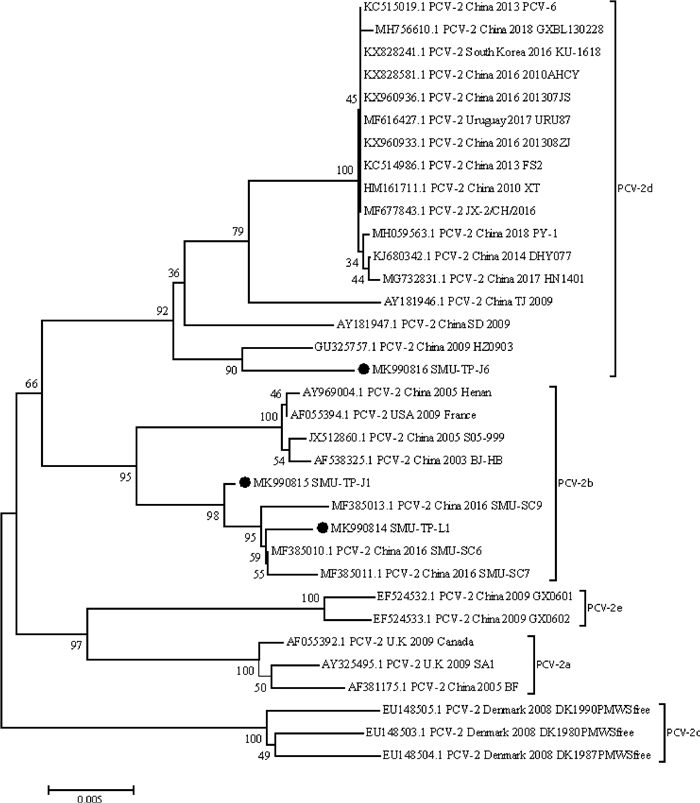
|
图 3 PCV-2全基因遗传进化分析(本研究病毒序列用黑点标记) Fig. 3 PCV-2 complete genetic evolution analysis (The viral sequences in this study are marked by black spots) |
通过Megalign软件分析,SMU-TP-L1、SMU-TP-J1和SMU-TP-J6之间的核苷酸相似性在98.4%~99.5%,与参考毒株的核苷酸相似性在96.9%~99.9%。运用MEGA 7.0软件构建遗传进化树(图 3),结果表明:SMU-TP-L1和SMU-TP-J1为PCV-2b型,SMU-TP-J6为PCV-2d型。如图 3所示,SMU-TP-L1和SMU-TP-J1与SMU-SC6株、SMU-SC7株和SMU-SC9株聚为一小支,SMU-SC6株、SMU-SC7株和SMU-SC9株为本实验室分离得到的四川流行株[19],这表明SMU-TP-L1和SMU-TP-J1与四川流行株较近亲缘关系;SMU-TP-J6与HZ0903株聚为一支,亲缘关系最近。
3 讨论目前病毒宏基因组技术广泛应用于病毒学领域研究,能清晰地描绘出各种特定环境中的病毒谱,有助于很好地了解自然界中病毒的分布动态,实时监测一些致病性病毒和潜在致病性病毒的变化情况,也成为挖掘潜在新发病毒的工具,该技术将是人类和动物预防、诊断和治疗新发病毒性传染病的最有效的途径。2002年,Breitbart等[20]首次用该技术分析加州海岸表层海水的病毒群落,发现海水中的病毒以噬菌体为主,开启了病毒宏基因组学技术无偏差鉴定病原的先河。2005年,Edwards与Rohwer[21]首次提出病毒宏基因组学这一概念。2007年,Delwart[22]阐述了利用病毒宏基因组学技术挖掘人和动物体内的新病毒群落。燕超等[23]在桂越边境采集蝙蝠样本并对其进行病毒宏基因组学分析。从青藏高原地区的16个藏猪场采集藏猪腹泻粪便样本进行检测,检测出分属于11个科的19种病毒,在内地猪腹泻粪便中检测出分属于8个科的15种病毒[16],这说明藏猪腹泻粪便样本病毒种类和群落组成更为复杂。在检测出的19种病毒中已经证实可以引起腹泻的病原有PEDV、PCV-2和BVDV-1,与腹泻相关的病毒主要是以小DNA病毒为主,这可能是引起藏猪腹泻相关病毒的一个特征,有待进一步证实。BVDV-1多发于牛,这一现象也佐证了藏猪与牛羊混群饲养造成交叉感染的可能性;拼接获得的HEV片段与人源HEV毒株亲缘关系最近,说明藏猪与人有相互感染的可能性,具有重要的公共卫生学意义。
通过病毒宏基因组学也发现3种可能导致藏猪腹泻的新发病毒,分别是swine pasivirus(SPaV)、bufavirus(BuV)和rabovirus。SPaV是在法国健康猪粪便中首次检测到的一种新病毒,属于小RNA病毒科[24]。SPaV感染仔猪不表现症状,有学者在罗马尼亚东部集约化养猪场调查发现,在粪便中检出率为37%(30/82),保育猪感染率高达50%[25],但是SPaV与腹泻之间的联系还有待进一步研究。最近在我国发现的一种新型病毒PLV-CHN(parechovirus-like virus),与法国SPaV-1具有82%的核苷酸相似性,被认为是pasivirus的不同基因型[26],这说明SPaV已经在中国流行。BuV有一个单链DNA基因组,为细小病毒科病毒[27]。目前在犬、猪等不同动物和人粪便样本中均有检出,Chieochansin等[28]报道在人腹泻样本检出率为(4/1 495, 0.27%),而健康样本没有检出(0/726);有报道称在犬腹泻粪便中检出率为42.15%[29];Liu等[30]对60份猪腹泻粪便样本进行了BuV检测,检出率为13.3%。以上结果说明BuV与腹泻有一定的联系。Rabovirus是一种新的小RNA病毒科病毒,在遗传基础上与enterovirus和Sapelovirus有关[31],有研究表明enterovirus和Sapelovirus与内地猪腹泻相关[16]。以上结果表明,上述三种病毒均与藏猪腹泻有一定联系,对于新发病毒在藏猪中流行情况和分子特征有待一步研究。
PCV-2为单股环状DNA病毒,是目前发现的最小的动物病毒之一。被认为是与猪的多种疾病及综合征相关的重要病原,主要引起断奶仔猪多系统衰竭综合征、猪呼吸道疾病综合征、繁殖障碍等,统称为猪圆环病毒相关疾病。该病已成为世界范围内影响养猪业发展的重要疾病,在不同个体及猪群中表现出不同的临床症状[32]。本研究首次在藏猪粪便中检出PCV-2,检出率为10.96%(16/146,95% CI: 6.4%~17.2%),检出率显著低于四川地区猪场组织、血清样本中检出率[19, 33],可能由于样本选择不同,造成本研究的PCV-2检出率显著低于以往数据,有待进一步研究。本研究获得三株病毒全基因组SMU-TP-L1、SMU-TP-J1和SMU-TP-J6,与参考毒株高度同源,核苷酸相似性在96.9%~99.9%;其中SMU-TP-L1和SMU-TP-J1为PCV-2b型,SMU-TP-J6为PCV-2d型,在遗传进化分析中,SMU-TP-L1和SMU-TP-J1与本实验室先前获得的四川地区PCV-2流行株[19]聚为一支,有较近的亲缘关系,这说明PCV-2可能是在引种改良藏猪品质和与内地贸易往来过程中带入,有待进一步研究证实。
4 结论利用病毒宏基因组学方法首次获得了腹泻藏猪粪便病毒群落信息,鉴定19种病毒,发现腹泻藏猪粪便病毒组成丰富。存在与人和其他畜种相互感染的情况,值得我们高度重视,具有重要的公共卫生学意义,这种差别可能与藏猪独特的饲养环境有关,可以为藏猪腹泻病防控提供一定的理论依据。
| [1] |
强巴央宗, 张浩, 纪素玲, 等. 藏猪屠宰性能和肉质测定与分析[J]. 中国畜牧杂志, 2008, 44(21): 10–11, 48.
CHAMBA Y Z, ZHANG H, JI S L, et al. Determination and analysis of slaughtering performance and meat quality of Tibetan pigs[J]. Chinese Journal of Animal Science, 2008, 44(21): 10–11, 48. (in Chinese) |
| [2] |
时新春.西藏林芝地区藏猪群6种传染病的血清流行病学调查[D].杨凌: 西北农林科技大学, 2013.
SHI X C. The epidemiological investigation of six pig contagious diseases in Tibetan pigs[D]. Yangling: Northwest A&F University, 2013. (in Chinese) http://cdmd.cnki.com.cn/article/cdmd-10712-1013347754.htm |
| [3] | GAO X, ZHANG L H, JIANG X, et al. Porcine epidemic diarrhea:an emerging disease in Tibetan pigs in Tibet, China[J]. Trop Anim Health Prod, 2019, 51(2): 491–484. DOI: 10.1007/s11250-018-1707-3 |
| [4] | WANG M, WANG Y, BALOCH A R, et al. Detection and genetic characterization of porcine deltacoronavirus in Tibetan pigs surrounding the Qinghai-Tibet Plateau of China[J]. Transbound Emerg Dis, 2018, 65(2): 363–369. DOI: 10.1111/tbed.12819 |
| [5] | BÁNYAI K, MARTELLA V, BOGDÁN Á, et al. Genogroup I picobirnaviruses in pigs:evidence for genetic diversity and relatedness to human strains[J]. J Gen Virol, 2008, 89: 534–539. DOI: 10.1099/vir.0.83134-0 |
| [6] | BLOMSTRÖM A L, BELÁK S, FOSSUM C, et al. Detection of a novel porcine boca-like virus in the background of porcine circovirus type 2 induced postweaning multisystemic wasting syndrome[J]. Virus Res, 2009, 146(1-2): 125–129. DOI: 10.1016/j.virusres.2009.09.006 |
| [7] | CHEN J W, CHEN F, ZHOU Q F, et al. Complete genome sequence of a novel porcine sapelovirus strain yc2011 isolated from piglets with diarrhea[J]. J Virol, 2012, 86(19): 10898. DOI: 10.1128/JVI.01799-12 |
| [8] | CHENG W X, LI J S, HUANG C P, et al. Identification and nearly full-length genome characterization of novel porcine bocaviruses[J]. PLoS One, 2010, 5(10): e13583. DOI: 10.1371/journal.pone.0013583 |
| [9] | CHEUNG A K, NG T F, LAGER K M, et al. A divergent clade of circular single-stranded DNA viruses from pig feces[J]. Arch Virol, 2013, 158(10): 2157–2162. DOI: 10.1007/s00705-013-1701-z |
| [10] | CULLEY A I, LANG A S, SUTTLE C A. Metagenomic analysis of coastal RNA virus communities[J]. Science, 2006, 312(5781): 1795–1798. DOI: 10.1126/science.1127404 |
| [11] | ZHANG Y Z, SHI M, HOLMES E C. Using metagenomics to characterize an expanding virosphere[J]. Cell, 2018, 172(6): 1168–1172. DOI: 10.1016/j.cell.2018.02.043 |
| [12] |
方肇寅, 谢华萍, 吕红霞, 等. 1999~2005年我国婴幼儿人杯状病毒腹泻研究[J]. 病毒学报, 2007, 23(1): 9–15.
FANG Z Y, XIE H P, LÜ H X, et al. Investigation of human calicivirus (HuCV) diarrhea among infantile and young children in China, 1999-2005[J]. Chinese Journal of Virology, 2007, 23(1): 9–15. DOI: 10.3321/j.issn:1000-8721.2007.01.002 (in Chinese) |
| [13] |
王新珍.东北稻田水体蓝藻病毒基因多样性与水体病毒宏基因组学研究[D].长春: 中国科学院大学(中国科学院东北地理与农业生态研究所), 2017.
WANG X Z. Analysis of cyanophage genetic diversity and viral metagenomics in paddy waters in Northeast China[D]. Changchun: University of Chinese Academy of Sciences (Northeast Institute of Geography and Agricultural, Chinese Academy of Sciences), 2017. (in Chinese) |
| [14] |
刘文彬, 张翠媛, 虞结梅, 等. 中国广西龙虎山野生猕猴粪便的病毒宏基因组学分析[J]. 病毒学报, 2016, 32(3): 273–282.
LIU W B, ZHANG C Y, YU J M, et al. Metagenomic analysis of wild rhesus monkey virome in Longhu Mountain in Guangxi Area, China[J]. Chinese Journal of Virology, 2016, 32(3): 273–282. (in Chinese) |
| [15] |
舒鹏.宏基因组测序技术在肠道菌群研究和临床样本检测中的应用[D].合肥: 安徽医科大学, 2016.
SHU P. The application of metagenomics sequencing technology in the research of intestinal flora and the detection of clinical samples[D]. Hefei: Anhui Medical University, 2016. (in Chinese) http://cdmd.cnki.com.cn/Article/CDMD-10366-1016144002.htm |
| [16] | ZHANG B, TANG C, YUE H, et al. Viral metagenomics analysis demonstrates the diversity of viral flora in piglet diarrhoeic faeces in China[J]. J Gen Virol, 2014, 95: 1603–1611. DOI: 10.1099/vir.0.063743-0 |
| [17] |
韩文, 罗玉子, 赵碧波, 等. 基于宏基因组学的猪群样本病毒探测方法的建立[J]. 微生物学报, 2013, 53(2): 197–203.
HAN W, LUO Y Z, ZHAO B B, et al. Metagenomics-based detection of swine viruses[J]. Acta Microbiologica Sinica, 2013, 53(2): 197–203. (in Chinese) |
| [18] |
单同领, 童光志.病毒宏基因组学分析腹泻仔猪肠道病毒群落及变异PEDV的分离和鉴定[C]//动物生态养殖、疫病防控、食品安全与人类健康——中国畜牧兽医学会家畜传染病学分会第八届全国会员代表大会暨第十五次学术研讨会论文集.徐州: 中国畜牧兽医学会家畜传染病学分会, 解放军军事医学科学院军事兽医研究所, 2013: 513.
SHAN T L, TONG G Z. Isolation and identification of enterovirus community and variant pedv in diarrhea piglets[C]//Animal Ecological Culture, Blight Prevention and Control, Food Safety and Human Health-the eighth National Congress and the 15th Symposium of the Society of Animal Infectious Diseases of the Chinese Society of Animal Husbandry and Veterinary Medicine. Xuzhou: Society of Livestock Infectious Diseases, Chinese Society of Animal Husbandry and Veterinary Medicine, Institute of Military Veterinary Medicine, Academy of Military Medical Sciences, PLA, 2013: 513. (in Chinese) http://cpfd.cnki.com.cn/Article/CPFDTOTAL-ZGXJ201310001149.htm |
| [19] | QIN S N, RUAN W Q, YUE H, et al. Viral communities associated with porcine respiratory disease complex in intensive commercial farms in Sichuan province, China[J]. Sci Rep, 2018, 8(1): 13341. DOI: 10.1038/s41598-018-31554-8 |
| [20] | BREITBART M, SALAMON P, ANDRESEN B, et al. Genomic analysis of uncultured marine viral communities[J]. Proc Natl Acad Sci U S A, 2002, 99(22): 14250–14255. DOI: 10.1073/pnas.202488399 |
| [21] | EDWARDS R A, ROHWER F. Viral metagenomics[J]. Nat Rev Microbiol, 2005, 3(6): 504–510. DOI: 10.1038/nrmicro1163 |
| [22] | DELWART E L. Viral metagenomics[J]. Rev Med Virol, 2007, 17(2): 115–131. DOI: 10.1002/rmv.532 |
| [23] |
燕超, 张畅, 徐琳, 等. 桂越边境地区蝙蝠病毒组研究[J]. 中国科学:生命科学, 2019, 49(3): 266–279.
YAN C, ZHANG C, XU L, et al. Metagenomic analysis of bat virome in the Guangxi-Vietnam border area[J]. Sci Sin Vitae, 2019, 49(3): 266–279. (in Chinese) |
| [24] | SAUVAGE V, AR GOUILH M, CHEVAL J, et al. A member of a new Picornaviridae genus is shed in pig feces[J]. J Virol, 2012, 86(18): 10036–10046. DOI: 10.1128/JVI.00046-12 |
| [25] | ZAULET M, PETROVAN V, BIRLADEANU A M, et al. Identification and prevalence of swine pasivirus 1 in eastern Romanian pig farms[J]. J Vet Diagn Invest, 2017, 29(3): 305–311. DOI: 10.1177/1040638717696044 |
| [26] | YU J M, LI X Y, AO Y Y, et al. Identification of a novel picornavirus in healthy piglets and seroepidemiological evidence of its presence in humans[J]. PLoS One, 2013, 8(8): e70137. DOI: 10.1371/journal.pone.0070137 |
| [27] | COTMORE S F, AGBANDJE-MCKENNA M, CHIORINI J A, et al. The family Parvoviridae[J]. Arch Virol, 2013, 159(5): 1239–1247. |
| [28] | CHIEOCHANSIN T, VUTITHANACHOT V, THEAMBOONLERS A, et al. Bufavirus in fecal specimens of patients with and without diarrhea in Thailand[J]. Arch Virol, 2015, 160(7): 1781–1784. DOI: 10.1007/s00705-015-2441-z |
| [29] | LI J J, CUI L, DENG X T, et al. Canine bufavirus in faeces and plasma of dogs with diarrhoea, China[J]. Emerg Microbes Infect, 2019, 8(1): 245–247. DOI: 10.1080/22221751.2018.1563457 |
| [30] | LIU L H, SCHWARZ L, ULLMAN K, et al. Identification of a novel bufavirus in domestic pigs by a viral metagenomic approach[J]. J Gen Virol, 2016, 97(7): 1592–1596. DOI: 10.1099/jgv.0.000476 |
| [31] | NG T F F, SACHSENRÖDER J, REUTER G, et al. Rabovirus:a proposed new picornavirus genus that is phylogenetically basal to enteroviruses and sapeloviruses[J]. Arch Virol, 2015, 160(10): 2569–2575. DOI: 10.1007/s00705-015-2523-y |
| [32] | MENG X J. Spread like a wildfire-the omnipresence of porcine circovirus type 2(PCV2) and its ever-expanding association with diseases in pigs[J]. Virus Res, 2012, 164(1-2): 1–3. DOI: 10.1016/j.virusres.2011.12.005 |
| [33] |
赵军, 周桐枫, 刘鹏娟, 等. 四川地区腹泻仔猪群猪圆环病毒Ⅱ型分子流行病学调查[J]. 中国预防兽医学报, 2016, 38(7): 546–549.
ZHAO J, ZHOU T F, LIU P J, et al. Molecular epidemiology of porcine circovirus type Ⅱ in diarrhea piglets in Sichuan region[J]. Chinese Journal of Preventive Veterinary Medicine, 2016, 38(7): 546–549. (in Chinese) |



