2. 天津市畜牧兽医研究所奶牛技术产业创新团队, 天津 300381;
3. 廊坊师范学院生命科学学院, 廊坊 065000
2. Dairy Technology Industry Innovation Team, Tianjin Animal Husbandry and Veterinary Research Institute, Tianjin 300381, China;
3. College of Life Sciences, Langfang Normal College, Langfang 065000, China
羊奶的品质与脂肪酸组成之间的内在联系已成为学术界和消费者关注的热点[1]。乳中不饱和脂肪酸的合成受激素和日粮结构的影响,因此在转录水平调控基因的表达可为羊奶乳脂合成的调控提供一个可操控的途径[2]。
乳脂代谢调控主要包括脂肪酸从头合成、脂肪酸的摄取和转运、脂肪酸去饱和以及转录因子调控等。CREB (cAMP response element binding protein)是细胞核内一类重要的转录因子,是在研究生长抑素基因的表达调控时从大鼠脑中分离出来的蛋白,是bZIP蛋白家族中的一员[3]。目前,在哺乳动物细胞广泛表达的主要有CREBA和CREB$A, 前者具有较高的刺激转录活性。据报道,CREB通过cAMP信号途径结合CRE诱导基因转录的发生,CRE是进化上高度保守且广泛存在于真核生物许多基因启动区的一段DNA序列[4],CREB与CRE的结合可大幅提高CRE下游基因的转录活性。Oliner等[5]在Hela细胞中的研究表明,CREB蛋白及包括CBP在内的共激活因子的氨基端通过与SREBP的激活结构域结合,增强一系列受SREBP靶向调控并且与脂质及固醇合成相关的关键基因的转录活性。甾醇调节因子结合蛋白(SREBPs)在细胞代谢中起着重要作用。哺乳动物SREBP1主要控制脂肪酸、甘油三酯和胆固醇的合成[6]。与脂肪合成相关的 FASN、LPL、ACACA和SCD1等关键基因的激活过程已经研究清楚[2]。Vankoningsloo等[7]在小鼠前脂肪细胞中敲低CREB基因的表达显著下调了一系列启动子中含有CRE结合位点的脂质代谢基因(包括SCD1等),并且显著降低了细胞中甘油三酯含量。综上所述,CREB很有可能作为重要的转录因子在脂质代谢调控中发挥重要作用。目前,该基因在奶山羊乳腺上皮细胞脂质代谢中的作用鲜有报道。
本研究通过从奶山羊乳腺组织中克隆到CREB基因完整的CDS区,并对不同泌乳时期CREB基因的表达差异进行分析。旨在筛选靶向CREB基因的siRNA,继而研究干扰该基因对乳腺上皮细胞中脂质合成相关基因表达及甘油三酯合成的影响,对进一步揭示该基因在乳腺组织脂质代谢中的调控机制,提高山羊的生产性能及山羊乳品质具有重要的指导意义。
1 材料与方法 1.1 材料奶山羊乳腺组织取自西北农林科技大学萨能羊原种场,试验羊为生长状况良好,年龄、体重相似且处于泌乳高峰和干奶期的奶山羊(n=3),泌乳盛期奶山羊乳腺上皮细胞购自赛百慷(上海)生物技术股份有限公司。
RNA提取Trizol试剂盒、PrimeScript® RT Reagent Kit试剂盒、实时定量的试剂SYBR® Premix Ex TaqTM和LA TaqDNA聚合酶均购自宝生生物工程大连有限公司(TaKaRa)。pMD19-T载体连接试剂盒、质粒提取试剂盒、DNA凝胶纯化回收试剂盒及DNA片段快速回收试剂盒均购自天根生化科技有限公司(北京)。DEPC水购自索莱宝生物科技有限公司(北京)。T4 DNA连接试剂盒、氨苄青霉素、IPTG、X-gal、限制性内切酶(Sal Ⅰ、EcoR Ⅰ)及DNA markerⅢ、DMEM/F-12培养基、胎牛血清和Lipofectamine® RNAiMAX转染试剂购自赛默飞公司(北京)。甘油三酯检测试剂盒和BCA蛋白检测试剂盒购自普利来基因技术有限公司(北京)。
1.2 方法 1.2.1 奶山羊CREB基因CDS区的克隆根据GenBank中绵羊(XM004004864)、牛(BT026534)和人(BC095407)等物种的CREB基因序列进行核苷酸同源性比对,利用Premier 5.0和Oligo7软件针对保守区域设计特异性扩增引物:F:5′-GAGAAGCGGAGTGTTGGT-3′;R:5′-AGGTGTTGGAGCATTCACAG-3′。引物由上海生工生物工程公司合成。
以泌乳盛期乳腺组织cDNA为模板,对CREB基因进行PCR扩增,连接pMD19-T载体。PCR反应体系及反应程序参照文献[8]。退火温度为55 ℃,延伸时间为90 s。产物用1%的琼脂糖凝胶电泳进行检测。回收约1 300 bp的目的条带,纯化后与pMD19-T载体进行连接,构建TA克隆载体pMD19-T-CREB,转化E.coli Top10大肠杆菌感受态细胞,37 ℃培养13~16 h后,挑选阳性克隆,进行Sal I/EcoR I双酶切鉴定正确后送测序。
1.2.2 CREB基因序列生物信息学分析利用NCBI中的Blastn(http://www.ncbi.nlm.nih.gov/BLAST/)进行奶山羊CREB基因核苷酸序列与绵羊、牛和人的差异比对。通过Clustal Omega(https://www.ebi.ac.uk/Tools/msa/clustalo/)进行物种间CREB氨基酸序列多重比对,利用ProtParam(http://www.expasy.org/tools/protparam.html)、ExPASy Proteomics Server(http://us.expasy.org/cgi-bin/protscale.pl)和TMHMM(http://www.cbs.dtu.dk/services/TMHMM/)对奶山羊CREB蛋白质的分子量、理论等电点、蛋白疏水性和跨膜结构域进行预测。
1.2.3 CREB基因在奶山羊不同泌乳时期表达水平分析分别将奶山羊干奶期和泌乳盛期3个个体的乳腺组织提取总RNA, 并反转录成cDNA,具体参照PrimeScript®RT Regent Kit(TaKaRa)说明书。在CFX96型实时荧光定量PCR仪上进行定量分析,定量引物由上海生工生物工程公司合成。按照TaKaRa的SYBR®Primix Ex TaqTMⅡ试剂盒说明书,检测CREB基因在两个时期的表达情况(荧光定量引物见表 1),反应体系:SYBR®Premix Ex TaqTM(2×)PCR 5 μL,10 μmol·L-1上下游引物各0.4 μL,模板0.6 μL,ddH2O补至10 μL。反应步骤:95 ℃预变性30 s;95 ℃ 5 s,60 ℃ 3 0s,共40个循环。扩增效率的计算均参照文献[9]。选择UXT、MRPL39作为内参,两个内参的Ct值为几何平均值。采用2-ΔΔCt法进行相对定量分析,每个样品设置3个重复。其中ΔΔCt=[(Ctgene-CtUXT、MRPL39)]试验组-[(Ctgene-CtUXT、MRPL39)]对照组。
|
|
表 1 实时荧光定量PCR引物 Table 1 Primers for qRT-PCR |
利用Invitrogen公司BLOCK-iTTM RNAi Express( https://rnaidesigner.invitrogen.com/rnaiexpress/)在线设计靶向CREB基因CDS序列的siRNA(siCREB)及对照组siRNA(negative control, 表 2), 并由锐博生物科技有限公司(广州)进行合成。
|
|
表 2 针对山羊CREB基因的siRNA序列 Table 2 Sequence of siRNA for goat CREB |
分别将对照组和试验组siRNA以20 nm浓度转染至泌乳盛期乳腺上皮细胞中,具体步骤参照Lipofectamine®RNAiMAX转染试剂说明书,每组重复3次。48 h后用PBS润洗两遍,采用Trizol法提取总RNA,检测浓度和纯度后反转录为cDNA,用于实时荧光定量PCR, 最后检测CREB基因的表达量,根据干扰效率确定有效siRNA。
1.2.6 qRT-PCR检测干扰CREB基因对奶山羊乳脂代谢相关基因表达的影响将siRNA转染至泌乳盛期乳腺上皮细胞,48 h后采用Trizol一步法提取总RNA并反转录成cDNA,用于荧光定量PCR检测。利用Primer Premier5.0软件,根据NCBI已公布的基因序列设计脂质代谢相关基因的qRT-PCR引物(表 1),定量引物由上海生工生物工程公司合成。反应体系:SYBR®Premix Ex TaqTM(2×)PCR 5 μL,10 μmol·L-1上下游引物个各0.4 μL,模板0.6 μL,ddH2O补至10 μL。反应步骤:95 ℃预变性30 s;95 ℃ 5 s,60 ℃ 3 0s,共40个循环。每个样品设置3个重复。采用2-ΔΔCt法进行相对定量分析,选择UXT、MRPL39作为内参,Ct值为两个内参的几何平均值,ΔΔCt=[(Ctgene-CtUXT、MRPL39)]试验组-[(Ctgene-CtUXT、MRPL39)]对照组。通过SPSS统计软件对数据进行显著性分析,结果用“平均值±标准差”表示,P < 0.05表示差异显著,P < 0.01表示差异极显著。
1.2.7 检测干扰CREB基因对奶山羊乳腺上皮细胞甘油三酯含量的影响在乳腺上皮细胞中分别将对照组和干扰组(n=3)siRNA转染后,弃去培养基,将细胞用PBS清洗3次,加入甘油三酯检测试剂盒中的裂解液室温轻柔摇晃20 min后,用细胞刮铲将细胞碎片和裂解液全部转移至离心管中进行超声破碎,根据试剂盒说明书利用酶比色法检测甘油三酯含量。细胞中的总蛋白含量使用BCA蛋白检测试剂盒进行检测。甘油三酯的总含量使用每孔对应的蛋白含量进行标定校准,其单位为μg·mg-1蛋白。
2 结果 2.1 奶山羊CREB基因CDS区的克隆及生物信息学分析以奶山羊乳腺组织总RNA为模板,通过RT-PCR扩增得到了奶山羊CREB基因,经1%的琼脂糖凝胶电泳检测为约1 300 bp的片段(图 1A),与预期结果基本相符,与pMD19-T载体连接后进行双酶切鉴定(图 1B), 得到两条清晰的条带,其中一条与目的片段一致,确认连接成功并进行测序,结果通过Blast比对,确认克隆得到西农萨能奶山羊CREB基因的CDS区。西农萨能奶山羊CDS区全长984 bp, 编码327个氨基酸,提交到GenBank,登录号为MK158073。

|
M.DNA相对分子质量标准;1.CREB基因CDS区PCR扩增产物;2.pMD19-T-CREB经Sal I和EcoR I的双酶切产物 M.DNA marker Ⅲ; 1.Amplification product of CREB CDS region by PCR; 2.pMD19-T-CREB vector digested by Sal I and EcoR I 图 1 CREB基因的克隆与鉴定 Fig. 1 Cloning and identification of CREB gene |
经Blast进行同源性分析发现,奶山羊CREB基因的CDS区核苷酸序列与绵羊(XM004004864)、牛(BT026534)、人(BC095407)的相似度分别为99%、99%、95%,氨基酸序列相似性均在99%以上(图 2),说明CREB基因在哺乳动物中有很高的保守性。利用ProtParam软件对CREB的氨基酸序列分析表明,CREB蛋白分子量为36.69 ku,理论等电点为4.80。进行蛋白疏水性预测发现,CREB疏水最大值为2.022,最小值为-0.533,该蛋白具有较强的疏水性。且不存在跨膜结构域。

|
gCREB为山羊(Capra hircus)CREB氨基酸序列,oCREB为绵羊(Ovis aries)CREB氨基酸序列,bCREB为牛(Bos taurus)CREB氨基酸序列,hCREB为人(Homo sapiens)CREB氨基酸序列。黑色部分表示所比对的氨基酸序列完全一致,灰色部分表示所比对的氨基酸中出现了保守替换,白色部分表示所比的氨基酸中出现了非保守替换 gCREB is the goat (Capra hircus) CREB amino acid sequence, oCREB is the sheep (Ovis aries) CREB amino acid sequence, bCREB is the cattle (Bos taurus) CREB amino acid sequence, hCREB is the human (Homo sapiens) CREB amino acid sequence. The black part indicates the indentical amino acid, the gray part indicates a conservative substitution in the aligned amino acid, the white part indicates a nonconservative substitution in the aligned amino acid 图 2 奶山羊与绵羊、牛、人CREB基因的氨基酸序列比对结果 Fig. 2 Comparison of amino acid sequences of CREB among dairy goat, sheep, cattle and human |
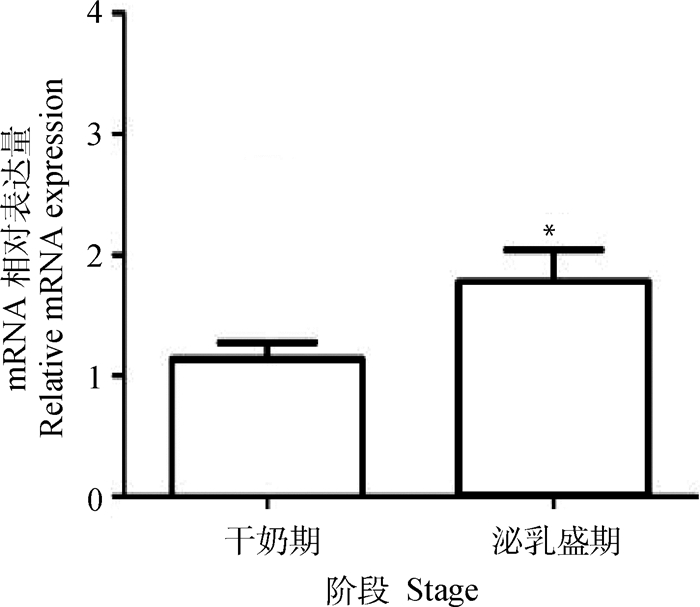
以UXT和MRPL39作为内参基因,采用荧光定量PCR技术检测CREB基因在奶山羊泌乳盛期和干奶期乳腺组织的表达量差异。结果表明,CREB基因在泌乳盛期乳腺组织中的表达量为干奶期的1.93倍,且差异显著(P < 0.05,图 3)。
|
*.P < 0.05;**.P < 0.01, the same as below 图 3 奶山羊不同泌乳时期乳腺组织CREB基因的表达量分析 Fig. 3 Analysis of CREB gene expression in mammary gland tissue in different lactation stages of dairy goats |
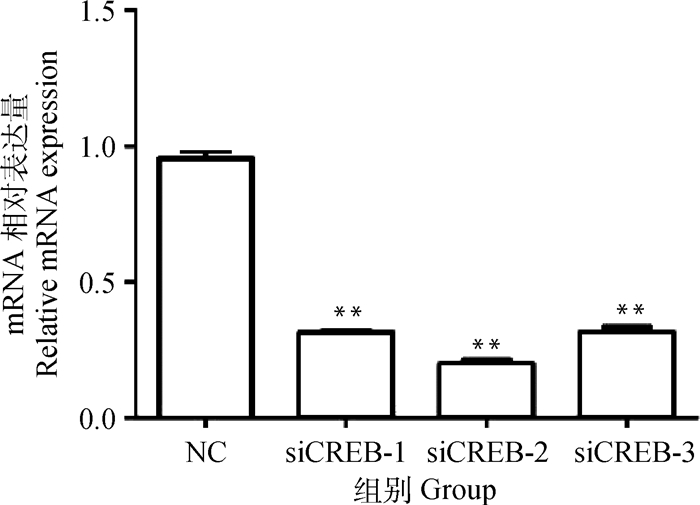
将3对干扰组的siRNA及对照组siRNA分别转染至乳腺上皮细胞,培养48 h后,采用Trizol法提取总RNA,反转录为cDNA进行荧光定量PCR。结果表明,3对siRNA均有显著的干扰效果,其中siCREB-2干扰效率最高(图 4),经计算siCREB-2的干扰效率为72%。
|
图 4 奶山羊CREB基因siRNAs序列的干扰效率检测 Fig. 4 Detection of interference efficiency of siRNA sequences of CREB gene in dairy goats |
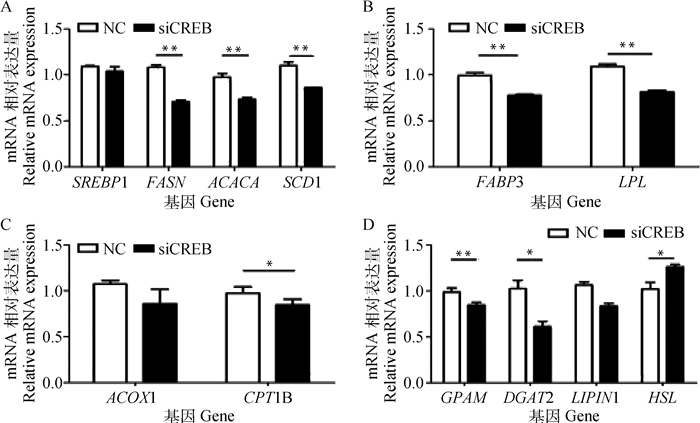
将筛选到的有效siRNA转染至乳腺上皮细胞48 h后,进行总RNA提取并反转录成cDNA,以此为模板通过荧光定量PCR检测脂质合成相关基因的表达情况。结果表明,与对照组相比,干扰CREB基因后,进行乳脂合成的重要转录因子SREBP1的表达量无显著变化,而其参与调控的脂肪酸从头合成和去饱和过程的关键基因FASN、ACACA及SCD1的表达量均被极显著下调(P < 0.01,图 5A);参与脂肪酸摄取和转运的关键基因FABP3和LPL的表达受到极显著抑制(P < 0.01,图 5B);参与脂肪酸氧化的CPT1B基因的表达量显著下调(P < 0.05,图 5C);催化甘油三酯合成的关键基因GPAM和DGAT2被显著下调(P < 0.05),水解甘油三酯的重要基因HSL表达量显著上调(P < 0.05,图 5D)。
|
A.脂肪酸从头合成及去饱和相关基因;B.脂肪酸摄取和转运相关基因;C.脂肪酸氧化相关基因;D.甘油三酯合成与水解相关基因 A. Fatty acid de novo synthesis and desaturation related genes; B. Fatty acid uptake and transport related genes; C. Fatty acid oxidation related genes; D. Triglyceride synthesis and hydrolysis related genes 图 5 干扰CREB基因后对脂质合成相关基因mRNA表达的影响 Fig. 5 Effect of interfering with CREB gene on the mRNA expression of lipid synthesis related genes |
在乳腺上皮细胞中进行对照组与干扰组siRNA转染,培养48 h后,弃去培养基,将细胞用PBS清洗3次,采用试剂盒进行细胞内甘油三酯含量的测定并以每孔对应的蛋白含量进行标定校准,结果表明,干扰CREB基因后显著降低了乳腺上皮细胞中的甘油三酯含量(P < 0.05,图 6)。

|
图 6 干扰CREB基因对奶山羊乳腺上皮细胞中甘油三酯含量的影响 Fig. 6 Effect of interfering with CREB gene on triglyceride content in mammary epithelial cells of dairy goats |
本研究克隆得到了西农萨能奶山羊乳腺组织CREB基因的CDS区,与绵羊、牛和人的核苷酸同源性分别为99%、99%和95%,氨基酸同源性在99%以上。CREB基因广泛存在于真核细胞中, 且有8种剪接方式[10],但在结构上高度保守,主要分为N端调节转录功能区域和C端结合启动子区域,在细胞核内通过磷酸化和去磷酸化改变其亚细胞定位、DNA结合来调节许多真核转录因子的活性。据报道,乳腺上皮细胞PTEN基因过表达的小鼠模型中,处于泌乳期的小鼠不能正常泌乳,泌乳量显著下降[11]。PTEN是以一种依赖于磷酸酶的方式去磷酸化的CREB,通过抑制CREB与靶基因启动子顺式作用元件的结合来降低泌乳相关基因的表达[12]。泌乳是奶山羊的主要性能,乳腺组织又是泌乳时期脂质代谢发挥重要作用的组织之一。本研究通过检测CREB基因在不同泌乳时期的表达情况,发现其在泌乳盛期乳腺组织中的表达量高出干奶期约1.9倍。因此,该基因很可能在奶山羊泌乳时期发挥一定生理功能,参与调控泌乳时期的相关代谢途径。本研究旨在通过RNAi技术进一步阐明CREB基因在奶山羊泌乳高峰时期进行乳脂代谢的调控机制。通过在线设计并合成siRNA, 转染至奶山羊乳腺上皮细胞中。以cDNA作为模板,根据荧光定量PCR结果得到CREB基因的表达量,从而筛选到有效siRNA,并计算其干扰效率为72%,效果显著,可为后续对该基因的功能研究提供理想的试验材料。
SREBP1是参与脂肪酸代谢调控的重要转录因子[13]。在小鼠和牛乳腺组织中的研究表明,SREBP1从内质网运输到高尔基体进行加工剪接,释放出成熟的nSREBP,才能进入细胞核,结合在下游靶基因的启动子(如SRE位点及E-box上),进而调节转录。Jie等[14]和Han等[4]的试验均表明,CREB通过结合其一系列转录共激活因子参与SREBP的加工成熟,并对SREBP下游靶基因进行调控。因此,CREB基因很可能在转录复合物中作为重要枢纽,调控SREBP1的成熟过程,对脂质代谢调控发挥重要作用[15]。本研究发现,在乳腺上皮细胞干扰CREB基因后,SREBP1基因表达水平无显著变化,却使参与脂肪酸从头合成及去饱和的FASN、ACACA及SCD1基因表达量均极显著下调。表明在乳腺上皮细胞中干扰CREB基因有可能通过降低SREBP蛋白的活性及其靶基因的转录,从而抑制细胞内脂肪酸从头合成及去饱和过程,具体调控机制有待证实。有研究在小鼠前脂肪细胞中同样用siRNA干扰CREB基因,结果显著下调了启动子中含有CRE结合位点的SCD1基因的表达, 并且显著降低了细胞中甘油三酯含量。Yao等[16]在奶山羊乳腺上皮细胞中证实,SCD1基因催化的主要产物MUFA和PUFA将参与乳腺中甘油三酯的合成和细胞内信号通路的转导。本研究干扰CREB基因显著下调参与甘油三酯合成的GPAM和DGAT2等关键基因的表达,显著上调参与甘油三酯水解的HSL基因的表达;并且显著抑制细胞内甘油三酯的合成,这与前人的研究结果是一致的。从头合成过程是构成合成甘油三酯脂肪酸库的主要部分[17],另一部分来源于摄取的长链脂肪酸,从外周循环血液摄取的脂肪酸需要被脂解,再由转运蛋白使特定的长链脂肪酸转运到特定的细胞器上[18]。本研究干扰CREB基因显著下调了参与脂肪酸脂解和转运的关键基因LPL和FABP3的表达。Yao等[19]的研究表明,FABP3的启动子序列存在CREB结合位点,并在牛、绵羊、猪中高度保守。因此,干扰CREB基因很可能阻碍了长链脂肪酸的摄取及转运,具体机制有待进一步证实。
乳脂代谢调控从脂肪酸的摄取和转运、从头合成及去饱和再到甘油三酯的合成都需要耗能,这些能量主要来自线粒体中的脂肪酸β-氧化[20]。本研究干扰CREB基因显著下调了参与脂肪酸肉碱转运系统的重要基因CPT1B的表达。由此,干扰CREB基因很可能通过抑制能量代谢途径对脂质代谢产生一定影响。Oishi等[21]在KLF5基因敲除小鼠中的试验表明,KLF5基因通过诱导CREB结合蛋白(CBP)的激活上调了CPT1B基因表达,而KLF5基因正是能量代谢的关键调节器[22]。该结果为研究CREB基因在能量代谢中的具体作用提供了参考。
4 结论本研究克隆了奶山羊CREB基因,对其在不同泌乳时期的表达水平进行了分析,结果表明,CREB基因在泌乳盛期乳腺组织的表达量约是干奶期的1.93倍, 并且成功筛选到能够有效干扰奶山羊CREB基因的siRNA(干扰效率为72%),通过qRT-PCR检测脂质合成相关基因的表达,与对照组相比,干扰CREB基因后显著下调了FASN、ACACA、SCD1、FABP3、LPL、CPT1B、GPAM和DGAT2基因表达量,显著上调了HSL基因表达量,并且细胞内甘油三酯含量显著下降。该结果为研究CREB基因在奶山羊乳腺脂酯合成中的调控机理奠定了基础。
| [1] | HAN L Q, GAO T Y, YANG G Y, et al. Overexpression of SREBF chaperone (SCAP) enhances nuclear SREBP1 translocation to upregulate fatty acid synthase (FASN) gene expression in bovine mammary epithelial cells[J]. J Dairy Sci, 2016, 101(7): 6523–6531. |
| [2] | WANG C M, YUAN R S, ZHUANG W Y, et al. Schisandra polysaccharide inhibits hepatic lipid accumulation by downregulating expression of SREBPs in NAFLD mice[J]. Lipids Health Dis, 2016, 15(1): 195. DOI: 10.1186/s12944-016-0358-5 |
| [3] | GU T T, ZHANG Z, WANG J L, et al. CREB is a novel nuclear target of PTEN phosphatase[J]. Cancer Res, 2011, 71(8): 2821–2825. DOI: 10.1158/0008-5472.CAN-10-3399 |
| [4] | HAN J B, LI E W, CHEN L Q, et al. The CREB coactivator CRTC2 controls hepatic lipid metabolism by regulating SREBP1[J]. Nature, 2015, 524(7564): 243–246. DOI: 10.1038/nature14557 |
| [5] | OLINER J D, ANDRESEN S K, HANSEN S K, et al. SREBP transcriptional activity is mediated through an interaction with the CREB-binding protein[J]. Genes Dev, 1996, 10(22): 2903–2911. DOI: 10.1101/gad.10.22.2903 |
| [6] | CARCANGIU V, MURA M C, DAGA C, et al. Association between SREBP-1 gene expression in mammary gland and milk fat yield in Sarda breed sheep[J]. Meta Gene, 2013, 1: 43–49. DOI: 10.1016/j.mgene.2013.10.001 |
| [7] | VANKONINGSLOO S, DE PAUW A, HOUBION A, et al. CREB activation induced by mitochondrial dysfunction triggers triglyceride accumulation in 3T3-L1 preadipocytes[J]. J Cell Sci, 2006, 119(7): 1266–1282. DOI: 10.1242/jcs.02848 |
| [8] | SHI H B, WU M, ZHU J J, et al. Fatty acid elongase 6 plays a role in the synthesis of long-chain fatty acids in goat mammary epithelial cells[J]. J Dairy Sci, 2017, 100(6): 4987–4995. DOI: 10.3168/jds.2016-12159 |
| [9] | BIONAZ M, LOOR J J. Identification of reference genes for quantitative real-time PCR in the bovine mammary gland during the lactation cycle[J]. Physiol Genomics, 2007, 29(3): 312–319. DOI: 10.1152/physiolgenomics.00223.2006 |
| [10] | OTANI T, MATSUDA M, MIZOKAMI A, et al. Osteocalcin triggers Fas/FasL-mediated necroptosis in adipocytes via activation of p300[J]. Cell Death Dis, 2018, 9(12): 1194. DOI: 10.1038/s41419-018-1257-7 |
| [11] | SUN J H, LIU X, CONG L X, et al. Metabolomics study of the therapeutic mechanism of Schisandra chinensis lignans in diet-induced hyperlipidemia mice[J]. Lipids Health Dis, 2017, 16(1): 145. DOI: 10.1186/s12944-017-0533-3 |
| [12] | ISLAM M A, XU Y J, TAO W, et al. Restoration of tumour-growth suppression in vivo via systemic nanoparticle-mediated delivery of PTEN mRNA[J]. Nat Biomed Eng, 2018, 2(11): 850–864. DOI: 10.1038/s41551-018-0284-0 |
| [13] | WU S, NÄÄR A M. SREBP1-dependent de novo fatty acid synthesis gene expression is elevated in malignant melanoma and represents a cellular survival traits[J]. Sci Rep, 2019, 9(1): 10369. DOI: 10.1038/s41598-019-46594-x |
| [14] | JIE Z W, XIE Z A, XU W B, et al. SREBP-2 aggravates breast cancer associated osteolysis by promoting osteoclastogenesis and breast cancer metastasis[J]. Biochim Biophys Acta, 2019, 1865(1): 115–125. DOI: 10.1016/j.bbadis.2018.10.026 |
| [15] | LU J, CHENG B J, FANG B C, et al. Protective effects of allicin on 1, 3-DCP-induced lipid metabolism disorder in HepG2 cells[J]. Biomed Pharmacother, 2017, 96: 1411–1417. DOI: 10.1016/j.biopha.2017.10.125 |
| [16] | YAO D W, LUO J, HE Q Y, et al. Characterization of the liver X receptor-dependent regulatory mechanism of goat stearoyl-coenzyme A desaturase 1 gene by linoleic acid[J]. J Dairy Sci, 2016, 99(5): 3945–3957. DOI: 10.3168/jds.2015-10601 |
| [17] | TRACZ-GASZEWSKA Z, DOBRZYN P. Stearoyl-CoA desaturase 1 as a therapeutic target for the treatment of cancer[J]. Cancers, 2019, 11(7): 948. DOI: 10.3390/cancers11070948 |
| [18] | LI A N, WU L J, WANG X, et al. Tissue expression analysis, cloning and characterization of the 5'-regulatory region of the bovine FABP3 gene[J]. Mol Biol Rep, 2016, 43(9): 991–998. DOI: 10.1007/s11033-016-4026-7 |
| [19] | YAO D W, LUO J, HE Q Y, et al. Liver X receptor α promotes the synthesis of monounsaturated fatty acids in goat mammary epithelial cells via the control of stearoyl-coenzyme A desaturase 1 in an SREBP-1-dependent manner[J]. J Dairy Sci, 2016, 99(8): 6391–6402. DOI: 10.3168/jds.2016-10990 |
| [20] | SANCHEZ-GURMACHES J, TANG Y F, JESPERSEN N Z, et al. Brown fat AKT2 is a cold-induced kinase that stimulates ChREBP-mediated de novo lipogenesis to optimize fuel storage and thermogenesis[J]. Cell Metabol, 2018, 27(1): 195–209. DOI: 10.1016/j.cmet.2017.10.008 |
| [21] | OISHI Y, MANABE I, TOBE K, et al. SUMOylation of Krüppel-like transcription factor 5 acts as a molecular switch in transcriptional programs of lipid metabolism involving PPAR-δ[J]. Nat Med, 2008, 14(6): 656–666. DOI: 10.1038/nm1756 |
| [22] | BALLESTRI S, ZONA S, TARGHER G, et al. Nonalcoholic fatty liver disease is associated with an almost twofold increased risk of incident type 2 diabetes and metabolic syndrome.Evidence from a systematic review and meta-analysis[J]. J Gastroenterol Hepatol, 2016, 31(5): 936–944. DOI: 10.1111/jgh.13264 |



