2. 西南民族大学生命科学与技术学院 青藏高原动物遗传资源保护与利用四川省重点实验室, 成都 610041;
3. 四川省畜牧科学研究院, 成都 610066
2. Qinghai-Tibetan Plateau Animal Genetic Resource Reservation and Utilization Key Laboratory of Sichuan Province, College of Life Science and Technology, Southwest Minzu University, Chengdu 610041, China;
3. Sichuan Animal Science Academy, Chengdu 610066, China
藏鸡是一种生活在海拔为1 700~3 000 m的青藏高原高寒河谷地区的珍贵家禽[1]。该品种肉质优良、口感鲜美、营养丰富[2]。随着生活水平的不断提高,人们不仅对肉类的口感有较高要求,更是对肉类产品的营养价值有了更高的标准。所以,藏鸡作为青藏高原上开发的绿色禽类有着巨大的市场潜力。但由于藏鸡地处高寒缺氧的地区,造成其受精率低、繁殖性能不高、生长速度缓慢等。70日龄的藏鸡体重大约为941 g,而泸宁鸡到63日龄体重便可达986 g左右[3-4]。可见,相对于泸宁鸡这种肉蛋兼用型品种来说,藏鸡显得生长速度较慢,相对肉鸡则更甚之。对藏鸡生长发育规律的研究发现,藏鸡在14周龄左右达到生长高峰,增重的速度最快,14周龄以后生长速度逐步减慢。到21周龄左右时,藏鸡体重的增长量极小[3]。
肌肉发育受多种因素的影响,比如营养、年龄、饲养管理、基因等。其中基因作为影响其发育的内在因素,受到遗传育种学者们的高度关注。例如肌肉生长抑制素(myostain,MSTN)突变造成的双肌现象,将大大增加动物体内的肌肉组织含量[5]。相较于改善其他外部条件,改变动物的基因或基因型,会是一条促进肌肉快速发育的“捷径”。
转录组是特定组织或细胞在某一生理条件下所表达的所有RNA,是连接基因组和蛋白质组的桥梁[6]。miRNA是近年来生物学研究的一个重要方向,它能够作用于mRNA来抑制或促进靶基因的表达[7]。研究发现,当骨骼肌发育时,C2C12细胞表达Pax7基因[8]。而miR-1和miR-206通过抑制Pax7可调节C2C12细胞的增殖和分化[9]。一个miRNA还可以作用于多个靶基因发挥作用,miRNA-1除了靶向Pax7发挥作用,还可靶向HDAC4促进成肌细胞分化,miRNA-133可影响SRF和TGFBR1的表达,从而促进鸭成肌细胞增殖[10]。可知,miRNA在调节肌肉增殖和分化方面有着不可忽视的作用。随着高通量测序技术的不断发展,RNA测序(RNA sequencing,RNA-Seq)为大范围的转录组学和miRNA研究提供了强有力的支持,相对于一些传统方法,其优势相当明显[11]。研究者已成功利用该技术研究了牦牛[12]、猪[13]、鸡[14]等物种的肌肉组织。迄今为止,针对藏鸡的研究多集中于其生长性状测量和基因与体重相关性分析方面,而对藏鸡肌肉发育尚无系统而全面的研究。另外, 肌肉发育是一个复杂的过程,这个过程涉及大量基因的转录调控,单个基因的研究无异于“杯水车薪”。因此,为进一步了解藏鸡的肌肉发育,需要对其转录组和miRNA进行研究。本研究使用RNA-Seq技术从转录组和miRNA角度探究藏鸡的肌肉发育,深入了解藏鸡肌肉组织在不同发育时期中的差异表达基因和miRNA及其调控通路,为藏鸡肌肉发育的进一步研究提供基础。
1 材料与方法 1.1 材料 1.1.1 藏鸡及样品采集本试验选取成都益生康健农业有限公司藏鸡养殖基地的120和150日龄健康藏鸡各3只为试验动物,屠宰前禁食12 h,然后通过放血法屠宰,屠宰后迅速采集腿肌组织并装入无RNA酶的冻存管中,120日龄的腿肌组织标记为TC120,150日龄为TC150,接着迅速置于液氮,带回实验室-80 ℃保存备用。
1.1.2 主要试剂Trizol购自Invitrogen公司(上海),PrimeScriptTM RT reagent Kit with gDNA Eraser(Perfect Real Time)和TB GreenTM Premix Ex TaqTM Ⅱ(Tli RNaseH Plus)购自TaKaRa公司(大连)。
1.2 方法 1.2.1 藏鸡腿肌总RNA的提取及cDNA的合成按照Trizol试剂盒说明书提取肌肉组织总RNA,检测总RNA质量后,使用NanoDrop ND-2000(Agilent)对RNA的纯度及浓度进行检测。利用PrimeScriptTM RT reagent Kit with gDNA Eraser(Perfect Real Time)将得到的总RNA反转录合成cDNA第一条链,miRNA采用特异性反转录引物合成cDNA(表 1),-80 ℃保存备用。
|
|
表 1 miRNA特异性反转录引物 Table 1 miRNA-specific reverse transcription primer |
测序文库构建及测序由美吉生物医药科技有限公司(上海)完成。从腿肌组织中提取总RNA;利用带有Oligo(dT)的磁珠与mRNA的ployA尾进行A-T碱基配对,从总RNA中分离mRNA;加入fragmentation buffer,将mRNA随机断裂成200 bp左右的小片段;在逆转录酶的作用下,加入六碱基随机引物,以mRNA为模板反转合成一链cDNA,随后进行二链合成,形成稳定的双链结构;加入end repair mix将cDNA补成平末端,随后在3′末端加上一个A碱基,用于连接Y字形的接头;文库富集,PCR扩增15个循环;2%琼脂糖凝胶回收目的条带;TBS380定量,按数据比例混合上机;cBot上进行桥式PCR扩增,生成cluster;Hiseq4000对构建好的PE文库进行2×150 bp测序。Small RNA文库利用Illumina公司的TruseqTM Small构建,步骤与转录组文库类似,进行SE50测序。
1.2.3 藏鸡腿肌转录组数据分析为保证后续生物信息分析的准确性,先对原始测序数据进行质控检验。当数据符合要求后,使用SeqPrep和Sickle软件对raw reads进行过滤,去除接头序列、空读序列和低质量序列(含N比率超过10%和质量值小于20的序列),得到clean reads。利用TopHat2将clean reads比对到鸡参考基因组( ftp://ftp.ensembl.org/pub/release-84/fasta/gallus_gallus/dna/)。使用RSEM软件计算基因的表达量,表达定量的结果以FPKM(fragments per kilobase of exon per million reads mapped)为单位。采用edgeR软件进行基因的表达差异分析,以P-value < 0.05和|log2FC|≥1作为显著差异表达基因(differentially expression genes,DEGs)的筛选标准。将筛选出的DEGs向GO(gene ontology, http://www.geneontology.org/)数据库各个GO term进行映射,统计GO注释结果。用Goatools软件进行富集分析,使用方法为Fisher精确检验,并使用Bonferroni、Holm、Sidak和False discovery rate对P值进行校正,校正后,P(P-FDR)≤0.05时认为此GO功能存在显著富集情况。利用KEGG(kyoto encyclopedia of genes and genomes, http://www.genome.jp/kegg/)数据库,将基因按照参与的通路或行使的功能分类。用KOBAS进行KEGG通路富集分析,计算方式为Fisher精确检验,并采用BH(FDR)进行多重检验,校正后P < 0.05的KEGG通路定义为在差异表达基因中显著富集的KEGG通路。
1.2.4 藏鸡腿肌small RNA数据分析对raw reads进行过滤,去除reads中的3′接头序列、由于接头自连等原因导致没有插入片段的reads、长度不符的reads(<18 nt或>32 nt)和低质量序列(含未知碱基和质量值小于20的reads),得到clean reads。使用最新的Rfam数据库(http://Rfam.sanger.ac.uk/)对测得的small RNA进行注释,去除其中非miRNA序列,同时也对比对上的非miRNA序列进行种类和数目统计。使用Bowtie、RNAfold和miRDeep2软件对已知miRNA进行鉴定及其表达量统计分析,并利用TPM(transcripts per million)进行表达量的均一化处理。使用DEGseq鉴定样本间差异表达miRNA。利用miRanda软件对miRNA的靶基因进行预测, 并进行mRNA联合分析。
1.2.5 藏鸡腿肌转录组和small RNA测序结果的实时荧光定量PCR验证为了进一步保证测序结果的可靠性,以GAPDH和5S rRNA分别作为mRNA和miRNA的内参,挑选5个基因和5个miRNAs,采用实时荧光定量PCR(qRT-PCR)验证其表达量。用Primer Premier 5设计引物,基因和miRNA名称及引物信息见表 2。反应体系为10 μL:上、下游引物各0.4 μL,cDNA模板1 μL,ddH2O 2.2 μL, TB Green Premix Ex Taq II(Tli RNaseH Plus)6 μL。qRT-PCR扩增程序:95 ℃预变性3 min;95 ℃变性10 s,60 ℃(根据实际引物退火温度进行调整)退火15 s,72 ℃延伸30 s,39个循环;最后72 ℃延伸5 min;4 ℃保存。采用2-ΔΔCt计算得到相对表达量。
|
|
表 2 实时荧光定量PCR引物信息 Table 2 The qRT-PCR primer information |
原始测序数据显示,两样本的碱基A-T、C-G含量基本相等,说明碱基组成稳定平衡; 两样本的绝大部分碱基质量超过40,低质量碱基少; 两样本大部分碱基的错误率低于0.02%,这些均说明测序质量高。原始测序数据经过滤后,在转录组中,两样本一共得到184 726 208条clean reads,TC120和TC150的Q30的百分比分别为95.46%和95.81%,说明测序质量高,得到的数据准确可靠,可用于后续的分析;small RNA数据中,两样本总共得到24 203 705条clean reads。
2.2 藏鸡腿肌转录组测序结果比对分析比对结果显示,TC120和TC150分别有78.08%和76.66%的clean reads比对到鸡参考基因组上,比对到基因组多个位置的序列比例分别为1.07%和1.10%,符合试验要求(表 3)。
|
|
表 3 比对统计结果 Table 3 The results of mapping |
统计small RNA文库中的所有序列长度分布,大多数序列集中在21~24 nt(图 1)。其中,22 nt长度的序列频率最高,然后为23 nt长度的序列。miRNA通常集中在22 nt[15],符合试验要求。

|
图 1 小RNA的长度分布 Fig. 1 Length distribution of small RNA |
TC120和TC150测得的序列中,miRNA共占69%(图 2)。

|
图 2 小RNA分类注释 Fig. 2 Annotation of small RNAs |
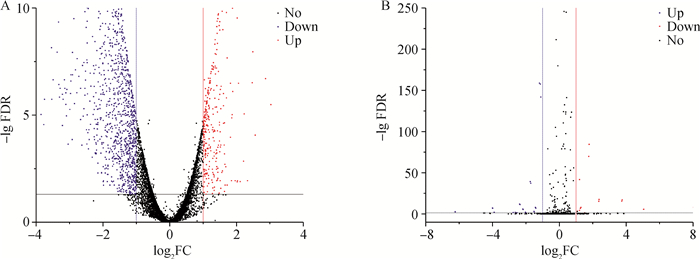
edgeR分析结果显示,两个发育阶段的DEGs有1 691个,以TC120为对照样,TC150为处理样,发现其中上调DEGs有330个,下调DEGs有1 361个(图 3A)。其中与肌肉发育相关的基因载脂蛋白B mRNA编辑酶催化多肽家族2(apolipoprotein B mRNA-editing enzyme catalytic subunit 2,APOBEC2)、肌肉骨骼胚胎核蛋白1(musculoskeletal embryonic nuclear protein 1,MUSTN1)、生长分化因子8(growth and differentiation factor-8,GDF-8)和肌肉特异Bop基因(SET and MYND domain containing 1,SMYD1)显著下调,钙蛋白酶3(calpain 3,CAPN3)显著上调。qRT-PCR结果与RNA-Seq结果基本一致(图 4)。
|
图 3 差异表达基因(A)和miRNA(B)火山图 Fig. 3 Volcano chart of differentially expressed genes (A) and miRNAs (B) |

|
图 4 RNA-Seq差异表达基因和miRNA的qRT-PCR验证 Fig. 4 Validation of qRT-PCR result to RNA-Seq result |
同样以TC120为对照样,TC150为处理样,发现显著差异表达miRNA共有22个,其中9个上调,13个下调(图 3B)。qRT-PCR结果与RNA-Seq结果基本一致(图 4)。
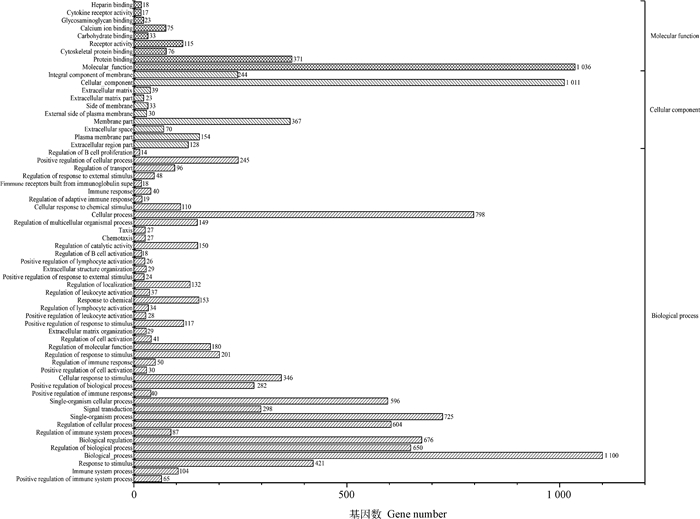
2.5 藏鸡腿肌差异表达基因的GO富集分析GO富集分析显示,DEGs在分布于生物过程(biological process,BP)、细胞组分(cellular component,CC)和分子功能(molecular function,MF)3大类中的62个GO条目上均出现显著富集(图 5)。富集前10的条目列于表 4中,其中最为富集的是免疫系统过程的正调节,其次是免疫系统过程。
|
图 5 差异表达基因的GO富集分析 Fig. 5 GO enrichment analysis of differentially expressed genes |
|
|
表 4 前10富集GO分类 Table 4 Top 10 enrichment GO categories |
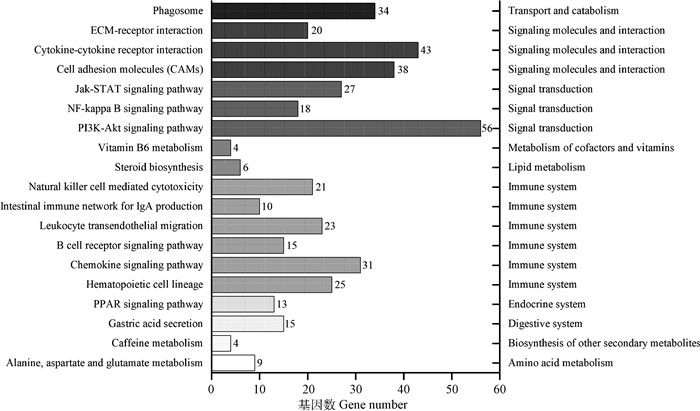
KEGG富集分析表明,DEGs中共有19个通路出现显著富集,见图 6。
|
图 6 差异表达基因显著富集的KEGG通路 Fig. 6 KEGG pathway enrichment analysis of differentially expressed genes |
22个差异表达miRNAs预测到的靶基因中,有83个出现在DEGs显著富集的KEGG通路上,如表 5所示。
|
|
表 5 miRNAs靶基因的KEGG通路 Table 5 KEGG pathways of miRNAs target genes |
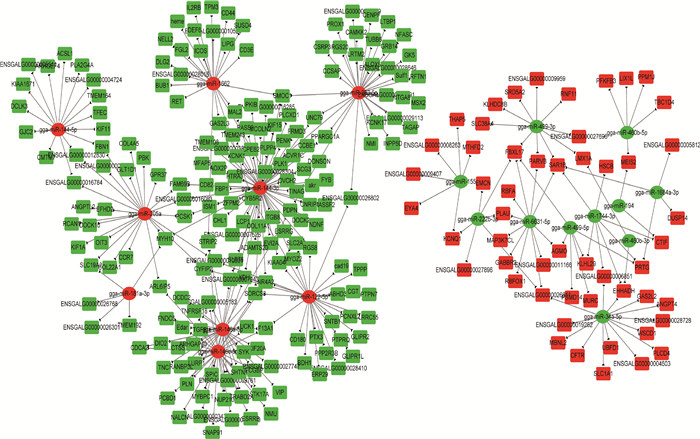
在动物中,miRNA与其预测的靶基因之间大多数为负调控。在本研究中,343个miRNA-mRNA对(涉及20个差异miRNAs和246个差异mRNAs)的表达模式为负相关(图 7)。
|
图 7 miRNA-mRNA负相关网络图 Fig. 7 miRNA-mRNA negative correlation network |
本研究发现多个在肌肉发育过程中起着重要调控作用的基因在藏鸡生长的两个节点出现显著性差异表达。比如下调基因MyoG、MUSTN1、APOBEC2、SMYD1和上调基因CAPN3。本研究中,促进肌肉发育的基因(MyoG、MUSTN1、APOBEC2、SMYD1)在120日龄的表达量显著高于150日龄,抑制肌肉发育的基因(CAPN3)在150日龄的表达量相对于120日龄显著上调,推测可能是120日龄藏鸡的生长速度大于150日龄的生长速度造成的[3]。MyoG属于生肌调节因子家族中的一员,它能使肌原细胞退出细胞周期并进一步融合形成多核的肌原纤维[16]。它还能联合MyoD和MRF4,一起激活肌源性分化系统[17]。MyoG在胎儿的肌发生中扮演着独特的作用,试验证明MyoG被敲除的小鼠,胎儿时期无法进行肌生成[18]。沉默大鼠的MUSTN1基因则会导致肌源性的融合和分化被抑制,这表明该基因在肌肉发育中扮演重要角色[19]。Xu等[20]研究发现,北京鸭中MUSTN1基因在1~3周龄表达量上升,随后下降,同时腿肌纤维的相对生长速率也在1~3周龄达到峰值。李娟[21]研究发现,MUSTN1基因在二郎山山地鸡胸腿肌组织中的表达和肌纤维直径与密度呈极显著正相关。APOBEC2是载脂蛋白B mRNA编辑酶催化多肽家族的一员。Ohtsubo等[22]发现,APOBE2缺陷可导致肌肉质量下降。进一步的研究表明,APOBEC2缺陷会导致线粒体的缺失,从而增加肌肉细胞的自我吞噬,然后产生肌病和肌肉萎缩。证明了APOBEC2是维持正常骨骼肌功能的必需基因[23]。Tan等[24]已证明,敲除SMYD1基因会完全阻断骨骼肌中肌原纤维的形成。在敲除SMYD1基因的胚胎中,所有关键的肌节结构被破坏[25]。过表达SMYD1会上调肌肉特异标记基因并且促进成肌细胞分化和肌管的形成[26]。CAPN3是一种只在骨骼肌中特异表达的钙蛋白酶[27]。朱文奇等[28]对鸭进行了CAPN3基因表达与肌纤维性状的关联分析,发现CAPN3 mRNA的表达与肌纤维直径和面积存在负相关关系。陶璇等[29]发现,藏猪CAPN3 mRNA表达量显著高于杜洛克猪,且与肌纤维面积、剪切力呈负相关,说明CAPN3基因会抑制肌纤维生长。
本研究中,GO富集分析显示,在前10富集的GO条目中,大多数与免疫相关。在KEGG显著富集的通路中,其中有6条通路也都与免疫相关。这暗示某些免疫基因和通路对生长发育具有刺激作用。这与Li等[30]之前在泸宁鸡和白羽肉鸡上的研究结果相似,大量的差异表达基因富集条目与免疫有关。Dou等[31]研究发现,生长速度更快且体型更大的鸡的肌肉组织中与免疫应答相关的基因有更高的表达量。在高生长发育压力状态下,儿茶酚胺通路被上调,并受免疫应答影响[32-34]。多巴胺和单胺作为神经递质分别刺激奖励动机行为和情绪/唤醒行为。在体内,多巴胺还参与肠胃运动(通过去甲肾上腺素机制)调节血管扩张。这些机制对于生长更快的鸡调节饲料成分的吸收和分配是必要的[31]。还有研究发现,大鼠在受伤后,炎性细胞能够促进卫星细胞的激活,从而促进骨骼肌的再生[35]。巨噬细胞在健康的肌肉中呈静止状态,但肌肉的创伤导致其迅速活化并参与肌肉再生[36]。肌肉损伤会引发炎症反应,在反应过程中,肌内白细胞的数量可迅速增加100倍以上[37]。本研究中KEGG结果显示,最为富集的正是造血细胞系通路。
miR-499是一个与骨骼肌纤维类型相关的miRNA,它能减少Sox6的表达从而促进红肌的形成且在红肌纤维中大量表达[38]。在猪的卫星细胞中,miR-499促进MyHC I和MyHC IIa mRNA的表达[39]。Wu等[39]做了进一步研究,发现miR-499能抑制C2C12细胞的分化和促进该细胞的增殖。miR-499的靶基因为TGFβR1,敲除该基因显著抑制C2C12细胞增殖而促进该细胞分化。本研究中,藏鸡的miR-499最为显著差异表达,在120日龄大量表达,推测可能是为达到肌肉的快速发育,卫星细胞的大量增殖造成的。Wang等[40]发现,gga-miR-205a能通过结合钙粘蛋白11(cadherin-11, CDH11)影响成肌细胞的增殖和分化。CDH11是出生后肌肉生长、骨量维持和成骨细胞分化的关键调节因子[41]。miR-205a能与CDH11结合且过表达能抑制鸡原代成肌细胞的增殖[40]。本研究中,miR-205a在藏鸡150日龄表达上调,同时转录组结果显示CDH11表达下调,说明miRNA-205a抑制了靶基因CDH11的表达,进而影响了肌肉的发育。这一结果也对应了本研究在KEGG的结果,细胞粘附通路与肌肉发育密切相关。进一步研究发现,MyoG能刺激miR-205的表达[40]。本研究转录组和miRNA结果显示,MyoG在藏鸡120日龄表达量高于150日龄,而miR-205在120日龄的表达量低于150日龄,两个为负相关表达,这与上一说法相矛盾,具体原因还有待进一步的研究。miR-194在增重较快的约克夏猪中的表达量高于增重较慢的莱芜猪,暗示miR-194在肌肉生长中发挥重要作用[42]。同时发现,miR-194在C2C12成肌细胞分化时被上调[43]。Wang等[44]研究发现,miR-194能增加Myf5和MyoG的表达,暗示miR-194促进成肌细胞的分化。联合转录组结果来看,确实本研究中miR-194和MyoG在藏鸡120日龄的表达量都高于150日龄。
4 结论本研究利用RNA-Seq技术对不同生长日龄藏鸡的肌肉组织转录组和miRNA进行测序和分析,获得了大量差异表达基因和miRNA以及有关通路的富集。这为进一步解析藏鸡肌肉发育特点和改良提供了丰富的数据基础。并发现藏鸡在生长发育快速时期,免疫相关基因大量表达,但背后的具体机制还有待进一步的探究。
| [1] |
徐珑洋, 梅寒, 王志敏, 等. 藏鸡、泸宁鸡POU1F1基因外显子2多态性与生长性能的关联研究[J]. 黑龙江畜牧兽医, 2018(16): 67–71.
XU L Y, MEI H, WANG Z M, et al. Polymorphism analysis of POU1F1 gene exon 2 and its correlation study on growth performance in Tibetan chicken and Luning chicken[J]. Heilongjiang Animal Science and Veterinary Medicine, 2018(16): 67–71. (in Chinese) |
| [2] |
王英明, 徐亚欧, 王志敏, 等. 藏鸡KLF15基因克隆、组织表达谱及其表达与肌内脂肪含量相关性的研究[J]. 畜牧兽医学报, 2019, 50(2): 261–270.
WANG Y M, XU Y O, WANG Z M, et al. Studies on the cloning of KLF15 gene, tissue expression profile and the association between its expression and intramuscular fat content in Tibetan chicken[J]. Acta Veterinaria et Zootechnica Sinica, 2019, 50(2): 261–270. (in Chinese) |
| [3] |
张燕, 吴锦波, 何世明, 等. 阿坝州藏鸡生长曲线拟合与分析[J]. 中国家禽, 2017, 39(19): 56–59.
ZHANG Y, WU J B, HE S M, et al. Fitting and analysis of growth curve of Tibetan chicken in Aba prefecture[J]. China Poultry, 2017, 39(19): 56–59. (in Chinese) |
| [4] |
刘光伟, 赵燕英, 王海, 等. 泸宁鸡不同饲养期屠宰性能与肉品质特性测定与分析[J]. 黑龙江畜牧兽医, 2015(5): 87–90.
LIU G W, ZHAO Y Y, WANG H, et al. Determination and analysis of slaughter performance and meat quality characteristics of Luning chicken in different rearing periods[J]. Heilongjiang Animal Science and Veterinary Medicine, 2015(5): 87–90. (in Chinese) |
| [5] | KAMBADUR R, SHARMA M, SMITH T P L, et al. Mutations in myostatin (GDF8) in double-muscled belgian blue and piedmontese cattle[J]. Genome Res, 1997, 7(9): 910–915. DOI: 10.1101/gr.7.9.910 |
| [6] |
字向东, 罗斌, 夏威, 等. 基于RNA-Seq技术的牦牛体外受精胚胎发育转录组分析[J]. 中国农业科学, 2018, 51(8): 1577–1589.
ZI X D, LUO B, XIA W, et al. Transcriptomic analysis of IVF embryonic development in the Yak (Bos grunniens) via RNA-Seq[J]. Scientia Agricultura Sinica, 2018, 51(8): 1577–1589. (in Chinese) |
| [7] | MOTAMENY S, WOLTERS S, NVRNBERG P, et al. Next generation sequencing of miRNAs- strategies, resources and methods[J]. Genes, 2010, 1(1): 70–84. DOI: 10.3390/genes1010070 |
| [8] | LEPPER C, PARTRIDGE T A, FAN C M. An absolute requirement for Pax7-positive satellite cells in acute injury-induced skeletal muscle regeneration[J]. Development, 2011, 138(17): 3639–3646. DOI: 10.1242/dev.067595 |
| [9] | CHEN J F, TAO Y Z, LI J, et al. microRNA-1 and microRNA-206 regulate skeletal muscle satellite cell proliferation and differentiation by repressing Pax7[J]. J Cell Biol, 2010, 190(5): 867–879. DOI: 10.1083/jcb.200911036 |
| [10] |
吴宁昭.miRNA-1和miRNA-133在鸭骨骼肌发育中的表达及功能初步研究[D].扬州: 扬州大学, 2017.
WU N Z.The expression of miRNA-1 and miRNA-133 and its function on duck skeletal cell proliferation and differentiation[D]. Yangzhou: Yangzhou University, 2017.(in Chinese) http://cdmd.cnki.com.cn/Article/CDMD-11117-1017238883.htm |
| [11] | MIAO X Y, LUO Q M. Genome-wide transcriptome analysis between small-tail Han sheep and the Surabaya fur sheep using high-throughput RNA sequencing[J]. Reproduction, 2013, 145(6): 587–596. DOI: 10.1530/REP-12-0507 |
| [12] |
纪会, 王会, 柴志欣, 等. 牦牛不同年龄肌肉组织microRNA表达谱及生物信息学分析[J]. 畜牧兽医学报, 2019, 50(5): 957–971.
JI H, WANG H, CHAI Z X, et al. Differential expression profile and bioinformatics analysis of miRNAs in Yak muscle tissue during development[J]. Acta Veterinaria et Zootechnica Sinica, 2019, 50(5): 957–971. (in Chinese) |
| [13] |
成志敏, 张宁芳, 王媛媛, 等. 基于RNA-Seq技术筛选影响猪肌纤维性状的候选基因[J]. 畜牧兽医学报, 2019, 50(5): 918–929.
CHENG Z M, ZHANG N F, WANG Y Y, et al. Screening of candidate genes for muscle fiber characteristics in pig using RNA-Seq[J]. Acta Veterinaria et Zootechnica Sinica, 2019, 50(5): 918–929. (in Chinese) |
| [14] |
张敏, 王杰, 孙艳发, 等. 肉鸡肌肉与脂肪组织基因组差异剪接基因分析[J]. 畜牧兽医学报, 2018, 49(10): 2124–2132.
ZHANG M, WANG J, SUN Y F, et al. Differential splicing gene analysis between muscle and fat tissues in broilers[J]. Acta Veterinaria et Zootechnica Sinica, 2018, 49(10): 2124–2132. DOI: 10.11843/j.issn.0366-6964.2018.10.008 (in Chinese) |
| [15] | RIFFO-CAMPOS Á L, RIQUELME I, BREBI-MIEVILLE P. Tools for sequence-based miRNA target prediction:what to choose?[J]. Int J Mol Sci, 2016, 17(12): 1987. DOI: 10.3390/ijms17121987 |
| [16] | WRIGHT W E, SASSOON D A, LIN V K. Myogenin, a factor regulating myogenesis, has a domain homologous to MyoD[J]. Cell, 1989, 56(4): 607–617. DOI: 10.1016/0092-8674(89)90583-7 |
| [17] | COMAI G, TAJBAKHSH S. Molecular and cellular regulation of skeletal myogenesis[J]. Curr Top Dev Biol, 2014, 110: 1–73. DOI: 10.1016/B978-0-12-405943-6.00001-4 |
| [18] | VENUTI J M, MORRIS J H, VIVIAN J L, et al. Myogenin is required for late but not early aspects of myogenesis during mouse development[J]. J Cell Biol, 1995, 128(4): 563–576. DOI: 10.1083/jcb.128.4.563 |
| [19] | LIU C, GERSCH R P, HAWKE T J, et al. Silencing of Mustn1 inhibits myogenic fusion and differentiation[J]. Am J Physiol Cell Physiol, 2010, 298(5): C1100–C1108. DOI: 10.1152/ajpcell.00553.2009 |
| [20] | XU T S, GU L H, SUN Y, et al. Characterization of MUSTN1 gene and its relationship with skeletal muscle development at postnatal stages in Pekin ducks[J]. Genet Mol Res, 2015, 14(2): 4448–4460. DOI: 10.4238/2015.May.4.2 |
| [21] |
李娟.鸡生长发育和肌纤维生长的影响因素与相关基因表达研究[D].成都: 四川农业大学, 2013.
LI J.Study on the influence factors about growth and muscle fiber related gene expression of chickens[D]. Chengdu: Sichuan Agricultural University, 2013.(in Chinese) http://cdmd.cnki.com.cn/Article/CDMD-10626-1014103189.htm |
| [22] | OHTSUBO H, SATO Y, SUZUKI T, et al. APOBEC2 negatively regulates myoblast differentiation in muscle regeneration[J]. Int J Biochem Cell Biol, 2017, 85: 91–101. DOI: 10.1016/j.biocel.2017.02.005 |
| [23] | SATO Y, OHTSUBO H, NIHEI N, et al. Apobec2 deficiency causes mitochondrial defects and mitophagy in skeletal muscle[J]. FASEB J, 2018, 32(3): 1428–1439. DOI: 10.1096/fj.201700493R |
| [24] | TAN X G, ROTLLANT J, LI H Q, et al. SmyD1, a histone methyltransferase, is required for myofibril organization and muscle contraction in zebrafish embryos[J]. Proc Natl Acad Sci U S A, 2006, 103(8): 2713–2718. DOI: 10.1073/pnas.0509503103 |
| [25] | LI H Q, ZHONG Y W, WANG Z F, et al. Smyd1b is required for skeletal and cardiac muscle function in zebrafish[J]. Mol Biol Cell, 2013, 24(22): 3511–3521. DOI: 10.1091/mbc.e13-06-0352 |
| [26] | LI D L, NIU Z, YU W S, et al. SMYD1, the myogenic activator, is a direct target of serum response factor and myogenin[J]. Nucleic Acids Res, 2009, 37(21): 7059–7071. DOI: 10.1093/nar/gkp773 |
| [27] | NAKAJIMA T, FUKIAGE C, AZUMA M, et al. Different expression patterns for ubiquitous calpains and Capn3 splice variants in monkey ocular tissues[J]. Biochim Biophys Acta, 2001, 1519(1-2): 55–64. DOI: 10.1016/S0167-4781(01)00212-3 |
| [28] |
朱文奇, 徐文娟, 束婧婷, 等. 鸭骨骼肌早期发育过程中钙蛋白酶3(CAPN3)基因的表达及其与肌纤维性状的关联[J]. 畜牧兽医学报, 2014, 45(3): 385–390.
ZHU W Q, XU W J, SHU J T, et al. Expression of CAPN3 gene in skeletal muscles and its association with myofiber traits during embryonic and early post-hatching development in ducks[J]. Acta Veterinaria et Zootechnica Sinica, 2014, 45(3): 385–390. (in Chinese) |
| [29] |
陶璇, 顾以韧, 梁艳, 等. 藏猪和杜洛克猪CAPN3基因表达差异及其与肉质性状相关性分析[J]. 养猪, 2016(4): 76–78.
TAO X, GU Y R, LIANG Y, et al. Expression differences of CAPN3 gene and its correlation analysis with meat quality traits in Tibet and Duroc pigs[J]. Swine Production, 2016(4): 76–78. DOI: 10.3969/j.issn.1002-1957.2016.04.037 (in Chinese) |
| [30] | LI Z X, XU Y O, LIN Y Q. Transcriptome analyses reveal genes of alternative splicing associated with muscle development in chickens[J]. Gene, 2018, 676: 146–155. DOI: 10.1016/j.gene.2018.07.027 |
| [31] | DOU T F, ZHAO S M, RONG H, et al. Biological mechanisms discriminating growth rate and adult body weight phenotypes in two Chinese indigenous chicken breeds[J]. BMC Genomics, 2017, 18(1): 469. DOI: 10.1186/s12864-017-3845-9 |
| [32] | ARNSTEN A F T. Stress signalling pathways that impair prefrontal cortex structure and function[J]. Nat Rev Neurosci, 2009, 10(6): 410–422. DOI: 10.1038/nrn2648 |
| [33] | SAWCHENKO P E, LI H Y, ERICSSON A. Circuits and mechanisms governing hypothalamic responses to stress:a tale of two paradigms[J]. Prog Brain Res, 2000, 122: 61–78. DOI: 10.1016/S0079-6123(08)62131-7 |
| [34] | MADDEN K S, SANDERS V M, FELTEN D L. Catecholamine influences and sympathetic neural modulation of immune responsiveness[J]. Annu Rev Pharmacol Toxicol, 1995, 35: 417–448. DOI: 10.1146/annurev.pa.35.040195.002221 |
| [35] |
孙茹.骨骼肌损伤与修复过程中炎症反应与肌卫星细胞再生关系的研究[D].长春: 东北师范大学, 2009.
SUN R.Studies on the relationship between inflammatory response and satellite cell regeneration in skeletal muscle injury and repair process[D]. Changchun: Northeast Normal University, 2009.(in Chinese) http://cdmd.cnki.com.cn/Article/CDMD-10200-2009160627.htm |
| [36] | KRIPPENDORF B B, RILEY D A. Distinguishing unloading-versus reloading-induced changes in rat soleus muscle[J]. Muscle Nerve, 1993, 16(1): 99–108. |
| [37] | TIDBALL J G. Regulation of muscle growth and regeneration by the immune system[J]. Nat Rev Immunol, 2017, 17(3): 165–178. DOI: 10.1038/nri.2016.150 |
| [38] | NACHTIGALL P G, DIAS M C, CARVALHO R F, et al. microRNA-499 expression distinctively correlates to target genes sox6 and rod1 profiles to resolve the skeletal muscle phenotype in Nile tilapia[J]. PLoS One, 2015, 10(3): e0119804. DOI: 10.1371/journal.pone.0119804 |
| [39] | WU J Y, YUE B L, LAN X Y, et al. miR-499 regulates myoblast proliferation and differentiation by targeting transforming growth factor β receptor 1[J]. J Cell Physiol, 2019, 234(3): 2523–2536. DOI: 10.1002/jcp.26903 |
| [40] | WANG Z J, OUYANG H J, CHEN X L, et al. gga-miR-205a affecting myoblast proliferation and differentiation by targeting CDH11[J]. Front Genet, 2018, 9: 414. DOI: 10.3389/fgene.2018.00414 |
| [41] | DI BENEDETTO A, WATKINS M, GRIMSTON S, et al. N-cadherin and cadherin 11 modulate postnatal bone growth and osteoblast differentiation by distinct mechanisms[J]. J Cell Sci, 2010, 123(15): 2640–2648. DOI: 10.1242/jcs.067777 |
| [42] | CHEN W, FANG G F, WANG S D, et al. Characterization and differential expression of microRNA in skeletal muscle of Laiwu and Yorkshire pig breeds[J]. Genes Genomics, 2017, 39(2): 173–182. DOI: 10.1007/s13258-016-0484-5 |
| [43] | SIENGDEE P, TRAKOOLJUL N, MURANI E, et al. MicroRNAs regulate cellular ATP levels by targeting mitochondrial energy metabolism genes during C2C12 myoblast differentiation[J]. PLoS One, 2015, 10(5): e0127850. DOI: 10.1371/journal.pone.0127850 |
| [44] | WANG Y H, LI M L, WANG Y H, et al. A Zfp609 circular RNA regulates myoblast differentiation by sponging miR-194-5p[J]. Int J Biol Macromol, 2019, 121: 1308–1313. DOI: 10.1016/j.ijbiomac.2018.09.039 |



